星斑川鲽、黄盖鲽和石鲽线粒体基因Cytb和COⅠ片段序列的比较研究
2016-12-20郑风荣刘洪展郭湘云关洪斌
李 青,王 波,郑风荣,刘洪展,郭湘云,关洪斌
( 1. 山东大学 海洋学院,山东 威海 264209; 2. 国家海洋局 第一海洋研究所,山东 青岛 266061 )
星斑川鲽、黄盖鲽和石鲽线粒体基因Cytb和COⅠ片段序列的比较研究
李 青1,2,王 波2,郑风荣2,刘洪展1,郭湘云2,关洪斌1
( 1. 山东大学 海洋学院,山东 威海 264209; 2. 国家海洋局 第一海洋研究所,山东 青岛 266061 )
星斑川鲽、钝吻黄盖鲽和石鲽是我国北方重要的经济海水鱼类。为探究这3种鲽科鱼类间的亲缘关系及遗传信息,本文采用了PCR扩增和直接测序的技术,对这3种鲽科鱼类各24尾个体的线粒体基因Cytb和COⅠ进行初步研究。测序得到的序列经人工处理校正后分析,其中Cytb基因片段可用长度为358 bp,包含58个变异位点,20个转换位点,3个颠换位点;COⅠ基因片段可用长度为559 bp,包含变异位点56个,转换位点18个,颠换位点3个;Cytb基因片段和COⅠ基因片段均表明黄盖鲽和石鲽具有较高的单倍型多样性与较低的核苷酸多样性,而星斑川鲽具有较低的单倍型多样性和较低的核苷酸多样性。基于Cytb和COⅠ序列的两种系统发育树(邻接法和不加权对群法)均表明3种鲽科鱼类中星斑川鲽与石鲽亲缘关系更近,与黄盖鲽相对较远。鲽科鱼类物种遗传多样性及遗传结构的分析可以为鱼类的保护和管理提供重要的理论依据,为保障我国鲽科鱼类养殖业的健康可持续发展提供理论指导和科学依据。
星斑川鲽;黄盖鲽;石鲽;线粒体DNA
鲽形目鱼类常被称作比目鱼、偏口鱼。是我国重要的海水养殖鱼类。星斑川鲽(Platichthysstellatus)又名星突江鲽,川鲽属[1],是一种大型经济鱼类。星斑川鲽主要栖息在N 33°~73°和W 105°~E 127°附近的太平洋和北冰洋海域以及附近的河流[2],属于冷水广盐性鱼类。钝吻黄盖鲽(Pseudopleuronectesyokohamae),黄盖鲽属,冷温性鱼类,具有地方种群多、适应能力强、底栖杂食性等特点[3]。石鲽(Kareiusbicoloratus),石鲽属,主要分布于温带和寒带。这三种鲽科鱼类肉质鲜美,口感独特,深受消费者的喜爱。目前这三种鲽科鱼类已经成为我国北方重要的海水养殖鱼类,然而对三种鱼类养殖生理生化方面的研究报道较多,比较其遗传多样性的研究报道却很少,而正确的遗传结构鉴定对于重要渔业资源的管理起到积极带动作用[4]。
鱼类线粒体基因是长约15~20 kb的结构高度紧凑的共价双链闭环分子[5],是一个相对独立的单位[6]。因进化速率快、呈母系遗传、群体之间遗传差异极易被检出,线粒体基因常被作为有效的遗传标记应用于群体遗传多样性、遗传结构以及物种鉴定研究中[7-10]。在线粒体中,细胞色素b(Cytb)基因和细胞色素C氧化酶Ⅰ(COⅠ)基因具有比线粒体rDNA和非编码区更易排序[11]、进化速率适中、容易扩增和测序以及富含系统发育信息[12]等优点,而被广泛应用于鱼类种质资源状况和种群遗传多样性以及分子系统发育的探究中。本研究测定了日照星斑川鲽、江苏星斑川鲽、钝吻黄盖鲽和石鲽线粒体基因组的Cytb基因和COⅠ基因的部分序列,分析比较了3个鲽科鱼类在碱基组成、变异位点、转换位点、单倍型数目、单倍型多样性和核苷酸多样性等方面的异同,并计算了遗传距离和基于双参数法建立系统发育树。同时比较了源于同一野生群体经在不同地理位置养殖后的星斑川鲽后代在线粒体遗传信息上的异同,以及同属于野生群体的黄盖鲽和石鲽在线粒体遗传信息的不同。
1 材料与方法
1.1 试验材料
24尾日照星斑川鲽于2014年8月取自日照海洋水产资源增殖站,属于人工养殖群体;24尾江苏的星斑川鲽于2015年3月取自江苏赣县属于人工养殖群体,与日照星斑川鲽均是由生活在我国黄海北部的野生群体经人工养殖十年而繁殖的后代;24尾钝吻黄盖鲽于2015年1月取自烟台蓬莱近海,属于野生群体;24尾石鲽于2015年6月取自日照近海,属于野生群体。取尾鳍样品于95%酒精固定-80 ℃保存。
1.2 试验方法
基因组DNA提取,取每个样本的尾鳍约30 mg,分别用海洋动物组织基因组DNA提取试剂盒提取,将溶于130 μL TE(Tris和EDTA)缓冲液中,于-20 ℃保存。提取基因组水浴时需充分振荡使其混匀,以保证消化完全;溶解前应使漂洗液充分挥发,避免漂洗液中的乙醇残留影响后续的PCR反应。
目的片段PCR扩增用于扩增线粒体基因DNA的Cytb基因片段[13],上游引物Cytb F:5′-AACCACCGTTGTTATTCAACT-3′;下游引物Cytb R:5′-CTCAGAATGACATTTGTCCTCA-3′;线粒体CO Ⅰ基因片段引物,上游引物CO Ⅰ F:5′-CACAAAGACATTGGCACCCT-3′;下游引物CO Ⅰ R:5′-CCTCCTGCAGGGTCAAAGAA-3′。PCR反应总体积为50 μL:DNA模板,1.5 μL(15 ng/μL),上下游引物各2 μL(10 μmol/L),2×Reaction Mix 25 μL,Taq DNA 聚合酶0.6 μL(2.5 U/μL)(东盛生物),ddH2O 18.9 μL。Cytb基因PCR 反应条件为:94 ℃预变性4 min;94 ℃变性30 s,53 ℃退火30 s,72 ℃延伸1 min,第二步到第四步34个循环;72 ℃总延伸10 min。CO Ⅰ基因PCR 反应条件为:94 ℃预变性4 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,第二步到第四步34个循环;72 ℃总延伸10 min。PCR反应在TaKaRa PCR Thermal Cycler Dice上进行。取5 μL PCR扩增产物进行1%琼脂糖凝胶电泳检测(U=5 V/cm)。
目的片段的序列测定将含有目的条带的PCR产物中送北京美吉桑格生物医药科技有限公司用ABI3730XL自动测序仪进行单向测序,测序反应采用与相对应的正向引物。
序列分析测序结果经Dnastar软件包校对后人工截取有效片段。软件DNA Sequence Polymorphism 5.0[14]用来计算各个群体单倍型数、单倍型多样性[15]、核苷酸多样性[15]以及平均核苷酸差异数[16];利用软件Mega 6.0[17]碱基组成、碱基替换统计、种间种内遗传距离计算、用邻接法(分别设自展值为1000或内部分支检验次数是1000)和不加权对群法(内部分支检验次数1000)构建系统发育树,从而进行系统分化分析。
2 结 果
2.1 4个鱼类群体的线粒体基因Cytb
2.1.1 Cytb部分基因序列碱基组成分析
将返回的4个群体的测序结果经Bioedit Sequence Alignment Editor 校对后人工截取总长421 bp中的358 bp片段进行分析,应用Mega 6.0中Sequence Date Explore分别计算4个群体的碱基组成频率(表1)。由表1 可见,钝吻黄盖鲽相比其他3个鲽类群体碱基组成差异相对较大,GC含量相对较高(49.6%),而日照星斑川鲽、江苏星斑川鲽、石鲽的GC含量分别为46.6%、46.6%、46.5%。差异主要出现在第3位点,在第3位点日照星斑川鲽、江苏星斑川鲽、石鲽的GC含量分别为49.6%、49.6%、49.3%,钝吻黄盖鲽在第3位点的GC含量却高达57.7%。鸟嘌呤在第3位点的含量呈现一致的偏低,仅占8.4%,这与肖永双等[18]的研究结果一致。
运用Mega 6.0软件分别对4个鲽科鱼类Cytb部分基因序列间变异位点的分析统计(表2)。每个种群内部碱基序列差异很小,变异位点最多的是石鲽,仅仅只有5个。3种鱼共4个群体作为一个整体,在358个碱基中,保守位点(C)高达300个碱基,变异位点(V)58个,简约信息位点(Pi)57个,自裔位点(Si)1个。钝吻黄盖鲽的4倍简并位点最多,有64个,其他3类有63个。星斑川鲽2个群体各有63个2倍简并位点,钝吻黄盖鲽和石鲽分别有62个和61个。软件DnaSP 5计算得到的各个群体单倍型数目、单倍型多样性、核苷酸多样性以及平均核苷酸差异性表明,石鲽遗传多样性最高,单倍型数目是星斑川鲽的3倍,是钝吻黄盖鲽的2倍,可以初步得出养殖鱼类群体多样性低于野生群体。

表1 Cytb基因片段序列的碱基组成频率 %
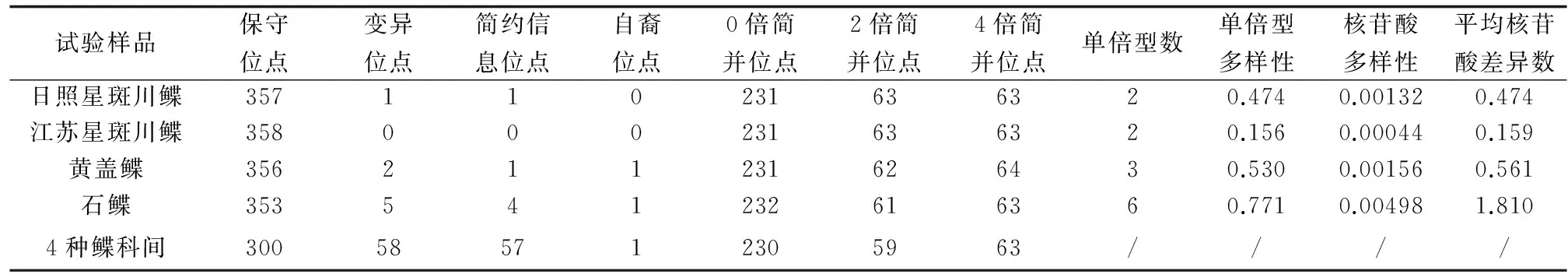
表2 Cytb基因片段平均位点分析
2.1.2 Cytb部分基因序列碱基替换统计
根据Mega 6.0中的Sequence Date Explore分别对4个群体鱼类的Cytb序列的碱基替换数进行统计分析(表3)。
经统计,日照星斑川鲽、江苏星斑川鲽、钝吻黄盖鲽、石鲽4个群体间转换取代的速率为5.97%,颠换取代的速率为0.89%。转换与颠换速率之比是6.67。转换速率明显高于颠换速率。

表3 Cytb基因序列碱基替换统计表
2.1.3 Cytb部分基因序列遗传距离计算
4个群体的Cytb基因通过双参数模型计算所获得的遗传距离见表4。由表4可见,4个鲽类群体内部遗传距离分别为:钝吻黄盖鲽0.002,标准差0.001;江苏星斑川鲽内部遗传距离最小,为0.000,标准差0.000;石鲽0.005,标准差0.002;日照星斑川鲽0.001,标准差0.001。群体间遗传距离最小的是江苏星斑川鲽和日照星斑川鲽,仅仅为0.001,星斑川鲽江苏群体与日照群体内部遗传距离相等。星斑川鲽和钝吻黄盖鲽间的遗传距离最大,江苏、日照2个星斑川鲽群体与钝吻黄盖鲽间的遗传距离分别为0.147、0.148。钝吻黄盖鲽和石鲽平均遗传距离为0.131,石鲽和日照星斑川鲽、江苏星斑川鲽群体的平均遗传距离分别为0.015、0.075。

表4 基于双参数模型模型4个群体的Cytb序列的遗传距离
注:表格对角线以下为种群间遗传距离,对角线以上为标准差.
2.1.4 Cytb系统进化树的构建
将测序所得的3种鲽的序列在NCBI数据库中进行Blast同源性检索,在GenBank中选取星斑川鲽、石鲽、钝吻黄盖鲽Cytb序列各一条,进行系统进化分析。另外选取马舌鲽(ReinhardtiushippoglossoidesKF445157.1)作为外群进行系统发育构建(表5)。由于每个群体内部差异很小,建树时仅选取每个群体的3尾个体。利用Mega 6.0软件中的邻接法(又分为内部分支检验和自展法检测)和不加权对群法构建系统发育树(图1~3)。结果显示,邻接法和不加权对群法具有基本相似的拓扑结构:星斑川鲽2个群体基本没有差异,与星斑川鲽KF445179.1聚为一支;石鲽与星斑川鲽的遗传关系相对更近一些,石鲽样品与石鲽AB987795.1聚为一支后与星斑川鲽聚为一大支;马舌鲽KF445157.1与钝吻黄盖鲽相对更近,聚为一大支,与星斑川鲽和石鲽的一大支相并列。

表5 GenBank下载的几种鲽的Cytb序列信息

图1 基于Cytb基因片段的邻接法(自展法检测)构建系统树节点上数值代表自展法检测的百分值(重复计算1000次).
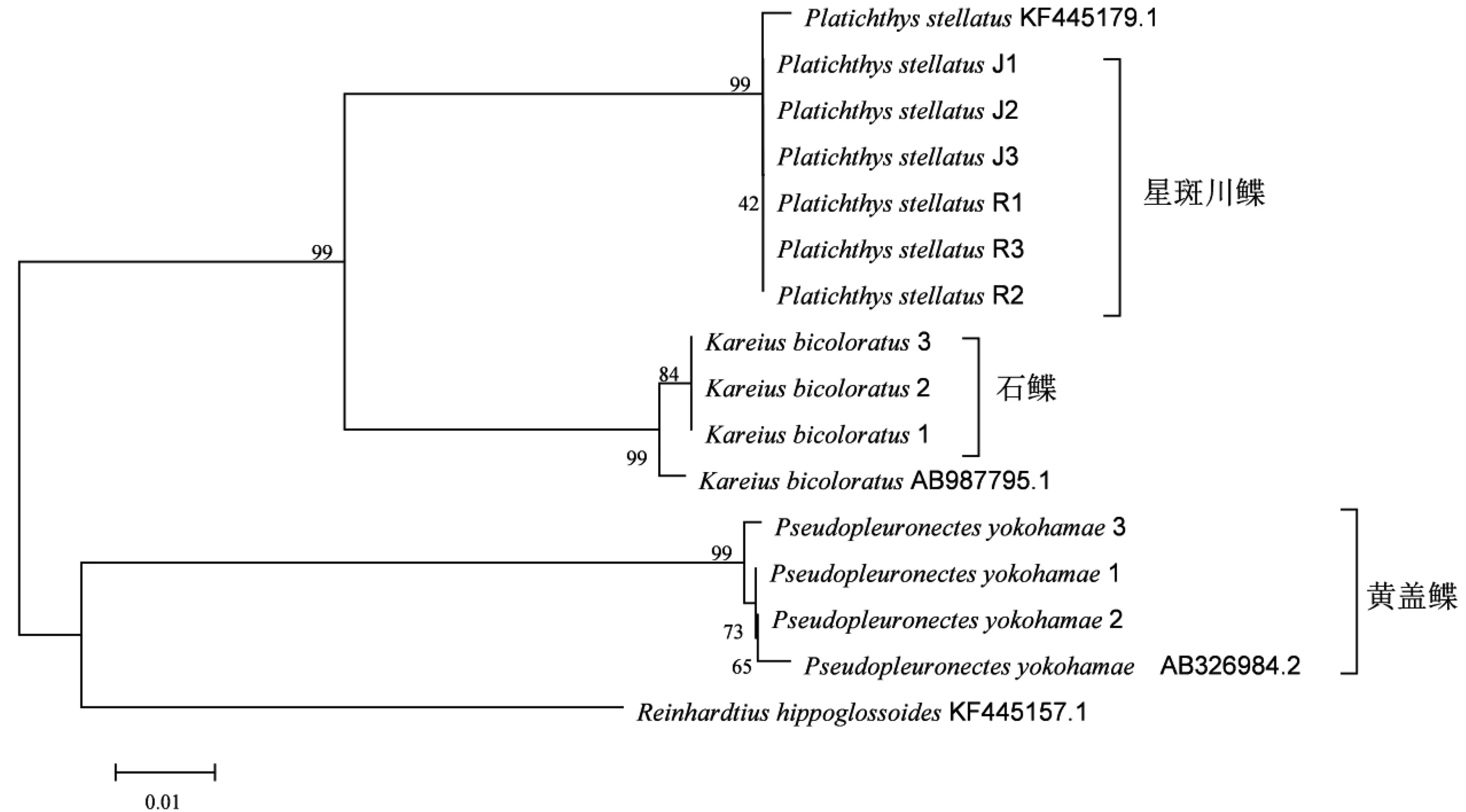
图2 基于Cytb基因片段的邻接法(内部分支检验)构建系统树节点上数值代表内部分支检验百分值(重复计算1000次).
2.2 4个鱼类群体的线粒体基因COⅠ
2.2.1 COⅠ部分基因序列碱基组成分析
将返回的4个群体的测序结果经Bioedit Sequence Alignment Editor 校对后人工截取总长604 bp中的559 bp片段进行分析,应用Mega 6.0中Sequence Date Explore分别计算4个群体的碱基组成频率(表6)。结果表明,在4个群体线粒体基因COⅠ片段中,(C+G)含量略低于(A+T),钝吻黄盖鲽中(C+G)含量为47.5%,星斑川鲽2个群体均为47.7%,石鲽相对最高,为48.1%。(C+G)在第一位点含量超过(A+T)含量,55.6%~56.1%;第2位点(C+G)最低,均为41.4%;第3位点(G+C)含量分别为:星斑川鲽2个群体均为46.3%,钝吻黄盖鲽45.1%,石鲽47.3%。
运用Mega 6.0软件对4个鲽科鱼类COⅠ部分基因序列间变异位点分析统计(表7)。钝吻黄盖鲽和石鲽相对星斑川鲽的变异位点要多,分别为5个、4个,分别占0.89%和0.71%。4个群体作为整体,共有56个变异位点,占10.02%,包括53个简约信息位点和3个自裔位点。0倍简并位点、2倍简并位点、4倍简并位点4个群体间相差不大。2个星斑川鲽群体共享1个单倍型,这与肖永双等[19]的研究一致。就变异位点和单倍型来说,钝吻黄盖鲽的多样性略高于石鲽,钝吻黄盖鲽24个个体共享6个单倍型,石鲽24个个体共享5个单倍型。

图3 基于Cytb基因片段的不加权对群法(内部分支检验)法构建系统树节点上数值代表内部分支检验的百分值(重复计算1000次).

试验样品总碱基频率第一位点第二位点第三位点TCAGTCAGTCAGTCAG日照星斑川鲽27.529.524.818.21626.228.929.44428.514.512.92333.930.612.4江苏星斑川鲽27.629.524.718.21626.228.929.44428.514.512.92333.930.612.4黄盖鲽26.830.225.617.31526.728.929.44428.514.512.92235.533.49.6石鲽27.629.324.318.81626.228.929.44428.514.512.92333.329.514.0

表7 COⅠ基因片段平均位点分析
2.2.2 COⅠ部分基因序列碱基替换统计
根据Mega 6.0中的Sequence Date Explore分别对4个群体鱼类的Cytb序列的碱基替换数进行统计分析(表8)。由表8可见,转换颠换主要发生在第3位点,发生在第1位点的转换仅有1个,而在第3位点转换位点有17个,颠换位点有3个。转换取代速率为3.35%,颠换取代速率为0.56%,转换与颠换位点的比值为R=6。

表8 COⅠ基因序列碱基替换统计表
2.2.3 COⅠ部分基因序列遗传距离计算
用双参数模型计算所获得的4个群体的遗传距离见表9。由表9可知,钝吻黄盖鲽与其他3个群体遗传距离最远,均为0.074;星斑川鲽两个群体几乎无差异,遗传距离为0;星斑川鲽与石蝶相对较近,为0.045。
表9基于双参数模型模型4个群体的COⅠ序列的遗传距离

群体黄盖鲽江苏星斑川鲽石鲽日照星斑川鲽黄盖鲽0.0110.0110.011江苏星斑川鲽0.0740.0080.000石鲽0.0740.0450.008日照星斑川鲽0.0740.0000.045
注:表格对角线以下是种群间遗传距离,对角线以上是标准差.
2.2.4 COⅠ系统进化树的构建
选取GenBank中的3条序列以及马舌鲽作为外群(表10),利用软件Mega 6.0分别使用邻接法和不加权对群法构建系统发育树(图4~6)。比较3种系统发育树,两个星斑川鲽群体均交叉混合为一支,与星斑川鲽EU266365.1为同一物种;石鲽与石鲽JQ738484.1为一物种;石鲽与星斑川鲽聚为一大支后再与钝吻黄盖鲽聚为一支,马舌鲽KC015868.1单独为一支。

表10 GenBank下载的几种鲽的COⅠ序列信息
3 讨 论
遗传多样性是物种适应生存、进化发展的必要条件,遗传多样性越高,遗传变异越丰富,对环境适应能力就越强,可适宜生存的环境就越广泛。由此,研究生物遗传多样性可以为采取积极有效的措施保护生物多样性提供科学指导。相对于同工酶、RAPD等分子标记,DNA序列分析在用于遗传多样性分析试验时更准确、更直接[20]。线粒体基因Cytb和COⅠ是两个非常保守的基因序列,常被用来检测群体遗传结构和遗传多样性[21-22]。本试验采用Cytb和COⅠ两对引物探究日照星斑川鲽、江苏星斑川鲽、钝吻黄盖鲽、石鲽4个鲽类群体在碱基组成、变异及保守位点、碱基替换等方面的异同,以及根据双参数模型建立的系统发育树。
4个鲽类群体在COⅠ和Cytb 两个基因片段上的碱基组成方面,GC含量与AT含量相差不大,这与无脊椎动物如三疣梭子蟹(Portunustritubercalatus)[23]、栉孔扇贝(Chlamysfarreri)[24]有区别。总体看4个群体的在这两个基因片段中的变异位点很少,反映了线粒体基因Cytb和COⅠ是两种很保守的基因序列。根据Grant[25]的4种分类标准[a,较低的单倍型多样性(<0.5),较低的核苷酸多样性(<0.005);b,较高的单倍型多样性,较低的核苷酸多样性;c,较低的单倍型多样性,较高的核苷酸多样性;d,较高的单倍型多样性,较高的核苷酸多样性],从整个试验结果来看,石鲽和钝吻黄盖鲽具有较高的单倍型多样性与较低的核苷酸多样性,这与海水中大多数鱼类相同,如银鲳(Pampusargenteus)[26],小黄鱼(Pseudosciaenapolyactis)[27],西北太平洋的花鲈(Lateolabraxjaponicus)与斑点鲈鱼(L.maculatus)[28]等,这可能是种群受瓶颈效应影响后又经历建立者效应的结果[29]。而星斑川鲽属于较低单倍型与较低核苷酸多样性结合类,这可能与我国引进星斑川鲽时间较晚,分化程度较低有关。
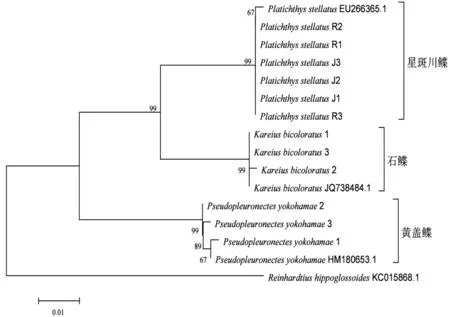
图4 基于COⅠ基因片段的邻接法(自展法检测)构建系统树节点上数值代表自展法检测的百分值(重复计算1000次).

图5 基于COⅠ基因片段的邻接法(内部分支检验)构建系统树节点上数值代表内部分支检验的百分值(重复计算1000次).

图6 基于COⅠ基因片段的不加权对群法 (内部分支检验)构建系统树节点上数值代表内部分支检验的百分值(重复计算1000次).
从遗传距离可以看出,星斑川鲽2个群体的群体内遗传距离与群体间遗传距离相等,可视为同一个群体。可以推测星斑川鲽进化速率较慢或二者分布地区环境的区别不够大到影响种群的生存。石鲽和星斑川鲽相对更近,与黄盖鲽较远,这与6个系统发育树反应的结果一致。几个系统发育树的区别是,在基于Cytb基因片段序列建立的系统发育树中,钝吻黄盖鲽与马舌鲽成为一支,与星斑川鲽和石鲽相聚的一支并列;而在基于COⅠ基因序列建立的系统发育树中,星斑川鲽与石鲽聚为一支后,与钝吻黄盖鲽聚为一大支,然后与马舌鲽相聚。这可能与Cytb基因序列较短,区别太小有关。
[1] 王波,孙萍,方华华,等.星斑川鲽形态特征及相关参数的观测[J].海洋学报,2010,32(2):139-147.
[2] Miao G, Shao C, Ma H,et al. Isolation and characterization of polymorphic microsatellite loci from a dinucleotide-enriched genomic library of starry flounder (Platichthysstellatus) and cross-species amplification[J]. Conservation Genetics,2009,10(3):631-633.
[3] 周勤,王迎春.温度对黄盖鲽仔鱼生长、发育、摄食及PNR的影响[J]. 中国水产科学,1998,5(1):30-37.
[4] Utter F M. Biochemical genetics and fishery management: an historical perspective[J]. Journal of Fish Biology,1991,39(Suppl.A):1-20.
[5] 龚理,时伟,司李真,等.鱼类线粒体DNA重排研究进展[J]. 动物学研究,2013,34(6):666-673.
[6] 郭新红,刘少军,刘巧,等,鱼类线粒体DNA研究新进展[J]. 遗传学报, 2004,31(9):983-1000.
[7] Brown W. Evolution of animal mitochondrial DNA[J]. Evolution of Genes and Proteins, 1983(1):62-88.
[8] Harrison R G. Animal mitochondrial DNA as a genetic marker in population and evolutionary biology[J]. Trends in Ecology & Evolution, 1989,4(1):6-11.
[9] Miya M,Takeshima H,Endo H, et al. Major patterns of higher teleostean phylogenies: a new perspective based on 100 complete mitochondrial DNA sequences[J]. Molecular Phylogenetics and Evolution,2003,26(1):121-138.
[10] Pepe T,Trotta M, Di Marco I,et al. Mitochondrial cytochrome b DNA sequence variations: an approach to fish species identification in processed fish products[J]. Journal of Food Protection,2005,68(2):421-425.
[11] 俞建中. 黄海鲽形目鱼类系统发生与分子进化的初步研究[D].青岛:中国海洋大学,2004.
[12] 张迪,雷光春,龚成,等.基于COⅠ基因序列的太湖新银鱼遗传多样性[J]. 湖泊科学,2012,24(2):299-306.
[13] 张岩,肖永双,高天翔,等.两种黄盖鲽线粒体DNA部分片段比较分析[J]. 水产学报,2009,33(2):201-207.
[14] Librado P,Rozas J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics,2009,25(11):1451-1452.
[15] Nei M. Molecular evolutionary genetics[M]. New York: Columbia University Press,1987:1009-1021.
[16] Tajima F. Evolutionary relationship of DNA sequences in finite populations[J]. Genetics,1983,105(2):437-460.
[17] Tamura K, Stecher G, Peterson D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution,2013,30(12):2725-2729.
[18] 肖永双,张岩,高天翔. 基于线粒体DNA部分片段探讨石鲽与星突江鲽的亲缘关系[J]. 中国海洋大学学报:自然科学版,2010,40(6):69-76.
[19] 肖永双,肖志忠,刘清华,等.星斑川鲽、大菱鲆及其杂交后代的线粒体DNA序列比较分析[J].海洋科学,2014,38(6):5-9.
[20] Buonaccorsi V, McDowell J,Graves J. Reconciling patterns of inter-ocean molecular variance from four classes of molecular markers in blue marlin (Makairanigricans)[J]. Molecular Ecology, 2001,10(5):1179-1196.
[21] 赵峰,庄平,章龙珍,等.基于线粒体Cyt b基因的黄海南部和东海银鲳群体遗传结构分析[J]. 水生生物学报,2011,35(5):745-752.
[22] 赵亮,谢本贵,刘志瑾,等.太湖新银鱼线粒体D-loop和Cyt b片段序列结构与进化速率比较[J]. 动物学杂志,2010,45(2):27-38.
[23] 郭天慧,孔晓瑜,陈四清,等.三疣梭子蟹线粒体DNA16S rRNA和COⅠ基因片段序列的比较研究[J]. 中国海洋大学学报:自然科学版,2004,34(1):22-28.
[24] 刘亚军,喻子牛,姜艳艳,等.栉孔扇贝16S rRNA基因片段序列的多态性研究[J]. 海洋与湖沼,2002,33(5):477-483.
[25] Grant W, Bowen B. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity,1998,89(5):415-426.
[26] 彭士明,施兆鸿,侯俊利. 基于线粒体D-loop区与COⅠ基因序列比较分析养殖与野生银鲳群体遗传多样性[J]. 水产学报,2010,34(1):19-25.
[27] 彭博,章群,赵爽,等.中国近海小黄鱼遗传变异的细胞色素b序列分析[J]. 广东农业科学,2010(2):131-135.
[28] Liu J X,Gao T X,Yokogawa K,et al. Differential population structuring and demographic history of two closely related fish species, Japanese sea bass (Lateolabraxjaponicus) and spotted sea bass (Lateolabraxmaculatus) in Northwestern Pacific[J]. Molecular Phylogenetics and Evolution,2006,39(3):799-811.

ComparativeAnalysisofMitochondrialCytbandCOⅠSequencesofFloundersPlatichthysstellatus,PseudopleuronectesyokohamaeandKareiusbicoloratus
LI Qing1,2,WANG Bo2, ZHENG Fengrong2,LIU Hongzhan1, GUO Xiangyun2,GUAN Hongbin1
( 1. Marine College, Shandong University,Weihai 264209,China;2. First Institute of Oceanography, State Oceanic Administration,Qingdao 266061,China )
FloundersPlatichthysstellatus,PseudopleuronectesyokohamaeandKareiusbicoloratuswere economically important marine fish in north China. To evaluate the relationship among the three members in Pleuronectidae and genetic information, mitochondrial DNA cytochrome b gene (Cytb) and mitochondrial cytochrome c oxidase subunit Ⅰ gene (COⅠ) fragments of the three species(24 individuals in each species) were preliminarily studied by PCR amplification and direct sequencing. By analyzing the sequence obtained by manual processing, we found that the Cytb gene fragment was 358 bp, which contained 58 variable sites, 20 transitionsal pairs, and 3 transversional pairs, while the available length of the COⅠ gene fragment was 559 bp, including 56 variable sites, 18 transitionsal pairs and 3 transversional pairs. Analysis of Cytb and COⅠ gene fragments revealed that bothP.yokohamaeandK.bicoloratushad a high haplotype diversity and low nucleotide diversity whileP.stellatushad low haplotype diversity and low nucleotide diversity. Two kinds of phylogeny trees (Neighbor-Joining Tree and UPGMA tree) based on cytb and COⅠ sequences showed thatP.stellatusandK.bicoloratusis were closer thanP.yokohamaein genetic relationship. Analysis of genetic diversity and genetic structure can provide an important theoretical basis for the conservation and management of fish, and provide theoretical guidance and scientific basis for the healthy and sustainable development of pleuronectid aquaculture.
Platichthysstellatus;Pseudopleuronectesyokohamae;Kareiusbicoloratus;mtDNA
10.16378/j.cnki.1003-1111.2016.06.004
S917.4
A
1003-1111(2016)06-0625-08
2015-12-14;
2016-03-07.
国家高技术发展计划(“863”)项目(2012AA10A408,2012AA10A413);国家海洋公益性行业科研专项(201405010).
李青(1990—),女,硕士研究生;研究方向为:鱼类遗传.E-mail: liqingshdwh@163.com.通讯作者:关洪斌(1961—),男,副教授,博士;研究方向:基因表达和动物营养. E-mail: guanhongbin@sdu.edu.cn.
