杜洛克猪全基因组连锁不平衡分析
2016-12-19刁淑琪罗元宇陈桂华陈赞谋李加琪
刁淑琪,罗元宇,蔡 迪,陈桂华,陈赞谋,张 豪,李加琪,张 哲
(华南农业大学动物科学学院,广东 广州 510642)
杜洛克猪全基因组连锁不平衡分析
刁淑琪,罗元宇,蔡 迪,陈桂华,陈赞谋,张 豪,李加琪,张 哲
(华南农业大学动物科学学院,广东 广州 510642)
利用猪Illumina Porcine SNP60K芯片对福建某核心种猪场杜洛克猪216个个体进行基因型检测,基于该高密度SNP芯片数据,运用Haploview软件计算全基因组连锁不平衡并构建杜洛克猪连锁不平衡图谱。结果表明,该杜洛克猪群体不同染色体上相邻标记间r2存在波动,波动范围为0.46~0.59,相邻标记间的平均连锁不平衡程度r2为0.52,SSC10的r2最低(平均为0.46),SSC6的r2最高(平均为0.59),连锁不平衡水平随着标记间距的增加而衰减、变异程度随之减小。该研究结果可为杜洛克猪遗传分析及全基因组选择研究提供参考。
猪;连锁不平衡;杜洛克;SNP
连锁不平衡(Linkage Disequilibrium,LD)又称等位基因关联,是指同一条染色体上,等位基因间广泛存在的非随机组合现象[1]。连锁不平衡是群体遗传学研究的重要内容之一,在分子生物学研究与动物遗传育种领域,群体基因组连锁不平衡信息是数量性状基因座定位、群体遗传结构分析和全基因组选择的基础。连锁不平衡受选择、突变和遗传漂变等因素的影响。随着科学技术的发展,覆盖全基因组的高密度遗传标记已广泛运用于人和动植物的连锁不平衡研究,并取得了很大的研究进展。在人类基因组学研究中,将单核苷酸多态性(Single Nucleotide Polymorphisms,SNP)作为遗传标记来深入研究连锁不平衡已成为一种趋势。结合关联分析和功能研究,将包括复杂疾病及其中间表型在内的人类各种表型特征,与基因组上以SNP为主的多态性位点联系起来,这对疾病预测、预防和治疗等的突破具有重要作用[2]。
目前,在畜禽方面有很多基于连锁不平衡的研究。在牛的连锁不平衡研究中,Mckay等[3]利用2 670个SNP标记构建了8个不同品种的连锁不平衡图谱,并发现其LD区域长度不超过500 kb。Lu等[4]利用SNP标记对安格斯牛、夏洛莱牛和杂交牛3个群体进行LD分析,发现当标记间距小于30 kb时,3个品种的LD分别为0.29、0.22和0.21,认为不同品种和不同染色体间LD强度具有显著差异。Ross等[5]以r2作为LD的度量指标,利用SNP标记估计了澳大利亚荷斯坦牛、澳洲安格斯牛、新西兰黑白花奶牛和娟姗牛4个群体的LD,其大小分别为0.35、0.25、0.22和0.14。
在猪的连锁不平衡研究方面,Amaral等[6]通过分析中国家猪、欧洲家猪和欧洲野猪的371个SNP,发现欧洲猪种的LD程度高于中国猪种。Badke等[7]以r2作为度量指标对长白猪、约克夏、杜洛克和汉普夏的LD进行比较,发现在标记间距较小时,杜洛克猪的LD较高;而标记间距较大时,汉普夏猪的LD较低。此外,Ai等[8]比较了中国地方猪和西方家猪的LD,发现西方家猪的LD高于中国地方猪。
本研究以杜洛克猪为研究对象,基于SNP芯片基因组数据分析杜洛克猪群体的连锁不平衡水平及其衰减规律,旨在为杜洛克猪的连锁不平衡研究提供一定的参考。
1 材料与方法
1.1 试验材料
试验对象为福建某核心种猪场的216头杜洛克猪(公猪20头,母猪196头),试验猪均在常规条件下饲养,体况正常。使用OMEGA Tissue DNA Kit对所有个体的耳组织抽提基因组DNA。采用Illumina Porcine SNP60K芯片[9]进行SNP基因分型[10],共得到61 565个SNP。
1.2 基因型质量控制
采用美国农业部和爱荷华州立大学于2010年9月联合组装的猪基因组(Sus scrofa Build 10)作为参考序列(http://www.animalgenome. org/blast/),利用Plink软件[11]对供试的216个个体和61 565个SNPs进行基因型质量控制。质控标准为:个体检出率(call rate)>0.95,SNP检出率>0.95,最小等位基因频率(Minor allele frequency,MAF)>0.05和哈迪-温伯格平衡P值(Hardy-Weinberg equilibrium,HWE)≥10-6。经过质控后,剩余215个个体和32 186个有效SNP用于后续研究。
1.3 LD度量
连锁不平衡程度通常用D'[12]和r2[13]进行度量,本研究选用r2作为LD的度量标准。r2表示两个位点统计上和遗传上相关程度的关系(0< r2<1),其表现较稳定r2,对基因频率的变化不敏感。r2的计算公式为:

式中,PA1和PB1是两个标记位点上第1个等位基因的频率,PA1B1是等位基因之间形成的单倍型频率。
采用Haploview软件[15]计算各SNP之间的r2值,采用R软件进行后续的统计分析。
2 结果与分析

图1 杜洛克猪质量控制后SNP最小等位基因频率分布
2.1 基因型质量控制
供试的216个个体和61 565个SNPs进行基因型质量控制后,剩余215个个体和32 186个有效SNP进行后续分析。质控后的SNP最小等位基因频率(MAF)分布情况见图1。从图1可以看出,MAF呈均匀分布,本研究中稀有突变位点极少。其中,70%以上SNP的MAF>0.20,说明本研究中杜洛克猪群体中的SNP多态性较高,分型数据适用于LD的计算和图谱的构建。
2.2 杜洛克猪各染色体LD
杜洛克猪18条常染色体和X染色体r2的衰减趋势如图2所示。从图2可以看出,相邻标记间的LD水平随标记间物理距离的增大而衰减。此外,除了SSC6、SSC14、SSC16、SSC17和SSC18等染色体外,其他染色体上个别间距较大的标记之间也存在高r2现象。
2.3 杜洛克猪全基因组LD及LD图谱

图2 杜洛克猪各染色体的r2分布
杜洛克猪全基因组r2的衰减趋势如图3所示,在全基因组水平上,LD变化趋势与各染色体LD一致,即LD水平随标记间距的增加而衰减,LD的变异程度也随之减小。但在某些距离较远的SNP标记之间,LD程度也很高。
本研究还构建了杜洛克猪全基因组连锁不平衡图谱(图4),图4中颜色越深代表LD程度越强。结果表明,除了在相同染色体上的标记间存在LD,不同染色体上的标记间也存在一定程度的LD。
2.4 杜洛克猪不同标记间距的LD分布
本研究统计了杜洛克猪各染色体上相邻标记间的r2分布情况,从表1可见,杜洛克猪不同染色体上相邻标记间的r2存在波动,波动范围为0.46~0.59。
对杜洛克猪的r2统计发现,不同染色体上标记间的LD程度存在差异。本研究分别统计了杜洛克猪在标记间距为50 kb、500 kb、1 Mb和5 Mb时各染色体的LD分布,结果(表1)表明,SSC10的连锁不平衡程度较低,SSC6的连锁不平衡程度较高。
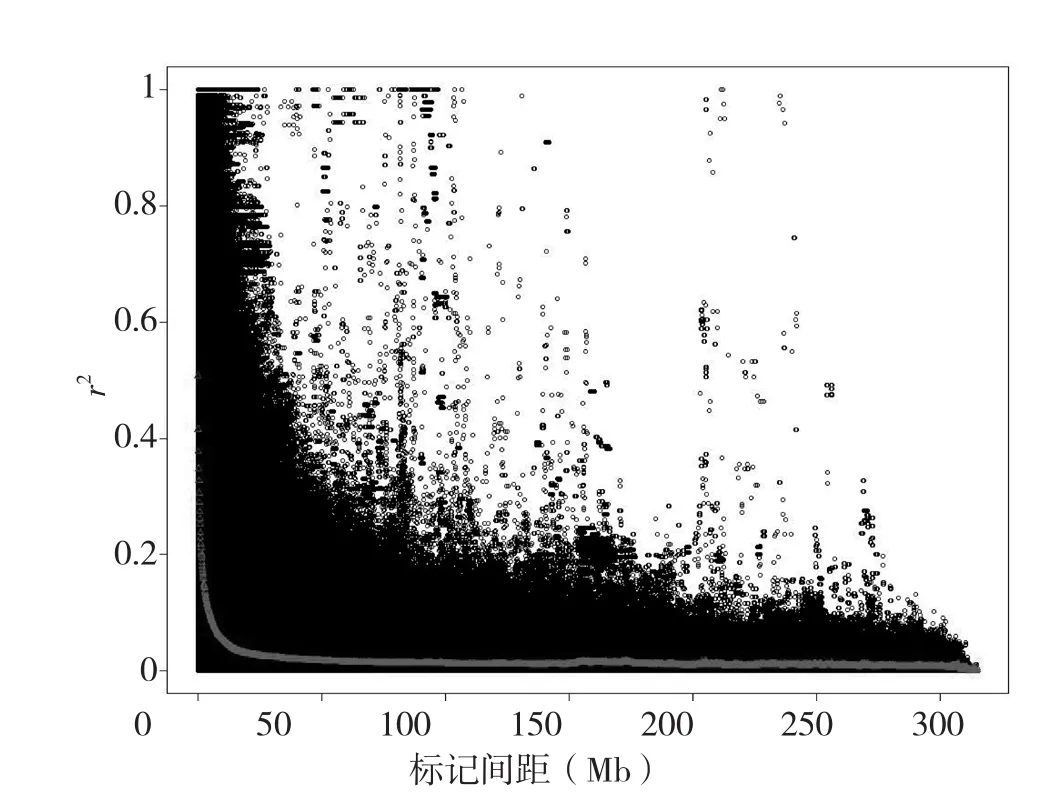
图3 杜洛克猪全基因组的r2分布

图4 杜洛克猪全基因组连锁不平衡图谱
3 结论与讨论
本研究利用Illumina Porcine SNP60K芯片,对216头杜洛克猪进行了全基因组LD分析,揭示了该杜洛克猪群体中LD的变化规律,成功构建了杜洛克猪的高密度连锁不平衡图谱,为杜洛克猪QTL定位、群体遗传结构分析及基因组选择等研究提供了参考。
在相邻标记(标记间距平均为82 kb)之间,该杜洛克猪群体的平均LD程度为0.52,结果与Badke等[16]对4个外种猪的LD研究结果(r2=0.46,相邻标记间距约为70 kb)相似。在每条染色体之间,杜洛克的LD程度波动范围为0.46~0.59。这说明杜洛克拥有较高的连锁不平衡水平,推测其在进化过程中受到高强度的人工选择,长期的选育也导致了杜洛克猪种的高度纯化,从而使其LD程度较高。此外,本研究比较了标记间距为50 kb、500 kb、1 Mb和5 Mb时各染色体的r2分布,发现SSC10的r2较小,SSC6的r2较大。这与Uimari等[17]对芬兰长白猪和芬兰约克夏猪LD进行分析的研究中SSC10 r2偏小的结果相同。同时,也与Lei等[18]对3个丹麦猪种进行LD分析研究得到SSC10的r2偏小的结果相同。
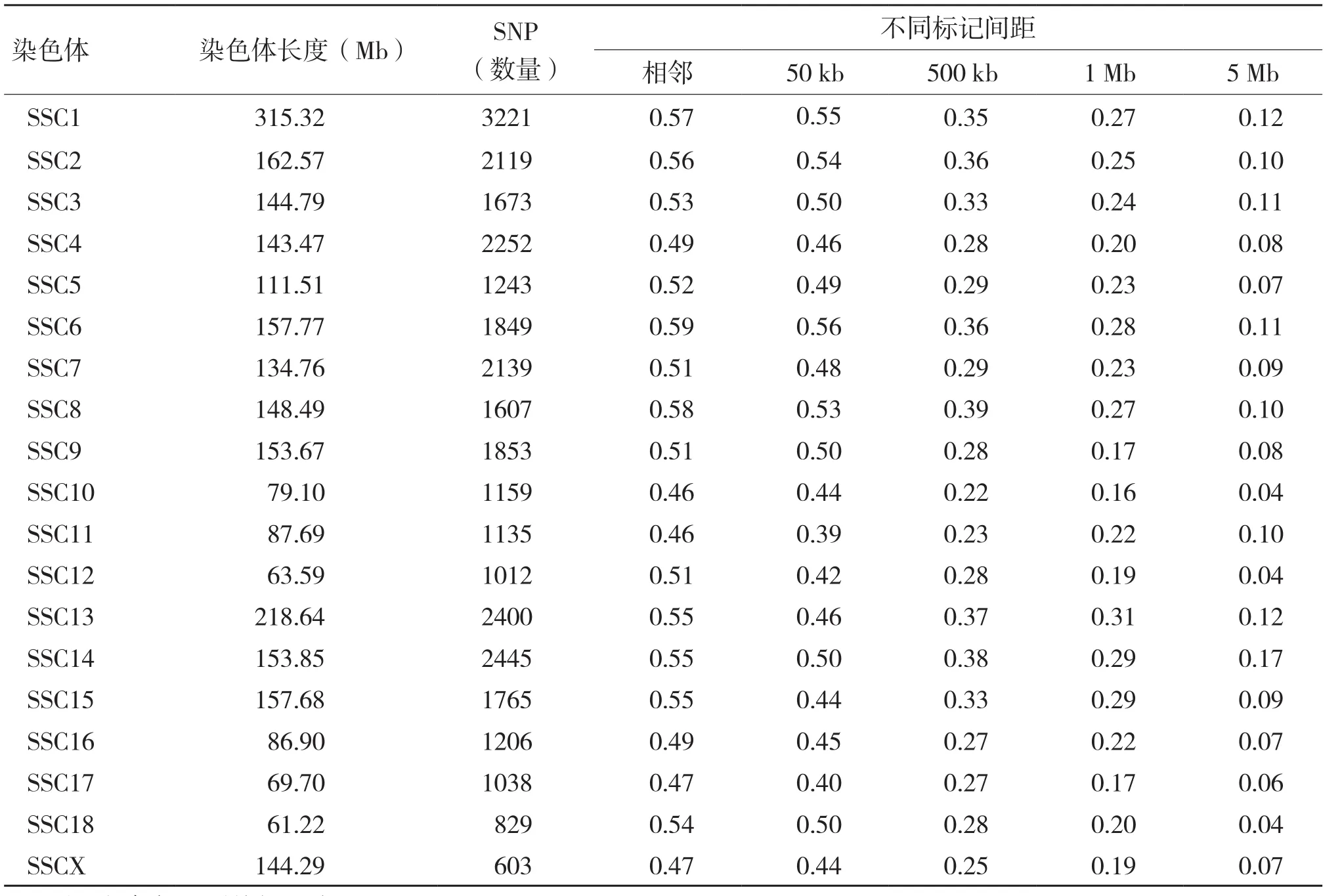
表1 杜洛克猪各染色体上标记间的r2分布情况
LD随着标记间距的增加呈衰减趋势,标记间r2逐渐降低,本研究结果与上述规律一致。但在本研究中发现某些距离较远的标记间也存在强LD,这可能和参考基因组序列有关[19-20]。通过比较不同物种间相邻标记间的连锁不平衡发现,杜洛克猪的LD程度高于人类的LD水平[21-22],也高于绵羊的LD水平[23]。参考GWAS和GS的研究,将有用的LD界定为相邻标记间平均r2>0.3[24],这在本研究中SNP密度是充足的,但质控后的SNP在基因组上呈非均匀分布,这可能会影响到SNP芯片在相关研究中的应用效果。
[1]Hill W G,Robertson A. Linkage disequilibrium in finite populations[J]. TAG. Theoretical and applied genetics. Theoretische und angewandteGenetik,1968,38(6):226-231.
[2]Wall J D,Pritchard J K. Haplotype blocks and linkage disequilibrium in the human genome[J]. Nat Rev Genet,2003,4(8):587-597.
[3]McKay S D,Schnabel R D,Murdoch B M,et al. Whole genome linkage disequilibrium maps in cattle[J]. BMC GENET,2007,8(1):74.
[4]Lu D,Sargolzaei M,Kelly M,et al. Linkage disequilibrium in Angus,Charolais,and Crossbred beef cattle[J]. Frontiers in Genetics,2012,3.
[5]de Roos Al APWD,Hayes B J,Spelman R J ,et al. Linkage Disequilibrium and Persistence of Phase in Holsten-Friesian,Jersey and Angus Cattle[J]. Genetics,2008,179(3):1503-1512.
[6]Amaral A J,Megens H J,Crooijmans R P M A,et al. Linkage Disequilibrium Decay and Haplotype Block Structure in the Pig[J]. Genetics,2008,179(1):569-579.
[7]Badke Y M,Bates R O,Ernst C W,et al Estimation of linkage disequilibrium in four US pig breeds[J]. Bmc Genomics,2012,1(13):1-10.
[8]Ai H S,Huang L S,Ren J. Genetic Diversity,Linkage Disequilibrium and Selection Signatures in Chinese and Western Pigs Revealed by Genome-Wide SNP Markers[J]. Plos One,2013,2(8):e56001.
[9]Ramos A M,Crooijmans R P M A,Affara N A,et al. Design of a High Density SNP Genotyping Assay in the Pig Using SNPs Identified and Characterized by Next Generation Sequencing Technology[J]. Plos One,2009,8(4):e6524.
[10]Heaton M P,Harhay G P,Bennett G L,et al. Selection and use of SNP markers for animal identification and paternity analysis in U.S. beef cattle[J]. Mamm Genome,2002,13(5):272-281.
[11]Purcell S,Neale B,Todd-Brown K,et al. PLINK:A Tool Set for Whole-Genome Association and Population-Based Linkage Analyses[J]. The American Journal of Human Genetics,2007,81(3):559-575.
[12]Lewontin R C. The Interaction of Selection and Linkage. I. General Considerations;Heterotic Models[J]. Gnentics,1964,1(49):49-67.
[13]Hill W G. Estimation of linkage disequilibrium in randomly mating populations.[J]. Heredity,1974,33(2):229-239.
[14]Zhao H,Nettleton D,Soller M,et al. Evaluation of linkage disequilibrium measures between multi-allelic markers as predictors of linkage disequilibrium between markers and QTL[J]. Genet Research,2005,86(1):77-87.
[15]Barrett J C,Fry B,Maller J,et al. Haploview: analysis and visualization of LD and haplotype maps[J]. Bioinformatics,2005,21(2):263-265.
[16]Badke Y M,Bates R O,Ernst C W,et al. Estimation of linkage disequilibrium in four US pig breeds[J]. Bmc Genomics,2012,1(13):1-10.
[17]Uimari P,Tapio M. Extent of linkage disequilibrium and effective population size in Finnish Landrace and Finnish Yorkshire pig breeds[J]. J Anim Sci,2011,89(3):609-614.
[18]Wang L,Rensen P S,Janss L,et al. Genomewide and local pattern of linkage disequilibrium and persistence of phase for 3 Danish pig breeds[J]. Bmc Genet,2013,1(14):1-11.
[19]Farnir F,Coppieters W,Arranz J J,et al. Extensive Genome-wide Linkage Disequilibrium in Cattle[J]. Genome Res,2000,10(2):220-227.
[20]Smith E M,Wang X,Littrell J,et al. Comparison of linkage disequilibrium patterns between the HapMap CEPH samples and a family-based cohort of Northern European descent[J]. Genomics,2006,88(4):407-414.
[21]Dunning A M,Durocher F,Healey C S,et al. The extent of linkage disequilibrium in four populations with distinct demographic histories[J]. Am J Hum Genet,2000,67(6):1544-1554.
[22]Kruglyak L. Prospects for whole-genome linkage disequilibrium mapping of common disease genes.[J]. Nat Genet,1999,2(22):139-144.
[23]Zhao F P,Wang G K,Zeng T,et al. Estimations of genomic linkage disequilibrium and effective population sizes in three sheep populations[J]. Livest Sci,2014,170:22-29.
[24]Ai H S,Huang L S,Ren J. Genetic Diversity,Linkage Disequilibrium and Selection Signatures in Chinese and Western Pigs Revealed by Genome-Wide SNP Markers[J]. Plos One,2013,8:e560012.
(责任编辑 崔建勋)
Genome-wide linkage disequilibrium analysis in Duroc pigherd
DIAO Shu-qi,LUO Yuan-yu,CAI Di,CHEN Gui-hua,CHEN Zan-mou,ZHANG Hao,LI Jia-qi,ZHANG Zhe
(College of Animal Science,South China Agricultural University,Guangzhou 510642,China)
In this study,we collected 216 Duroc pigs from a breeding herd in Fujian province and genotyped them with Illumina Porcine SNP60K Bead Chip. Based on the high-density SNP data,the genome-wide LD was calculated with Haploview. Results showed that LD reduced as the marker intervals increasing. The r2between adjacent markers ranged from 0.46 to 0.59 in the Duroc population. The mean r2was 0.52 among adjacent markers across the genome,with the minimum in SSC10 (r2=0.46),and the maximum in SSC6 (r2=0.59). This study provides useful information for further genome-wide analysis of Duroc pig.
pig;linkage disequilibrium;Duroc;SNP
S828.2;Q343.1
A
1004-874X(2016)11-0116-06
2016-08-10
广东省自然科学基金(2014A03031345);国家现代农业产业技术体系项目(CARS-36);华南农业大学大学生创新训练项目(201410564155)
刁淑琪(1993-),女,在读硕士生,E-mail:saradiao@126.com
张哲(1984-),男,博士,副教授,E-mail:zhezhang@scau.edu.cn
刁淑琪,罗元宇,蔡迪,等.杜洛克猪全基因组连锁不平衡分析[J].广东农业科学,2016,43(11):116-121.
