血清磷酸化肽的分离富集和质谱定量及其作为潜在肿瘤标志物的评价
2016-12-14翟贵金汪福意
翟贵金, 吴 魁, 汪福意,3*
(1. 中国科学院化学研究所, 中国科学院活体分析化学重点实验室, 北京 100190; 2. 天津市医学表观遗传学重点实验室, 天津医科大学生物化学与分子生物学系, 天津 300070; 3. 中国科学院大学, 北京 100049)
血清磷酸化肽的分离富集和质谱定量及其作为潜在肿瘤标志物的评价
翟贵金1,2, 吴 魁1, 汪福意1,3*
(1. 中国科学院化学研究所, 中国科学院活体分析化学重点实验室, 北京 100190; 2. 天津市医学表观遗传学重点实验室, 天津医科大学生物化学与分子生物学系, 天津 300070; 3. 中国科学院大学, 北京 100049)
血清中磷酸化肽种类和浓度的变化既能反映人体内蛋白质水解酶活性的变化,又能反映蛋白质翻译后磷酸化的水平,业已成为肿瘤标志物寻找和发现的重要目标。因而,血清中磷酸化肽的鉴定及其定量分析在具有临床应用价值的肿瘤标志物的筛选与发现中起着重要作用。由于血清中的内源性磷酸化肽丰度极低,在质谱分析中的离子化效率不高,且受到来自高丰度非磷酸化肽和蛋白质的信号抑制及干扰,血清中磷酸化肽的质谱定量分析是分析化学研究中的一个巨大挑战。文章对血清磷酸化肽的分离富集、质谱定量分析及其作为肿瘤标志物的筛选和评价等3个方面的研究进展进行总结、评述,并展望该领域的未来研究趋势和应用前景。
磷酸化肽;分离富集;质谱定量;肿瘤标志物;血清;综述
蛋白质的磷酸化是细胞信号转导过程的中心事件,几乎调节着包括细胞增殖、分化、代谢、凋亡以及侵袭等过程在内的所有生命活动[1-5]。蛋白质磷酸化的异常可引发和促进癌症等重大疾病的发生和发展[6]。血清中的磷酸化肽是血液蛋白质组的重要组成部分,一般源自细胞分泌和磷酸化蛋白质水解或者降解产生的肽段[7]。因而,血清中磷酸化肽种类和丰度的变化既能反映人体内蛋白质水解酶活性的变化,又能反映蛋白质翻译后磷酸化的水平,业已成为肿瘤标志物(tumor markers, TMs)寻找和发现的重要目标[8-10]。所以,对血清中磷酸化肽进行鉴定以及定量分析对具有临床应用价值和前景的肿瘤标志物的筛选与发现具有重要意义。
目前,分析技术尤其是质谱技术的快速发展,使得磷酸化蛋白质和磷酸化肽的发现与鉴定成为可能。质谱技术在复杂生物样品(如血清)中磷酸化肽段的定性与定量研究中的出色表现为发展用于肿瘤诊断的新技术提供了很好的机会[11]。但是,由于血清中内源性磷酸化肽丰度极低,在质谱分析中的离子化效率不高,且受到来自高丰度非磷酸肽和蛋白质的信号抑制及干扰[12-15],因而,血清中磷酸化肽的质谱定量分析是分析化学研究面临的一个巨大挑战。为解决磷酸化肽质谱定量分析中的这些突出问题,分析化学家们致力于发展对磷酸化肽具有特异性作用的高效分离富集材料,为磷酸化肽的质谱定量分析奠定了基础,同时也为血清磷酸化肽作为肿瘤标志物的筛选和评价提供了方法学保证。作为潜在肿瘤标志物的血清磷酸化肽的分析流程主要包括:(1)血清样品中磷酸化肽的高效分离富集;(2)磷酸化肽的质谱定性和定量分析;(3)基于大量临床样本定量分析数据的生物统计学分析,评价血清磷酸化肽作为肿瘤标志物的特异性和灵敏度。本文将对近年来在血清磷酸化肽分离富集、质谱定量分析以及其作为潜在肿瘤标志物的筛选和评价3个方面的研究进展进行总结和评述,并分析该领域存在的问题,展望其未来的发展趋势。
1 血清磷酸化肽的分离富集
血清是一个非常复杂的生物体系,其中含有大量的蛋白质,其浓度范围可达10个数量级,并且90%的总蛋白质由20多个高丰度的蛋白质组成[16]。血清中蛋白质与肽段等的复杂性以及高丰度蛋白质的影响使得基于质谱的血清磷酸化肽的分析表征变得异常困难。为了克服血清的复杂性以及来自非磷酸化蛋白质(肽段)的离子抑制效应,高效选择性地分离、富集磷酸化肽显得十分必要[17-19]。为此,分析化学家们发展了一系列的方法分离富集复杂体系中的磷酸化肽。其中最为常用的就是固定金属离子亲和色谱(immobilized metal affinity chromatography, IMAC)和金属氧化物亲和色谱(metal oxide affinity chromatography, MOAC)[4,20]。
1.1 固定金属离子亲和色谱
固定金属离子亲和色谱(IMAC)是目前广泛应用的磷酸化肽段富集方法。其原理为带正电的金属离子(如Fe3+、Al3+、Ga3+和Zr3+等)与带负电的磷酸基团产生静电相互作用而结合,这种结合能力受到pH值、离子强度以及溶液中有机相比例的影响。在低pH条件下,金属离子与磷酸基团稳定结合,将磷酸化肽从复杂体系中分离出来;在高pH值条件下,金属离子与磷酸基团间的结合被破坏,磷酸化肽被洗脱释放出来,经过必要的浓缩处理再进行质谱分析[21-23]。富集缓冲液中三氟乙酸(TFA)的浓度也会影响金属离子与磷酸化肽的结合。从理论上讲,如果TFA浓度过低,则不能有效地将非磷酸化肽段中所含氨基酸上的羧基质子化,但TFA浓度过高又会导致磷酸化肽中磷酸基团负电荷的减少。因而,针对不同的分析对象,需要对TFA的使用浓度进行优化。此外,在富集缓冲液中加入乙腈等有机相可以有效消除非磷酸化肽的非特异性吸附,但乙腈含量过高或过低,都会影响磷酸化肽的质谱检测信号强度,乙腈等有机相的用量同样需要仔细的实验优化[24]。
目前,IMAC技术不仅广泛用于磷酸化蛋白质组学研究中,也已成为血清中磷酸化肽分离富集的有力工具。Cirulli等[25]应用改进的固定金属离子亲和色谱法从人血清中成功富集并利用质谱鉴定了4条内源性磷酸化肽段。Li等[26]在Fe3O4@SiO2磁性硅球表面键合上螯合金属离子的官能团,然后采用IMAC原理,首次将Ce4+固定在磁球表面并应用于实际血样中磷酸化肽的富集。最近,Li等[27]利用一种磁性介孔硅将Ti4+嫁接在磁球上,制备了一种新的IMAC材料——Fe3O4@Ti-mSiO2磁性微球,并将其应用于脱脂牛奶以及人血清中内源性磷酸化肽的富集与分离。Lin等[18]将聚多巴胺覆盖在磁性石墨烯表面,将Hf4+固定在石墨烯上,合成了一种新的IMAC材料(magG@PDA-Hf4+),这种新型材料集成了石墨烯的大比表面积、多巴胺的亲水性和生物相容性以及Fe3O4的超顺磁性等特点,对磷酸化肽表现出良好的选择性和灵敏性。
但是,IMAC技术也有其局限性,其中最突出的问题是复杂肽段混合物中高丰度非磷酸化肽段的非特异性吸附。一些含有多个酸性氨基酸残基的非磷酸化肽段可以与磷酸化肽段一起吸附在材料上,从而造成对磷酸化肽段富集分离的选择性降低。邹汉法课题组发现了磷酸酯锆(钛)与磷酸化肽之间的高特异性相互作用,并利用这一相互作用建立了以磷酸基团为螯合配体的新一代固定化金属离子亲和色谱技术[19,28-30]。研究表明该新型IMAC材料对磷酸化肽富集的特异性高,可以有效地避免酸性非磷酸化肽段的非特异性吸附。与传统的IMAC材料相比,其对磷酸化肽的富集能力提高了3~10倍,大大提高了磷酸化蛋白质(肽段)的检测灵敏度和鉴定覆盖率(见图1)[31]。最近,冯钰琦课题组[32]合成了一种类似的新型IMAC材料——磁性砷酸酯锆纳米颗粒。应用这种纳米颗粒分离富集磷酸化肽,有效减少了非磷酸化肽的干扰,富集血清磷酸化肽的特异性和后续质谱检测的灵敏度都得到大幅度的提升。
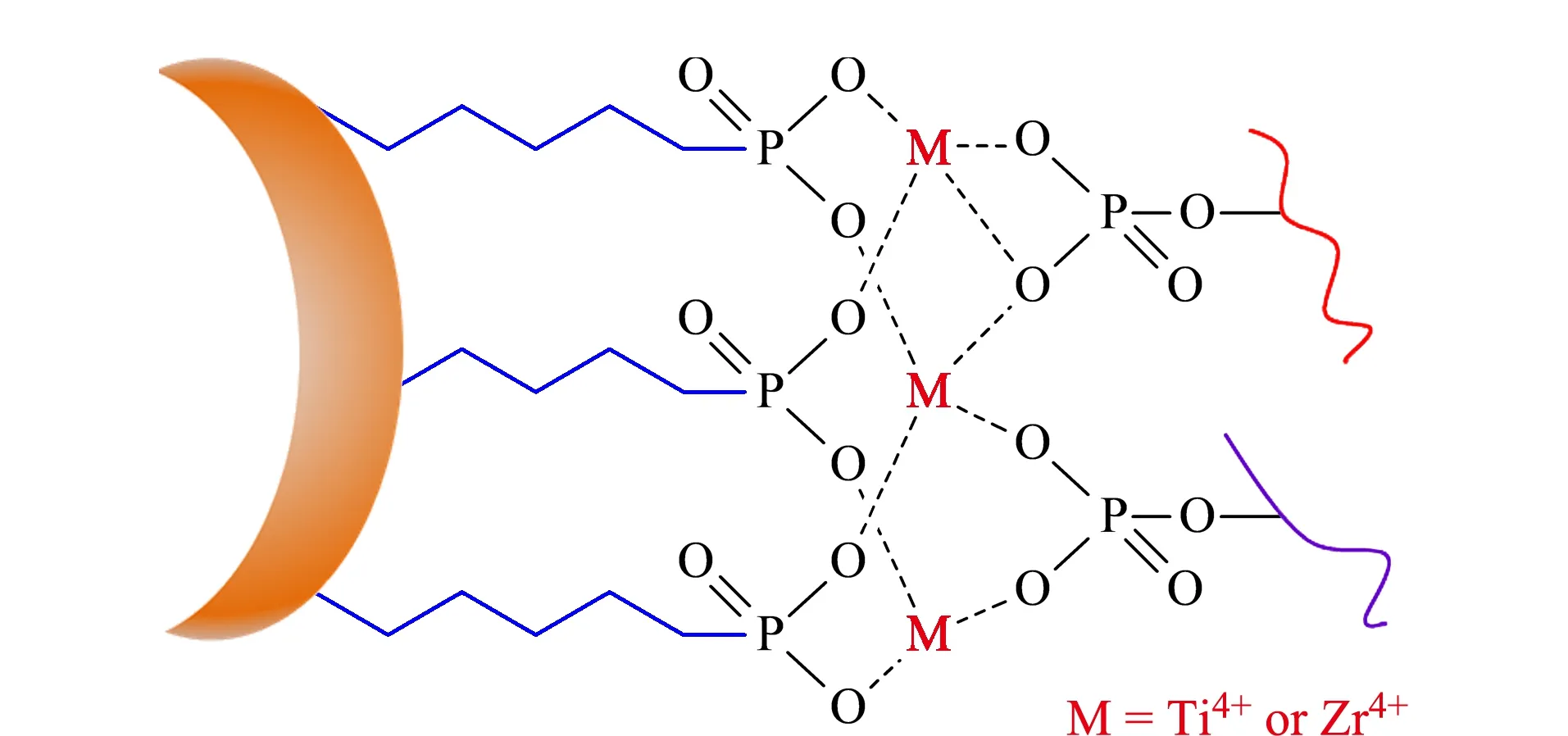
图 1 以磷酸基团为螯合配体的IMAC材料吸附 磷酸化肽段的机理Fig. 1 Mechanism of the phosphonate based-immobilized metal affinity chromatography (IMAC) adsorbents for phosphopeptide enrichment
IMAC材料的发展和改进显著提升了富集分离磷酸化肽的选择性,大大减少了非磷酸化肽的干扰,提高了磷酸化肽的富集效率。但是,IMAC材料从血清中富集的磷酸化肽的数量还是相对较少。最近,Zhu等[33]综合利用Ti (IV)-MCM-41[34]的分子排阻和富集功能、二维液相色谱分离以及集成碰撞诱导裂解(CID)-电子转移解离(ETD)的新型检索策略,从人血清中总共鉴定了143条可靠性较高的内源性磷酸化肽(涵盖133个磷酸化位点),大大提高了血清内源性磷酸化肽的鉴定效率。
1.2 金属氧化物亲和色谱
与IMAC相比,金属氧化物亲和色谱(MOAC)对磷酸化肽具有更高的富集能力以及更好的选择性,是近年来受到广泛关注的新型磷酸化蛋白/肽段富集技术。二氧化钛、二氧化锆以及三氧化二镓等材料都已经广泛应用于磷酸化肽的分离富集[26,35-37]。其中TiO2富集法是目前最为成熟的金属氧化物富集方法。TiO2在不同的pH值条件下,表现为路易斯酸或路易斯碱特性,在酸性条件下,钛原子带正电表现为路易斯酸,可以与阴离子结合;在碱性条件下,则表现为路易斯碱,可以与阳离子结合。这样磷酸化肽段的磷酸基在酸性条件下与之结合,在碱性条件下洗脱从而达到富集分离磷酸化肽的目的。冯钰琦课题组[37]构建了一种TiO2涂层的磁性空心多孔硅球(TiO2/MHMSS)材料,实现了复杂肽段混合物中磷酸化肽的高效分离富集。我们[35]应用这种TiO2/MHMSS材料从表皮生长因子受体(EGFR)介导的磷酸化反应混合物中分离富集痕量的磷酸化底物,发展了一种高效、低成本的EGFR抑制剂筛选方法。
MOAC材料一个突出的问题是金属氧化物(TiO2、ZrO2等)亲水性较差,导致其磷酸化肽富集效率降低。为解决这一问题,朱俊杰等[38]合成了一种Fe3O4-石墨烯-ZrO2三元复合网状材料,借助石墨烯网格结构提升ZrO2与溶液中磷酸化肽的接触,提高MOAC材料对磷酸化肽的富集能力。另一种克服金属氧化物亲水性差的方法是以纳米尺度的金属氧化物构建MOAC材料,通过纳米材料较大的比表面积增加金属氧化物与磷酸化肽的接触。如冯钰琦等[39]采用液相沉积(LPD)技术制备了一种TiO2纳米颗粒涂层的毛细管柱,显著提高了MOAC材料分离富集磷酸化肽的效率,还实现了磷酸化肽分离富集和质谱分析的在线联用。我们课题组[40]将这种TiO2纳米颗粒涂层的毛细管应用于基于质谱分析的蛋白激酶抑制剂筛选,大大提高了筛选通量。最近,Liu等[41]将1,6-己二胺修饰在TiO2纳米颗粒表面,构建了一种亲和型TiO2杂交材料(NH2@TiO2),并作为一种生物功能吸附剂用于血清磷酸化肽的选择性富集,磷酸基团与氨基基团之间的作用大大提高了磷酸化肽的富集效率。另外,从血清等复杂生物样品中分离富集磷酸化肽时,TiO2纳米颗粒的富集能力容易受到其他高丰度蛋白质的影响;为此,Yao等[42]基于介孔硅的分子排阻特性,构建了一种TiO2杂交材料(G@TiO2@mSiO2)。该材料用于血清内源性磷酸化肽富集分析时,表现出了很好的灵敏度、选择性以及分子排阻效应,为生物标志物的发现以及临床诊断应用提供了有效的分析平台。
最近,冯钰琦课题组[43]发展了一种新型的磷酸化肽段分离富集技术:阴离子交换色谱法(anion-exchange chromatography, AEX)。他们设计、合成了一种磁性石墨氮化碳材料,由于该材料表面含有大量氮以及碱性功能基团,可以在较低pH条件下保持对磷酸化肽段的较强吸附特性。他们应用该方法成功实现了人血清以及兔脑中磷酸化肽的分离富集。
血清磷酸化肽分离富集原理和流程与磷酸化肽(或蛋白质)类似:在低pH条件下磷酸化肽与富集材料特异性结合,然后在高pH条件下实现所富集磷酸化肽的洗脱。但在血清磷酸化肽富集材料的设计上,引入了分子排阻的理念并结合具有较小孔径的多孔硅材料,以去除血清中大量高丰度蛋白质的非特异性吸附干扰[34];而对磷酸化蛋白质的分离富集往往很少直接利用IMAC或者MOAC,因为清洗过程中会出现大量低吸附磷酸化蛋白质的丢失,从而导致磷酸化蛋白质富集效率的降低。因此,在磷酸化蛋白质富集材料的制备方面,常常引入介孔纳米材料(具有可控的纳米孔径),以提高材料的比表面积和亲水性,降低非特异性吸附,达到提高磷酸化蛋白质分离富集效率的目的[44]。
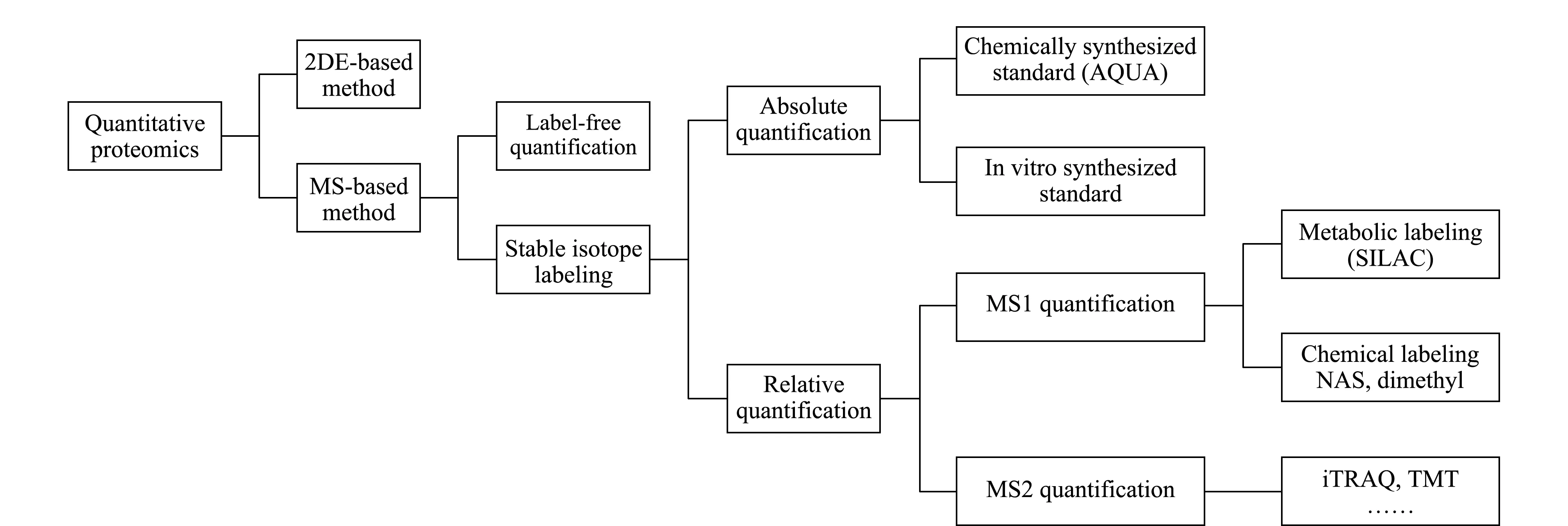
图 2 定量蛋白质组学常用的分析方法Fig. 2 Strategies of quantitative proteomic analysis
2 血清磷酸化肽的质谱定量分析
随着分子生物学、细胞生物学及肿瘤生物学等生命学科的快速发展,人们认识到生物体内磷酸化蛋白质/肽段丰度变化更能反映生命活动过程或疾病的发生、发展的状态,表达量或丰度发生显著变化的磷酸化蛋白质/肽段有可能成为药物靶标或者疾病诊断的生物标志物。因此,分析化学家们发展了很多用于蛋白质以及蛋白质翻译后修饰定量研究方法,以期深入探索蛋白质磷酸化以及磷酸化多肽水平的动态变化与癌症等重大疾病发生、发展的关系。图2列出了定量蛋白质组学分析中常用的方法。
2.1 相对质谱定量
稳定同位素标记方法与质谱技术的结合广泛应用于定量蛋白质组学研究领域,这些方法主要包括同位素代谢标记(如stable isotope labeling by amino acids in cell culture, SILAC[45])、基于化学反应的同位素标记方法(如isotope coded affinity tag[46](ICAT)、isobaric tags for relative and absolute quantification[47](iTRAQ)和tandem mass tags[48](TMT)等)以及无同位素标记的相对定量法等。在这些方法中,同位素代谢标记技术或许是最精确的质谱定量方法。但是,该方法通常比较昂贵。Hu等[34]利用iTRAQ对分离富集获得的磷酸化肽进行了标记,并利用MALDI-TOF MS进行定量分析。
相反,化学标记通常简便、高效并且经济,在定量蛋白质组学领域中的应用更为广泛。其中一种化学标记策略是利用甲醛反应在肽段的N端和赖氨酸侧链引入两个甲基,反应完成后,轻标试剂可以在肽段N端引入两个CH3,而中/重标分别引入CHD2和13CD3。最后3组(轻、中、重)肽段之间均有4 Da的质量差异[49-51]。最近,Chen等[52]利用此化学标记策略对从血清中分离富集的磷酸化肽进行原位标记,实现了白血病患者与正常个体血清样品中磷酸化肽的质谱定量分析。
另外一个化学反应标记试剂,N-乙酸基琥珀酰亚胺(NAS),能够特异性地与肽段N端和赖氨酸残基侧链反应而引入乙酰基对肽段进行标记。此策略简单、经济,在蛋白质组学定量研究方面有着广阔的应用前景[53-58]。我们在利用NAS对人血清中的磷酸化肽进行标记时发现,当标记只发生在肽段N端时,因其轻重同位素标签仅有3 Da的差别,质谱分析时轻重标记肽段的质谱峰存在重叠,给定量工作带来了一定的困难,必须结合同位素峰模拟去除重叠部分予以校正处理[59]。
2.2 绝对质谱定量
绝对质谱定量的基本原理就是在待测样品中加入已知量的稳定同位素标记的内标,类似于同位素稀释分析法。因为磷酸化肽的相对定量分析并不能完全反映生物基体状态或者对外界刺激的响应程度,所以特定位点磷酸化的绝对定量分析是必要的。磷酸化肽的绝对定量分析可以实现不同条件下,目标肽段上多个磷酸化位点的定量分析。近年来,分析化学家们也发展了多种绝对定量的分析方法。其中最为常用的当属Gygi课题组发展的AQUA(the absolute quantification)方法[60]。该方法直接将合成的同位素标记肽加入到样品中,基于内源性目标肽段与同位素标记肽段的色谱峰面积比率来进行绝对定量分析。尽管该方法非常直接简便,但其依据单点校正,容易引起分析误差,尤其是在不同样品之间内源性肽段动态范围较宽或者肽段浓度不在线性响应之内的情况下。我们基于化学标记策略,利用合成的磷酸化肽标准物质建立了多点标准曲线,实现了血清中内源性磷酸化肽的绝对定量分析,提高了质谱绝对定量分析的准确性[59]。最近,我们在合成磷酸化肽标准物时,直接在肽段中引入两个轻、重同位素标记的甘氨酸残基,既避免了肽段化学标记反应不完全可能导致的误差,也避免了单一N-端化学标记时轻重肽段同位素质谱峰的重叠[59],进一步提高了血清磷酸化肽质谱绝对定量分析的准确性。
目前,血清中磷酸化肽的质谱定量工作,主要针对其中源于磷酸化蛋白原(fibrinogen)的4条磷酸化肽段,对其他139条[33]已经被明确鉴定的磷酸化肽段几乎没有涉及。这主要是因为血清中绝大部分磷酸化肽的丰度极低,富集后的质谱分析仍然不能给出超低丰度磷酸化肽的定量结果,从一个侧面给磷酸化肽的分离富集提出了更高的要求。另一方面,当前的磷酸化蛋白质组学主要集中于对蛋白质翻译后磷酸化过程、磷酸化位点及其生物学意义的研究,血清磷酸化肽作为潜在肿瘤标志物的筛选和评价研究还处于起始阶段,缺乏大规模临床样品分析数据,这为我们在这一领域更深入的研究提供了机遇。
3 血清磷酸化肽作为肿瘤标志物的筛选
肿瘤标志物是一类在肿瘤发生、发展过程中由肿瘤细胞所产生、分泌并释放到血液、体液及组织中,能反映肿瘤存在和生长的一类物质[61,62]。过去50年来,通过科研工作者们的不断努力,许多肿瘤标志物得以应用于临床诊断。如前列腺特异性抗原(PSA)用于诊断前列腺癌,甲胎蛋白(AFP)用于诊断肝癌,癌抗原CA-125用于诊断卵巢癌,癌胚抗原(CEA)和癌抗原CA19-9用于诊断结直肠癌及胃癌等。但是,它们在应用于癌症早期临床诊断时都缺乏足够高的特异性与灵敏度。因此,为更有效地筛查和诊断癌症,寻找新的具有更高临床诊断特异性与灵敏度的生物标志物已经迫在眉睫。
血清中磷酸化肽段同时包含了蛋白质磷酸化修饰水平和蛋白酶活性动态变化的信息,为肿瘤标志物研究提供了一种全新的视角。但是,关于血清磷酸化肽作为肿瘤标志物的研究还较少。Hu等[34]利用磷酸钛修饰的介孔硅颗粒对血清中的磷酸化肽进行分离富集,并结合iTRAQ标记,对12位肝癌患者与12位健康志愿者血清中的磷酸化肽进行了定量研究。与健康志愿者相比,其中两条磷酸化肽在癌症患者血清中的水平有显著变化,反映了某些相关酶有可能成为肝癌检测的生物标志物。Qin等[63]发展了一种磷酸功能化的介孔硅纳米材料,实现了血清中内源性磷酸化肽段的原位富集与标记,通过质谱相对定量的方法对10个肝癌患者与10个健康者血清中的内源性磷酸化肽进行了比较分析,发现其中3条磷酸化肽段在两组之间的丰度差异显著。最近,Min等[64]利用TiO2纳米管阵列与同位素标记技术对7个卵巢癌与7个正常个体血清中磷酸化肽进行了定量分析,发现其中两条磷酸化肽的丰度有明显变化;Chen等[52]合成了一种新型纳米颗粒——Fe3O4-TiNbNS用于磷酸化肽的分离富集,并通过化学标记结合质谱定量分析对8个白血病患者与8个健康志愿者血清磷酸化肽水平进行了表征。但是,上述研究都是基于质谱相对定量分析进行的,不适合生物标志物筛选和发现研究中大规模、多样品之间的比较分析。
我们发展了一种基于化学标记的质谱绝对定量分析方法,分析了20个胃癌患者与20个正常个体血清中磷酸化肽的绝对浓度[59]。我们发现其中一条磷酸化肽(F3)的丰度在两组之间差异显著(见图3),有可能成为胃癌诊断的生物标志物。但其在临床诊断方面的应用前景还需要通过对更多样本、更具代表性人群的分析,对其诊断灵敏度与特异性进行进一步的系统验证。此外,鉴于血清中的磷酸化肽来自磷酸化蛋白质酶解,其浓度水平反映了蛋白酶、外肽酶、激酶、磷酸酶等在体内的水平和活力,是各种酶复杂作用的平衡结果,所以,磷酸化肽浓度的变化与肿瘤发生发展之间的关系以及更明确的分子机理还需要进一步的研究。
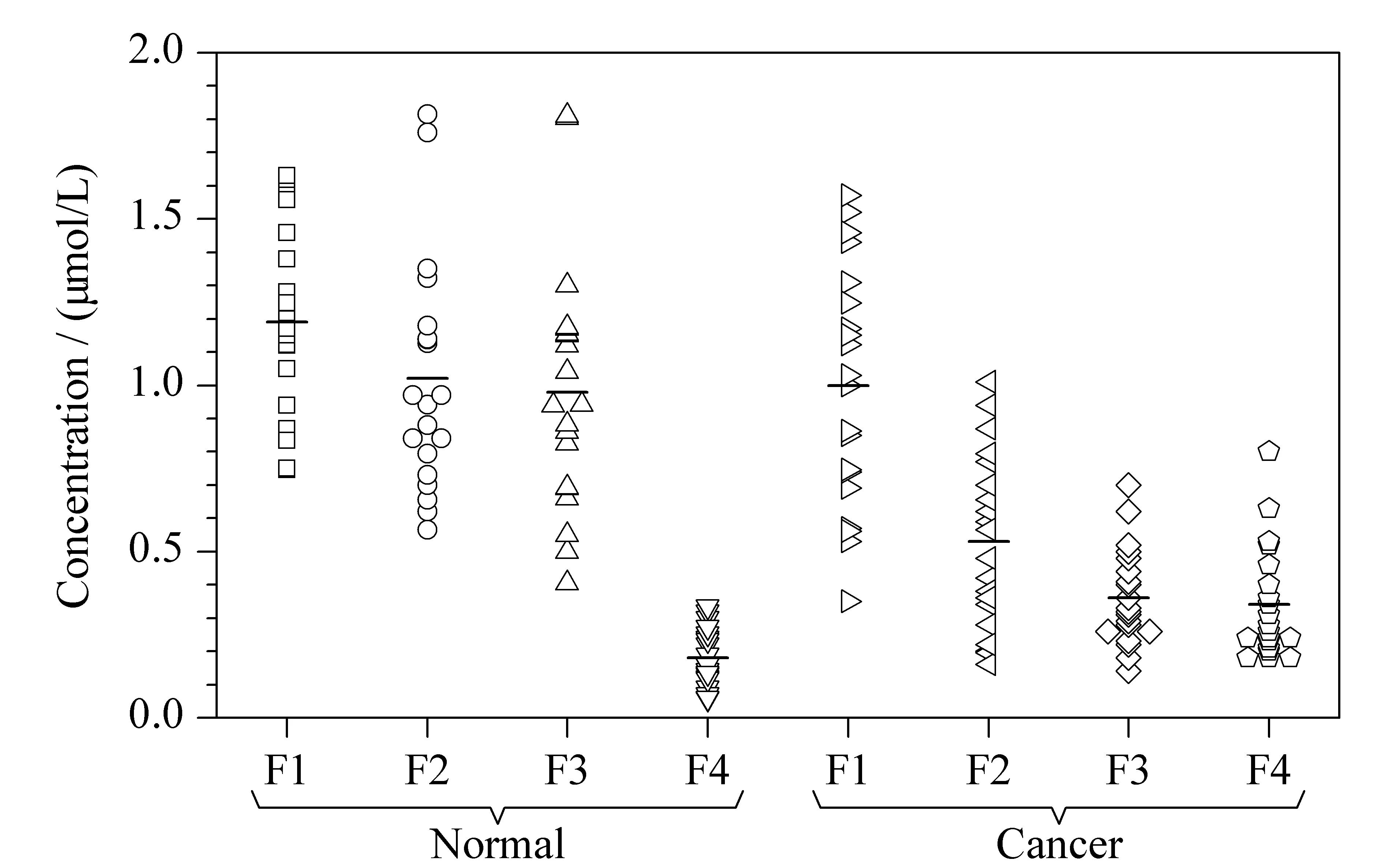
图 3 血清中磷酸化肽浓度散点分布图Fig. 3 Scatter plots of the concentration distribution of the four endogenous phosphopeptides F1-F4 in serum samples
4 结论与展望
综上所述,磷酸化肽在质谱分析中容易受到高丰度非磷酸化肽等基体物质的干扰和离子抑制,因此,在进行质谱分析前,需要对其进行有效的分离富集。在过去的10多年中,磷酸化肽的分离富集新方法大量涌现。这些方法在磷酸化肽的富集效率、选择性以及灵敏度等方面都有很大的提升,对复杂生物体系(如血清)中磷酸化肽的分析表征带来了诸多益处,使得大规模磷酸化肽组的分析成为可能。
质谱定量方法,特别是基于化学反应标记策略的质谱定量分析法,在血清磷酸化肽的定量分析中应用广泛。另外,质谱绝对定量分析方法的建立,使得大规模、多样品之间的分析比较成为可能,为血清磷酸化肽作为肿瘤标志物的筛选和评价提供了技术平台。
从血清中筛选和发现有效的肿瘤生物标志物,特别是对早期癌症有高特异性与灵敏度的诊断标志物,是一项具有挑战性的工作。关于血清磷酸化肽作为肿瘤标志物的筛选和评价的报道还较少,系统性的研究尤其缺乏。这也给血清磷酸化肽质谱定量-肿瘤标志物的筛选留下了进一步的研究空间。未来重点应在几个方面予以开展:加大样本量,使样本更具代表性,包括增加良性肿瘤患者、炎症患者、患有凝血方面疾病的患者的血清样本等;目前的研究大多是推断潜在磷酸化肽肿瘤标志物浓度的变化与体内酶活性变化有关,但没有对特定酶的活性进行表征,还需要相关的实验去证实;此外,检测治疗前后或手术前后患者血清中潜在磷酸化肽肿瘤标志物浓度的变化,可以发现标志物与临床治疗之间的关系,验证其作为一种肿瘤治疗标志物的可能性。我们相信,通过这些系统性的分析和验证,未来一定有高特异性与灵敏度的血清磷酸化肽被筛选出来用于肿瘤的临床筛查与诊断。
[1] Hunter T. Cell, 2000, 100: 113
[2] Paradela A, Albar J P. J Proteome Res, 2008, 7: 1809
[3] Reinders J, Sickmann A. Proteomics, 2005, 5: 4052
[4] Thingholm T E, Jensen O N, Larsen M R. Proteomics, 2009, 9: 1451
[5] Zanivan S, Meves A, Behrendt K, et al. Cell Rep, 2013, 3: 552
[6] Zhang Y Y, Fonslow B R, Shan B, et al. Chem Rev, 2013, 113: 2343
[7] He K, Wen X Y, Li A L, et al. PLoS One, 2013, 8: e63724
[8] Villanueva J, Martorella A J, Lawlor K, et al. Mol Cell Proteomics, 2006, 5: 1840
[9] Liotta L A, Petricoin E F. J Clin Invest, 2006, 116: 26
[10] Lee H J, Na K, Kwon M S, et al. Proteomics, 2009, 9: 3395
[11] Jensen O N. Nat Rev Mol Cell Bio, 2006, 7: 391
[12] Zhou W D, Ross M M, Tessitore A, et al. J Proteome Res, 2009, 8: 5523
[13] Ficarro S B, McCleland M L, Stukenberg P T, et al. Nat Biotechnol, 2002, 20: 301
[14] Lu Z D, Duan J C, He L, et al. Anal Chem, 2010, 82: 7249
[15] Zou Y, Jiang W H, Zou L J, et al. Chinese Journal of Chromatography, 2013, 31(4): 367
邹瑶, 姜武辉, 邹丽娟, 等. 色谱, 2013, 31(4): 367
[16] Anderson N L, Anderson N G. Mol Cell Proteomics, 2002, 1: 845
[17] Oda Y, Nagasu T, Chait B T. Nat Biotechnol, 2001, 19: 379
[18] Lin H Z, Deng C H. Talanta, 2016, 149: 91
[19] Zhou H J, Ye M L, Dong J, et al. J Proteome Res, 2008, 7: 3957
[20] Li X S, Yuan B F, Feng Y Q. TrAC-Trends Anal Chem, 2016, 78: 70
[21] Posewitz M C, Tempst P. Anal Chem, 1999, 71: 2883
[22] Trojer L, Stecher G, Feuerstein I, et al. J Chromatogr A, 2005, 1079: 197
[23] Zhao Y Y, Guo Z M, Li X L, et al. Chinese Journal of Chromatography, 2013, 31(8): 763
赵艳艳, 郭志谋, 李秀玲, 等. 色谱, 2013, 31(8): 763
[24] Kokubu M, Ishihama Y, Sato T, et al. Anal Chem, 2005, 77: 5144
[25] Cirulli C, Chiappetta G, Marino G, et al. Anal Bioanal Chem, 2008, 392: 147
[26] Li Y, Lin H Q, Deng C H, et al. Proteomics, 2008, 8: 238
[27] Li X S, Pan Y N, Zhao Y, et al. J Chromatogr A, 2013, 1315: 61
[28] Zhou H J, Xu S Y, Ye M L, et al. J Proteome Res, 2006, 5: 2431
[29] Feng S, Ye M L, Zhou H J, et al. Mol Cell Proteomics, 2007, 6: 1656
[30] Yu Z Y, Han G H, Sun S T, et al. Anal Chim Acta, 2009, 636: 34
[31] Zhou H J, Ye M L, Dong J, et al. Nat Protoc, 2013, 8: 461
[32] Li X S, Xu L D, Zhu G T, et al. Analyst, 2012, 137: 959
[33] Zhu J, Wang F J, Cheng K, et al. J Proteomics, 2013, 78: 389
[34] Hu L H, Zhou H J, Li Y H, et al. Anal Chem, 2009, 81: 94
[35] Ji L Y, Wu J H, Luo Q, et al. Anal Chem, 2012, 84: 2284
[36] Kweon H K, Hakansson K. Anal Chem, 2006, 78: 1743
[37] Wu J H, Li X S, Zhao Y, et al. Chem Commun, 2010, 46: 9031
[38] Min Q H, Zhang X X, Zhang H Y, et al. Chem Commun, 2011, 47: 11709
[39] Lin B, Li T, Zhao Y, et al. J Chromatogr A, 2008, 1192: 95
[40] Lu S, Luo Q, Li X C, et al. Analyst, 2010, 135: 2858
[41] Liu H L, Yang T Y, Dai J Y, et al. Analyst, 2015, 140: 6652
[42] Yao J, Sun N, Deng C, et al. Talanta, 2016, 150: 296
[43] Zhu G T, He X M, Chen X, et al. J Chromatogr A, 2016, 1437: 137
[44] Lu Z D, Ye M M, Li N, et al. Angew Chem Int Edit, 2010, 49: 1862
[45] Ong S E, Blagoev B, Kratchmarova I, et al. Mol Cell Proteomics, 2002, 1: 376
[46] Gygi S P, Rist B, Gerber S A, et al. Nat Biotechnol, 1999, 17: 994
[47] Ross P L, Huang Y L N, Marchese J N, et al. Mol Cell Proteomics, 2004, 3: 1154
[48] Thompson A, Schafer J, Kuhn K, et al. Anal Chem, 2003, 75: 1895
[49] Boersema P J, Foong L Y, Ding V M Y, et al. Mol Cell Proteomics, 2010, 9: 84
[50] Hsu J L, Huang S Y, Chow N H, et al. Anal Chem, 2003, 75: 6843
[51] Wu C J, Chen Y W, Tai J H, et al. J Proteome Res, 2011, 10: 1088
[52] Chen X Q, Li S Y, Zhang X X, et al. Nanoscale, 2015, 7: 5815
[53] Chakraborty A, Regnier F E. J Chromatogr A, 2002, 949: 173
[54] Ji J Y, Chakraborty A, Geng M, et al. J Chromatogr B, 2000, 745: 197
[55] Zhang R J, Sioma C S, Wang S H, et al. Anal Chem, 2001, 73: 5142
[56] Kindy J M, Taraszka J A, Regnier F E, et al. Anal Chem, 2002, 74: 950
[57] Xun Z Y, Sowell R A, Kaufman T C, et al. Mol Cell Proteomics, 2008, 7: 1191
[58] Du Z F, Luo Q, Yang L P, et al. J Am Chem Soc, 2014, 136: 2948
[59] Zhai G J, Wu X Y, Luo Q, et al. Talanta, 2014, 125: 411
[60] Gerber S A, Rush J, Stemman O, et al. P Natl Acad Sci USA, 2003, 100: 6940
[61] Zhang X W, Li L, Wei D, et al. Trends Biotechnol, 2007, 25: 166
[62] Diamandis E P. J Natl Cancer I, 2010, 102: 1462
[63] Qin H Q, Wang F J, Wang P Y, et al. Chem Commun, 2012, 48: 961
[64] Min Q H, Chen X Q, Zhang X X, et al. Lab Chip, 2013, 13: 3853
Separation, enrichment, mass spectrometric quantification and evaluation of serum phosphopeptides as potential tumor biomarkers
ZHAI Guijin1,2, WU Kui1, WANG Fuyi1,3*
(1.CASKeyLaboratoryofAnalyticalChemistryforLivingBiosystems,InstituteofChemistry,ChineseAcademyofSciences,Beijing100190,China; 2.TianjinKeyLaboratoryofMedicalEpigenetics,DepartmentofBiochemistryandMolecularBiology,TianjinMedicalUniversity,Tianjin300070,China;3.UniversityofChineseAcademyofSciences,Beijing100049,China)
Serum phosphopeptides has been a vital objective for the discovery of tumor biomarkers as their levels can reflect the changes in the activities of both endo- and exo-proteases, and the level of protein phosphorylation. Comprehensive analysis of serum phosphopeptides may lead to discovery of novel tumor biomarkers for clinical diagnosis. However, the extremely low abundance of serum phosphopeptides as well as high background level of highly abundant non-phosphoproteins/peptides makes the mass spectrometry (MS)-based phosphopeptide profiling a great challenge. In this review, we will summarize and review update advances in the separation, enrichment and MS quantification of phosphopeptides as well as the evaluation of serum phosphopeptides as potential tumor biomarkers. We will also prospect the future tendency and applications of this multi-disciplinary research field.
phosphopeptides; separation and enrichment; mass spectrometric quantification; tumor biomarkers; serum; review
10.3724/SP.J.1123.2016.08037
2016-08-30
国家自然科学基金项目(21135006,21127901,21275148,21575145,21321003);国家重点基础研究发展计划项目(2013CB531805).
Foundation item: National Natural Science Foundation of China (Nos. 21135006, 21127901, 21275148, 21575145, 21321003); National Program on Key Basic Research Project of China (“973” Program) (No. 2013CB531805).
O658
A
1000-8713(2016)12-1192-07
邹汉法研究员纪念专辑(上)·专论与综述
* 通讯联系人.Tel:(010)62529069,E-mail:fuyi.wang@iccas.ac.cn.
