单克隆抗体药物糖基化修饰分析研究进展
2016-12-14丛宇婷胡良海
丛宇婷, 胡良海
(吉林大学生命科学学院, 吉林 长春 130012)
单克隆抗体药物糖基化修饰分析研究进展
丛宇婷, 胡良海*
(吉林大学生命科学学院, 吉林 长春 130012)
单克隆抗体药物是一类以免疫球蛋白G的结构为基础的大分子糖蛋白药物,为癌症、自身免疫疾病以及病毒感染等多种疾病的治疗提供了全新的途径。单抗药物的糖基化修饰类型及水平对其稳定性、清除率、免疫原性、抗体依赖细胞毒性及补体依赖细胞毒性等都有一定的影响。单抗药物的迅速发展及其在多种疾病治疗中日益凸显的重要性都对单抗药物的研发及用药安全等方面提出了更高的要求。因此,建立规范可靠的单抗药物糖基化修饰分析方法有着十分重要的意义。该综述将简要介绍单克隆抗体药物糖基化修饰及相关的定性、定量分析方法。
单克隆抗体药物;糖基化修饰;质谱;蛋白质组学;综述
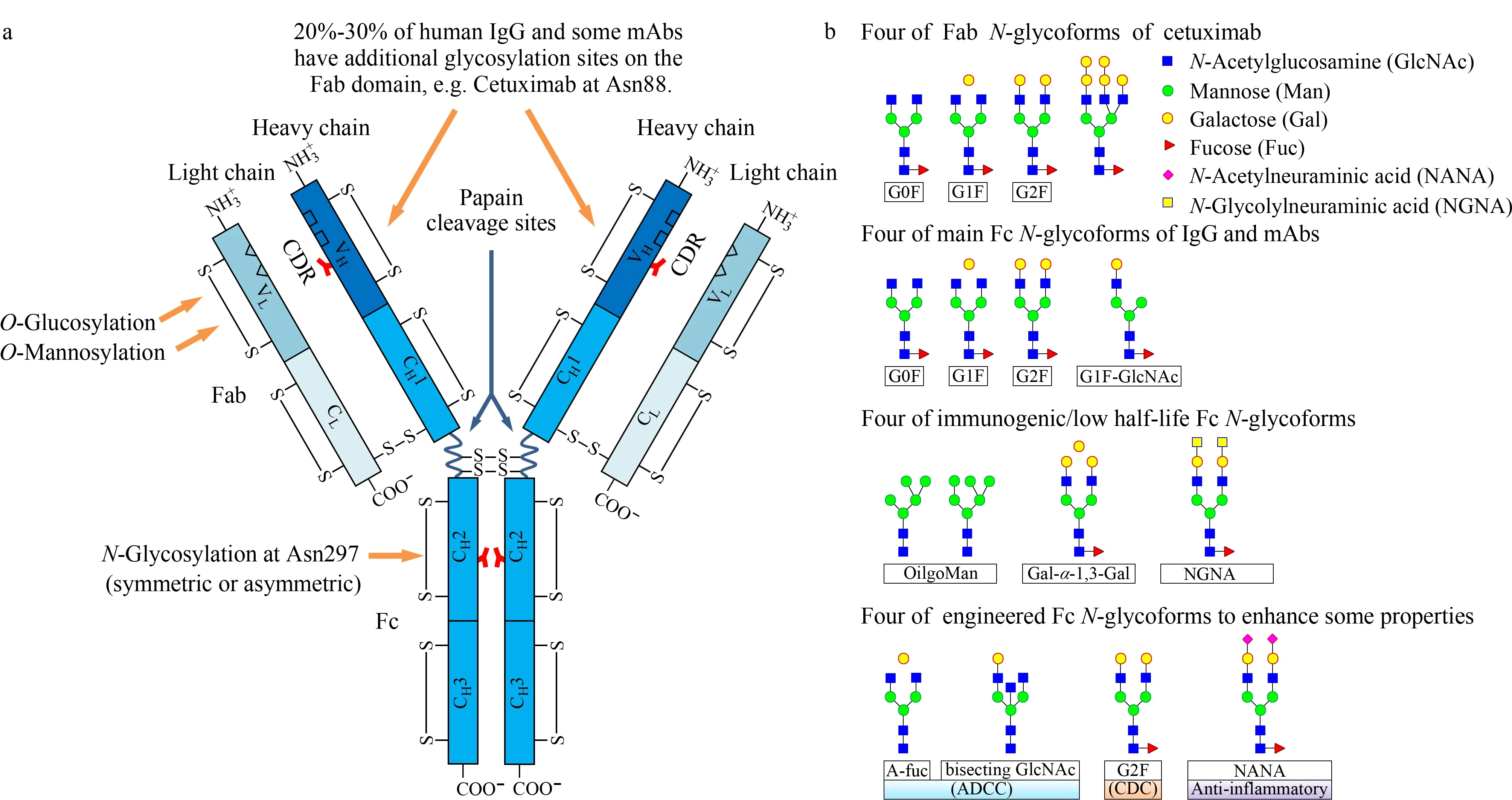
图 1 单抗药物分子结构及其糖基化修饰示意图Fig. 1 (a) General structure of therapeutic monoclonal antibodies and (b) the diagram of glycosylation of therapeutic monoclonal antibodies IgG: immunoglobulin G; mAb: therapeutic monoclonal antibody; VH: variable domain of heavy chain; VL: variable domain of light chain; CH1, CH2, and CH3: constant domains of heavy chain; CL: constant domain of light chain; CDR: complementarity determining regions; Fab: fragment antigen binding part; Fc: fragment crystallizable part; ADCC: antibody-dependent cellular cytotoxicity; CDC: complement-dependent cytotoxicity; G0F: GlcNAc4Man3Fuc1; G1F: GlcNAc4Man3Gal1Fuc1; G2F: GlcNAc4Man3Gal2Fuc1; A-fuc: GlcNAc4Man3Gal1.
1 单克隆抗体药物及其糖基化修饰简介
单克隆抗体药物(therapeutic monoclonal antibody,mAb;以下简称单抗药物)是一类以γ型免疫球蛋白G(immunoglobulin G,IgG)为结构基础的大分子蛋白类药物,结构如图1a所示。1975年,Kohler和Milstein[1]证明,骨髓瘤细胞与经抗原免疫的动物脾细胞融合,形成的融合细胞能分泌针对该抗原的均一、高特异性的抗体——单克隆抗体,这种技术称为杂交瘤技术[1-3],这一发现实现了一个世纪前Ehrlich提出的通过抗体靶向治疗肿瘤的设想[4]。随着杂交瘤技术的发展,第一支单抗药物——莫罗单抗(muromomab)于1986年由美国食品和药品管理局(United States Food and Drug Administration,FDA)批准上市[1],从此,单抗药物逐渐走入人们视野并得到了越来越多的关注。作为最重要的生物药物之一,单抗药物为多种疾病的治疗提供了全新的途径,包括心血管疾病、中枢神经紊乱、自身免疫疾病、病毒感染以及癌症等[4-11]。最初的单抗药物为鼠源单抗,该类型单抗药物具有典型的免疫原性且不易诱导免疫效应反应,因此限制了它们的临床应用。随着抗体工程的发展,嵌合单抗以及人源化单抗相继问世,最终B淋巴细胞cDNA库和噬菌体展示技术的发展使完全人源单抗成为可能,第一支完全人源单抗——阿达木单抗(adalimumab)已成功研制并于2002年由FDA批准上市[12]。完全人源单抗具有良好的功效,并且无副作用,已成为当前发展最迅速的一类单抗药物。目前,已有超过50种单抗药物由FDA和欧盟医药产品管理机构(European Medicine Agency,EMA)批准上市,还有四十余种单抗药物处于临床Ⅲ期研究阶段[13]。根据Ecker等[14]的推测,预计以每年批准4支单抗药物上市的速度,到2020年将有大约70支单抗药物投入市场使用,全球市场销售额将达1 250亿美元。
作为复杂的大分子糖蛋白药物,单抗药物两条重链的Fc片段上297位的天冬氨酸(依据Kabat编号)上具有一个高度保守的N-糖基化修饰位点[15]。有趣的是,尽管单抗药物的轻链与重链的氨基酸序列是对称的,但是Fc片段上N-糖基化修饰有些是对称的而有些则不是[16]。单抗药物的N-糖基化修饰通常是复杂的双天线糖链结构,可能发生岩藻糖化和唾液酸化。许多单抗药物的糖型中高甘露糖型处于较低的水平,这同样也发生在人免疫球蛋白上。人免疫球蛋白中也经常存在平分型N-乙酰葡糖胺。此外,20%~30%的人免疫球蛋白及个别单抗药物在Fab(fragment antigen binding part)片段上也存在N-糖基化修饰位点,比如西妥昔单抗(cetuximab)重链的第88位天冬氨酸[17]。已发现一些单抗药物存在O-糖基化修饰,如在通过毕赤酵母生产的单抗药物中发现存在单独一个甘露糖修饰的O-糖基化修饰[18];由中国仓鼠卵巢(Chinese hamster ovary,CHO)细胞生产的某人源化单抗药物存在单独一个葡萄糖修饰的O-糖基化修饰[19]。单抗药物的糖基化修饰与单抗药物的生产系统、选择的细胞系种类以及孵育过程密切相关[20]。目前最常用的细胞孵育体系是哺乳动物细胞,包括CHO细胞、鼠NS0细胞、鼠Sp2/0细胞和鼠杂交瘤细胞等[20-22]。其中CHO细胞系生产出的单抗药物的糖基化修饰最接近人IgG的糖基化修饰情况,而鼠NS0或Sp2/0细胞生产出的单抗药物的糖基化修饰情况则相对不同。例如通过鼠NS0或Sp2/0细胞生产出的单抗药物糖基化修饰通常存在少量的非人源化α-1,3-半乳糖修饰以及大量的非人源化N-羟乙酰神经氨酸(N-glycolyl neuraminic acid,NGNA)唾液酸结构,而这些糖基化修饰在人体内易引起免疫原性[23-26]。此外,大肠杆菌被用于非糖基化修饰的Fab片段的生产[20]。而酵母、真菌、昆虫细胞和植物等生产体系则由于受到产生糖基化修饰的限制而仍未得到广泛应用[20,27]。大量研究表明单抗药物的N-糖基化修饰对其稳定性、清除率、免疫原性、抗体依赖细胞毒性(antibody-dependent cellular cytotoxicity,ADCC)及补体依赖细胞毒性(complement-dependent cytotoxicity,CDC)等都有一定的影响[28]。例如,缺少核心岩藻糖的糖型能够增强对FcγRⅢa受体的亲和力,从而提高ADCC[29];而高水平的唾液酸化的糖型则会减少对FcγRⅢa受体的亲和力,从而降低ADCC[30];此外,高甘露糖型会降低血浆半衰期,提高免疫原性以及提高ADCC[30]。图1b中列举了一些糖型影响单抗药物理化特性的实例。
单抗药物的迅速发展及其在多种疾病治疗中日益凸显的重要性都对单抗药物的研发及用药安全等方面提出了更高的要求。此外,FDA于2012年颁布了《生物仿制产品开发的指导意见草案》,给出了单抗药物的评价标准,并明确指出对于单抗药物这种大分子蛋白类药物要对能够影响其有效性及安全性的初级或二级结构进行评价,如N-糖型的表征[31]。因此,对单抗药物糖基化修饰的定性、定量研究对药物研发、改进以及安全用药等都具有十分重要的意义。本综述将简要介绍单抗药物糖基化修饰的分析研究进展。
2 单抗药物糖基化修饰分析方法
对单抗药物糖基化修饰的定性和定量研究已在各个层次上展开,包括在完整蛋白质水平、蛋白片段、糖肽以及糖链水平进行定性和定量表征。常用的技术手段包括毛细管电泳技术(capillary electrophoresis,CE)[32]、高效液相色谱技术(high-performance liquid chromatography,HPLC)[33,34]、电喷雾质谱技术(electrospray ionization mass spectrometry,ESI-MS)[33,35]及傅里叶离子回旋共振质谱技术(Fourier ion cyclotron resonance mass spectrometry,FT-ICR-MS)[36]等。本节将简要介绍各个层次上单抗药物糖基化修饰分析的常用方法及研究现状。
2.1 完整蛋白质水平的MS分析和CE分析
ESI-MS偶联HPLC或者尺寸排阻色谱(size-exclusion chromatography, SEC)技术常用于完整蛋白质层次的单抗糖基化修饰的表征[37]。HPLC-MS对蛋白质具有较好的分离能力,但常伴有吸附性强、形成多峰等问题,可以通过色谱柱材料选择、流动相调节和升高柱温等方式进行改进[38-40]。SEC相对简单,只需在室温条件下进行分离[41,42],但分离度和柱效较低,并且同样存在蛋白质吸附等问题。ESI-MS通常具有较高的分辨率及质量精度,在单抗糖基化修饰的表征研究中发挥了重要作用,已成功用于多种单抗药物糖基化修饰的表征。FT-ICR-MS由于其极高的分辨率(>106)和质量精度(<1 ppm(1×10-6))而被越来越多地用于完整蛋白质水平的单抗糖基化修饰分析[36],但是操作复杂、价格高昂等不足使其仍未得到广泛应用。2000年,Makarov等[43]发展了一种通过离子围绕中心电极的轨道旋转而捕获离子的装置—静电场轨道阱(orbitrap)质量分析器。随后Thermo公司推出了系列Orbitrap质谱,除了具有高达5×105的分辨率、高质量精度(<1 ppm)等优点外,它的完整蛋白质模式能提供对完整蛋白质的灵敏、准确的分析,对于50 kDa内的蛋白质能实现同位素分辨,是分析完整蛋白质水平的糖基化修饰的强有力工具。此外,基质辅助激光解析电离-飞行时间质谱技术(matrix-assisted laser desorption ionization-time of flight mass spectrometry, MALDI-TOF MS)可用于完整蛋白质糖基化的快速分析,但是由于较低的分辨率使其更适合于相对分子质量较小的蛋白质而在完整单抗水平的糖基化分析中不具备优势。为了提高质谱分析的灵敏度,可以通过酶辅助手段在单抗片段层次进行糖基化表征。这种基于middle-up策略的糖基化表征通常结合蛋白质还原和酶解手段生成Fab、Fc(fragment crystallizable part)、Fc/2、重链和轻链等蛋白质片段。常用的还原试剂包括二硫苏糖醇(DL-dithiothreitol,DTT)、三(2-羧乙基)膦(tris(2-carboxyethyl)phosphine,TCEP)、β-巯基乙醇(β-ME)和巯基乙胺(mercaptoethylamine,MEA)等,常用的酶包括木瓜蛋白酶、胃蛋白酶、IdeS和IdeZ等。Sinha等[16]的研究表明,与完整单抗水平的糖基化修饰分析结果相比,单抗片段水平的糖基化修饰分析能够获得更多的糖基化修饰信息,具有更高的灵敏度。
CE-MS技术也常被用于糖基化修饰的分析中[44-46]。此外,蛋白水平的唾液酸化分析也可通过基于电荷差异的电泳策略实现,包括等电聚焦电泳技术(isoelectric focusing electrophoresis, IEF)[44,47]、CE技术[48]及离子交换色谱技术(ion-exchange chromatography, IEX)[46,49]等。这些方法通常被用于蛋白药物的质量控制监测中[50],不过传统的IEF和IEX方法不能和质谱技术兼容且耗时,而CE技术因其快速、高分辨率、低样本量以及能与质谱兼容等优势而更适用于蛋白药物糖基化修饰的表征研究[51]。
2.2 糖链水平的分析
N-糖基化修饰的糖链可以通过酶切作用或者化学方法从糖蛋白上释放出来,最常用的是通过肽糖苷酶F(peptide-N-glycosidase F,PNGase F)或肽糖苷酶A(peptide-N-glycosidase A,PNGase A)酶切作用,或者肼解作用释放糖链。O-糖链的释放通常采用化学方法,包括肼解法[52]、β-消除法[53]和三氟甲磺酸法[54]等。释放的糖链可以利用MALDI-TOF MS技术进行简单、方便的快速定性、定量分析,但是唾液酸化糖链易被破坏,使其难以表征,因此有研究通过将糖链甲基化实现了提高离子化效率、同时保护唾液酸化糖链的目的[37,55,56]。HPLC方法常用于对糖链的定性、定量分析,因为糖链的紫外吸收较弱,通常会在分析前对糖链进行荧光标记以提高检测灵敏度,2-氨基苯甲酰胺(2-aminobenzamide,2-AB)和2-氨基苯甲酸(2-aminobenzoic acid,2-AA)是最常用的荧光标记试剂。标记后的糖链可以通过亲水作用色谱(hydrophilic interaction chromatography,HILIC)、反向高效液相色谱、阴离子交换色谱(anion-exchange chromatography,AEC)以及CE等技术进行分析[57]。而释放的糖链也可以不进行荧光标记直接通过高效阴离子交换色谱结合脉冲安培检测技术或者热裂解色谱技术进行分析[58,59],但是方法的稳定性及重现性较差[59]。最近的一项研究通过7种正交方法(包括HILIC、CE、高效阴离子交换色谱结合脉冲安培检测法(high-performance anionic exchange chromatography-air pulsing ampere detectors,HPAEC-PAD)等)在糖链水平对单抗的糖基化修饰进行了分析,7种方法的分析结果基本一致[15]。通过和质谱技术的联用,上述方法不仅可用于糖基化的定性和定量分析,同样适用于对未知糖型的表征,但是解析过程却相对繁琐且成本较高。随着质谱技术的发展,高分辨质谱在糖链结构的解析方面为我们提供了一种更简单、快速、灵敏度的方法[60-62]。但是糖链的同分异构体及三维结构则需通过核磁共振(nuclear magnetic resonance, NMR)技术和X射线晶体学(X-ray crystallography)进行分析[63,64]。
2.3 糖肽水平的分析
糖肽水平的蛋白质糖基化修饰位点鉴定及糖基化修饰情况分析通常是通过ESI-MS技术或MALDI-TOF MS技术完成的[65],而ESI-MS是进行糖肽表征最有力的工具。通过MS/MS谱图可以获取大量糖链信息,结合糖肽分析软件可以简单、高效地完成糖基化位点鉴定及糖链组成的确定。目前有许多免费的糖肽分析软件可以使用,包括GlycoMod[66]、GlycoPep DB[67]、GlycoMiner[68]和ArMone[69]等。
基于MRM策略的糖肽水平定量分析也展现出了强大的优势。Toyama等[70]建立了一种通过“能量依赖型”氧鎓离子监测模式对单抗药物的N-糖型进行定量表征的方法,系统地研究了质谱能量与N-糖链氧鎓离子碎裂情况之间的关系,实现了对N-糖链的定量表征,糖肽的检测下限为30 amol,线性范围达4个数量级,并可区分糖链的同分异构体。该研究为单抗药物N-糖型的定量研究提供了一个新思路。Hong等[71]建立了一种利用MRM策略同时对IgG蛋白质及其N-糖型进行绝对定量的方法。该方法中IgG蛋白质的检测下限为60 amol,检测动态范围达3个数量级。对IgG蛋白质进行绝对定量的同时,可在不经过富集的情况下直接对血清中26种高丰度N-糖肽进行测定。实现了对IgG蛋白质及N-糖型的同时测定。可参考该方法对生物基质中的单抗及其N-糖型的定量分析。Yang等[72]通过MRM方法对6种单抗药物的糖基化修饰进行了定量研究,并同时考察了单抗药物糖基化修饰的位点占有率。该方法快速、高效,具有良好的特异性,为单抗药物糖基化修饰的定量研究、药物研发及质量监控提供了新途径。
在糖肽水平的定量分析过程中,一方面由于糖肽丰度往往远低于普通肽段,另一方面则由于糖肽的离子化易受共洗脱物的抑制,尤其在复杂样品中抑制效应更显著,因此常辅以分离富集策略以排除干扰、提高检测灵敏度。常见的糖肽分离富集方法包括酰肼化学法(hydrazide chemistry)[73]、凝集素亲和色谱法(lectin affinity chromatography, LAC)[74]、HILIC[75]等。酰肼化学法的原理是利用高碘酸钠氧化糖链上的顺式邻位二羟基生成醛基,而酰肼基团可以与醛基发生化学键合,从而富集出糖肽[76]。该方法特异性好,但是会不可逆地破坏糖链结构,因此不适合用于糖链的结构解析及定量研究。LAC的原理是利用植物或动物细胞合成并分泌的凝集素能与特定的糖单元结合的特点富集糖肽[75],最早被用于糖基化富集研究中。如伴刀豆凝集素(concanavalin A,Con A)能特异性地富集甘露糖,麦胚凝集素(wheat germ agglutinin,WGA)能特异性地富集N-乙酰葡糖胺。该方法的缺点在于,凝集素具有选择性,只能富集特定种类的糖,不适用于糖基化修饰的广谱性富集,并且凝集素与糖链之间的相互作用较弱,因此富集效率较低。HILIC的原理是以水-水溶性有机溶剂的混合物为流动相,亲水作用材料表面吸附水分子形成富水层,分析物中的亲水成分会浓缩在所形成的富水层中,疏水成分则留在高含量有机溶剂的流动相中[77]。糖肽相对于非糖肽具有更强的亲水性,容易被保留在亲水富集材料上,达到富集的目的。该方法对所有糖链都有富集作用,并且不会破坏糖链结构,而且随着该技术的不断发展,方法特异性也不断提高,因此是用于糖基化定量研究的较好选择[78]。
3 结论与展望
单抗药物是当前发展最为迅速的蛋白类药物之一,作为大分子糖蛋白药物,单抗药物的糖基化修饰对其稳定性、有效性和安全性等各方面均有不同程度的影响,因此对其糖基化修饰进行全面表征具有十分重要的意义。这就需要及时建立高效、快速、可靠的糖基化修饰定性、定量分析方法。而糖基化修饰本身的复杂性以及对糖基化修饰检测的难度,给相关研究带来了巨大的挑战。尽管对单抗药物糖基化修饰的定量研究已在各个层次上展开并且在快速发展,但仍存在一些问题,如糖肽富集的回收率和重复性、样品处理的通量、方法灵敏度、数据分析处理流程的简化、标准化流程的建立等,这有待于分析化学家发展新的方法和技术,为单抗药物的研究提供高效可靠的分析手段。
[1] Kohler G, Milstein C. Nature, 1975, 256(5517): 495
[2] Liu J K. Ann Med Surg (Lond), 2014, 3(4): 113
[3] Hnasko R M, Stanker L H. Methods Mol Biol, 2015, 1318: 15
[4] Weiner L M, Surana R, Wang S. Nat Rev Immunol, 2010, 10(5): 317
[5] Chen Z, Wang J, Bao L, et al. Nat Commun, 2015, 6: 6714
[6] Oliphant T, Engle M, Nybakken G E, et al. Nat Med, 2005, 11(5): 522
[7] Isaacs J D. Arthritis Res Ther, 2009, 11(3): 225
[8] Bayry J, Lacroix-Desmazes S, Kazatchkine M D, et al. Nat Clin Pract Rheumatol, 2007, 3(5): 262
[9] Hogarth P M, Pietersz G A. Nat Rev Drug Discov, 2012, 11(4): 311
[10] Mauri C, Mars L T, Londei M. Nat Med, 2000, 6(6): 673
[11] Berman D, Korman A, Peck R, et al. Pharmacol Ther, 2015, 148: 132
[12] Weiner L M. J Immunother, 2006, 29(1): 1
[13] Reichert J M. MAbs, 2015, 7(1): 1
[14] Ecker D M, Jones S D, Levine H L. MAbs, 2015, 7(1): 9
[15] Reusch D, Haberger M, Maier B, et al. MAbs, 2015, 7(1): 167
[16] Sinha S, Pipes G, Topp E M, et al. J Am Soc Mass Spectrom, 2008, 19(11): 1643
[17] Janin-Bussat M C, Tonini L, Huillet C, et al. Methods Mol Biol, 2013, 988: 93
[18] Ha S, Wang Y, Rustandi R R. MAbs, 2011, 3(5): 453
[19] Tanaka M, Koga A, Kobe A, et al. Eur J Pharm Biopharm, 2013, 83(2): 123
[20] Beck A, Wagner-Rousset E, Bussat M C, et al. Curr Pharm Biotechnol, 2008, 9(6): 482
[21] Song T, Ozcan S, Becker A, et al. Anal Chem, 2014, 86(12): 5661
[22] Jefferis R. Arch Biochem Biophys, 2012, 526(2): 159
[23] Hokke C H, Bergwerff A A, Van Dedem G W, et al. FEBS Lett, 1990, 275(1/2): 9
[24] Hamilton S R, Gerngross T U. Curr Opin Biotechnol, 2007, 18(5): 387
[25] Sheeley D M, Merrill B M, Taylor L C. Anal Biochem, 1997, 247(1): 102
[26] Beck A, Klinguer-Hamour C, Bussat M C, et al. J Pept Sci, 2007, 13(9): 588
[27] Triguero A, Cabrera G, Royle L, et al. Anal Biochem, 2010, 400(2): 173
[28] Beck A, Wagner-Rousset E, Ayoub D, et al. Anal Chem, 2013, 85(2): 715
[29] Chung S, Quarmby V, Gao X, et al. MAbs, 2012, 4(3): 326
[30] Scallon B J, Tam S H, McCarthy S G, et al. Mol Immunol, 2007, 44(7): 1524
[31] Anonymous. Biotechnol Law Rep, 2012, 31: 171
[32] Said N, Gahoual R, Kuhn L, et al. Anal Chim Acta, 2016, 918: 50
[33] Reusch D, Haberger M, Falck D, et al. MAbs, 2015, 7(4): 732
[34] Geyer H, Geyer R. Biochim Biophys Acta, 2006, 1764(12): 1853
[35] Stadlmann J, Pabst M, Kolarich D, et al. Proteomics, 2008, 8(14): 2858
[36] Tran B Q, Barton C, Feng J, et al. J Proteomics, 2016, 134: 93
[37] Zhang Z, Pan H, Chen X. Mass Spectrom Rev, 2009, 28(1): 147
[38] Beck A, Debaene F, Diemer H, et al. J Mass Spectrom, 2015, 50(2): 285
[39] Xie H, Chakraborty A, Ahn J, et al. MAbs, 2010, 2(4): 379
[40] Ayoub D, Jabs W, Resemann A, et al. MAbs, 2013, 5(5): 699
[41] Xu C F, Zang L, Weiskopf A. J Chromatogr B, 2014, 960: 230
[42] Liu H, Gaza-Bulseco G, Chumsae C. J Am Soc Mass Spectrom, 2009, 20(12): 2258
[43] Makarov A. Anal Chem, 2000, 72(6): 1156
[44] Zhao S S, Chen D D. Electrophoresis, 2014, 35(1): 96
[45] Haselberg R, de Jong G J, Somsen G W. Anal Chem, 2013, 85(4): 2289
[46] Thakur D, Rejtar T, Karger B L, et al. Anal Chem, 2009, 81(21): 8900
[47] Reichel C, Thevis M. Bioanalysis, 2013, 5(5): 587
[48] De Kort B J, De Jong G J, Somsen G W. Electrophoresis, 2012, 33(19/20): 2996
[49] Schiestl M, Stangler T, Torella C, et al. Nat Biotechnol, 2011, 29(4): 310
[50] Federici M, Lubiniecki A, Manikwar P, et al. Biologicals, 2013, 41(3): 131
[51] Staub A, Guillarme D, Schappler J, et al. J Pharm Biomed Anal, 2011, 55(4): 810
[52] Merry A H, Neville D C, Royle L, et al. Anal Biochem, 2002, 304(1): 91
[53] Wada Y, Dell A, Haslam S M, et al. Mol Cell Proteomics, 2010, 9(4): 719
[54] Edge A S, Faltynek C R, Hof L, et al. Anal Biochem, 1981, 118(1): 131
[55] Sanchez-De Melo I, Grassi P, Ochoa F, et al. J Proteomics, 2015, 127: 225
[56] Marino K, Bones J, Kattla J J, et al. Nat Chem Biol, 2010, 6(10): 713
[57] Zhang L, Luo S, Zhang B. MAbs, 2016, 8(2): 205
[58] Pabst M, Altmann F. Proteomics, 2011, 11(4): 631
[59] Grey C, Edebrink P, Krook M, et al. J Chromatogr B, 2009, 877(20/21): 1827
[60] Harvey D J. J Am Soc Mass Spectrom, 2005, 16(5): 647
[61] Harvey D J. J Am Soc Mass Spectrom, 2005, 16(5): 631
[62] Harvey D J. J Am Soc Mass Spectrom, 2005, 16(5): 622
[63] Wormald M R, Petrescu A J, Pao Y L, et al. Chem Rev, 2002, 102(2): 371
[64] Halbeek H. Methods Mol Biol, 1993, 17: 115
[65] Chen Y H, Yan G Q, Zhou X W, et al. Chinese Journal of Chromatography, 2010, 28(2): 135
陈瑶函, 晏国全, 周新文, 等. 色谱, 2010, 28(2): 135
[66] Cooper C A, Gasteiger E, Packer N H. Proteomics, 2001, 1(2): 340
[67] Go E P, Rebecchi K R, Dalpathado D S, et al. Anal Chem, 2007, 79(4): 1708
[68] Ozohanics O, Krenyacz J, Ludanyi K, et al. Rapid Commun Mass Spectrom, 2008, 22(20): 3245
[69] Cheng K, Chen R, Seebun D, et al. J Proteomics, 2014, 110: 145
[70] Toyama A, Nakagawa H, Matsuda K, et al. Anal Chem, 2012, 84(22): 9655
[71] Hong Q, Lebrilla C B, Miyamoto S, et al. Anal Chem, 2013, 85(18): 8585
[72] Yang N, Goonatilleke E, Park D, et al. Anal Chem, 2016, 88(14): 7091
[73] Zhang H, Li X J, Martin D B, et al. Nat Biotechnol, 2003, 21(6): 660
[74] Kaji H, Saito H, Yamauchi Y, et al. Nat Biotechnol, 2003, 21(6): 667
[75] Alpert A J. J Chromatogr, 1988, 444: 269
[76] O'Shannessy D J, Hoffman W L. Biotechnol Appl Biochem, 1987, 9(6): 488
[77] Alpert A J. J Chromatogr, 1990, 499: 177
[78] Ma C, Pan Y T, Zhang Q, et al. Chinese Journal of Chromatography, 2013, 31(11): 1057
马成, 潘一廷, 张琪, 等. 色谱, 2013, 31(11): 1057
Recent advances on the analysis of glycosylation of therapeutic monoclonal antibodies
CONG Yuting, HU Lianghai*
(SchoolofLifeSciences,JilinUniversity,Changchun130012,China)
Therapeutic monoclonal antibodies are the variants of immunoglobulin G, most commonly based on human or mouse immunoglobulin G. They provide a new platform to treat a wide range of diseases, such as cancers, inflammatory diseases and virus infection. A lot of studies demonstrated thatN-glycosylation of therapeutic monoclonal antibodies has effects on their stability, solubility, and clearance as well as immunogenicity, antibody-dependent cellular cytotoxicity and complement-dependent cytotoxicity. The fast development and increasing importance of their therapeutic action highlight the importance of drug research and medication security. Therefore, it has great significance to establish reliable qualitative and quantitative analytical methods of glycosylation of therapeutic monoclonal antibodies. This review introduces the glycosylation of therapeutic monoclonal antibodies and the method developments for qualitative and quantitative analyses of their glycosylation.
therapeutic monoclonal antibody (mAb); glycosylation; mass spectrometry (MS); proteomics; review
10.3724/SP.J.1123.2016.08035
2016-08-29
国家自然科学基金项目(81373374).
Foundation item: National Natural Science Foundation of China (No. 81373374).
O658
A
1000-8713(2016)12-1186-06
邹汉法研究员纪念专辑(上)·专论与综述
* 通讯联系人.E-mail:lianghaihu@jlu.edu.cn.
