基于主动分析的基因芯片图像网格定位
2016-12-07李铁军邵桂芳上官亚力吴顺祥
李铁军,孙 跃,邵桂芳,上官亚力,吴顺祥
(1.重庆大学自动化学院,重庆400044;2.厦门大学航空航天学院,福建厦门361005)
基于主动分析的基因芯片图像网格定位
李铁军1,孙 跃1,邵桂芳2*,上官亚力2,吴顺祥2
(1.重庆大学自动化学院,重庆400044;2.厦门大学航空航天学院,福建厦门361005)
基因芯片图像处理对解读成千上万个基因点的杂交信息,建立可信疾病预测具有重要的意义,其分析的可靠性和准确性将直接影响到芯片的推广与使用.而基因芯片制备过程复杂,图像种类多样,单一的网格定位方法无法满足各种类型基因芯片图像处理的需求.在分析现有网格定位方法的基础上,基于“仿人”思想,将认知科学的主动分析引入到图像处理中,提出通过自主感知来明确基因芯片图像处理过程中不确定性因素的存在及其程度,并采取相应方法来降低或消除不确定因素的影响.最后通过6个数据集上的384幅网格图像实验,证明所提出的自动对比度增强和自适应网格定位调整方法无需参数输入和人工干预,能较大程度提高网格定位准确率.
基因芯片;图像处理;网格定位
重大疾病(如癌症)的预防与控制是世界各国面临的重大科技难题[1].例如,全球每年大约有100万新发大肠癌病例,至少50万患者死亡,而中国大肠癌发病率以4.71%的速度逐年递增[2].传统的仪器检测筛查方法具有介入性、单疾病、费用高和周期长等诸多缺点.20世纪90年代发展来的基因芯片(cDNA microarray)以高通量、微型化、自动化及低成本等优点成为后基因组时代最重要的基因检测工具之一,能够有效地应用于基因测序、生物学研究和疾病预防与控制[3].包括《Science》和《Nature》在内的国际权威刊物近年来发表了大量应用基因芯片进行癌症预防与控制的研究成果.基因芯片分析包含样本采集、芯片制备、扫描成像、图像处理、数据分析和生物学应用等[4],如图1所示.
基因芯片的图像处理是提取基因表达水平信息的关键步骤,其准确性和可靠性将直接影响基因表达谱数据的有效性,并最终决定生物学分析和病理分析的成败.因此,基因芯片图像处理对解读成千上万个基因点的杂交信息,建立可信疾病预测具有重要的意义,其分析的可靠性和准确性将直接影响到芯片的推广与使用.基因芯片图像处理包括图像增强、网格定位、基因点分割和信息点提取4个步骤[5],其目的是提取基因表达谱数据,建立基因与其对应功能的关联,用于后续基因数据分析.基因芯片图像处理是以整个芯片为单位的(每张芯片可能包含12×4或4×4个子网格,每个子网格又包含22×22、40×40或14×18个基因点不等),即输入是2幅基因芯片图像(对应荧光标记Cy3和Cy5),输出是芯片里每一个基因点的表达值,一次输出结果可能包含23 232或12 096个基因点不等(与不同芯片制备公司相关).由于基因芯片图像处理过程存在众多不确定性,如芯片制作过程复杂(会带来操作失误或仪器误差),图像质量多变(会出现噪声等级不同、基因点形态各异和基因点分布疏密不一),图像分析方法多样等,对基因芯片图像处理造成巨大挑战.
针对网格定位问题,以完全自动化处理为目标,其算法经历了如下发展历程(如图2所示):
1)Angulo等[5]将二维图像处理问题转换为一维信号处理,提出一种最简单快速的形态学投影法,但该方法容易受噪声影响;
2)Katzer等[6]提出马尔科夫随机场法,该方法需要输入网格的行列参数;
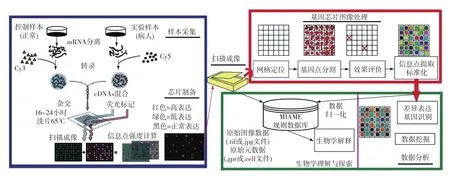
图1 基因芯片分析流程图Fig.1 The flowchart for cDNA microarray analysis
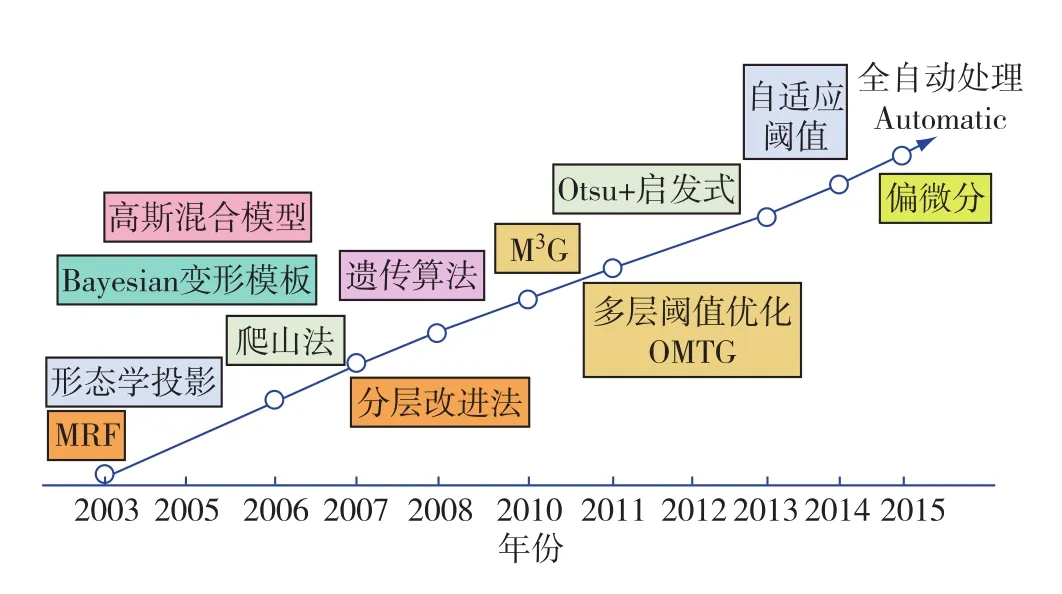
图2 网格定位方法发展趋势Fig.2 Development trend of gridding methods
3)齐飞等[7]和Ceccarelli等[8]通过高斯混合模型和贝叶斯推理方式辨识参数来实现网格定位,但需要有参考模板;
4)Rueda等[9]和Zacharia等[10]将网格定位问题转化为优化问题,提出分别利用爬山法和遗传算法来求解,该类方法比较耗时并且受算法初始参数设置影响;
5)很多学者[11-20]考虑在算法内加入最优阈值搜索功能,提出一系列具有自适应调节能力的完全自动处理方法.
虽然不断有各种网格定位方法涌现,但由于基因芯片图像的复杂不确定性,导致每种方法都无法取得精确结果.因此,本研究在分析基因芯片图像特点的基础上,依据近5年来从事基因芯片图像处理研究的经验,分析了影响其结果的原因,并引入“仿人”思想,建立基于主动分析的网格定位方法.该方法能基于四阶矩进行图像自动对比度自动感知与增强,并利用最大内间方差法(Otsu)进行阈值的自动获取,初步实现网格定位后,依据结果的统计分析进行自调整.在6个数据集上通过对比本文方法与投影法[5]和最大类间方差方法[16]的网格定位效果,证明了主动分析引入的有效性和必要性.
1 网格定位方法
1.1基因芯片图像分析
基因芯片图像具有显著不同于其他图像处理问题的特点,决定了其处理分析的不确定性.这些特点主要有:
1)全局性,即关注每个基因点区域.不同于人脸识别和目标检测等问题,由于基因点相对均匀地分布在图像中的每一个区域,所以整幅图像都是算法关注的焦点.
2)多样性,算法要同时处理多个芯片、多个网格或多个基因点,但是不同芯片、不同网格或不同基因点上的图像都存在各种动态变化的特点.
3)精确性,要求结果完全准确.基因芯片图像处理,不是根据特征去寻找图像中的某个目标或区域,而是提取每一个基因点上的信息,来精确地估计基因表达水平.
近5年的研究实践表明[16,21],造成基因芯片图像处理结果不确定的来源虽然可能是多方面的,但可以分为以下两个层面:
1)数据层面,即图像质量,与实验设备、实验设计及材料等有关,如针对不同病症、不同病人群体,采用不同技术不同手段制备的基因芯片.由于芯片制备厂商不同,以及芯片制备过程中的取样、标记、杂交条件和扫描仪器等带来的误差影响,导致实际基因芯片图像质量各异,图3展示了部分具有不同质量的子网格图像和基因点图像.这些质量差异可以从以下3个方面来看:
(i)基因芯片层面.(a)分布各异,即每个芯片图像包含的子网格数不同,每个子网格包含的基因点数也不同;(b)分辨率各异,即芯片图像和基因点均具有不同的分辨率,如一幅基因芯片图像大小为4 325像素×11 388像素,其基因点大概为25像素×25像素.
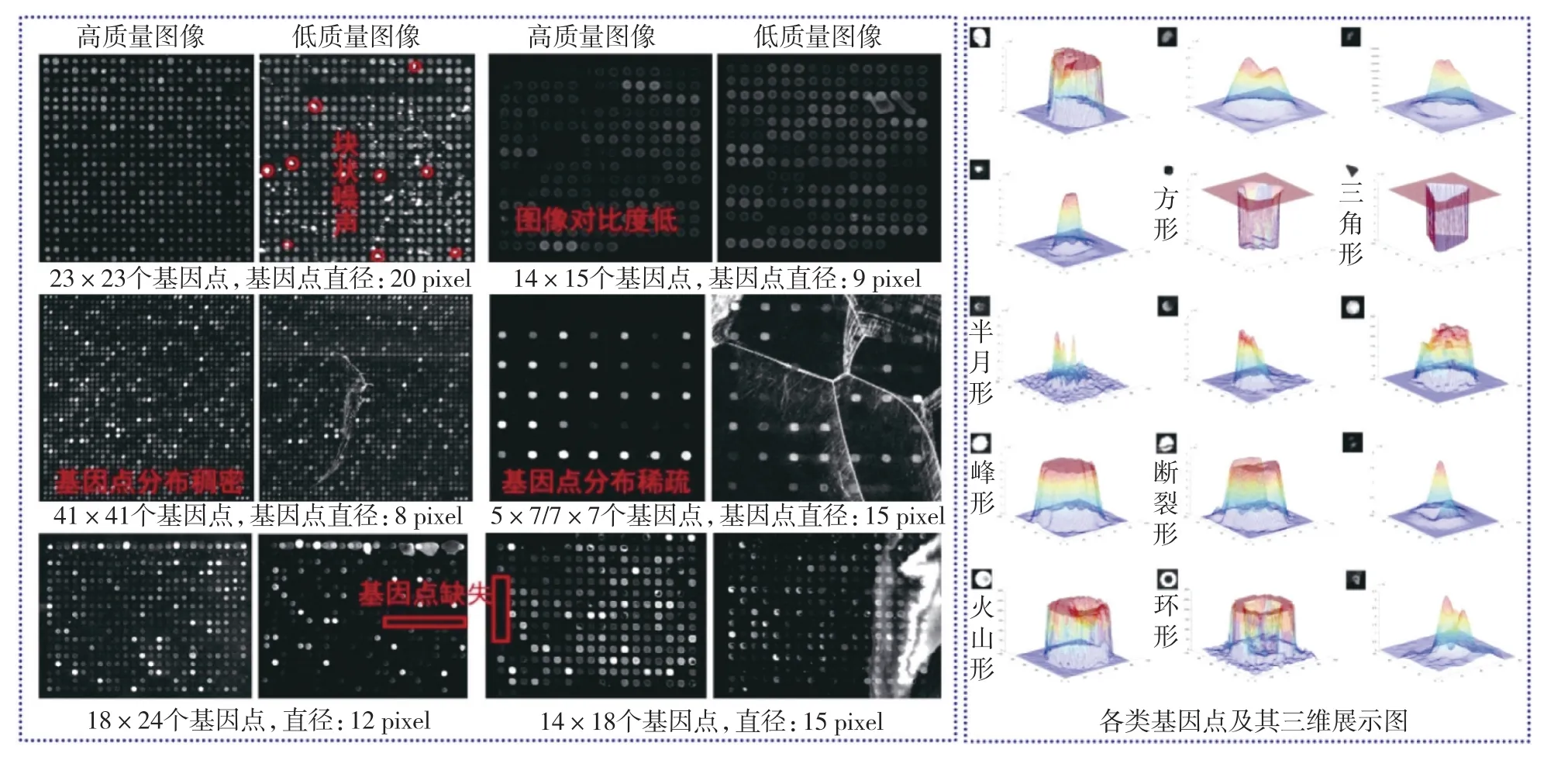
图3 子网格及各类基因点示意Fig.3 Examples of sub-grids and spots
(ii)子网格角度.(a)基因点分布疏密不一,有的基因点排列非常紧凑,网格定位时容易压到基因点边缘,而有的基因点分布很稀疏,容易受噪声影响;(b)噪声等级不同,有的包含少量噪声,有的包含大量噪声;(c)噪声类型也不同,如块状噪声、彗星尾噪声等,而且噪声有些是和基因点混在一起的,导致基因点无法分割或影响其表达值计算;(d)缺失基因点比例不同;(e)有时会存在倾斜,整个子网格倾斜或部分基因点区域倾斜.
(iii)基因点自身.(a)基因点形态各异,其基本形态可能因基因芯片制备公司的不同,有圆形、方形和三角形等;(b)即使基本形态固定,也会有峰形、火山形、半月形、断裂形和环形等变化;(c)基因点粘连,有些基因点可能会连在一起,导致无法分割.
上述众多不确定因素可能会同时发生在一次处理过程中,造成基因芯片图像质量动态变化,导致基因芯片图像处理精度低.
2)方法层面.研究者采用的不同图像处理算法,具有局限适应性(适用于某类图像或某种处理)等差异,难以适应基因芯片图像处理的复杂过程,增加了基因芯片图像处理的难度.基因芯片图像处理具有串行分级特性,即前面步骤是后面的基础,其误差会向后累积,使得仅采用单一方法来解决全局处理具有极大的挑战性.同时,已有的图像处理方法在某些方面表现出独特的优势是值得借鉴的.
与基因芯片数据的复杂性形成鲜明对比的是,现有的图像处理方法只能针对单个或若干个问题,尽可能地提升处理过程的单一目标或者多个目标的准确率,而不适用于其他问题或其他图像区域,无法满足基因芯片图像处理的特殊要求.
我们认为,想从根本上防止或降低基因芯片图像处理中的不确定性,仅改进或提高局部算法的性能是不够的.提高基因芯片图像处理精确性的有效办法应该是结合实际问题的特点,对造成结果不确定的内在原因进行深入挖掘和分析,有针对性的采取方法进行处理,即不同问题不同对待,实现从“被动分析”到“主动处理”的转变.
1.2网格定位的主动分析
主动分析网格定位框架如图4所示.
为实现主动分析,基因芯片网格定位主要包括3个环节:1)自适应对比度增强,根据图像自身数据感知其对比度情况,并进行增强;2)网格定位,依据自适应阈值方法进行分割;3)网格定位结果的自调整,即依据分割结果的统计分析,自动判断分割结果正确与否,并进行调整.

图4 网格定位主动分析框架Fig.4 The active analysis flowchart for gridding
1.3自适应对比度增强
因为基因芯片图像普遍存在对比度较低现象,并且低对比度对网格定位影响较大.为此,本研究引入四阶矩来建立图像对比度自动感应与增强算法[21].设含有N个像素点的基因芯片图像灰度值可用f(x,y)表示,首先将二维图像信号转换为一维列信号x= f(:),再通过下式得到自动增强后的图像g(x,y):

上述操作有如下优点:1)不需要任何参数输入; 2)自适应实现对比度增强;3)可大幅度提高基因芯片图像处理精度;4)适应性广,可以适用于各种类型基因芯片图像.
1.4网格定位结果自调整
由于采用任何网格定位方法,都无法达到100%准确率,为此研究提出网格定位结果的自调整,即统计分析网格定位结果,得到每个网格图像的网格线平均间距,计算每根网格线误差及方差,从而判断该网格线定位正确与否,并进行调整.
以水平网格定位为例,hj代表网格定位获取的垂直方向网格线坐标,p为垂直方向网格线数,可统计出网格线间距并得到其平均距离从而计算网格线误差和方差
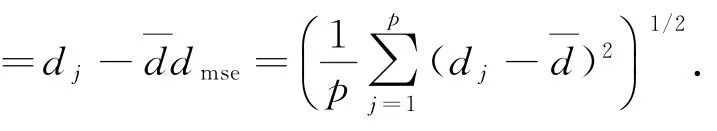
1)der(m)<0,|der(m-1)|<ε,|der(m+1)|<ε,多出来一条线,如图5(a)所示;
2)der(m)>0,|der(m-1)|<ε,|der(m+1)|<ε,少了一条线,如图5(b)所示;
3)画错了位置der(m)>0,der(m+1)>0或der(m)>0,der(m+1)<0,如图5(c)所示.
其中,ε用于描述示网格线间距与平均网格线间距之间的差异程度,取值为如前所述平均网格间距和网格线方差dmse是根据每次网格定位结果自动统计出来的,故ε无需人工设置.
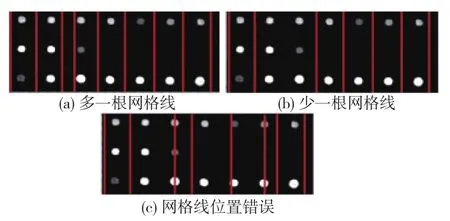
图5 网格线分割错误示意Fig.5 Examples of wrong gridding lines
2 实验分析
为验证本研究方法的有效性,分别在来自6个不同数据集的基因芯片图像上进行了对比实验(Stanford Microarray Database,SMD;University of California,San Francisco,UCSF;Gene Expression Omnibus,GEO;Computational Cancer Genomics group of the Swiss Institute of Bioinformatics,SIB; Joe DeRisi′s individual tiff files;Baylor College of Medicine,BCM)[16].图6给出了在加入主动分析前后基于形态学硬阈值和Otsu自适应阈值的网格定位准确率对比情况.
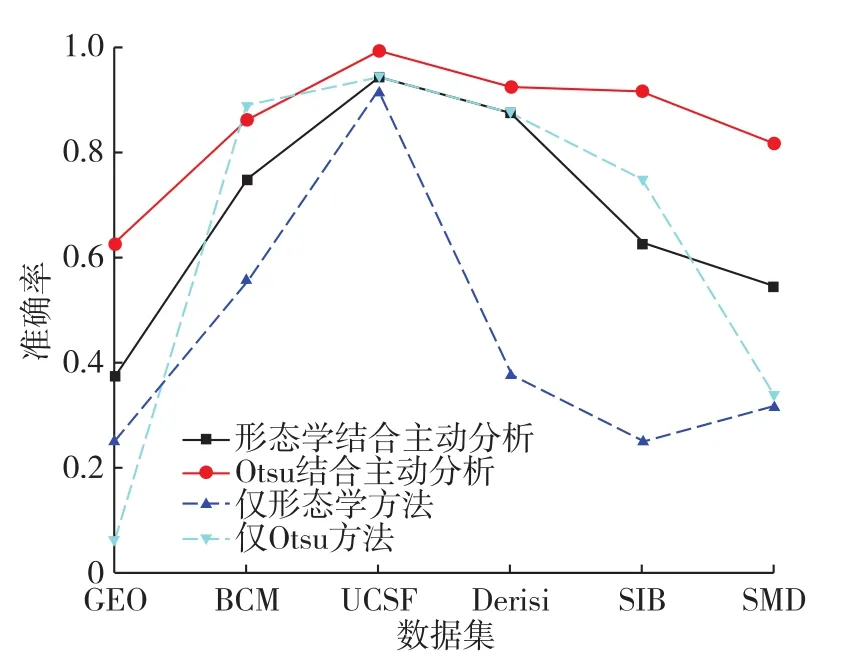
图6 加入主动分析前后的网格定位准确率对比Fig.6 Comparison of gridding accuracy when applied active analysis or not
可以看出主动分析的引入较大程度提高网格定位准确率,GEO和SMD数据集准确率提高最大.此外,Otsu自适应阈值网格定位比基于形态学的硬阈值具有更高准确率.并且在6个数据集上,GEO和SMD准确率相对较低,主要原因是其图像对比度普遍很低.图7展示了GEO数据集下网格定位效果,可以看出受低对比度及噪声影响,只有在引入主动分析的Otsu网格定位方法上才取得了准确定位.
SIB数据集上的网格定位准确率也比较低,主要是其基因点分布比较稀疏,使得网格定位很容易受噪声影响,如图8所示,只有引入主动分析的Otsu网格定位方法取得了准确定位.
而图9展示了BCM数据集图像网格定位结果,由于该数据集基因点比较密集,分辨率较大,因此,即使有噪声影响,各种方法也都能取得准确定位.换句话说,就是主动分析对BCM数据集的作用比较小.

图7 GEO数据集网格定位示例Fig.7 Examples of gridding on GEO data set

图8 SIB数据集网格定位示例Fig.8 Examples of gridding on SIB data set
图10给出了UCSF数据集图像网格定位结果,由于该数据集原始图像对比度很低,在引入主动分析,特别是对比度增强后,网格定位准确率明显提高.
3 结 论
基因芯片图像的网格定位对基因点表达谱分析具有重要作用,而基因芯片图像获取过程复杂,图像类型多样,导致网格定位方法准确率不高.此外,目前大多数网格定位方法仅考虑了图像去噪的预处理.为此,本研究在分析导致基因芯片图像网格定位准确率不高的原因及现有网格定位方法发展的基础上,提出引入主动分析,即通过对图像自身数据的挖掘分析,进行自动增强处理.通过在来自6个数据集的不同类型基因芯片图像上对比实验,证明了引入主动分析的有效性.本研究提出的主动分析方法不需要人工干预及参数输入,可以适应各类图像,准确率也较未加入主动分析方法有较大提高.后续工作将针对噪声等级进行自动分析,并建立快速有效的去噪方法.

图9 BCM数据集网格定位示例Fig.9 Examples of gridding on BCM data set

图10 UCSF数据集网格定位示例Fig.10 Examples of gridding on UCSF data set
[1] 夏俊芳,刘箐.生物芯片应用概述[J].生物技术通报,2010 (7):73-77.
[2] 王媛媛.大肠癌干细胞基因表达谱特点及其信号通路的筛选鉴定[D].广州:南方医科大学,2011:1-9.
[3] 博思数据研究中心.2011—2015年中国生物芯片行业深度调研与投资前景研究报告[R/OL].[2011-03-01] [2015-11-15].http:∥www.bosidata.com/dianzi1103/ V2504375TQ.html.
[4] BAJCSY P.An overview of DNA Microarray image requirements for automated processing[C]∥Proceedings of the 2005 IEEE Computer Society Conference on Computer Vision and Pattern Recognition.Los Alamitos: IEEE,2005:147-149.
[5] ANGULO J,SERRA J.Automatic analysis of DNA microarray images using mathematical morphology[J]. Bioinformatics,2003,19(5):553-562.
[6] DEMIRKAYA O,ASYALI M H,SHOUKRI M M.Segmentation of cDNA microarray spots using markov random field modeling[J].Bioinformatics,2005,21(13): 2994-3000.
[7] QI F,LUO Y,HU D.Recognition of perspectively distorted planar grids[J].Pattern Recognition Letters, 2006,27(14):1725-1731.
[8] CECCARELLI M,ANTONIOL G.A deformable grid matching approach for microarray images[J].IEEE Transactions on Image Processing,2006,15(10): 3178-3188.
[9] RUEDA L,VIDYADHARAN V.A hill-climbing approach for automatic gridding of cDNA microarray images[J].IEEE Transactions on Computational Biology and Bioinformatics,2006,3(1):72-83.
[10] ZACHARIA E,MAROULIS D.An original genetic approach to the fully automatic gridding of microarray images[J].IEEE Transaction on Medical Imaging,2008, 27(6):805-813.
[11] WANG Y,MA M,ZHANG K,et al.A hierarchical refinement algorithm for fully automatic gridding in spotted DNA microarray image processing[J].Information Sciences,2008,177(4):1123-1135.
[12] BARIAMIS D,IAKOVIDIS D,MAROULIS D.M3G: maximum margin microarray gridding[J].BMC Bioinformatics,2010,11:49.
[13] RUEDA L,REZAEIAN I.A fully automatic gridding method for cDNA microarray images[J].BMC Bioinformatics,2011,12(113):1-17.
[14] DEEPA J,TESSAMMA T.A new gridding technique forhigh density microarray images using intensity projection profile of best sub image[J].Computer Engineering and Intelligent Systems,2013,4(1):7-18.
[15] FOUAD I A,MABROUK M S,SHARAWY A A.A new method to grid noisy cdna microarray images utilizing denoising techniques[J].International Journal of Computer Applications,2013,63(9):36-44.
[16] SHAO G F,YANG F,ZHANG Q,et al.Using the maximum between-class variance for automatic gridding of cdna microarray images[J].IEEE/ACM Transactions on Computational Biology and Bioinformatics,2013,10 (1):181-192.
[17] THAMARAIMANALAN P,DHINESH K D,NIRMALAKUMARI K.Effective gridding and segmentation for microarray images[J].International Journal of Computer Science and Mobile Computing,2014,3(2): 353-360.
[18] AHMAD M M,JAMBEK A B,MASHOR M Y.A study on microarray image gridding techniques for DNA analysis[C]∥2ndInternational Conference on Electronic Design.Penang:IEEE,2014:171-175.
[19] HARIKIRAN J,AVINASH B,LAKSHMI P V,et al. Automatic gridding method for microarray images[J]. Journal of Theoretical and Applied Information Technology,2014,65(1):235-241.
[20] BELEAN B,TEREBES R,BOT A.Low-complexity PDE-based approach for automatic microarray image processing[J].Medical and Biological Engineering and Computing,2015,53(2):99-110.
[21] SHAO G F,LI T J,ZUO W D,et al.A combinational clustering based method for cDNA microarray image segmentation[J].PLoS ONE,2015,10(8):1-23.
An Active Analysis Method for Microarray Image Gridding
LI Tiejun1,SUN Yue1,SHAO Guifang2*,SHANGGUAN Yali2,WU Shunxiang2
(1.School of Automation,Chongqing University,Chongqing 400044,China; 2.School of Aerospace Engineering,Xiamen University,Xiamen 361005,China)
Image processing is a key aspect of cDNA microarrays analysis and the aim of the whole experimental procedure is to obtain meaningful biological conclusions,which depend on the accuracy of this stage.Unfortunately,because the complicated hybridization procedure of microarrays will result in dynamic changing of microarray image quality,results of existing methods can not meet the need of overall accuracy and batch processing.Therefore,through analyzing the current gridding methods and inspired by the human simulated theory,we introduce the active analysis into image processing.First,via autonomous perception,we make sure the existence and degree of uncertainty factors in image processing procedure.Then we reduce or eliminate the affection by adopting corresponding methods.Finally,experiments on 384 sub-grids drawn from six different data sets prove that the effectiveness of the proposed automatic contrast enhancement method and gridding result self-adjusting.In addition,the proposed method needs no human intervention and parameter setting,and can improve the gridding accuracy to a large extent.
microarray;image processing;gridding
TP 391.41
A
0438-0479(2016)06-0888-07
10.6043/j.issn.0438-0479.201511006
2015-11-04 录用日期:2016-06-28
国家自然科学基金(61403318);福建省自然科学基金(2013J01255)
gfshao@xmu.edu.cn
李铁军,孙跃,邵桂芳,等.基于主动分析的基因芯片图像网格定位[J].厦门大学学报(自然科学版),2016,55(6): 888-894.
LI T J,SUN Y,SHAO G F,et al.An active analysis method for microarray image gridding[J].Journal of Xiamen University(Natural Science),2016,55(6):888-894.(in Chinese)
