微小RNA let-7a及其靶基因K-ras多态性与局部晚期食管鳞癌放化疗疗效的相关性研究
2016-12-06张建清汤旭山马晓丽
张建清, 汤旭山, 杨 媚, 白 鸽, 马晓丽, 张 莉
(新疆医科大学1第一附属医院肿瘤中心, 乌鲁木齐 830054; 2附属肿瘤医院消化内科, 乌鲁木齐 830011;
微小RNA let-7a及其靶基因K-ras多态性与局部晚期食管鳞癌放化疗疗效的相关性研究
张建清1, 汤旭山2, 杨 媚1, 白 鸽1, 马晓丽3, 张 莉3
(新疆医科大学1第一附属医院肿瘤中心, 乌鲁木齐 830054;2附属肿瘤医院消化内科, 乌鲁木齐 830011;
3第一附属医院VIP内科, 乌鲁木齐 830054)
目的 探讨微小RNA let-7a及其靶基因K-ras多态性与食管鳞癌患者放化疗疗效的关系。方法 收集局部晚期食管鳞癌患者(病例组,92例)和正常体检者(正常组,92例)血液标本。进行微小RNA let-7a rs10877887位点、rs13293512位点及靶基因K-ras rs712位点单核苷酸多态性检测。分析两组间各位点多态性的分型与分布特点及与放化疗疗效、预后及中位PFS的相关性,并运用荧光素酶报告基因实验K-ras是否为微小RNA let-7a的直接靶基因。结果 放化疗疗效与食管鳞癌分期及有无淋巴结转移有关(P<0.05)。两组基因多态性分析显示,rs10877887位点多态性无明显差异(P>0.05), rs13293512 携带 CC基因型的个体以及非TT基因型的个体食管鳞癌的发病风险增加(P<0.05),rs712位点携带TT基因型和非GG基因型的个体食管鳞癌的发病风险明显增加(P<0.05)。同时荧光索酶报告实验证明K-ras是微小RNA let-7a的直接靶基因。预后分析发现,rs10877887位点对疗效和预后无影响(P>0.05);rs13293512 携带CC基因型的个体,rs712位点携带TT基因型和非GG基因型的个体,有效率明显降低,中位PFS生存时间明显减少(P<0.05)。COX回归分析,显示淋巴有效治疗转移、临床分期Ⅳ期、rs13293512位点CC基因型、rs712位点TT基因型是食管鳞癌预后的独立危险因素。结论 rs13293512位点CC基因型与rs712位点TT基因型增加局部晚期食管鳞癌术后放化疗抵抗性和患病风险,降低放化疗有效疗效和中位PFS生存时间。在放化疗前对患者进行基因型检测,在一定程度上可以预测患者对放化疗的敏感性。
微小RNA let-7a;K-ras; 食管鳞癌; 基因多态性; 放化疗; 预后; 靶基因; 相关性
食管癌(Esophagus cancer, EC)是消化道第三常见的癌症,也是全球因癌致死的第七大死因,中国是EC发病率最高的区域之一。食管鳞状细胞癌即食管鳞癌(esophageal squamous cell carcinoma, ESCC)则是世界上最常见的EC组织学类型之一,虽然在美国表现出不断下降的趋势,但在发展中国家的发病率较高[1-2]。目前,国内外食管癌缺乏早期诊断方法,确诊时80%已为中晚期,5年生存率仅为15%~20%,中位生存期为18个月[3]。根据美国国家癌症综合网(NCCN)指南,中晚期食管癌主要以放化疗为主的综合治疗,但总体有效率不高。由于食管癌的高度侵袭性、较差的临床疗效和预后、综合治疗模式众多的特点,很难对其进行有效的预后判断及预测治疗疗效来提高患者生存率。因此寻找合适的靶点判断ESCC患者放化疗疗效对于ESCC的治疗与预后具有重大意义。
微小RNAs(MicroRNAs, miRNAs)属于非蛋白编码RNAs,是由长的内源性表达的RNA前体(primary RNA, pri-miRNA)产生,一个miRNA能调控几个基因的表达,一个基因能被一系列不同的miRNAs所调控,而let-7a的异常表达与许多人类疾病有所关联[4],包括心血管疾病[5]、肺病[6]和癌症[7]。MiRNAs的let-7a家族首先是在秀丽线虫中被发现,该家族在人体中包含13个成员,分布于9条不同的染色体上,而RAS是其靶基因[8]。K-ras基因位于人类染色体12q12.1,定位于细胞膜内侧[9],在许多人类癌症中该基因的突变十分常见[10]。有多项研究指出K-ras基因突变在多种人类癌症的发展进程中扮演着重要角色,尤其是在密码子12、13和61上[11-12]。关于miRNA let-7a及其靶基因K-ras多态性与ESCC患者放化疗疗效关系的研究相对较少。本研究拟通过检测微小RNA let-7a rs10877887位点、rs13293512位点及靶基因K-ras rs712位点的单核苷酸多态性,进一步确定miRNA let-7a及其靶基因K-ras多态性是否能影响ESCC患者放化疗疗效。
1 材料与方法
1.1 临床资料 根据美国癌症联合委员会(AJCC)和国际抗癌联盟(UICC)联合制定的食管癌TNM分期标准[13],选择2013年1月-2015年1月经新疆医科大学第一附属医院及附属肿瘤医院病理证实的Ⅲ、Ⅳ期局部晚期食管鳞癌患者92例为病例组。纳入标准:(1)经病理学诊断的原发性食管鳞癌首诊患者;(2)有可测量的病灶,年龄30~65岁,性别不限,身体状况评分PS(WHO Performance Status)≤0~2,能耐受放疗或化疗,预计生存期≥6个月;(3)患者无主要器官的功能障碍,血常规、肝、肾功能及心脏功能基本正常。排除标准:(1)继发性食管鳞癌患者;(2)曾经接受过手术治疗、放疗及化疗;(3)合并其它恶性肿瘤的患者。随机选取92例健康体检者为对照组。本项研究得到新疆医科大学第一附属医院及附属肿瘤医院伦理委员会的批准,受试者均签署知情同意书并享有知情权。
病例组92例,男性55例,女性37例,平均年龄(50.00±5.46)岁。对照组92例,男性53例,女性39例,平均年龄(49.08±4.92)岁。两组患者年龄、性别构成、吸烟史和饮酒史差异均无统计学意义(P>0.05)(表1)。
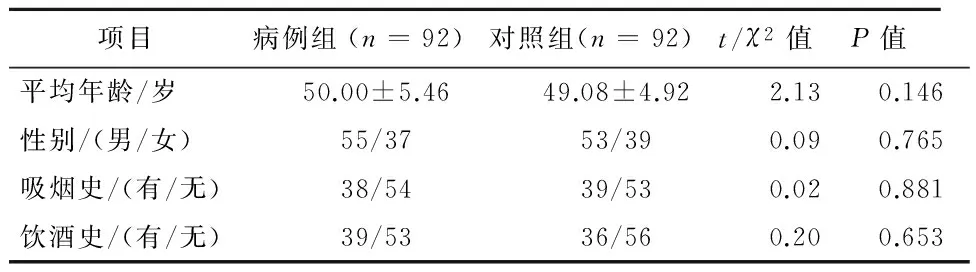
表1 病例组和对照组的一般资料比较
1.2 血清样本的采集及DNA的提取 收集病例组放化疗前和对照组患者的静脉血,取血后,37℃下血液凝固1~2 h(不加抗凝剂);4℃冰箱过夜;当血清自然析出后,以4℃、3 000 r/min离心10 min,分离血清,弃去不溶物;将血清移至干净试管,并分装成小份,储藏在-80℃超低温冰箱备用。血清样本收集和保存的整个操作过程中应尽可能缩短标本暴露时间,以防止标本中RNA降解,影响实验结果。采集所有入选研究对象外周静脉血2~3 mL,EDTA抗凝,酚/氯仿法提取基因组DNA。
1.3 微小RNA let-7a及其靶基因K-ras多态性分析 采用聚合酶链反应-限制性长度片段多态性进行微小RNA let-7a启动子区rs13293512(let-7a-1/let-7f-1/let-7d基因簇启动子区)和 rs10877887(let-7i启动子区)2 个多态性位点及靶基因K-ras rs712分型。根据文献引物合成序列设计引物,rs13293512上游引物:5′-GCAGGGAAGACAGTGAATGTTAAA-3′,下游引物: 5′-CCAAGAAAGCAGTATTATCAATGAAGTT-3[14], rs10877887上游引物5′-TGGTGTCTGACTGCGCTTT-3′,下游引物:5′-CCGAGAGCTACGGGGATGA-3′[15];K-ras rs712的引物为:5′-ATGACAGTGGAAGT-
TTTTTTTTCCTC-3′,下游:5′-GAATCATCAT-
CAGGAAGCCCAT-3′[16]。由生工生物工程(上海)股份有限公司合成。PCR反应体系为25 μL,含 10×PCR buffer 2.5 μL,dNTPs 2.5 μL,上、下游引物各1.0 μL,模板DNA 2.0 μL,氯化镁1.87 μL,Taq polymerase 0.23 μL,ddH2O 15 μL,PCR反应条件为:95.0℃ 2 min,95.0℃ 30 s,55℃ 30 s,72.0℃ 30 s,共40个循环;最终72.0℃ 5 min。PCR产物运用DNA全自动测序技术(上海铂尚生物有限公司测序),结果验证无误(图1)。
1.4 荧光素酶报告基因实验 本实验细胞为食管鳞癌细胞ECA109(中科院上海细胞库),对其进行传代培养后用于荧光素酶报告基因实验。合成含let-7a结合位点的K-ras 3′UTR启动子区序列,在pGL3 control vector(Promega, USA)的5′端Bg1Ⅱ位点插入,构建K-ras 3′UTR野生型(Wild)质粒(命名为K-ras 3′UTR-Wild )。并在该质粒基础上,突变let-7a结合位点(CUACUUUA),构建K-ras 3′UTR突变型(Mutant)质粒(命名为K-ras3′UTR-Mutant)。再将重组质粒与微小RNA let-7a模拟物(let-7a-mimics)或阴性对照序列(let-7a-NC)共同转染食管鳞癌细胞。具体为1.5×104个/孔接种于每孔100 μL培养基的96孔板中,于37℃、5% CO2及饱和湿度培养箱中培养24 h。次日更换每孔培养基至50 μL培养基,取10 μL减血清培养基OPTI-MEM (Thermo Fisher Scientific, 美国) 稀释let-7a-mimics至100 nmol/L,15 μL OPTI-MEM稀释重组质粒K-ras-Wild或K-ras-Mutant 100 ng,25 μL OPTI-MEM稀释Lipofectamine 2000 0.25 μL,5 min 后三者混合,轻轻摇匀,室温静置20 min。将上述混合液50 μL加入孔中,最终每孔总体积100 μL,每组设3个复孔,转染6 h后再加入100 μL新鲜培养基。转染48 h后使用荧光素酶报告基因检测试剂盒 (碧云天,中国) 进行双荧光素酶报告基因检测,并比较各组相对荧光素酶活性 (hRluc/hLuc)。
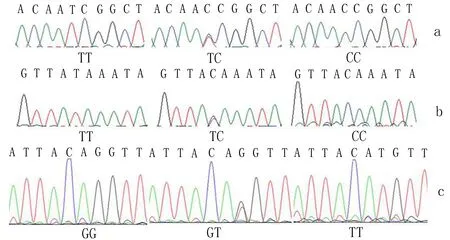
a:rs10877887位点测序结果;b:rs13293512位点测序结果;c:rs712位点测序结果
图1 微小RNA let-7a及其靶基因K-ras测序结果
1.5 放化疗治疗方法和疗效评价 采用同期放化疗联合治疗(根据NCCN及我国食管癌治疗指南):化疗方案:紫杉醇/多西他赛+卡铂/顺铂(剂量:紫杉醇(135~175)mg/m2·d,d1, 多西他赛75 mg/m2·d,d1;卡铂:AUC:4~5,d1,顺铂:75 mg/m2· d,d1~2),21 d1次;放疗方案:调强照射(IMRT),DT50~66 Gy/25~33 f。共2个周期,与放疗同期后序贯2~4个周期化疗。根据实体瘤评价标准(Response Evaluation Criteria in Solid Tumors RECIST 标准1.0)[17]:确定近期疗效为完全缓解(CR)、部分缓解(PR)、稳定(SD)或是进展(PD)。以CR+PR归为治疗有效组,以SD + PD归为治疗抵抗组。
1.6 随访 采用电话、门诊及查阅病历进行随访,随访截止时间为2016年6月,随访3~36个月,6例删失,随访率为93.0%。无进展生存时间(Progression Free Survival,PFS)定义为自治疗开始至癌症复发或患者的死亡时间。
2 结果
2.1 治疗有效组和抵抗组临床资料对比分析 根据患者的治疗情况,比较治疗有效组和抵抗组患者的临床资料,经统计学分析,放化疗疗效与食管鳞癌临床分期及有无淋巴结转移有关,与其他(年龄、性别、分化程度、食管癌部位)无关,差异有统计学意义(P均<0.05)(表2) 。
2.2 基因型分析及基因频率比较 rs10877887位点多态性分析,对照组和病例组基因型频率分布差异无统计学意义(P>0.05)。rs13293512位点多态性分析,病例组CC基因型和非TT基因型分布显著高于对照组,携带CC基因型的个体以及非TT基因型的个体患病风险增加(OR=4.26,95%CI=1.80~10.06,P=0.001;OR=2.27,95%CI=1.20
~4.32,P=0.017)。RS712位点多态性分析,病例组TT基因型和非GG基因型分布明显高于对照组,rs712携带TT基因型和非GG基因型的个体食管鳞癌的发病风险增加(OR=4.88,95%CI=1.80~13.25,P=0.002;OR=1.93,95%CI=1.08~3.47,P=0.039)(表3)。
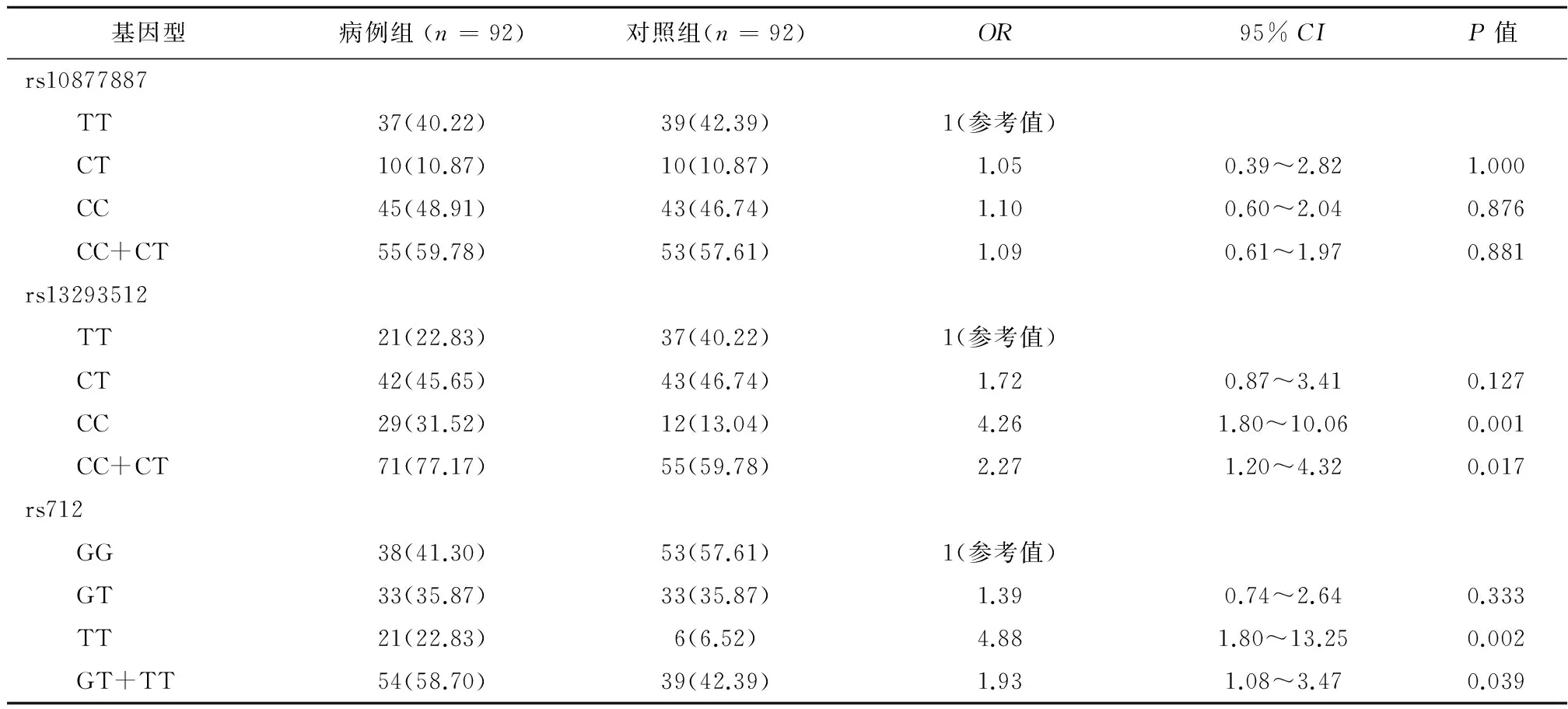
表3 两组基因频率分布比较/例(%)
2.3 K-ras是微小RNA let-7a的直接靶基因 利用生物医学数据库,根据基因互补结合原理分析基因结构,结合预试验初步确定K-ras为食管鳞癌细胞中微小RNA let-7a的靶基因之一(图2a),let-7a可能通过识别并结合K-ras mRNA发挥生物学作用。let-7a mimics与构建K-ras基因的3′UTR及突变的荧光索酶报告质粒共转染食管鳞癌细胞后,与NC组相比,let-7a 与野生型质粒共转染使海肾荧光索酶活性下降,海肾荧光索酶与萤火虫荧光素酶活性之比下降(P<0.05),而let-7a对突变质粒的作用无明显差异(P=0.404)(图2b)。
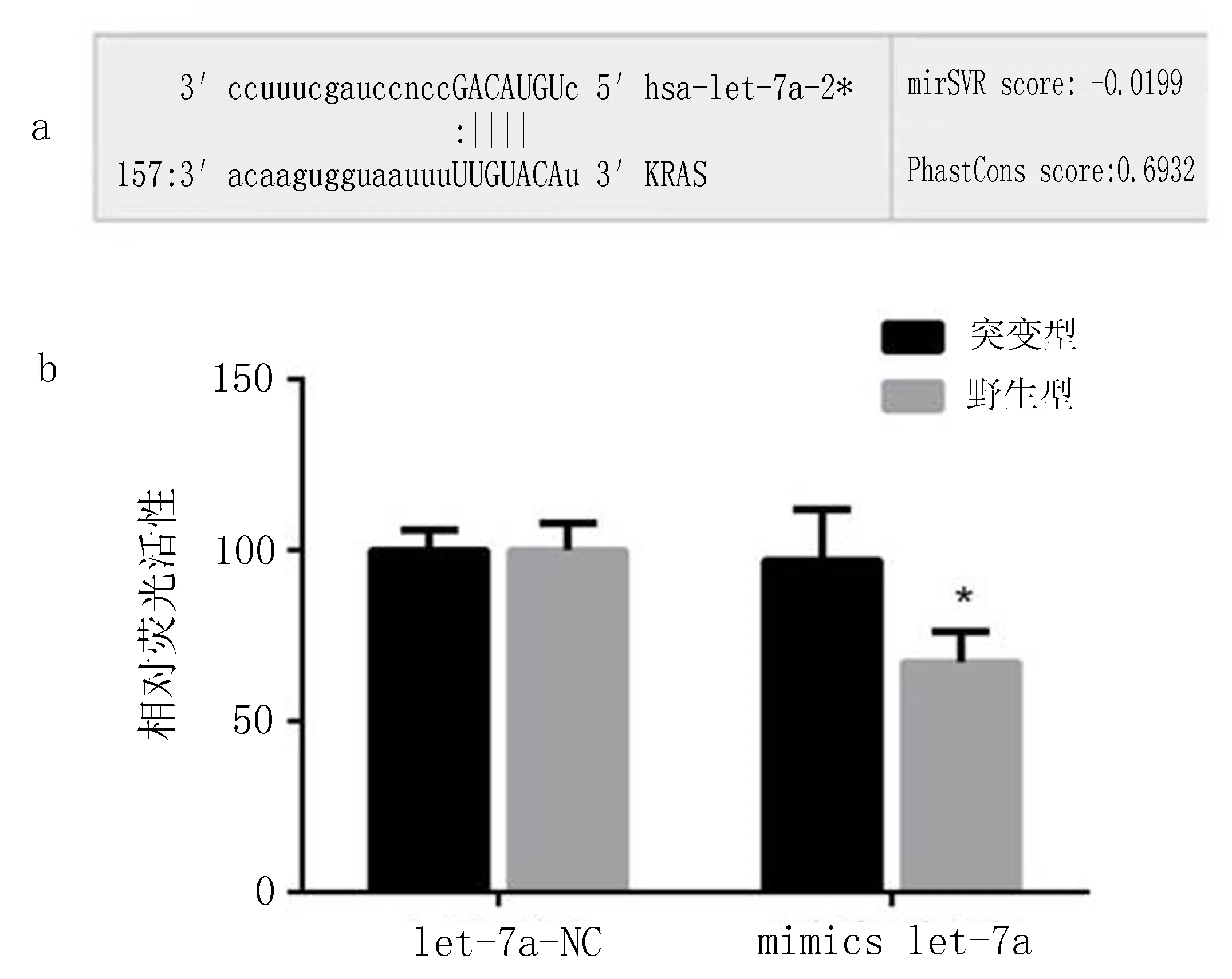
a: miRNA靶位预测软件预测K-ras存在let-7a的结合靶点; b: 荧光素酶报告基因实验检测食管鳞癌细胞各处理组荧光活性。 与NC相比,*P<0.05。
图2 let-7a与K-ras的靶向验证
2.4 基因多态性与食管鳞癌放化疗疗效的关系 rs10877887位点各基因型患者放化疗有效治疗率差异无统计学意义(P>0.05)。rs13293512位点携带CC基因型的个体与携带TT基因型的个体相比,食管鳞癌的放化疗抵抗能力增加,有效治疗率显著降低(P<0.05);rs712位点,与GG基因型相比,携带TT基因型和非GG基因型的个体显著增加食管鳞癌的放化疗抵抗性能,差异具有统计学意义(P<0.05)(表4)。
2.5 基因多态性与食管鳞癌预后的关系 rs10877887位点TT、TC、CC基因型中位PFS分别为21.9、20.7、20.8个月,各基因型组间差异不具有统计学意义(P>0.05)(图3a)。rs13293512位点TT、TC、CC基因型中位PFS分别为23.1、23.1、18.7个月,rs13293512携带CC基因型的个体中位生存时间明显降低(P<0.001)(图3b)。靶基因K-ras rs712位点GG、GT、TT基因型中位PFS分别为22.6、22.1、18.6个月,RS712携带非GG基因型的个体中位生存时间明显降低(P<0.001)(图3c)。
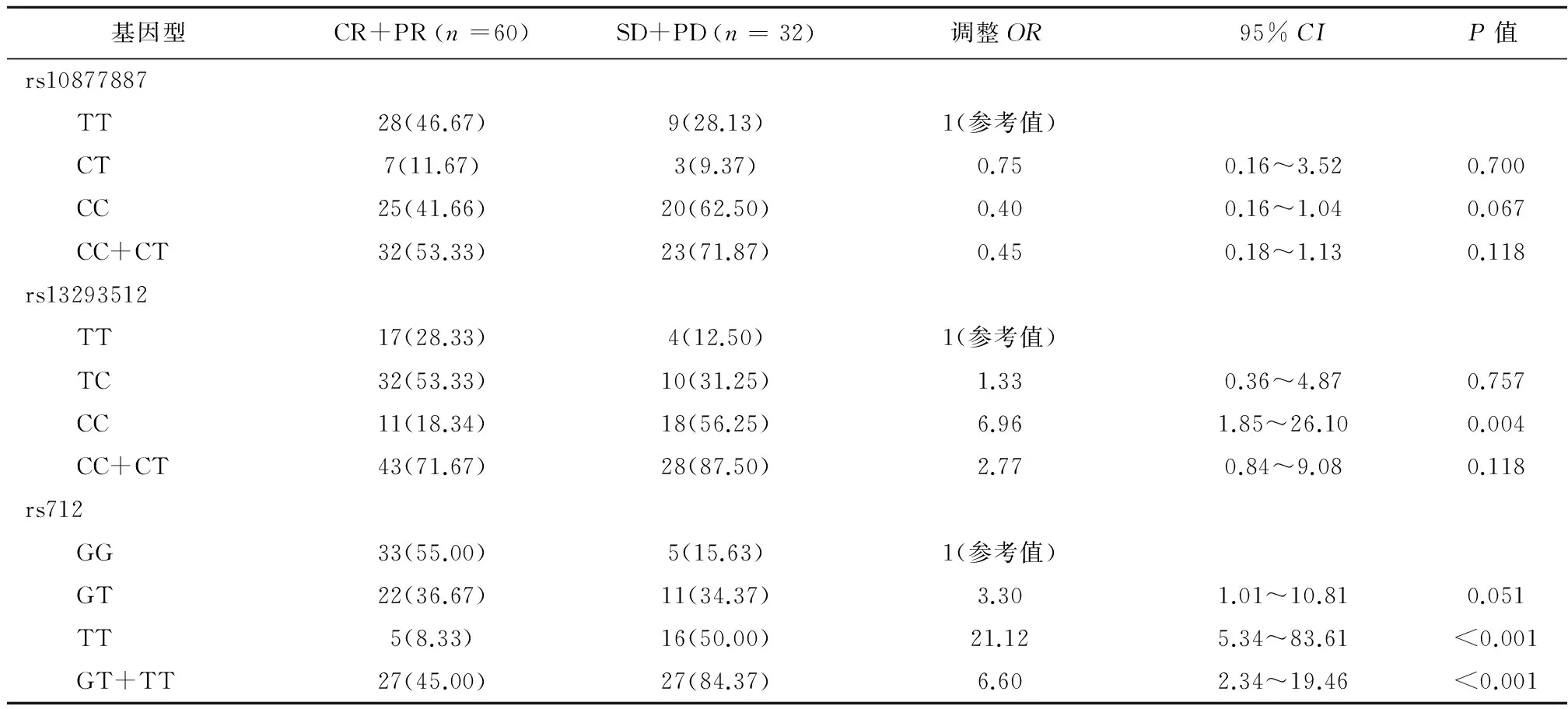
表4 基因多态性与食管鳞癌术后放化疗疗效的关系/例(%)
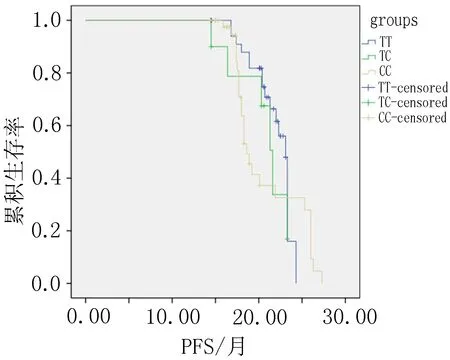
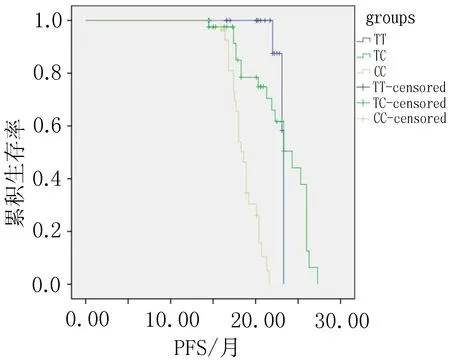
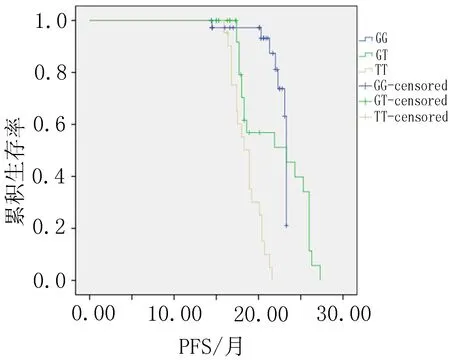
a: rs10877887位点基因型中位无进展生存时间曲线 b: rs13293512位点基因型中位无进展生存时间曲线 c: rs712位点基因型中位无进展生存时间曲线
图3 微小RNA let-7a及其靶基因K-ras基因多态性中位无进展生存时间曲线
2.6 食管鳞癌预后COX回归分析 将患者临床分期、淋巴转移情况、分化程度、rs13293512位点、rs712位点进行危险因素分析,结果显示有淋巴转移、临床分期Ⅳ期、rs13293512位点CC基因型、rs712位点TT基因型是影响食管鳞癌预后的独立危险因素(P<0.05)(表5)。
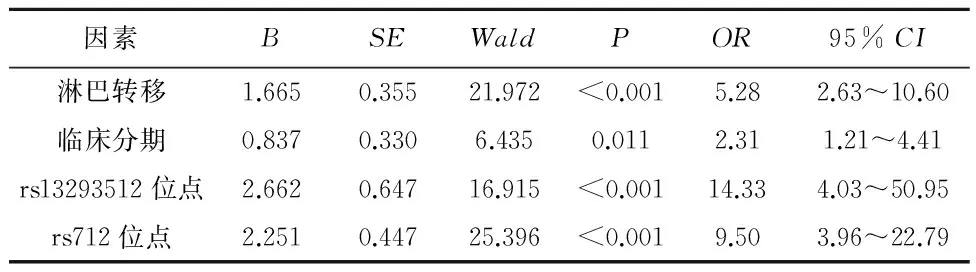
表5 食管鳞癌预后独立危险因素分析
3 讨论
ESCC是世界上最常见和致命的恶性肿瘤之一,与西方国家相比,东亚的发病率更高,基因组成可能促成食管上皮细胞的恶化[18]。近来,许多研究者把目光投向了miRNAs领域,有关let-7a、K-ras与直肠癌[19]、胰腺癌[20]等癌症的报道并不少见,但将上述基因与ESCC联系起来的研究相对较少。因此,本研究旨在探讨微小RNA let-7a及其靶基因K-ras多态性与ESCC患者放化疗疗效的关系,发现RNA let-7a的rs13293512位点CC基因型与K-ras的rs712位点TT基因型能增加局部晚期ESCC术后放化疗抵抗性和患病风险,降低放化疗有效疗效和中位PFS生存时间,提示在放化疗前对患者进行基因型检测,在一定程度上可以预测患者对放化疗的敏感性。
本研究发现病例组let-7a rs13293512位点CC基因型和非TT基因型分布高于对照组,表明携带CC基因型的个体以及非TT基因型的个体患病风险增加,且ESCC的有效治疗率显著降低。let-7a在许多癌症中存在表达下调[21],其miRNA表达水平与正常相比降低,且该miRNA的靶基因Ras又是原癌基因,意味着Ras受到let-7a的抑制作用减小,癌基因编码的蛋白增加,从而导致肿瘤的发生[22]。let-7a基因上的SNPs可能改变miRNA-mRNA的相互作用,从而改变其基因调节功能,影响多种恶性肿瘤的发生、发展。有研究显示let-7a rs13293512位点多态能够影响到中国人群患癌易感性[23]。Smits等[24]的研究也显示let-7a基因多态性可能成为癌症预后指标。此外,Shen等[25]的研究表明,let-7a结合位点的SNPs与癌病变和生存率密切相关,患者携带CC+CT基因型比起携带TT基因型更有可能患得癌症,C等位基因是风险因素,本研究与其报道基本一致。
本研究发现病例组K-ras rs712位点的TT基因型和非GG基因型分布明显高于对照组,表明携带TT基因型和非GG基因型的个体ESCC的发病风险增加。Ras基因人类肿瘤中相对常见的癌基因,其家族包含H-ras、K-ras和N-ras3个重要成员[26],而K-ras基因是癌症中最常见的突变基因之一[27]。Ras基因突变导致细胞增殖的持续激活,在许多人类肿瘤中,K-ras基因突变都发生频繁,且ras蛋白质的过度表达能抑制细胞凋亡,推动肿瘤的发生、发展[28]。有研究表明,与GG基因型相比,TT和GT/TT基因型显著增加了肿瘤的发病风险,T等位基因携带者肿瘤分期更高,提示K-ras rs712等位基因多态性很可能是肿瘤的易感因素[29]。此外,本研究经ESCC预后COX回归分析发现淋巴转移、临床分期Ⅳ期、rs13293512位点CC基因型、rs712位点TT基因型均是影响食管鳞癌预后的独立危险因素,进一步证实了上述结论。
综上所述,本研究发现了let-7a rs13293512位点CC基因型与rs712位点TT基因型能增加局部晚期食管鳞癌术后放化疗抵抗性和患病风险,从而降低放化疗有效疗效,提示在放化疗前对患者进行基因型检测,在一定程度上可以预测患者对放化疗的敏感性。然而,目前关于let-7a rs13293512位点CC基因型与rs712位点TT基因型之间有无相关性、两者同时存在是否患者预后更差的相关研究尚少,还需要进一步的研究来证实以及阐明内在机制。
[1] Napier KJ, Scheerer M, Misra S, et al. Esophageal cancer: A Review of epidemiology, pathogenesis, staging workup and treatment modalities[J]. 世界胃肠肿瘤学杂志:英文版, 2014, 6(5):112-120.
[2] Giri S, Pathak R, Aryal MR, et al. Incidence trend of esophageal squamous cell carcinoma: an analysis of Surveillance Epidemiology, and End Results (SEER) database[J]. Cancer Causes Control,2015, 26(1):159-161.
[3] Jemal A, Clegg LX, Ward E, et al.Annual report to the nation on the status of cancer, 1975-2001, with a special feature regarding survival[J].Cancer,2004, 101(1):3- 27.
[4] Wagner S, Ngezahayo A, Murua EH, et al. Role of miRNA let-7a and its major targets in prostate cancer[J]. Biomed Res Int,2014,2014:376326-376326.
[5] Bao MH, Feng X, Zhang YW, et al. let-7a in Cardiovascular Diseases, Heart Development and Cardiovascular Differentiation from Stem Cells[J]. Int J Mol Sci,2013,14(11):23086-23102.
[6] Polikepahad S, Knight JM, Naghavi AO, et al. Proinflammatory role for let-7a microRNAS in experimental asthma[J].J Biol Chem, 2010, 285(39):30139-30149.
[7] Johnson CD,Esquela-Kerscher A, Stefani G, et al. The let-7a microRNA represses cell proliferation pathways in human cells[J]. Cancer Res, 2007, 67(16):7713-7722.
[8] Boyerinas B, Park SA, Murmann AE, et al. The role of let-7a in cell differentiation and cancer[J].Endocr Relat Cancer , 2010, 17(1):19-36.
[9] 刘伟, 王丽, 余英豪,等. k-ras基因在中国结直肠癌患者中的突变状态[J]. 世界华人消化杂志, 2011,(13):1367-1374.
[10] Polipalli S. K-RAS Gene mutations at codon 12 and 13 in pancreatic cancer and chronic pancreatitis patients from north indian population[J]. Int J Bio, 2014, 3(9):3280-3285.
[11] Zhang AM, Wang R, Sun K, et al. Diagnosis value of detection K-Ras gene mutations in patients with gastric cancer[J].Adv Materials Res,2012, 396-398(7):1470-1474.
[12] Wegman P, Ahlin C, Sorbe B. Genetic alterations in the K-Ras gene influence the prognosis in patients with cervical cancer treated by radiotherapy[J]. Int J Gynecol Cancer,2011, 21(1):86-91.
[13] Rice TW. Esophageal Cancer Staging[J]. Korean J Thorac Cardiovasc Surg, 2015, 48(3):157-163.
[14] 黄芳,胡铃敏,刘继斌,等. let-7a启动子区基因多态性与肝细胞肝癌遗传易感性的关联[J].中华预防医学杂志,2011,45(12):1093-1095.
[15] Shen LQ, Xie YZ , Qian XF,et al. A single nucleotide polymorphism in the promoter region of let-7a family is associated with lung cancer risk in Chinese[J]. Genet Mol Res, 2015,14 (2): 4505-4512.
[16] Zhao HL, Pan XM, Han BW, et al. A let-7a binding site polymorphism rs712 in the KRAS 3′ UTR is associated with an increased risk of gastric cancer[J].Tumor Biol, 2013,34:3159-3163.
[17] Sugimura K, Miyata H, Tanaka K, et al. let-7a expression is a significant determinant of response to chemotherapy through the regulation of IL-6/STAT3 pathway in esophageal squamous cell carcinoma[J]. Clin Cancer Res, 2012, 18(18):5144-5153.
[18] Zhang X, Zhou L, Fu G, et al. The identification of an ESCC susceptibility SNP rs920778 that regulates the expression of lncRNA HOTAIR via a novel intronic enhancer[J]. Carcinogenesis, 2014, 35(9):2062-2067.
[19] Duldulao MPN, Lee W, Le M, et al. let-7a microRNA regulates p53 and K-ras in colorectal cancer cells[J].J Am Coll Surg, 2011, 213(3):S25.
[20] Appari M, Babu KR, Kaczorowski A, et al. Sulforaphane, quercetin and catechins complement each other in elimination of advanced pancreatic cancer by miR-let-7a induction and K-ras inhibition[J]. Int J Oncol, 2014, 45(4):1391-1400.
[21] 刘清. 食管鳞癌中微小RNAlet-7a的作用及其对高迁移率蛋白A2的影响[D]. 乌鲁木齐: 新疆医科大学, 2011.
[22] 朱龙彪, 袁华, 张宇,等. let-7a基因多态与中国汉族人群头颈癌易感性的关联研究[J]. 口腔生物医学, 2014,5(2):57-60.
[23] Smits KM, Paranjape T, Nallur S, et al. A let-7a microRNA SNP in the KRAS 3′UTR is prognostic in early-stage colorectal cancer[J]. Clin Cancer Res, 2011, 17(24):7723-7731.
[24] Shen LQ, Xie YZ, Qian XF, et al. A single nucleotide polymorphism in the promoter region of let-7a family is associated with lung cancer risk in Chinese.[J]. Genet Mol Res, 2015, 14(2):4505-4512.
[25] 夏晓明, 金玮韵, 施仁忠,等. 非小细胞肺癌组织中let-7a、K-ras基因的表达变化及意义[J]. 山东医药, 2010, 50(42):74-75.
[26] Dalay N, Yalniz Z, Gurbuz O. Abstract A48: The K-ras let-7a miRNA binding site variant and K-ras mutations in colon cancer[J]. Mol Cancer Res, 2014, 12(12 Supplement):A48-A48.
[27] Lyronis ID, Baritaki S, Bizakis I, et al. K-ras mutation, HPV infection and smoking or alcohol abuse positively correlate with esophageal squamous carcinoma[J]. Pathol Oncol Res, 2008, 14(14):267-273.
[28] Xia XM, Jin WY, Shi RZ, et al. Clinical significance and the correlation of expression between let-7a and K-ras in non-small cell lung cancer[J]. Oncol Lett,2010, 1(6):1045-1047.
[29] 岩亮, 汪庆伟, 田昆仑. let-7a靶基因KRAS结合区rs712多态性与脑胶质瘤发生相关性的研究[J]. 中华肿瘤防治杂志, 2013, 20(11):811-814.
本文编辑 杨晨晨)
Effects of miRNA let-7a and K-ras polymorphisms on chemoradio therapy efficiency in patients with locally advanced esophageal squamous cell carcinoma
ZHANG Jianqing1, TANG Xushan2, YANG Mei1, BAI Ge1, MA Xiaoli3, ZHANG Li3
(1CancerCenter,theFirstAffiliatedHospitalofXinjiangMedicalUniversity,Urumqi830054,China;2DepartmentofInternalMedicineofDigestiveTumor,theAffiliatedTumorHospitalofXinjiangMedicalUniversity,Urumqi830011,China;3VIPDepartmentofInternalMedicine,theFirstAffiliatedHospitalofXinjiangMedicalUniversity,Urumqi830054,China)
Objective To investigate relationship between miRNA let-7a, K-ras polymorphisms and efficacy of chemoradiotherapy for esophageal squamous cell carcinoma (ESCC) patients. Methods Blood specimens of 92 patients and 92 healthy controls were respectively collected. Single nucleotide polymorphisms (SNPs) of let-7a (rs10877887 and rs13293512) and K-ras rs712 were detected. The genotyping and distribution of SNPs and their relevance with chemoradiotherapy efficacy and median Progression Free Survival (PFS) time were analyzed. Luciferase reporter gene assay was used to determine whether K-ras is the target gene of let-7a. Results The efficacy of chemoradiotherapy was correlated with ESCC tumor-node-metastasis staging and lymph node metastasis (LNM) (P<0.05). As gene polymorphism analysis demonstrating, no significant difference was found in rs10877887, while individuals with rs13293512 CC and non-TT genotypes had increased ESCC risks (P<0.05), so did those with rs712 TT and non-GG (P<0.05). Luciferase reporter gene assay showed that K-ras is direct targeting gene of let-7a. As prognostic analysis showed, individuals with rs10877887 had no effect on efficacy and prognosis, while individuals with rs13293512 CC genotype or rs712 TT and non-GG genotypes had a lower effective treatment rate and shorter median PFS time (P<0.05). Cox logistic analysis revealed that LNM , Ⅳstage, rs13293512 CC and rs712 TT genotypes are ESCC risk factors. Conclusion Rs13293512 CC and rs712 TT genotypes increase the risk and the resistance of chemoradiotherapy for locally advanced ESCC, and lower the efficacy of chemoradiotherapy and median PFS time, which implicated sensitivity of patients to chemoradiotherapy can be potentially predicted by genotyping before the chemoradiotherapy.
MiRNA let-7a; K-ras; esophageal squamous cell carcinoma; polymorphism; chemoradiotherapy; prognosis; target gene
新疆维吾尔自治区自然科学基金(2013211B61)
张建清(1982-),女,在读博士,主治医师,研究方向:食管癌的个体化治疗。
张 莉,女,教授,博士生导师,主任医师,研究方向:食管癌的个体化治疗,E-mail: zhangli9514@126.com。
R735.1
A
1009-5551(2016)12-1547-07
10.3969/j.issn.1009-5551.2016.12.016
2016-07-06]
