陆地棉CAD基因家族的进化和表达分析
2016-12-02张经博李波杨洋胡文冉陈方圆谢丽霞范玲
张经博,李波,杨洋,胡文冉,陈方圆,谢丽霞,范玲
(1. 新疆师范大学生命科学学院,乌鲁木齐 830054;2. 新疆农业科学院核技术生物技术研究所,乌鲁木齐 830091)
陆地棉CAD基因家族的进化和表达分析
张经博1,2,李波2,杨洋2,胡文冉2,陈方圆1,2,谢丽霞2,范玲2
(1. 新疆师范大学生命科学学院,乌鲁木齐 830054;2. 新疆农业科学院核技术生物技术研究所,乌鲁木齐 830091)
【目的】对陆地棉CAD基因家族成员进行进化和表达分析,了解其在棉纤维发育中的作用。【方法】应用生物信息学方法,从棉花基因组中筛选出21个CAD基因,并对CAD的基因结构、染色体分布、基因倍增模式以及系统进化进行分析。利用深度表达数据分析CAD基因在棉纤维发育各时期的表达。对比观察拟南芥cad5突变体和野生型根毛表型。 【结果】同源性高的CAD基因复制对之间有相似的基因结构;片段重复和串联重复是CAD基因扩增的主要方式。进化分析可知,CAD基因可分为七个亚组,且功能相近的基因聚类在相同亚组。利用深度测序表达数据分析可知,CAD基因在棉纤维各个发育时期均有表达。对比拟南芥cad5突变体和野生型根毛发现,突变体相比于野生型有更长的根毛。【结论】CAD基因参与了棉纤维的发育的过程,并对棉花纤维发育起到一定的调控作用。
棉纤维;CAD基因家族;系统进化分析;表达分析;cad5突变体;根毛
0 引 言
【研究意义】棉花是一种重要的经济作物,在全球100多个国家广泛种植,由它生产的棉纤维是全球重要的纺织原料之一,具有极其重要的经济价值[1]。陆地棉(Gossypiumhirsutum)是栽培面积及经济效益最大的栽培棉种,它贡献了超过90%的棉花产量。此外棉花还是研究染色体加倍,细胞伸长和细胞壁合成的重要模式植物[2]。棉花纤维是由胚珠表皮单细胞分化而成, 在纤维发育过程初期,纤维细胞以极快的速度同步化延伸,并最终分化成为超长的单一极性细胞[3],棉纤维需要经历纤维原始细胞的分化和突起、初生壁伸长、次生壁增厚和脱水成熟 4 个依次并相互重叠的过程,一般 45~50 d,涉及上千个基因的特异表达和蛋白相互作用[4]。研究棉纤维发育为棉纤维品质改良提供依据,阐明植物细胞伸长的重要手段,具有显著的学术理论价值和潜在经济价值。随着陆地棉的基因组测序工作已经完成,为在陆地棉中识别不同的基因家族以及了解其在棉花生长发育过程中的作用提供了可能[5]。【前人研究进展】细胞壁是植物区别与动物的主要器官之一。成熟棉花纤维细胞主要的组成部分就是细胞壁,细胞壁的沉积与改变对植物的生长、发育以及对外界环境的响应都有重要的作用,它最终决定了植物细胞的大小与形状。木质素是高等植物细胞壁的重要组成成分[6]。棉花纤维是单一超长的极性细胞,成熟的棉花纤维细胞壁中存在有木质素,木质素通常在次生细胞壁中积累[7, 8]。肉桂醇脱氢酶(cinnamyl alcohol dehydrogenase, CAD) 是一类参与木质素合成途径中的酶[9], 通常情况下, 它能够将木质素前体和木脂素类物质转化成木质素单体。 因此,CAD基因在植株生长发育过程中的作用尤为重要。CAD蛋白都具保守结构域 ADH_N 和ADH_zinc_N[10, 11]。根据种间核苷酸和氨基酸序列同源性的差异可将CAD基因分成 3 类, 其中第 1 类主要是石松类 、单子叶 、真双子叶(eudicots) 和裸子植物的CAD基因; 第 2 和第 3 类主要是单子叶和真双子叶植物的CAD基因。 第一类CAD基因都能够参与到维管束的发育和和木质素的合成过程,在木质素合成过程中它们将松柏醛和芥子醛还原成相应醇[12], 同第 1 类CAD基因相比第 2 和第3 类CAD基因在木质素合成中的作用并不突出, 可能是第1类CAD基因的冗余基因,也不能排除在植物发育其它方面的功能作用。 【本研究切入点】目前已经在多种植物中对CAD基因成员进行了鉴定[13]。全面的基因家族分析尚未在棉花中研究。随着陆地棉基因组测序的完成,能够在全基因组水平上对CAD基因家族进行分析和阐述。【拟解决的关键问题】利用生物信息学方法,对陆地棉全基因组的CAD基因进行鉴定和系统进化分析,并对其在棉纤维发育过程中的基因表达模式进行分析,为改良棉花纤维品质等重要农艺性状提供候选基因,为全基因组水平上的棉花分子育种提供参考和基础。
1 材料与方法
1.1 材 料
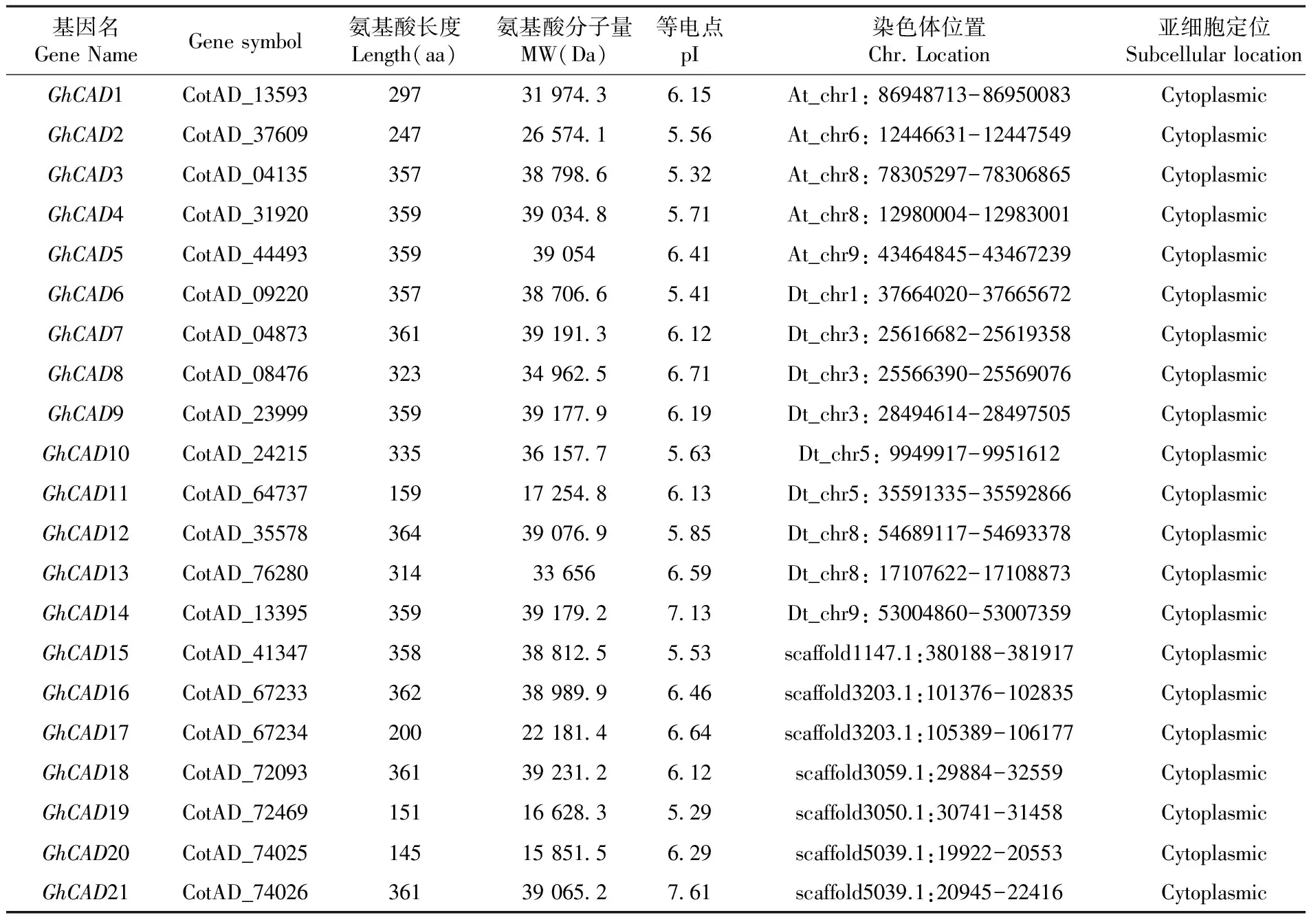
陆地棉的基因组数据和蛋白质数据均从cotton genome project(http://cgp.genomics.org.cn/page/species/download.jspategory=hirsutum) 处下载,利用本地blastp的方法以拟南芥的CAD蛋白质序列为探针,预测陆地棉的CAD基因家族成员,E值设定 1.2 方 法 1.2.1CAD基因的系统进化分析 将棉花、拟南芥、高粱、水稻、杨树的CAD蛋白质序列利用Cluster X19进行比对。随后将比对结果利用MEGA6.0软件根据比对结果构建系统进化树,构建系统树的方法采用邻接法,Bootstrap 重复次数 (Replications)设置为1 000[16],然后将生成的无根树提交到ITOL(http://itol.embl.de/) 形成环形进化树。 1.2.2CAD基因染色体定位和复制共线性分析 利用本地BLASTN从陆地棉基因组数据库中获取各CAD基因的染色体定位信息,通过MapDraw 2.2 软件作图[17]。随后根据文献[18,19]研究方法检查陆地棉CAD基因的复制情况,根据蛋白序列比对对结果识别同源基因对,经过比对后短序列如果能覆盖超过长序列70%的区域,认为这对同源基因为重复基因(duplication genes)。利用DnaSp软件计算重复基因对的ka(非同义突变率)、ks(同义突变率)值,确定其在进化过程中所受何种选择。 1.2.3CAD基因结构分析 将GhCAD基因的编码序列和对应的全基因组序列呈送到(http://gsds.cbi.pku.edu.cn/)[20]获得各基因的内含子、外显子个数和排布情况。 1.2.4CAD基因在棉纤维不同发育时期的表达分析 为了获得GhCAD基因在棉纤维发育过程中的表达情况,利用陆地棉标准系 TM-1 纤维的深度测序数据来分析CAD基因在纤维中的表达情况。数据下载与SRA 数据库(http://www.ncbi.nlm.nih.gov/sra/)。登录号为:5 dpa(SRX797917), 10 dpa(SRX797918),20 dpa(SRX797919),25 dpa(SRX797920)。随后利用Tophat将转录组数据mapping到GhCAD基因上,然后利用cufflinks估计CAD基因的表达水平[21]。 1.2.5 棉花CAD基因和拟南芥CAD基因的同源性分析 利用进化分析和序列比对的方法,找出GhCAD3和GhCAD6在拟南芥中的同源基因。并利用ClustlX2对三个基因进行同源性分析。 1.2.6 拟南芥纯和突变体的筛选和鉴定 拟南芥(Arabidopsisthaliana)野生型(Columbia-0型)和T-DNA插入突变系(Salk_019355)T1代种子由美国俄亥俄州立大学ABRC (Arabidopsis Biological Resource Center)提供。使用三引物法筛选 T-DNA 插入纯合突变体(以下简称纯合突变体)基因型的鉴定需要 3 条引物(LBb1+LP+RP)[22],PCR 所用引物的设计参照 SIAGnAL 的相关资料(http://signal.salk.edu/isectprimers.html),3条引物序列为LP:5' TCACCGCAATCAAATAAAACC3';RP:5' AGGTCAA GAATCAACAGCTGG 3';LBb1:5' GCGTGGA CCGCTTGCTGCAACT 3'。 将购买的突变体cad5 种子播种于盛有蛭石的小钵内,待植株生长 30 d 后,随机选取 10 株,以 Col 野生型拟南芥为对照,分别提取全基因组 DNA,分别采用LP/RP 和 BP/RP 引物组合,以全基因组 DNA 为模板进行 PCR 扩增, 根据电泳结果判断该植株是否为纯合突变体。LP/RP 和 BP/RP 预期 PCR 产物大小分别为 1 287 bp 和 1 272 bp。 1.2.7 野生型和突变体AtCAD5基因转录水平比较 为了获得野生型和突变体植株AtCAD5转录情况信息,取适当生长时期,已初步鉴定的纯合突变体和 Col 野生型拟南芥的叶片组织作材料,采用 Trizol 法提取适当组织的总 RNA,反转录为cDNA,根据 TAIR 网站(http://www.arabidopsis.org)上公布的拟南芥AtCAD5基因 cDNA 序列, 采用Primer Premier 5 引物设计软件设计了引物,引物采用Primer Premier 5 引物件设计,序列为:AtCAD5-F: 5‘CAACGGTAAACTTGTTCTACTCGGT3’,AtCAD5-R: 5‘AGATCGAGTAGCAGCCAATGTATTA33’,AtUBQ5-F:5‘GGTGCTAAGAAGAGGAAGAAT3’,AtUBQ5-R:5‘CTCCTTCTTTCTGGTAAACGT3’。进行RT-PCR和Quantitative RT-PCR检测。 荧光定量PCR使用仪器为BIO-RAD CFX Connect 荧光定量系统。使用试剂Power SYBR Green PCR Master Mix (Applied Biosystems Cat:4367659)。 1.2.8 野生型和突变体植株根毛表型观察 将生长有拟南芥野生型和突变体幼苗的培养皿置于体式镜(MZFLlll,LeicaMicrosystem,wetslar,Ge~ay)载物台上, 对距根尖第2和第3 mm处根上的根毛进行拍照,每个处理重复三次, 每个重复15棵苗,利用软件MoticIinagesplus2.0分析根毛长度[23]。 2.1 陆地棉基因组中CAD基因的鉴定 利用已知的拟南芥9个CAD基因作为模板,运用本地BlastP, 设E值 2.2CAD基因的系统进化 下载了已经报道了的拟南芥、水稻、高粱以及杨树的CAD基因的氨基酸序列同棉花的CAD蛋白进行系统进化分析。系统发育树显示植物CAD基因家族分成了7个亚组,棉花CAD基因分别属于其中的五个亚组。图1 图1 5个物种CAD基因家族的系统进化 基因名GeneNameGenesymbol氨基酸长度Length(aa)氨基酸分子量MW(Da)等电点pI染色体位置Chr.Location亚细胞定位SubcellularlocationGhCAD1CotAD_13593297319743615At_chr1:86948713-86950083CytoplasmicGhCAD2CotAD_37609247265741556At_chr6:12446631-12447549CytoplasmicGhCAD3CotAD_04135357387986532At_chr8:78305297-78306865CytoplasmicGhCAD4CotAD_31920359390348571At_chr8:12980004-12983001CytoplasmicGhCAD5CotAD_4449335939054641At_chr9:43464845-43467239CytoplasmicGhCAD6CotAD_09220357387066541Dt_chr1:37664020-37665672CytoplasmicGhCAD7CotAD_04873361391913612Dt_chr3:25616682-25619358CytoplasmicGhCAD8CotAD_08476323349625671Dt_chr3:25566390-25569076CytoplasmicGhCAD9CotAD_23999359391779619Dt_chr3:28494614-28497505CytoplasmicGhCAD10CotAD_24215335361577563Dt_chr5:9949917-9951612CytoplasmicGhCAD11CotAD_64737159172548613Dt_chr5:35591335-35592866CytoplasmicGhCAD12CotAD_35578364390769585Dt_chr8:54689117-54693378CytoplasmicGhCAD13CotAD_7628031433656659Dt_chr8:17107622-17108873CytoplasmicGhCAD14CotAD_13395359391792713Dt_chr9:53004860-53007359CytoplasmicGhCAD15CotAD_41347358388125553scaffold1147.1:380188-381917CytoplasmicGhCAD16CotAD_67233362389899646scaffold3203.1:101376-102835CytoplasmicGhCAD17CotAD_67234200221814664scaffold3203.1:105389-106177CytoplasmicGhCAD18CotAD_72093361392312612scaffold3059.1:29884-32559CytoplasmicGhCAD19CotAD_72469151166283529scaffold3050.1:30741-31458CytoplasmicGhCAD20CotAD_74025145158515629scaffold5039.1:19922-20553CytoplasmicGhCAD21CotAD_74026361390652761scaffold5039.1:20945-22416Cytoplasmic 2.3CAD基因在棉花染色体上的分布和基因复制 通过本地blastn获得了CAD基因在棉花染色体上的分布情况,其中有14个CAD基因被定位在特定的染色体上,还有7个CAD基因未被定位到染色体上,研究表明,CAD基因在棉花染色体上呈现出不均匀分布,棉花的26条染色体上只有八个染色体具有CAD基因。在棉花CAD基因中共有16基因形成19对复制基因,有的基因参与多次基因复制事件,例如GhCAD3、GhCAD7、GhCAD8分别参与三次基因复制。其中确定GhCAD7和GhCAD8为串联重复基因对,还有5对复制基因确定为片段复制基因对,其余部分基因未能定位到染色体上不能确定是何种复制方式。这19对复制基因中有12对复制基因的ka/ks小于1,说明这些基因在进化过程受到净化选择,即都是功能基因且功能保守,因此进化速率较慢。而还有7对复制基因ka/ks大于1,说明在进化过程中受到积极选择,即可能是功能冗余基因,因此进化速度较快[24]。表2,图2 表2 重复基因对的ka/ks 图2 CAD基因在染色体上的分布和基因复制 2.4CAD基因结构 对GhCAD基因的外显子和内含子排布进行分析,发现复制基因对之间拥有相似的基因结构,这也说明了在进化过程中是由同一祖先基因扩增而来。CAD基因有三种extron-intron类型,三种类型分别有5、5、和6个外显子组成,其中类型I和II的区别在于3和4号外显子长度不同,3号外显子长度长于4号外显子的属于类型I[25, 26]。而棉花CAD基因除具有这三种类型的基因结构外还具有3个外显子组成的基因结构类型。这说明与其他物种相比,棉花CAD基因有更为多样的基因结构类型。图3 图3 CAD基因家族的系统进化和基因结构 2.5CAD基因在陆地棉纤维不同发育时期的表达 对陆地棉标准系 TM-1 中CAD家族基因进行表达分析,深度测序数据选择棉纤维发育的 4 个时期(5、10、20及 25 dpa)。研究表明,在21个CAD基因中共有14个基因在纤维中检测到表达。其中GhCAD4和GhCAD9在5和10 dpa两个发育时期有很高的表达量,GhCAD3和GhCAD6在20和25 dpa,此外GhCAD5,GhCAD8和GhCAD12也在棉花纤维发育中个各个时期或某个时期有较高的表达。图4 2.6GhCAD3和GhCAD6与拟南芥CAD基因的同源性 根据多序列比对分析,得到GhCAD3和GhCAD6在拟南芥中的共同同源基因是AtCAD5,多序列比对分析显示三个基因之间有很高的序列相似性。而AtCAD5已经明确为可以参与木质素的合成[27],为将AtCAD5基因作为研究对象提供了理论支持。图5 图4 CAD基因家族在棉花纤维不同发育时期表达 2.7 拟南芥cad5纯合突变体的筛选 利用三引物法筛选拟南芥cad5纯合突变体, PCR鉴定结果显示野生型植株中只有利用LP+RP引物组合可以获得1 287 bp左右的PCR产物条带,而在纯合突变体中只有利用LB1+RP引物组合可以获得1 272 bp的PCR产物条带(图6A)。然后再分别以突变体和野生型DNA为模板,利用AtCAD5特异性引物进行PCR检测,结果表明在野生型中检测到AtCAD5基因而在突变体中未检测到AtCAD5基因(图6B)。这些结果说明获得了T-DNA插入的纯合突变体植株。图6 图5 AtCAD5、GhCAD3和GhCAD6的氨基酸序列比对 2.8 拟南芥cad5突变体和野生型植株AtCAD5转录水平比较 通过RT-PCR和Quantitative RT-PCR检测AtCAD5在突变体和野生型植株中转录水平。研究表明,野生型中AtCAD5转录水平明显高于突变体。突变体中AtCAD5转录水平明显下降。图7 图6 拟南芥Atcad5纯合突变体的PCR鉴定 A:RT-PCR结果比较 B: Quantitative RT-PCR结果比较 2.9 拟南芥cad5突变体的筛选和根毛表型观察 经过PCR筛选的得到拟南芥cad5基因的纯合突变体,研究表明,从5 d开始突变体植株的根毛长度逐渐长于野生型植株的根毛长度,而后随着生长天数的增加时两者的根毛长度差异更加明显。图8 A: cad5突变体和野生型植株不同天数根毛表型观察。 B: cad5突变体和野生型植株不同天数根毛长度统计。 3.1CAD基因家族鉴定和系统进化 在陆地棉全基因组水平上对CAD基因家族进行了坚定和功能分析。陆地棉中共鉴定到21个CAD基因,是拟南芥该家族成员2倍多,这也说明陆地棉染色体相对于拟南芥染色体经历了多次全基因组复制事件。利用wolf和targetP对CAD蛋白进行了亚细胞定位预测,发现所有的CAD基因均定位于细胞质当中,这可能和CAD基因的功能有一定的关系。将棉花、高粱、水稻、拟南芥和杨树的CAD基因进行系统进化分析,CAD基因分为了7个亚组且功能相近的基因在同一亚组中聚集,棉花CAD基因分别属于其中的五个亚组。其中GhCAD1、GhCAD3、GhCAD6、GhCAD10和GhCAD15同属于GroupII,这类CAD基因族中的AtCAD4、AtCAD5、AtCAD5、SbCAD2和OsCAD2的基因特性已经明确,参与了木质素的合成过程[24]。拟南芥AtCAD4和AtCAD5具有很高的同源性,当对AtCAD4和AtCAD5进行双突变后,木质素的含量大量减少[28],棉花中GhCAD3和GhCAD6也具有很高的同源性且在棉纤维20 和25 dpa有很高表达量,而又与AtCAD4和AtCAD5在同一亚组中,这说明GhCAD3和GhCAD6在棉花的木质素合成中发挥作用并且参与棉纤维的发育。GhCAD5、GhCAD14和AtCAD1同属于一个亚组,AtCAD1已经被报道在拟南芥AtCAD4和AtCAD5双突变体中,对木质素合成过程中起到功能补偿作用[25],这说明GhCAD5和GhCAD14也有相似的功能特性。基因家族中属于同一亚组的基因之间通常具有相同的功能特点,通过与其他物种的CAD基因进行系统的进化分析有助于了解棉花CAD基因功能,为以后研究棉花CAD基因的功能提供参考。在这几种植物来说,同属于单子叶植物纲的高粱和水稻与其他三种植物相比有更近的亲缘关系,可以看到亲缘关系较近的植物间,CAD基因并没有显示出较近的进化关系,这说明CAD基因家族在不同植物中的分布是不保守的。对GhCAD基因家族成员进行基因结构分析发现亲缘关系相近的基因拥有相似的基因结构,这些序列相似度极高的基因与基因组进化过程中的串联重复和片段复制有关,这些复制在基因家族成员扩增中起到重要作用。现代陆地棉由大约150万年前的两个祖先种二倍体棉花木本棉(AA)、雷蒙德氏棉(DD)杂交而成[5],研究陆地棉CAD基因的多次复制现象有助于了解棉花多倍化的进程。陆地棉基因组经历了全基因组重复、染体重排和串联重复等复杂的进化过程。研究结果表明,染色体大片段复制和串联重复在CAD基因家族的扩增中起到了重要的作用。在陆地棉中共有15个基因形成19个基因复制对,其中确定GhCAD7和GhCAD8为串联重复基因对,还有5对复制基因对确定为片段复制,而GhCAD3和GhCAD6是一对复制基因且受到净化选择,因此是保守的功能基因,这为将这两个基因作为后续的研究对象提供了依据。此外从棉花纤维表达数据来看,由同一祖先基因扩增出来的基因表达模式并不相同,这说明在进化过程中表达特性发生了分化。 近年来,对植物CAD基因家族的研究逐渐增多,已经在多种植物中鉴定了CAD基因的数量和中累,其中拟南芥中12个,水稻中12个,高粱中14个,葡萄中18个,苜蓿中17个,杨树中14个[2]。并且已经确定拟南芥CAD基因家族 中有6个可以催化5种肉桂醛生成肉桂醇,另外3个只有在底物浓度很高时才表现出活性,催化能力很低。拟南芥AtCAD4和AtCAD5具有很高的同源性,当对AtCAD4和AtCAD5进行双突变后,木质素的含量大量减少[28],而AtCAD1对其缺失的功能有一定的补偿作用[29],说明植物体内的CAD基因家族是协同起作用的。 3.2CAD基因家族表达 Han等在研究棉纤维细胞的伸长和次生壁时发现,木质素和木质素类似酚类化合物能够显著的影响棉纤维的质量性状,并且在棉纤维发育后期CAD6的表达量显著提高,该CAD6与研究提到的GhCAD3同源性最高[30]。木质素是苯丙烷代谢途径的一种产物,Hovav等[31]在研究棉花纤维发育相关基因的表达时发现,苯丙烷代谢途径相关基因在参与棉纤维细胞的伸长进程。此外Li等在研究棉花纤维发育相关的QTL时发现,CAD6基因定位在与棉花发育相关的的QTL位点上。由此推测CAD基因家族成员参与到棉花纤维发育的进程当中[32]。 从纤维表达数据发现多个CAD基因在棉花纤维中有较高的表达,其中GhCAD4和GhCAD9在5和10 dpa有很高的表达,GhCAD3和GhCAD6在20和25 dpa中有很高的表达,此外GhCAD5,GhCAD8和GhCAD12也在棉花纤维发育中各个时期或某个时期有较高的表达。这些结果表明CAD基因家族参与了棉花纤维发育进程。木质素的积累通常发生在细胞发育的次生壁时期[7],GhCAD3和GhCAD6在棉纤维发育次生壁增厚期有很高的表达,推断GhCAD3和GhCAD6可能在棉纤维细胞次生壁增厚期起到重要的作用,观察对比了GhCAD3和GhCAD6的同源基因AtCAD5突变体和野生型植株根毛长度的差异,发现突变体相较于野生型有更长的根毛,而AtCAD5已经确定可以参与木质素的合成,这些结果说明CAD基因参与了棉纤维的发育进程,并可能对其发育进程有一定的调控作用。 在陆地棉基因组中共鉴定到21个CAD基因,串联重复和片段重复是CAD基因家族主要的扩增方式,进化分析显示CAD基因家族可以分为7个亚族,且功能相近的基因在同一亚族。深度测序数据分析显示,大多数CAD基因均在棉纤维不同发育时期表达,说明CAD基因家族参与了棉纤维的发育,其中GhCAD4和GhCAD9在5和10 dpa两个发育时期有很高的表达,说明参与了棉纤维细胞的起始和伸长。GhCAD3和GhCAD6是保守的功能基因,也是在棉纤维中表达量最高的两个基因,且这两个基因都是20和25 dpa表达量显著升高,因此这两个基因应该在棉纤微次生壁增后期发挥重要作用。观察对比了GhCAD3和GhCAD6的同源基因AtCAD5突变体和野生型植株根毛长度的差异,发现突变体相较于野生型有更长的根毛,而AtCAD5已经确定可以参与木质素的合成,CAD基因参与了棉纤维的发育进程,并可能对其发育进程有一定的调控作用,为利用CAD基因改良棉纤维品质奠定了基础。 References) [1]Sunilkumar, G., Campbell, L. M., Puckhaber, L., Stipanovic, R. D., & Rathore, K. S. (2006). From the cover: engineering cottonseed for use in human nutrition by tissue-specific reduction of toxic gossypol.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 103(48):18,054-18,059. [2] Ruan, Y. L., Llewellyn, D. J., & Furbank, R. T. (2003). Suppression of sucrose synthase gene expression represses cotton fiber cell initiation, elongation, and seed development.PlantCell, 15(4):952-64. [3] Qin, Y. M., & Zhu, Y. X. (2011). How cotton fibers elongate: a tale of linear cell-growth mode.CurrentOpinioninPlantBiology, 14(1):106-111. [4] Hu, G., Jin, K., Yoo, M. J., Grupp, K., Chen, S., & Wendel, J. F. (2013). Proteomic profiling of developing cotton fibers from wild and domesticated gossypium barbadense.InteractiveCardiovascular&ThoracicSurgery, 200(2):570-582. [5] Li, F., Fan, G., Lu, C., Xiao, G., Zou, C., & Kohel, R. J., et al. (2015). Genome sequence of cultivated upland cotton (Gossypiumhirsutumtm-1) provides insights into genome evolution.NatureBiotechnology, 33(5):524-530. [6] Reiter, W. D., & Vanzin, G. F. (2001). Molecular genetics of nucleotide sugar interconversion pathways in plants.PlantMolecularBiology, 47(1-2):95-113. [7] Lerouxel, O., Cavalier, D. M., Liepman, A. H., & Keegstra, K. (2006). Biosynthesis of plant cell wall polysaccharides - a complex process.CurrentOpinioninPlantBiology, 9(6):621-630. [8] Ling, F., Shi, W. J., Hu, W. R., Hao, X. Y., Wang, D. M., & Hui, Y., et al. (2009). Molecular and biochemical evidence for phenylpropanoid synthesis and presence of wall-linked phenolics in cotton fibers.JournalofIntegrativePlantBiology, 51(7):626-637. [9] Mansell, R. L., Gross, G. G., Stckigt, J., Franke, H., & Zenk, M. H. (1974). Purification and properties of cinnamyl alcohol dehydrogenase from higher plants involved in lignin biosynthesis.Phytochemistry, 13(13):2,427-2,435. [11] Grosse, W., & Klapheck, S. (2010). Effects of 1-mcp and ethylene on expression of three cad genes and lignification in stems of harvested tsai tai (brassicachinensis).FoodChemistry, 123(1):32-40. [12] C, Halpin, & W, Schuch. (1992). Identification and characterization of cdna clones encoding cinnamyl alcohol dehydrogenase from tobacco.PlantMolecularBiology, 19(5):793-801. [13] Gasteiger, E., Hoogland, C., Gattiker, A., Duvaud, S., Wilkins, M. R., & Appel, R. D., et al. (2005). Protein identification and analysis tools on the expasy server.ProteomicsProtocolsHandbook, 112(112):571-607. [14] Emanuelsson, O., Nielsen, H., Brunak, S., & Heijne, G. V. (2000). Predicting subcellular localization of proteins based on their n-terminal amino acid sequence.JournalofMolecularBiology,300(4):1,005-1,016. [15] Horton P; Park KJ; Obayashi T; Fujita N; Harada H; Adams-Collier CJ; Nakai K. (2007). Wolf psort: protein localization predictor.NucleicAcidsResearch, 35(Web Server issue):585-587. [16] Ma, & Jun. (2015). Genome-wide identification and expression analysis of TCP transcription factors in cotton. [17] Liu, R. H., MENG Jin Ling ral University, Wuhan, & Chi. (2003). Mapdraw:a microsoft excel macro for drawing genetic linkage maps based on given genetic linkage data.Hereditas, 25(3):317-321. [18] Yang, S., Zhang, X., Yue, J. X., Tian, D., & Chen, J. Q. (2008). Recent duplications dominate nbs-encoding gene expansion in two woody species.MolecularGenetics&GenomicsMgg, 280(3):187-198. [19] Gu Z, Cavalcanti A, Chen FC, Bouman P, & Li WH. (2002). Extent of gene duplication in the genomes of drosophila, nematode, and yeast.MolecularBiology&Evolution, 19(3):256-262. [20] Guo, A. Y., Zhu, Q. H., & Chen, X. (2007). Gsds: a gene structure display server.Hereditas, 29(8):1,023-1,026. [21] Trapnell, C., Roberts, A., Goff, L., Pertea, G., Kim, D., & Kelley, D. R., et al. (2012). Differential gene and transcript expression analysis of rna-seq experiments with top hat and cufflinks.Psychopharmacology, 7(3):562-578. [22] 李敏, 杨双, 阮燕晔, 等. 拟南芥 T-DNA 插入突变体 atsuc3 的 PCR 鉴定[J]. 植物生理学通讯,2006,42(1):91-94. LI Min, YANG Shuang, Ruan Yan-hua, et al. (2006). Identification of ATSUC3 with T-DNA Insertion by PCR [J].PlantPhysiologyCommunications, 42(1):91-94. (in Chinese) [23] Zhu, C., Gan, L., Shen, Z., & Kai, X. (2006). Interactions between jasmonates and ethylene in the regulation of root hair development in Arabidopsis.JournalofExperimentalBotany, 57(6):1,299-1,308. [24] Morant, M., Hehn, A., & Werck-Reichhart, D. (2002). Conservation and diversity of gene families explored using the codehop strategy in higher plants.BmcPlantBiology, 2(1):1-12. [25] Barakat, A., Bagniewska-Zadworna, A., Choi, A., Plakkat, U., Diloreto, D. S., & Yellanki, P., et al. (2009). The cinnamyl alcohol dehydrogenase gene family in populus: phylogeny, organization, and expression.BMCPlantBiology, 9(1):26. [26] 张鲁斌, 谷会, 弓德强, 等. 植物肉桂醇脱氢酶及其基因研究进展[J]. 西北植物学报, 2011, 31(1):204-211. ZHANG Lu-bin, GU Hui, GONG De-qiang, et al. (2011). Research Progress of Cinnamyl Alcohol Dehydrogenase and Its Gene [J].ActaBotanicaBoreali-OccidentaliaSinica, 31(1):204-211. (in Chinese) [27] Ma, Q. H. (2010). Functional analysis of a cinnamyl alcohol dehydrogenase involved in lignin biosynthesis in wheat.JournalofExperimentalBotany, 61(10):2,735-2,744. [28] Sibout, R., & Séguin. (2005). Cinnamyl alcohol dehydrogenase-c and -d are the primary genes involved in lignin biosynthesis in the floral stem of Arabidopsis.PlantCell, 17(7):2,059-2,076. [30] Han, L. B., Li, Y. B., Wang, H. Y., Wu, X. M., Li, C. L., & Luo, M., et al. (2013). The dual functions of wlim1a in cell elongation and secondary wall formation in developing cotton fibers.PlantCell, 25(11):4,421-4,438. [31] Ran, H., Udall, J. A., Hovav, E., Rapp, R., Flagel, L., & Wendel, J. F. (2008). A majority of cotton genes are expressed in single-celled fiber.Planta, 227(2):319-329. [32] Li, X., Yuan, D., Zhang, J., Lin, Z., & Zhang, X. (2012). Genetic mapping and characteristics of genes specifically or preferentially expressed during fiber development in cotton.PlosOne, 8(1):65-65. Fund project:Supported by High-tech research and development program of Xinjiang "Research on the technology innovation of cotton fiber quality molecular Improvement (201111116) and NSFC "The function and regulation mechanism of cotton GhCAD6 gene in the development of cotton fiber" (31460386) Genomi-wide Investigation and Expression Analysis of CAD Gene Family inGossypiumhirsutum ZHANG Jin-bo1,2,LI Bo2,YANG Yang2,HU Wen-ran2,CHEN Fan-yuan1.2,XIE Li-xia2,FAN Ling2 (1. College of Life Sciences, Xinjiang Normal University, Urumqi 830054, China;2 Research InstituteofNuclearandBiotechnologies,XinjiangAcademyofAgriculturalSciences,Urumqi830091,China) 【Objective】 This study aims to conduct for the first time a genome-wide analysis ofCADgene family in cotton.【Method】A total of 21CADencoding genes were identified inGossypiumhirsutum. The phylogenic evolution, chromosome distribution, gene duplication and gene structure ofCADgene family went through phylogenetic analysis.【Result】The result revealed thatCADgene family could be clustered into 7 major subgroups and the genes which had similar functions were in the same subgroup. Both tandem duplications and segmental duplications were found to attribute to the expansion ofCADgene family.CADgene family had expression at different stages of development fiber. TheAtcad5 mutant had a longer root hair than that of the wild type.【Conclusion】CADgenes maybe are involved in developing fiber and play a role in regulating the development of fiber. cotton fiber;CADgene family; phylogenic analysis; expression pattern; cad5 mutant; root hair 10.6048/j.issn.1001-4330.2016.07.001 2016-03-05 新疆维吾尔自治区高技术项目“棉花纤维品质分子改良技术创新研究”(201111116);国家自然科学基金项目“棉花GhCAD6基因在棉花纤维发育中的功能及调控机制”(31460386) 张经博(1991- ),男,新疆人,硕士研究生,研究方向为作物分子生物学,(E-mail)952880259@qq.com 范玲(1958- ),女,新疆人,研究员,博士生导师,研究方向为棉花纤维品质分子改良,(E-mail)fanling@xaas.ac.cn S562;S188 A 1001-4330(2016)07-1177-112 结果与分析









3 讨 论
4 结 论
