西瓜抗枯萎病相关基因ClMYB转录因子的克隆及表达分析
2016-12-01韩金桓王丽霞高洪波吕桂云
韩金桓,王丽霞,高洪波, 吕桂云
(河北农业大学园艺学院,河北保定071001)
西瓜抗枯萎病相关基因ClMYB转录因子的克隆及表达分析
韩金桓,王丽霞,高洪波, 吕桂云
(河北农业大学园艺学院,河北保定071001)
【目的】克隆西瓜抗枯萎病相关基因ClMYB转录因子,进行生物信息学及表达模式分析,为进一步解析ClMYB在西瓜抗枯萎病机制中的作用提供理论依据。【方法】根据西瓜与枯萎病菌不亲和互作的抑制差减文库和Microarray数据分析,获得与西瓜抗枯萎病相关的基因ClMYB,采用RT-PCR技术分离克隆ClMYB cDNA全长序列;应用生物信息学方法分析该基因的保守结构域及序列特征;使用MEGA5.0对ClMYB蛋白序列及其同源序列进行多序列比对,并构建同源物种间系统进化树;采用GFP标记的方法进行亚细胞定位,分析编码蛋白表达位置;将该基因片段通过Nde I和Xba I双酶切连接至原核表达载体pCzn1,重组质粒转化至大肠杆菌Arctic Express,经终浓度为0.5 mmol·L-1IPTG诱导4 h,用SDS-PAGE分析融合蛋白的表达;利用实时荧光定量PCR方法检测目的基因在西瓜与枯萎病菌互作中及在茉莉酸(jasmonate,JA)诱导下的表达情况。【结果】利用RT-PCR方法从西瓜野生材料PI296341-FR根系组织中克隆该基因片段(GenBank:KT751229),序列比对及生物信息学分析表明,其基因编码的氨基酸序列具有MYB转录因子R2R3型的典型特征,其N端具有R2、R3两个MYB结构域,C端高度变异。多序列比对及进化树分析表明,该基因编码的蛋白与甜瓜MYB(GenBank:XM_008440304)和黄瓜MYB(GenBank:XM_011652633)同源性最高,其编码的氨基酸一致性达到86%,这与它们同属葫芦科植物有关;亚细胞定位显示ClMYB定位于细胞核,为典型的转录因子;成功构建了该基因的原核表达载体pCzn1-ClMYB,转化至大肠杆菌得到36 kD左右蛋白;ClMYB受枯萎病菌诱导,在高抗枯萎病菌材料PI296341-FR中,相对感病品种表达量高峰出现的早,且表达量高。50 μmol·L-1的MeJA处理可以显著提高感病材料Black diamond对枯萎病的抗病水平,同时诱导ClMYB表达,与抗病材料PI296341-FR相比表达趋势一致,但表达量更高。【结论】ClMYB为典型的R2R3-MYB转录因子,亚细胞定位于细胞核中;基因的原核表达得到36 kD的融合蛋白,在西瓜中的表达受枯萎病菌和茉莉酸诱导,推测ClMYB可能参与JA介导的西瓜抗枯萎病防卫反应的信号通路,在西瓜抗病中起一定的作用。
西瓜;枯萎病;ClMYB转录因子;基因克隆;表达分析
0 引言
【研究意义】西瓜(Citrullus lanatus)是世界第五大水果,中国西瓜的栽培面积、总产量与人均消费量均居世界首位,在高效农业中占有重要地位。西瓜枯萎病是由半知菌亚门镰孢属尖镰孢(Fusarium oxysporum f. sp. niveum)寄生引起的一种真菌土传病害,是世界范围内导致西瓜产量和品质降低的最严重障碍,病田一般减产20%—30%、严重田块可达50%—60%,甚至绝产[1]。野生西瓜材料PI296341-FR(Citrulls lanatus var. citrodes)是国际上公认的抗枯萎病菌3个生理小种的抗源[2]。在前期研究中,通过抑制差减文库和Microarray技术从PI296341-FR筛选出参与西瓜抗枯萎病响应的转录因子ClMYB和茉莉酸代谢途径[3-4]。明确ClMYB、茉莉酸(JA)与西瓜抗枯萎病的关系及其调控抗性反应途径,对解析该基因调控西瓜抗病性的机理和西瓜枯萎病的可持续控制具有重要的理论和实际意义。【前人研究进展】MYB在植株中的过表达能够显著提高植物的抗病能力,并具有一定的广谱效应。YANG等[5]从烟草中分离到一个TMV诱导基因NtMYB1,外源水杨酸(SA)可迅速诱导该基因表达,并且先于PR21的诱导,该基因编码蛋白可与PR21基因启动子中的MYB结合位点特异性结合,从而调节PR21的表达。在拟南芥和烟草中过量表达AtMYB30,可以看到转基因植株对不同的病原细菌表现过敏反应(HR)或类似过敏反应,并能增强对多种细菌和烟草蛙眼病菌(Cercospora nicotianae)的抗性。在反义表达AtMYB30的拟南芥株系中,能强烈抑制对不同病原细菌的抗性和HR细胞死亡反应,HR和防卫反应相关基因的表达也发生改变[6]。这些结果表明,AtMYB30是过敏性细胞死亡的正向调节子。AtMYB15与烟草(Nicotiana tabacum)的NtMYB2高度相似,它们能被创伤和调控PR基因表达的激发子诱导[7]。反义表达MYB72的株系在受到不同病原物侵染时无法形成ISR,表明MYB72作为一个新的ISR信号传导成员是建立广谱ISR所必需的[8]。偃麦草中的R2R3-MYB基因TiMYB2R-1过量表达可增强小麦和其他谷类作物对全蚀病的抗性[9],另一种R2R3-MYB基因TaPIMP1则表现出对小麦青枯病菌(Ralstonia solanacearum)病原体的抗性[10]。拟南芥MYB蛋白AtMYB30与黄单胞杆菌Ⅲ型效应子XopD互作,通过翻译后修饰来调控植物抗病性[11]。从华东葡萄中克隆的VpMYBR1能够提高转基因植株白粉病的抗性[12]。【本研究切入点】MYB转录因子是植物中最大的转录因子之一,在植物生长发育及抗逆境胁迫过程中发挥着极其重要的作用,已在拟南芥[6-8]、烟草[5]、偃麦草[8,11]、玉米[13]等多个植物抗病性方面得到证实。笔者课题组在西瓜中也检测到和枯萎病相关MYB有cluster133Contig1、08081903T1O_95_G12、cluster377Contig1,利用Microarray进一步分析发现,其中08081903T1O_95_ G12 在西瓜与枯萎病菌的非亲和互作前期中显著上调表达[3-4]。李猷等[14]在西瓜与枯萎病非亲和互作筛选出相关的MYB基因CH235。但这些相关的MYB都是初步筛选出的EST序列,在西瓜抗枯萎病研究中尚未见MYB转录因子克隆的报道。【拟解决的关键问题】在08081903T1O_95_G12序列的基础上,通过克隆西瓜ClMYB转录因子基因,对其序列进行生物信息学分析、通过亚细胞定位和原核表达,同时利用qRT-PCR检测该基因在不同抗性品种及JA处理下的表达模式,为深入解析该基因调控西瓜抗枯萎病性应答反应的分子机理提供理论基础。
1 材料与方法
试验于2014—2015年在河北农业大学园艺学院进行。
1.1 材料
以高抗枯萎病菌生理小种1 的野生西瓜种质PI296341-FR,高感枯萎病菌西瓜品种Black diamond为材料;病源材料枯萎病菌生理小种1(race1)采自北京市通州区西瓜枯萎病病株,由北京市农林科学院蔬菜研究中心许勇研究员惠赠。
限制性内切酶、SYBR®Premix Ex TaqTMⅡ(Tli RNaseH Plus)、10×PCR Buffer、dNTPs、Taq酶、蛋白质Marker、T4DNA连接酶购自宝生物工程(大连)有限公司;pCzn1载体、TOP10菌株、Arctic Express菌株、IPTG、SDS购自钟鼎生物公司;RNAprep Pure Plant Kit(Polysaccharides &Polyphenolics-rich)试剂盒、FastQuant RT Kit with gDNase试剂盒、质粒小提中量试剂盒购自北京天根公司;DNA胶回收试剂盒购自生工生物工程(上海)股份有限公司。
1.2 方法
1.2.1 试材培养 枯萎病菌人工接种采用浸根接种法,待西瓜幼苗2—3片真叶时,用枯萎病菌孢子悬浮液浓度为5×106个孢子/mL浸根15 min,中间摇动2—3次,防止孢子沉降。浸根完成后,定植到营养钵中,同时设立对照,接种清水。用50 μmol·L-1浓度茉莉酸甲酯(methyl jasmonate,MeJA)分别均匀喷洒于PI296341-FR和Black diamond叶片表面,以清水喷洒的为对照。接种后在温室26—30℃条件下培养,根据发病进程统计病情指数=Σ(病级株数×代表数值)× 100/调查总数×发病最重级的代表数值。处理后 0、6、12、24、48、72 h分别采集各组西瓜根系组织液氮冻存。用RNA提取试剂盒提取西瓜总RNA,按FastQuant RT Kit with gDNase试剂盒说明进行反转录。
1.2.2 ClMYB克隆 以野生西瓜材料PI296341-FR接种西瓜枯萎病菌后0、6、12、24、48、72 h根系组织各时间点总RNA等量混合物的反转录产物为模板,根据笔者课题组前期对抑制差减文库和Microarray数据分析得到的与枯萎病相关08081903T1O-95-G12 EST序列,设计分别带Xba I和Sma I酶切位点的上下游引物T-F:5′-GCTCTAGA GCATGGTGAGAGCTCC TTGTTGTGAGA-3′(下划线表示引入的Xba I酶切位点),T-R:5′-TCCCCCGGGTCCAAATACAGGGTCAT TTGGCGACCCA-3(′下划线表示引入的Sma I酶切位点),克隆该基因。PCR体系为:10×PCR Buffer 2.5 μL,dNTPs(2.5)4 μL,Taq酶0.25 μL,上下游引物(10 μmol·L-1)各0.5 μL,cDNA 1.25 μL,ddH2O补齐至25 μL。PCR程序为:94℃变性3 min,然后94℃30 s,60℃30 s,72℃2 min共35个循环,最后72℃延伸10 min,4℃保温。经2%琼脂糖凝胶电泳,回收目标片段,克隆至pMD19-T载体,转化大肠杆菌DH5α,筛选出阳性克隆进行测序,测序正确的克隆命名为T-ClMYB,获得目的基因序列命名为ClMYB(GenBank:KT751229)。
1.2.3 ClMYB序列的生物信息学分析 利用NCBI Blast(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行同源性分析;利用ExPASy ProtParam(http://expasy.org/ tools/pi_tool.html)进行蛋白理化性质进行预测;利用SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat. pl)在线软件进行蛋白质二级结构预测;采用DNAMAN软件进行氨基酸多序列比对分析;系统进化树采用MEGA5.0软件构建。
1.2.4 ClMYB蛋白的亚细胞定位 对1.2.2获得克隆T-ClMYB提取质粒,与PYBA1332-GFP(北京市农林科学院张春秋博士惠赠)分别经Xba I和Sma I双酶切,回收目的片段并用T4 DNA连接酶过夜连接,构建ClMYB融合表达载体,转化大肠杆菌DH5α,筛选出阳性克隆并进行测序验证。
分别用70%乙醇和灭菌蒸馏水清洗金粉,再加入无水乙醇制成金粉悬浮液,再顺序加入质粒、氯化钙、亚精胺振荡。取10 μL悬浮液加到基因枪中,基因枪PDS-1000He型轰击时真空度26 mmHg,可裂膜压力1 100 psi。将轰击好后的洋葱表皮放于25℃中暗培养24 h。然后于激光共聚焦显微镜在488 nm波长下观察GFP在洋葱表皮细胞内的表达情况。
1.2.5 pCzn1-ClMYB原核表达载体质粒构建及表达根据ClMYB cDNA序列设计分别带Nde I和Xba I酶切位点的上下游引物pCzn1-F:5′-CATATG GTGAGA GCTCCTTGTTGTGAGAA-3′(下划线表示引入的Nde I酶切位点),pCzn1-R:5′-TCTAGA AAATACAGGGT CATTTGGCGACCCA-3′(下划线表示引入的Xba I酶切位点),克隆该基因,将获得的目的片段PCR产物纯化后用Nde I和Xba I双酶切,在T4 DNA连接酶的作用下将目的片段连接至以相同酶切后回收的pCzn1载体上,16℃过夜连接。将连接产物转入大肠杆菌DH5α,筛选出阳性克隆进行测序。构建成功的表达载体质粒命名为pCzn1-ClMYB。将构建成功的表达载体质粒pCzn1-ClMYB,转化至表达菌株大肠杆菌Arctic Express,经50 μg·mL-1氨苄抗性筛选,挑取转化平板上的单克隆接种至含50 μg·mL-1氨苄的LB液体培养基中,37℃振荡培养至OD600为0.6—0.8,以终浓度为0.5 mmol·L-1的IPTG在37℃条件下诱导表达4 h,以不加IPTG诱导的pCzn1-ClMYB作为对照。诱导完成后,MYB-His包涵体经过变复性的方式,重溶目标蛋白,通过Ni柱亲和纯化获得目标蛋白,进行12% SDS-PAGE分析。
1.2.6 基因表达分析 采用real-time PCR分析不同处理后基因的表达情况,以18S rRNA为内参,反应引物如表1(18S rRNA,qClMYB),按照SYBR® Premix Ex TaqTMⅡ使用说明进行。每个样品设有3次重复。反应条件为:95℃预变性30 s,95℃变性5 s, 52℃复性30 s,72℃延伸30 s,变性到延伸循环40次;END(熔解曲线):95℃ 15 s,60℃15 s,荧光检测20 min,95℃15 s。采用2-△△CT法分析结果。
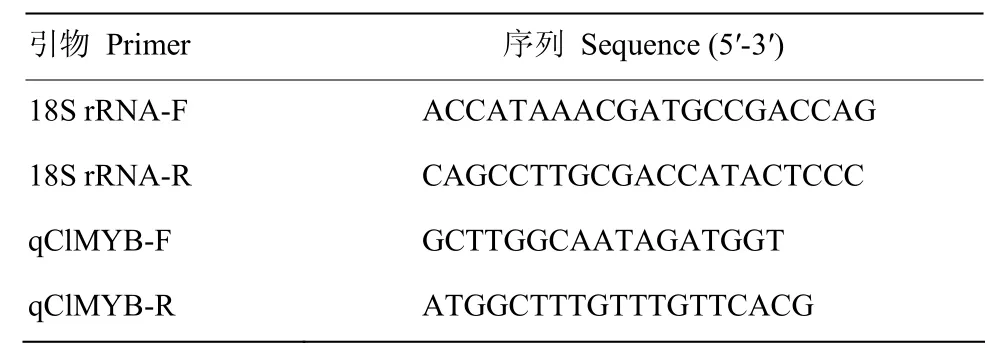
表1 ClMYB表达分析所用引物Table 1 Primers for expression analyses of ClMYB
2 结果
2.1 ClMYB DNA、cDNA序列的克隆与分析
ClMYB DNA序列全长955 bp,cDNA序列全长792 bp,开放阅读框编码263个氨基酸残基。对其DNA和cDNA序列分析表明,该基因在其起始密码子后第134、388位核苷酸处分别插入一段长74、89 bp的内含子,蛋白保守结构域预测发现,其N端第13—63、66—114位分别具有R2、R3两个MYB结构域(图1),该结构域是R2R3型MYB家族基因所特有的,推测该基因是R2R3型MYB转录因子。根据ProtParam预测显示,ClMYB蛋白的理论分子量为30.5 kD,理论等电点为6.22,分子式为C1371H2O53N373O400S10,酸性氨基酸残基总数为37,碱性氨基酸残基总数为33,脂肪系数为61.52,平均亲水性为-0.763,预测为亲水性蛋白,不稳定指数为50.96,预测该蛋白为不稳定蛋白。
根据SOPMA在线软件预测ClMYB蛋白的氨基酸序列的二级结构包括α-螺旋、β-转角、无规则卷曲、延伸链。其中构成无规则卷曲的氨基酸数最多为108个,百分比为41.06%。α-螺旋和延伸链次之,分别为34.22%和15.97%。β-转角所占比例最少,仅为8.75%(图2)。
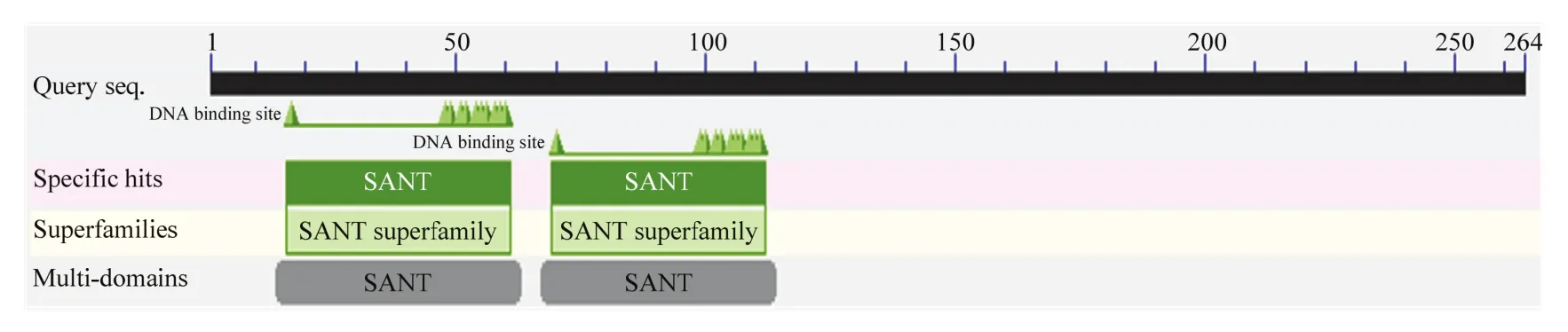
图1 ClMYB蛋白保守结构域Fig. 1 The predicted conservative domains of ClMYB protein

图2 ClMYB蛋白的二级结构预测Fig. 2 Predicted secondary structure of ClMYB protein
2.2 ClMYB编码氨基酸序列比对及系统进化树
将ClMYB的氨基酸序列与黄瓜(Cucumis sativus,XM_011652633)、甜瓜(Cucumis melo,XM_008440304)、烟草(Nicotiana tabacum,XM_009633531)、百脉根(Lotus japonicus,BT145262)、山嵛(Eutrema,XM_006405998)、醉蝶花(Tarenaya hassieriana,XM_010534986)、葡萄(Vitis vinifera,NM_001281203)、花生(Arachis hypogaea,KF208656)、甜菜(Beta vulgaris,XM_010676764)、大豆(Glycine max,XM_006573119)、油棕(Elaeis guineensis,XM_010918867)、海枣(Phoenix dactylifera,XM_008788388)12个物种中含有MYB结构域蛋白基因的氨基酸序列比对。分析显示,ClMYB与甜瓜、黄瓜等氨基酸序列相似性分别达到80%以上(图3)。利用MEGA5.0软件将ClMYB氨基酸序列与上述12个物种的MYB氨基酸序列进行聚类分析(图4),油棕和海枣被聚为一类,可能与它们同属于单子叶植物有关。ClMYB与甜瓜和黄瓜MYB基因被聚在一类,说明这可能与它们同属于葫芦科植物有关。
2.3 ClMYB的亚细胞定位
利用基因枪将构建好的重组质粒PYBA1332-GFP/ClMYB转入洋葱表皮细胞内,25℃暗培养24 h后制片,激光扫描共聚焦荧光显微镜观察。结果显示,在转化空载体PYBA1332-GFP的洋葱表皮绿色荧光分布整个细胞包括细胞膜、细胞质以及细胞核中(图5-A),说明没有目的基因空载是可以在细胞的细胞膜、细胞质以及细胞核中表达。而转化PYBA1332-GFP/ClMYB载体的洋葱表皮细胞内只有细胞核上有绿色荧光信号(图5-B),表明ClMYB蛋白定位于细胞核,为典型的转录因子蛋白。
2.4 pCzn1-ClMYB原核表达载体构建及表达
将ClMYB与原核表达载体pCzn1连接,得到融合表达载体pCzn1-ClMYB,经Nde I+ Xba I双酶切鉴定,得到798 bp 的目的片段(图6),测序结果表明无碱基突变,重组质粒构建成功。将重组质粒pCzn1-MYB转化大肠杆菌Arctic Express,经终浓度为0.5 mmol·L-1的IPTG在37℃条件下诱导表达4 h后进行SDS-PAGE电泳分析。结果表明,重组质粒pCzn1-ClMYB诱导产物出现一条特异蛋白带,未诱导表达及诱导上清液没有融合蛋白的表达(图7),则目标蛋白主要存在于包涵体中。融合蛋白纯化后蛋白理论分子量为36 kD左右(含His-tag)(图8),与预测的ClMYB蛋白分子量30.5 kD相近。
2.5 ClMYB在不同抗性材料上接种枯萎病菌后的表达分析
不同抗性材料接种枯萎病菌后ClMYB表达量有显著差异,根部表达量在高抗枯萎病菌材料PI296341-FR中接菌6 h后达到最大值,达到接种前原始表达量的7.2倍。高感枯萎病菌材料Black diamond中接菌24 h后达到最大值,达到接种前原始表达量的2.2倍,PI296341-FR ClMYB的根部表达量明显高于Black diamond(图9)。在接种处理后13 d后,PI296341-FR病情指数为2.67,Black diamond病情指数为87.75。
2.6 MeJA处理对ClMYB表达影响
MeJA处理Black diamond和PI296341-FR后,ClMYB对MeJA处理产生响应表达趋势一致,但表达量差异显著。Black diamond分别在接种后6和48 h出现ClMYB表达高峰,表达量分别是对照的15和11倍,而PI296341-FR在接种后6 和48 h出现表达高峰,表达量分别是对照的4和6.2倍(图10)。接种处理13 d后,PI296341-FR无MeJA处理的病情指数为2.67,MeJA处理后病情指数为1.28;而Black diamond无MeJA处理的病情指数为87.75,MeJA处理后病情指数为19.44。

图3 ClMYB氨基酸序列与其他物种序列多重比对Fig. 3 Aligments of the deduced amino acid sequence of ClMYB from other MYB proteins
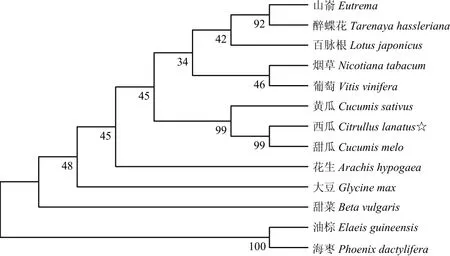
图4 ClMYB与其他MYB 蛋白的系统进化树Fig. 4 Phylogenetic tree of ClMYB and some other MYB proteins
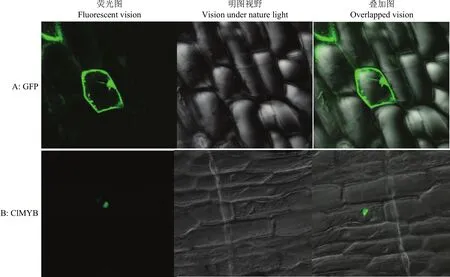
图5 ClMYB亚细胞定位分析Fig. 5 Subcellular localization of ClMYB protein in onion epidermal cells
3 讨论
R2R3亚类MYB转录因子主要参与次生代谢调节[15]、控制细胞的分化[16]、应答激素的刺激[17]及抵御外界胁迫和病原菌的侵染[18]。本研究对克隆的ClMYB序列分析发现,该基因含有两个MYB结构域,编码263个氨基酸残基的蛋白,属于典型的R2R3类型的MYB转录因子家族的成员。在拟南芥中,根据内含子在MYB 蛋白的BRH(the region between the conserved DNA-recognitionhelix)的插入位点的不同,将MYB 蛋白被分为A、B、C 3大类,其中A类MYB蛋白在BRH处没有插入内含子;B类MYB蛋白在BRH 的R3 处插入一个内含子;C类MYB蛋白占拟南芥蛋白的85%,主要参与苯丙烷类代谢途径,在BRH的R2中插入内含子[19]。许玲等[19]在大豆中分离一个C类MYB基因GmMYB111,与大豆的非生物胁迫和ABA信号转导途径有关。ClMYB在其起始密码子后第134、388位核苷酸处各插入一个内含子,属于C类MYB转录因子,结合笔者课题组前期筛选出参与西瓜与枯萎病菌非亲和互作的莽草酸-苯丙烷-木质素生物合成途径[4],ClMYB可能参与了此代谢途径,具体调控功能还有待后续研究证明。
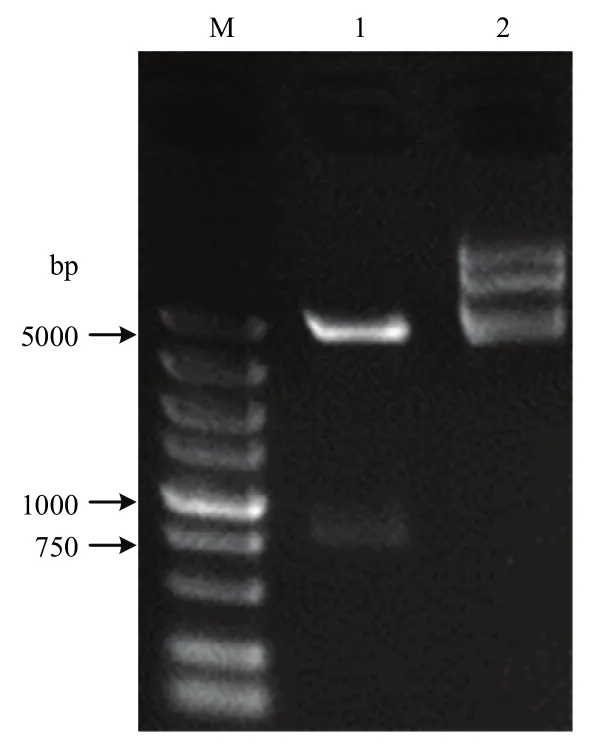
图6 重组质粒pCzn 1-ClMYB双酶切鉴定Fig. 6 Restriction enzyme analysis of recombinant plasmid pCzn 1-ClMYB
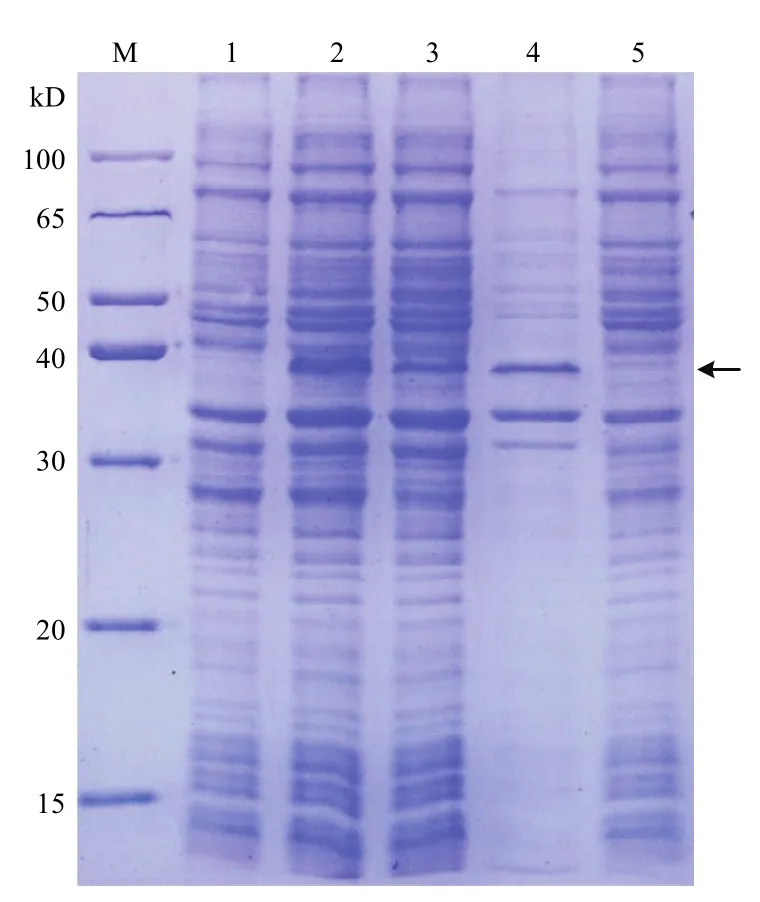
图7 ClMYB重组蛋白在大肠杆菌中表达Fig. 7 Expression of recombinant pCzn 1-ClMYB protein in E. coli Arctic Express
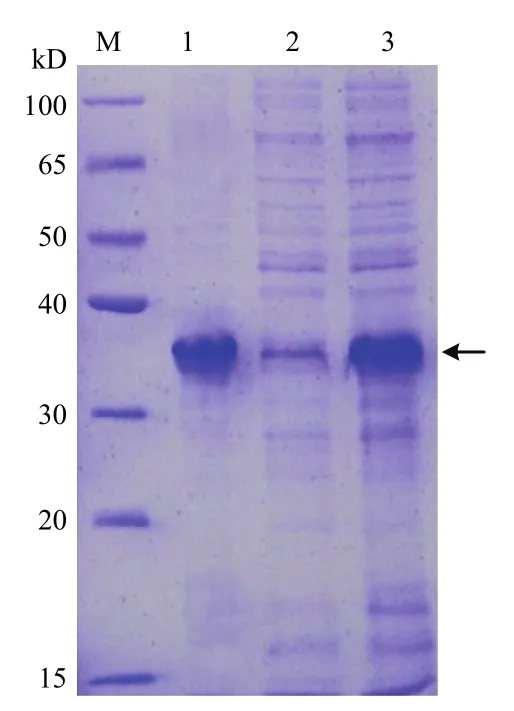
图8 ClMYB蛋白纯化SDS-PAGE分析Fig. 8 SDS-PAGE of ClMYB purification protein
SA、JA和乙烯是植物体内重要的抗病信号分子,它们在抵御生物类胁迫中具有重要作用[20]。AtMYB30是R2R3亚类MYB转录因子家族的成员,在拟南芥与病原细菌的非亲和互作中AtMYB30能被快速激活表达,而在起始细胞死亡受到影响的突变体中,该蛋白的表达被下调[21]。在细菌病原物诱导的HR中,AtMYB30表达依赖于SA积累,但与依赖于SA的防卫反应信号传导途径中的关键组分NPR1无关[22]。从中间偃麦草中克隆鉴定出R2R3MYB基因TiMYB2R-1,在小麦中异源表达,发现与全蚀病相关防御基因在转录水平上的表达均显著提高,表明超量表达TiMYB2R-1能用于提高小麦抵抗全蚀病的抗性[9]。ATR1 可引发HR细胞死亡反应从而增强植株对细菌病原物的抗性[23],ATR13 则可增强植株对卵菌、细菌和病毒的抗病能力[24]。大豆中的PsMYB1能够增强对大豆霉疫菌的抗性[25]。对拟南芥突变体中的BOS1的研究发现,BOS1诱导表达在coll突变体中被破坏,说明参与了茉莉酸抗病反应途径[26]。MYB还参与了依赖于JA的防御反应途径,在水稻幼苗中JA是抵御稻瘟病菌侵染的系统获得的重要抗病性诱导因子,从水稻中分离JAmyb,受稻瘟病和JA诱导,在敏感株系中受稻瘟病菌诱导水平更高[27]。本研究表明,ClMYB表达受与西瓜抗枯萎病和JA诱导,MeJA处理可以显著提高感病材料Black diamond对枯萎病的抗病水平,且诱导水平比在抗病材料PI296341-FR更高,推测ClMYB可能通过JA信号途径参与西瓜抗枯萎病的应答中。
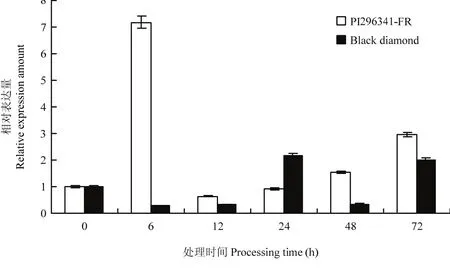
图9 枯萎病菌诱导ClMYB的表达分析Fig. 9 Expression analysis of ClMYB induced by F. oxysporum f. sp. niveum
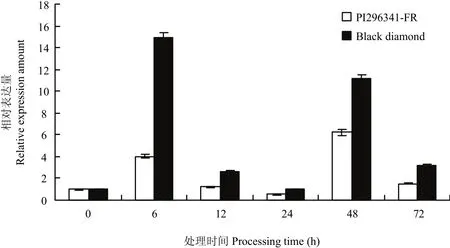
图10 ClMYB在MeJA处理后Black diamond和PI296341-FR的表达Fig. 10 Expression of ClMYB in response to MeJA treatment in Black diamond and PI296341-FR
4 结论
ClMYB编码的氨基酸序列具有MYB转录因子R2R3型的典型特征,其N端具有R2、R3 两个MYB结构域,C端高度变异。进化树分析表明,该基因编码的蛋白与甜瓜、黄瓜的MYB同源性最高,这与它们同属于葫芦科植物有关。亚细胞定位于细胞核中,基因的原核表达得到36 kD左右的融合蛋白,其表达可能与西瓜抗枯萎病和JA信号转导途径有关,推测其可能通过JA信号途径参与西瓜抗枯萎病的应答中。
[1] ZHANG Z G, ZHANG J Y, WANG Y C, ZHENG X B. Molecular detection of Fusarium oxysporum f. sp. niveum and Mycosphaerellamelonis in infected plant tissues and soil. FEMS Microbiology Letters, 2005, 249: 39-47.
[2] MARTYN R D, NETZER D. Resistance to race 0, 1, 2 of Fusarium wilt of watermelon in Citrullus sp. PI296341-FR. HortScience, 1991, 26(4): 429-432.
[3] 吕桂云, 郭绍贵, 张海英, 耿丽华, 许勇. 西瓜与枯萎病菌非亲和互作的表达序列标签分析. 中国农业科学, 2010, 43(9): 1883-1894. LÜ G Y, GUO S G, ZHANG H Y, GENG L H, XU Y. Analysis of expressed sequence tags in the incompatible interaction between watermelon and Fusarium oxysporum. Scientia Agricultura Sinica, 2010, 43(9): 1883-1894. (in Chinese)
[4] LÜ G Y, GUO S G, ZHANG H Y, GENG L H, SONG F G, FEI Z J, XU Y. Transcriptional profiling of watermelon during its incompatible interaction with Fusarium oxysporum f. sp. niveum. European Journal of Plant Pathology, 2011, 131: 585-601.
[5] YANG Y, KLESSING D F. Isolation and characterization of a tobacco mosaic virus-inducible myb oncogene homolog from tobacco. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93: 14972-14977.
[6] VAILLEAU F, DANIEL X, TRONCHET M, MONTILLET J L, TRIANTAPHYLIDES C, ROBY D. A R2R3-MYB gene, AtMYB30, acts as a positive regulator of the hypersensitive cell death program in plants in response to pathogen attack. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(15): 10179-10184.
[7] SUGIMOTO K, TAKEDA S, HIROCHIKA H. MYB-related transcription factor NtMYB2 induced by wounding and elicitors is regulator of the tobacco retrotransposon Tto1 and defense-related genes. The Plant Cell, 2000, 12: 2511-2527.
[8] VAN DER ENT S, VERHAGEN B W, VAN DOORN R, BAKKER D, VERLAAN M G, PEL J C, PROVENIERS C G, VAN LONG L C, TON J, PIETERSE C M J. MYB72 is required in early signaling steps of rhizobacteria-induced systemic resistance in Arabidopsis. Plant Physiology, 2008, 146: 1293-1304.
[9] LIU X, YANG L H, ZHOU X Y, ZHOU M P, LU Y. Transgenic wheat expressing Thinopyrum intermedium MYB transcription factor TiMYB2R-1 shows enhanced resistance to the take-all disease. Journal of Experimental Botany, 2013, 64(8): 2243-2253.
[10] LIU H X, ZHOU X Y, DONG N, LIU X, ZHANG H Y, ZHANG Z Y. Expression of a wheat MYB gene in transgenic tobacco enhances resistance to Ralstonia solanacearum, and to drought and salt stresses. Functional & Integrative Genomics, 2011, 11: 431-443.
[11] ZHANG Z Y, LIU X, WANG X D, ZHOU M P, ZHOU X Y, YE X G, WEI X. An R2R3 MYB transcription factor in wheat, TaPIMP1, mediates host resistance to Bipolaris sorokiniana and drought stresses through regulation of defense and stress-related genes. New Phytologist, 2012, 196: 1155-1170.
[12] 侯红敏, 王浩, 殷向静, 闫琴, 王跃进, 王西平. 华东葡萄抗白粉病VpMYBR1基因表达与功能分析. 中国农业科学, 2013, 46(7): 1408-1418. HOU H M, WANG H, YIN X J, YAN Q, WANG Y J, WANG X P. Expression and functional analysis of VpMYBR1 gene resistant to Uncinula necator from Vitis pseudoreticulata. Scientia Agricultura Sinica, 2013, 46(7): 1408-1418. (in Chinese)
[13] TIMMERMANS M C, HUDSON A, BECRAFT P W, NELSON T. ROUGH SHEATH2: a Myb protein that represses knox homeobox genes in maize lateral organ primordia. Science, 1999, 284: 151-153.
[14] 李猷, 王日升, 董登峰, 张曼, 刘文君, 陈振东, 董文斌. 西瓜与枯萎病菌非亲和互作相关基因的分离及表达分析. 植物病理学报, 2015, 45(1): 22-32. LI Y, WANG R S, DONG D F, ZHANG M, LIU W J, CHEN Z D, DONG W B. Isolation and expression analysis of resistance related genes in incompatible interaction between watermelon and Fusarium oxysporum f. sp. niveum. Acta Phytopathologica Sinica, 2015, 45(1): 22-32. (in Chinese)
[15] 邢文, 金晓玲. 调控植物类黄酮生物合成的MYB转录因子研究进展. 分子植物育种, 2015, 13(3): 689-696. XING W, JIN X L. Recent advances of MYB transcription factors involved in the regulation of flavonoid biosynthesis. Molecular Plant Breeding, 2015, 13(3): 689-696. (in Chinese)
[16] LAU S E, SCHWARZACHER T, OTHMAN R Y, HARIKRISHNA J A. dsRNA silencing of an R2R3-MYB transcription factor affects flower cell shape in a Dendrobium hybrid. BMC Plant Biology, 2015, 15: 194.
[17] DUBOS C, STRACKE R, GROTEWOLD E, WEISSHAAR B, MARTIN C, LEPINIEC L. MYB transcription factors in Arabidopsis. Trends in Plant Science, 2010, 15: 573-581.
[18] GAO F, ZHAO H X, YAO H P, LI C L, CHEN H, WANG A H, PARK S U, WU Q. Identification, isolation and expression analysis of eight stress-related R2R3-MYB genes in tartary buckwheat (Fagopyrum tataricum). Plant Cell Reports, 2016, 35: 1385-1396.
[19] 许玲, 卫培培, 张大勇, 徐照龙, 何晓兰, 黄益洪, 马鸿翔, 邵宏波.大豆转录因子基因GmMYB111的克隆及功能分析. 中国农业科学, 2015, 48(15): 3079-3089. XU L, WEI P P, ZHANG D Y, XU Z L, HE X L, HUANG Y H, MA H X, SHAO H B. Expression and function analysis of the transcriptionfactor Gm MYB111 in soybean. Scientia Agricultura Sinica, 2015, 48(15): 3079-3089. (in Chinese)
[20] LU J, ROBERT C A, RIEMANN M, COSME M, MÈNESAFFRANÉ L, MASSANA J, STOUT M J, LOU Y, GERSHENZON J, ERB M. Induced jasmonate signaling leads to contrasting effects on root damage and herbivore performance. Plant Physiology, 2015, 167(3): 1100-1116.
[21] DANIEL X, LACOMME C, MOREL J B, ROBY D. A novel myb oncogene homologue in Arabidopsis thaliana related to hypersensitive cell death. The Plant Journal, 1999, 20(1): 57-66.
[22] RAFFAELE S, RIVAS S, ROBY D. An essential role for salicylic acid in AtMYB30-mediated control of the hypersensitive cell death program in Arabidopsis. FEBS Letters, 2006, 580: 3498-3504.
[23] SOHN K H, LEI R, NEMRI A, JONES J D. The downy mildew effector proteins ATR1 and ATR13 promote disease susceptibility in Arabidopsis thaliana. The Plant Cell, 2007, 19: 4077-4090.
[24] RENTEL M C, LEONELLI L, DAHLBECK D, ZHAO B Y, STASKAWICZ B J. Recognition of the Hyaloperonospora parasitica effector ATR13 triggers resistance against oomycete, bacterial, and viral pathogens. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(3): 1091-1096.
[25] ZHANG M, LU J, TAO K, YE W W, LI A N, LIU X Y, KONG L, DONG S M, ZHENG X B, WANG Y C. A Myb transcription factor of Phytophthora sojae, regulated by MAP Kinase PsSAK1, is required for zoospore development. PLoS ONE, 2012, 7(6): e40246.
[26] MENGISTE T, CHEN X, SALMERON J, DIETRICH R. The BOTRYTIS SUSCEPTIBLE1 gene encodes an R2R3-myb transcription factor protein that is required for biotic and abiotic stress responses in Arabidopsis. The Plant Cell, 2003, 15(11): 2551-2565.
[27] LEE M W, QI M, YANG Y. A novel jasmonic acid-inducible rice myb gene associates with fungal infection and host cell death. Molecular Plant-Microbe Interactions, 2001, 14(4): 527-535.
(责任编辑 岳梅)
Cloning and Expression Analysis of Fusarium Wilt Resistance-Related Gene CIMYB Transcription Factor from CitruIIus Ianatus
HAN Jin-huan, WANG Li-xia, GAO Hong-bo, LÜ Gui-yun
(College of Horticulture, Agricultural University of Hebei, Baoding 071001, Hebei)
【Objective】The objective of this study is to clone ClMYB, which is a gene encoding MYB transcription factor, analyze its sequence features and expression characteristics, and to provide new insights into the understanding of the molecular mechanism in response of watermelon plants to Fusarium oxysporum f. sp. niveum infection.【Method】In a recent study, a novel EST of MYB transcription factors was obtained from data analysis of microarray and suppression subtractive hybridization (SSH) of incompatible interaction between watermelon and F. oxysporum f. sp. niveum. cDNA sequence of the MYB was isolated and clonedusing cDNA from watermelon roots with F. oxysporum f. sp. niveum infection by RT-PCR method, designated as ClMYB. The conserved domains and sequence features of ClMYB protein were analyzed by bioinformatics methods. Multiple sequence alignments and a phylogenetic tree of homologous species were done between ClMYB protein and its homologous ones from other species using MEGA5.0. The subcellular localization of ClMYB was analyzed using fusion expression vector PYBA1332-GFP. Then ORF of the cloned gene was inserted into the expression vector pCzn1 by Nde I and Xba I digestion. The recombinant plasmid was transformed into E. coli Arctic Express system and induced expression by 0.5 mmol·L-1IPTG for 4 h. The expression of the fusion protein was detected by SDS-PAGE. The expression pattern of target genes in watermelon with F. oxysporum f. sp. niveum were detected by real-time fluorescent quantitative PCR (qRT-PCR).【Result】Using RT-PCR method, cDNA sequence of ClMYB (GenBank: KT751229) was cloned from watermelon (PI296341-FR) root. Sequence alignment and bioinformatics analysis revealed that the deduced amino acids of ClMYB had common characteristics of MYB transcription factors with two MYB domains of R2 and R3 at the N-terminal and highly variation at the C-terminal. Phylogenetic tree analysis suggested that the encoded protein had the closest genetic relationship with Cucumis melo MYB (GenBank: XM_008440304) and Cucumis sativus MYB (GenBank: XM_011652633). Subcellular localization results showed that ClMYB protein was located in the nucleus which belongs to a typical transcription factor. The expression of fusion protein was obtained by inducing with IPTG in E. coli and its relative molecular weight was 36 kD. qRT-PCR showed that the expression of ClMYB was induced by F. oxysporum f. sp. niveum, and appeared peak earlier and higher in PI296341-FR than Black diamond. MeJA at 50 μmol·L-1could significantly improve the resistance of Black diamond to F. oxysporum f. sp. niveum and induce the expression of ClMYB. In PI296341-FR, the expression of ClMYB was also induced by MeJA, while its expression level was lower than Black diamond. 【Conclusion】ClMYB is a typical R2R3-MYB transcription factor and is located in the nucleus. The expression of fusion protein was obtained by prokaryotic expression and its relative molecular weight is 36 kD. The expression of ClMYB was induced by F. oxysporum f. sp. niveum and JA. It is speculated that ClMYB may be involved in JA-mediated resistance signal transduction network of F. oxysporum f. sp. niveum. It might play an important role in disease resistance of Citrullus lanatus.
watermelon; Fusarium wilt; ClMYB transcription factor; gene cloning; expression analysis
2016-05-06;接受日期:2016-06-16
国家自然科学基金(31201632)、河北省自然科学基金(C2016204138)
联系方式:韩金桓,E-mail:m15200099616@163.com。通信作者吕桂云,E-mail:guiyunlv@163.com
