小麦脱水素基因 WDHN1-2的克隆及其表达分析
2016-11-30朱维宁张大鹏张林生
刘 浩,朱维宁,张大鹏,张林生
(1.西北农林科技大学生命科学学院,陕西杨凌 712100; 2.西北大学生命科学学院,陕西西安 710069)
小麦脱水素基因 WDHN1-2的克隆及其表达分析
刘 浩1,朱维宁2,张大鹏1,张林生1
(1.西北农林科技大学生命科学学院,陕西杨凌 712100; 2.西北大学生命科学学院,陕西西安 710069)
为进一步明确小麦脱水素基因 WDHN1-2在逆境条件下的功能,以伞穗山羊草 DHN1基因为探针,通过电子克隆及RT-PCR技术获得 WDHN1-2基因后对其序列特征进行分析,同时利用基因表达综合数据库及半定量RT-PCR技术对该基因的表达模式进行解析。结果表明, WDHN1-2基因编码区(CDS)长为548 bp,编码的氨基酸具有脱水素保守序列K、Y和S片段,与山羊草脱水素EMT25371亲缘关系最近。WDHN1-2蛋白属于稳定且高度亲水蛋白,二级结构以α-螺旋和无规则卷曲为主;该蛋白在亚细胞中定位的可能性:过氧化物酶体>细胞核>线粒体基质,可能行使转录调控的功能。表达模式分析发现, WDHN1-2基因在小麦开花后22 d的胚乳中表达量最高,在ABA、PEG、NaCl及4 ℃低温胁迫下表达量均先上升后下降。
小麦;电子克隆;脱水素;生物信息学分析;半定量RT-PCR
脱水素,即胚胎发育晚期丰富蛋白第二家族蛋白,能够在植物胚胎发育晚期以及逆境胁迫(如低温、干旱和高盐等)下表达,部分脱水素的表达受到脱落酸 (ABA) 的诱导[1]。脱水素一般由85~574个氨基酸残基组成,其分子量从9.6~200 kDa不等[2-3]。脱水素富含极性氨基酸和亲水性氨基酸(甘氨酸和赖氨酸),缺乏疏水性氨基酸(色氨酸和半胱氨酸),具有高度亲水性和热稳定性,在90 ℃的水浴中仍可保持活性[4-5]。
根据脱水素中Y、S 和K 保守片段的组成,将其分为5种亚型:YnSKn、Kn、SKn、YnKn和KnS型[6-8]。其中,富含赖氨酸的K 片段是所有脱水素共有的,由15个氨基酸残基(EKKGIME/DKIKEKLPG)组成,一般有1~11个拷贝,位于蛋白质C端。这些K片段可以形成两亲性的α-螺旋,通过疏水作用与细胞内膜系统或蛋白的变性位点结合,阻止细胞结构的破坏及蛋白质变性[9-11]。多数脱水素的Y 片段位于其N端,其保守序列为(V/T)D(E/Q)YGNP,通常有1~3个拷贝,该片段与一些植物和细菌的分子伴侣的核酸结合位点具有同源性,但是Y 片段是否与核酸结合还需进一步的实验验证[12]。S片段由成串的丝氨酸组成,可被蛋白激酶磷酸化,使脱水素在信号肽的引导下定位在细胞质或细胞核[13-14]。
电子克隆是随着基因组计划和EST计划的实施而发展起来的,利用生物信息学手段获得基因部分或全长cDNA序列,并通过PCR技术进行基因的克隆及验证[15]。它具有投入低、速度快、针对性强、技术要求低等优点[16]。因此,电子克隆技术已成为植物基因工程中获得新基因的重要手段[17]。本研究以伞穗山羊草脱水素 DHN1基因为探针序列,通过电子克隆及RT-PCR技术获得 WDHN1-2基因后对其序列特征进行分析,同时利用基因表达综合数据库及半定量RT-PCR技术对该基因的表达模式进行解析,以期为后续 WDHN1-2基因的功能研究奠定基础。
1 材料与方法
1.1 材料及其处理
供试材料为普通六倍体小麦品种“中国春”,由西北农林科技大学生命科学学院提供。挑选籽粒饱满的小麦种子,蒸馏水冲洗3次后用75%的乙醇浸泡3~5 min,然后用去离子水冲洗乙醇,并浸泡24 h。取出种子置于铺有滤纸的培养皿上,加入适量水湿润,于人工培养箱中培养(光暗周期为16 h/8 h,昼夜温度为25 ℃/18 ℃,相对湿度为80%)。培养7 d后的小麦幼苗分别于4 ℃、20%PEG6000、800 mmol·L-1NaCl及100 μmol·L-1ABA条件下进行胁迫,并收集胁迫处理0、12、24、36、48、60 h后的小麦叶片,液氮速冻后,置于-80 ℃保存。
1.2 小麦叶片总RNA的提取及cDNA第一条链的合成
采用Invitrogen公司RNA提取试剂盒提取小麦叶片总RNA,并以此为模版,使用TaKaRa公司的反转录试剂盒,去除基因组DNA,合成单链cDNA,于-20 ℃保存备用。
1.3 小麦基因组DNA的提取
采用Tiangen公司植物基因组DNA提取试剂盒提取小麦基因组DNA。
1.4 小麦脱水素 WDHN1-2基因的克隆
以伞穗山羊草 (Aegilopsumbellulata) 脱水素 DHN1基因 (GenBank: AM180925) 为探针,在小麦EST数据库中Blast (http://blast.ncbi.nlm.nih.gov/Blast.cgi),下载相似度较高的EST序列,采用 DNAMAN进行序列拼接。通过NCBI提供的ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)在线软件,对电子克隆得到的基因序列进行开放式阅读框分析,初步确定所拼接序列为小麦 DHN1基因。利用小麦基因组数据库(http://plants.ensembl.org/Triticum_aestivum/Info/Inde) 对接接序列进行Blast分析,并通过Fgenesh软件 (http://linux1.softberry.com/berry.phtml) 进行基因预测,确保基因序列的准确性。
利用Primer 5.0对电子克隆获得的序列进行特异引物设计(上游引物5′-AGTAAGAGGCA AAGATGGAGTT-3′;下游引物5′-AAGCAGC CAGCCGAAGC-3′),再分别以小麦基因组 DNA和 cDNA 第一条链为模版进行PCR扩增。PCR产物回收纯化并连接到 pMD18-T载体,阳性克隆送上海生工生物公司进行测序。最后,将测序得到的cDNA序列与电子克隆得到的ORF序列比对,根据序列相拟度确定扩增序列是否为小麦 DHN1基因序列。
1.5 生物信息学分析
利用ExPASy服务器(http://www.expasy.org/resource)中的ProtParam和ProtScale软件预测蛋白的理化性质;通过DNAMAN V7和MEGA 5.0软件进行氨基酸的多序列比对和系统发育树的构建;利用Psort在线软件(http://psort.hgc.jp/form.html)对WDHN1-2蛋白进行亚细胞定位预测;通过Phyre 2 (http://www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id=index)同源建模软件预测蛋白质的二级和三级结构;通过ProtFun软件(http://www.cbs.dtu.dk/services/ProtFun/)分析WDHN1-2蛋白的功能;通过PLEXdb基因表达综合数据库(http://www.plexdb.org/index.php) Blast搜索 WDHN1-2基因所对应的探针,获得探针的表达量信息,并通过芯片数据在线分析软件鉴定小麦 WDHN1-2基因在发育过程中于不同器官中的表达模式。
1.6 WDHN1-2基因的半定量RT-PCR分析
以不同胁迫处理的小麦叶片cDNA为模板,以小麦18s rRNA基因作为内参(上游引物: 5′-CAGCACCGCCAGTGAATAG-3′;下游引物:5′-TGTAGGTGGCAAAGGTCTC-3′),通过TaKaRa公司的rTaq聚合酶进行PCR扩增,对 WDHN1-2基因进行半定量RT-PCR分析。 WDHN1-2半定量 RT-PCR引物与基因扩增引物一致。
2 结果与分析
2.1 小麦 WDHN1-2基因的克隆结果
通过伞穗山羊草脱水素 DHN1基因在NCBI中Blast得到20条相似度较高的小麦EST序列,利用DNAMAN软件将这些序列拼接,得到长为1 151 bp的conting。ORF Finder预测可知,该Conting存在长为459 bp的ORF序列,初步明确该基因序列为小麦 DHN1基因。在小麦基因组数据库中对可能的新基因进行Blsat分析,并通过Fgenesh软件进行基因预测,获得准确的基因序列。再以小麦cDNA第一条链和基因组DNA为模板进行PCR扩增,分别得到1条约500 bp和 550 bp的条带(图1),测序结果表明, WDHN1-2基因的cDNA 全长465 bp,DNA全长548 bp。将测序得到的cDNA序列与电子克隆得到的ORF序列进行比对,发现序列相似度为100%,说明扩增得到的序列为小麦 DHN1基因序列,故命名为 WDHN1-2基因。 WDHN1-2基因编码区序列(CDS)总长548 bp,含2个外显子(1~189 bp,273~548 bp)和1个内含子(190~272 bp),编码154个氨基酸残基(图2)。
2.2 WDHN1-2蛋白的生物信息学分析
2.2.1 WDHN1-2蛋白的理化性质
WDHN1-2蛋白分子式为C649H1033N213O220S7,分子量为15 564.00 kDa,理论等电点( pI )为9.52。脂肪族氨基酸指数为31.17,不稳定系数为25.34,为稳定蛋白。亲水性氨基酸(甘氨酸和赖氨酸)含量较高,缺乏疏水性氨基酸(色氨酸和半胱氨酸),总平均亲水性值 (GRAVY) 为-1.026,为高度亲水性蛋白(图3、图4)。
2.2.2 WDHN1-2蛋白的系统进化分析
利用NCBI数据库对WDHN1-2蛋白的氨基酸序列进行BlastP分析,并通过DNAMAN软件对其进行多重序列比对,结果表明,WDHN1与EMT25371 (山羊草,Aegilopstauschii)、CAJ56059 (圆锥小麦,Triticumturgidum)、AAF01697 (大麦,Hordeumvulgare)、ABO14410 (大麦,Hordeumvulgare)、EMS59889 (乌拉尔图小麦,Triticumurartu)、CAJ56055 (伞穗山羊草,Aegilopsumbellulata)和EMS58299 (乌拉尔图小麦,Triticumurartu) 的相似性分别为98.70%、96.64%、95.21%、94.52%、91.89%和96.00%,主要存在4个保守区域,即1个Y片段、1个S片段和2个K片段(图2、图5)。根据序列比对结果,利用MEGA 5.0对亲缘远近不同的脱水素序列进行系统进化分析(图6),结果表明,小麦WDHN1蛋白与山羊草EMT25371脱水素的亲缘关系最近。

M: DL2000 marker; 1: cDNA; 2: DNA
2.2.3 WDHN1-2蛋白的二级及三级结构
WDHN1-2蛋白由3种二级结构组成,其中α-螺旋、β-折叠及无规则卷曲分别占18%、3%和82%,即α-螺旋和无规则卷曲为主要的二级结构,且α-螺旋的位置主要位于蛋白的K片段保守区域(图7),这与三级结构预测结果(WDHN1-2蛋白含有2 个以K片段为核心的α-螺旋,其余序列为无规则卷曲)(图8)相吻合。

图2 WDHN1-2基因核酸序列及所编码蛋白的氨基酸序列分析

图3 WDHN1-2蛋白的氨基酸组成及含量分析

图4 WDHN1-2蛋白的氨基酸序列亲/疏水性预测
2.2.4 WDHN1-2蛋白的亚细胞定位及功能分析
蛋白在特定亚细胞位置才能行使其相应的功能,因此蛋白功能与其亚细胞定位紧密相关,蛋白的亚细胞定位也从一定程度反映了蛋白的功能。通过Psort软件对WDHN1-2进行预测,结果表明,该蛋白亚细胞定位的可能性:过氧化物酶体(0.635)> 细胞核(0.300)> 线粒体基质(0.100)。通过ProtFun软件对WDHN1-2的功能进行预测,结果(表1)表明,WDHN1-2蛋白主要参与转录调控、生长因子和信号转导等功能,推测WDHN1-2蛋白可能为转录因子。
2.2.5 不同器官中 WDHN1-2基因的表达情况
通过PLEXdb基因表达综合数据库Blast搜索 WDHN1-2 基因所对应的探针TaAffx.46097.1.S1_at,并在基因芯片数据库 (GEO accession:GSE12508)[19]中获得探针的表达量。结果(图9)表明, WDHN1-2 基因分别在小麦种子萌发期的根、幼苗期的叶、开花之前的雌蕊、开花后22 d的胚芽等器官中表达量均较高,其中,开花后22 d的胚芽中基因表达量最高。
2.3 逆境胁迫下叶片中 WDHN1-2基因的半定量RT-PCR分析结果
采用半定量RT-PCR对 WDHN1-2基因在非生物胁迫下于小麦叶片中的表达模式进行分析,结果(图10)表明,在 ABA、PEG、NaCl及4 ℃胁迫下 WDHN1-2基因表达量均先上升后下降。ABA、PEG和NaCl胁迫下,其表达量在24 h达到最高;低温胁迫下,12 h表达量达到最高。
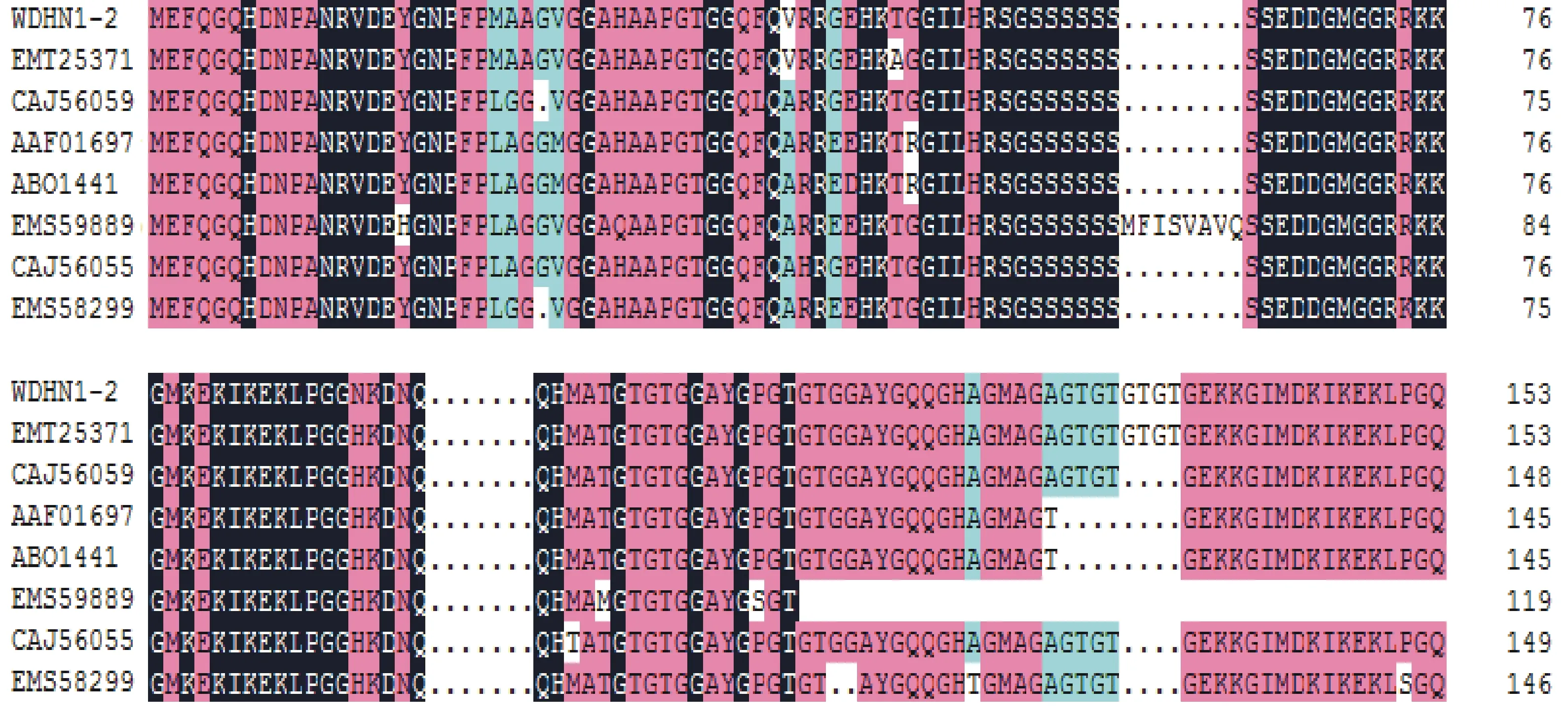
图5 WDHN1-2蛋白与其他植物脱水素氨基酸序列的多重序列比对结果

图6 WDHN1-2蛋白与其他植物脱水素蛋白的系统进化分析

波浪线:α-螺旋;箭头:β-折叠;问号:无规则卷曲。
Wavy line: Alpha helix; Arrow: Beta strand; Interrogation mark: Random coil.
图7 WDHN1-2蛋白的二级结构预测
Fig.7 Predicted secondary structure of WDHN1-2 protein

图8 WDHN1-2蛋白的三级结构预测

表1 WDHN1-2蛋白的主要功能预测

图9 基因芯片分析不同器官中 WDHN1-2基因表达模式
3 讨 论
电子克隆是依托于生物信息学克隆基因的一种技术,随着基因组计划和 EST 计划的实施而逐渐兴起,它具有投入低、速度快、针对性强和技术要求低等优点[17]。但由于某些基因剪切方式的多样性及生物大分子结构和功能的复杂性,分析软件的预测结果通常存在较大偏差,因此通过电子克隆获得的实验结果只能作为参考,必须进行试验验证。

图10 不同非生物胁迫下 WDHN1-2基因的表达模式
WDHN1-2蛋白分子量为15.56 kDa,其氨基酸序列中亲水性氨基酸(甘氨酸和赖氨酸)含量最高,缺乏疏水性氨基酸(色氨酸和半胱氨酸),总平均亲水性值 (GRAVY) 为-1.026,表明WDHN1-2蛋白为高度亲水蛋白。序列比对及系统进化树分析发现,WDHN1-2蛋白具有和脱水素家族一致的高度保守区域 (K、Y 和S 片段),其中K片段保守域还可以形成两亲性的α-螺旋,并且与山羊草 EMT25371脱水素的亲缘关系最近,由此推断WDHN1-2蛋白与山羊草EMT25371脱水素具有相似的生物功能。以上特征均表明 WDHN1-2属于YSK2型脱水素。
脱水素能够在植物胚胎发育晚期以及逆境胁迫(如低温、干旱和高盐等)下大量表达。 WDHN1-2基因在ABA、PEG、NaCl及4 ℃表达量均上升。通过功能预测推断,WDHN1-2蛋白可能行使转录调控的功能,调控下游信号转导以及逆境响应基因的表达,从而提高植物逆境胁迫下的耐受性[20];同时,亚细胞定位预测结果表明, WDHN1-2基因存在于细胞核的可能性较大,为此推论提供了间接的证据。部分YSK2型脱水素已被证明定位在细胞核,行使转录调控的功能,如RAB 15等[21],但是具体的调控机制仍然不清楚。
基因表达综合数据库是目前最大的且完全开放的高通量分子丰度数据库,主要存储基因表达数据[22-23]。通过该数据库Blast搜索 WDHN1-2基因发育过程中的表达模式,发现 WDHN1-2基因在小麦开花后22 d的胚芽中表达量最高,这与脱水素胚胎发育晚期大量表达一致。
本研究对小麦脱水素基因 WDHN1-2的克隆及生物功能的挖掘,为进一步提高小麦逆境耐受性及分子育种提供了新的线索和基础。
[1]KOSOVA K,VITAMVAS P,PRASIL I T.Wheat and barley dehydrins under cold,drought,and salinity-what can LEA-II proteins tell us about plant stress response? [J].FrontiersinPlantScience,2014,5:343.
[2]LABHILILI M,JOUDRIER P,GAUTIER M F.Characterization of cDNAs encodingTriticumdurumdehydrins and their expression patterns in cultivars that differ in drought tolerance [J].PlantScience,1995,112(2):219.
[3]KIM E C,LEE H S,CHOI D W.Sequence variability and expression pattern of the dehydrin gene family inPopulustremula×Populusalbavar.glandulosa[J].PlantOmics,2012,5(2):122.
[4]BATTAGLIA M,OLVERA-CARRILLO Y,GARCIARRUBIO A,etal.The enigmatic LEA proteins and other hydrophilins [J].PlantPhysiology,2008,148(1):6.
[5]ALLAGULOVA CH R,GIMALOV F R,SHAKIROVA F M,etal.The plant dehydrins: structure and putative functions [J].BiochemistryBiokhimiia,2003,68(9):945.
[6]BAE E K,LEE H,LEE J S,etal.Differential expression of a poplar SK2-type dehydrin gene in response to various stresses [J].BMBReports,2009,42(7):439.
[7]BRINI F,YAMAMOTO A,JLAIEL L,etal.Pleiotropic effects of the wheat dehydrin DHN-5 on stress responses inArabidopsis[J].Plant&CellPhysiology,2011,52(4):676.
[8]CLOSE T J.Dehydrins: A commonalty in the response of plants to dehydration and low temperature [J].PhysiologiaPlantarum,1997,100:291.
[9]KOAG M C,WILKENS S,FENTON R D,etal.The K-segment of maize DHN1 mediates binding to anionic phospholipid vesicles and concomitant structural changes [J].PlantPhysiology,2009,150(3):1503.
[10]RAHMAN L N,MCKAY F,GIULIANI M,etal.Interactions ofThellungiellasalsugineadehydrins TsDHN-1 and TsDHN-2 with membranes at cold and ambient temperatures-surface morphology and single-molecule force measurements show phase separation,and reveal tertiary and quaternary associations [J].Bba-biomembranes,2013,1828(3):967.
[11]DRIRA M,SAIBI W,BRINI F,etal.The K-segments of the wheat dehydrin DHN-5 are essential for the protection of lactate dehydrogenase and beta-glucosidase activitiesinvitro[J].MolecularBiotechnology,2013,54(2):643.
[12]CLOSE T J.Dehydrins: Emergence of a biochemical role of a family of plant dehydration proteins [J].PhysiologyPlantarum,1996,97(4):795.
[13]ALSHEIKH M K,SVENSSON J T,RANDALL S K.Phosphorylation regulated ion-binding is a property shared by the acidic subclass dehydrins [J].Plant,Cell&Environment,2005,28(9):1114.
[14]RIERA M,PHILLIPS R L.Protein kinase CK2 modulates developmental functions of the abscisic acid responsive protein Rab17 from maize [J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica,2004,101(26):9879.
[15]胡 皝,萧浪涛.生物信息学在新基因全长cDNA电子克隆中的应用[J].生物技术通报,2007(4):93.
HU H,XIAO L T.Application of bioinformatics in full-length cDNA sequence in silicon cloning of novel genes [J].BiotechnologyBulletin,2007(4):93.
[16]黄 骥,张红生,曹雅君,等.水稻功能基因的电子克隆策略 [J].中国水稻科学,2002,16(4):295.
HUANG J,ZHANG H S,CAO Y J,etal.Strategy of in silico cloning of functional genes in rice (Oryzasativa) [J].ChineseJournalofRiceScience,2002,16(4):295.
[17]王冬冬,朱延明,李 勇,等.电子克隆技术及其在植物基因工程中的应用[J].东北农业大学学报,2006,37(3):403.
WANG D D,ZHU Y M,LI Y,etal.Application of in silico cloning technique in plant gene engineering [J].JournalofNortheastAgriculturalUniversity,2006,37(3):403.
[18]KYTE J,DOOLITTLE R F.A simple method for displaying the hydropathic character of a protein [J].JournalofMolecularBiology,1982,157(1):105.
[19]SCHREIBER A W,SUTTON T,CALDO R A,etal.Comparative transcriptomics in the Triticeae [J].BMCGenomics,2009,10:285.
[20]HIRAYAMA T,SHINOZAKI K.Research on plant abiotic stress responses in the post-genome era: past,present and future [J].PlantJournal,2010,61(6):1041.
[21]THI V H P,HARTOMO T B,LEE M J,etal.Rab15 alternative splicing is altered in spheres of neuroblastoma cells [J].OncologyReports,2012,27(6):2045.
[22]余海浪,马文丽,郑文岭.用于基因数据挖掘的基因表达数据库GEO[J].中国生物工程杂志,2007,27(8):96.
YU H L,MA W L,ZHENG W L.Data mining procedures using GEO (Gene Expression Omnibus) [J].ChinaBiotechnology,2007,27(8):96.
[23]刘 华,马文丽,郑文岭.GEO (Gene Expression Omnibus):高通量基因表达数据库[J].中国生物化学与分子生物学报,2007,23(3):236.
LIU H,MA W L,ZHENG W L.GEO (Gene Expression Omnibus):High-throughput gene expression database [J].ChineseJournalofBiochemistryandMolecularBiology,2007,23(3):236.
Cloning and Expression Analysis of Dehydrin Gene WDHN1-2 in Wheat
LIU Hao1,ZHU Weining2,ZHANG Dapeng1,ZHANG Linsheng1
(1.College of Life Science,Northwest A&F University,Yangling,Shaanxi 712100,China;2.College of Life Science,Northwest University,Xi’an,Shaanxi 710069,China)
To investigate the function of WDHN1-2 gene in wheat, WDHN1-2 was cloned from wheat in silico and analyzed by bioinformatics using DHN1 gene ofAegilopsumbellulataas the probe. The CDS of WDHN1-2 gene was 548 bp,and the transcript levels of WDHN1-2 reached the highest level at 22 DAP in embryo. Semi-quantitative RT-PCR analysis indicated that transcript accumulation was first increased and then decreased under low temperature,sodium chloride (NaCl),abscisic acid (ABA) and polyethylene glycol (PEG) treatments.WDHN1-2 protein belonged to hydrophilic protein,which had typical features of DHNs that contains one Y segment,one S segment and two K segments. The secondary structure of this protein was mainly composed of alpha helix and random coil. This protein was likely to be located in the peroxisome,nucleus,and mitochondrial matrix,may play a function of transcriptional regulation.
TriticumaestivumL.;Insilicocloning; Dehydrin; Bioinformatics analysis; Semi-quantitative RT-PCR
时间:2016-10-08
2016-04-09
2016-05-21
旱区作物逆境生物学国家重点实验室资助课题(CSBA2015007);高等学校博士学科点专项科研基金项目(20120204110033)
E-mail:504885033@qq.com
张林生(E-mail:linszhang@nwsuaf.edu.cn)
S512.1;S332.2
A
1009-1041(2016)10-1291-08
网络出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20161008.0932.006.html
