肺炎克雷伯菌携带碳青霉烯酶KPC-2基因环境多态性研究
2016-11-29沈平华蒋晓飞
沈平华, 仉 英, 蒋晓飞
·论著·
肺炎克雷伯菌携带碳青霉烯酶KPC-2基因环境多态性研究
沈平华1, 仉 英2, 蒋晓飞1
目的 研究复旦大学附属华山医院耐碳青霉烯类肺炎克雷伯菌广泛播散初期携带blaKPC-2的完整基因结构,获得其播散的基因背景。方法 收集2006年8月―2009年12月连续不重复的碳青霉烯类耐药肺炎克雷伯菌42株,进行质粒抽提、blaKPC-2筛选、多位点序列分型(MLST);随后设计一系列PCR引物,对blaKPC-2基因结构进行完整测序。结果 MLST分型显示:34株属于ST11型,5株属于ST423,2株属于ST65,1株属于ST977。主要发现两种携带blaKPC-2基因结构,A型(8/42):Tn1721-blaKPC-2-Tn3;B型(34/42):Tn1721-blaKPC-2-ΔTn3-IS26,且B型结构的两端序列存在差异。结论 该组中肺炎克雷伯菌携带blaKPC-2的基因结构存在多态性,Tn1721-blaKPC-2-ΔTn3-IS26样结构占优势。研究获得的携带blaKPC-2基因结构及侧翼的完整序列,为进一步正确认识和理解blaKPC-2的播散模式和机制提供了完整的基因背景。
肺炎克雷伯菌; 碳青霉烯类耐药; KPC-2; 基因环境
碳青霉烯类耐药肺炎克雷伯菌的全球蔓延,已经成为临床治疗的巨大难题。KPC (Klebsiella pneumoniae carbapenemases)是导致肺炎克雷伯菌对碳青霉烯类耐药的主要原因[1-2],而KPC-2是KPC中最常见的类型之一[3]。
2007年,中国浙江杭州地区出现了首个关于KPC-2的报道,该报道中blaKPC-2位于一株临床肺炎克雷伯菌的质粒pKP048(GenBank accession no: FJ628167.2)上[4],通过全质粒测序可知blaKPC-2位于一个完整的Tn1721样转座子中。此后,在上海地区的一株临床肺炎克雷伯菌中发现质粒pKPHS2 (GenBank accession no: CP003224.1) 上Tn1721结构被IS26在Tn3的tnpR处打断,造成tnpATn3和IRL2Tn1721的丢失,形成了Tn1721-blaKPC-2-ΔTn3-IS26样的新结构[5]。
blaKPC-2的基因环境是了解blaKPC-2播散途径的重要信息,是进行有效医院感染控制的基础。本研究将华山医院自第1株碳青霉烯类耐药肺炎克雷伯菌的检出到该耐药细菌广泛播散的这段时期内耐碳青霉烯类肺炎克雷伯菌作为一个完整的样本,应用PCR及测序获得携带blaKPC-2完整的基因结构及侧翼序列,希望为下一步深入研究该耐药基因的播散机制提供全面的基因背景。
1 材料与方法
1.1菌株来源和药敏试验
收集上海华山医院2006年8月-2009年12月连续不重复的耐碳青霉烯类肺炎克雷伯菌,共42株。全部菌株使用法国生物梅里埃公司VITEKAMS 60全自动细菌鉴定系统鉴定菌种。肺炎克雷伯菌ATCC 700603和大肠埃希菌ATCC 25922购自卫生部临床检验中心。按照CLSI推荐的纸片扩散法(K-B法)进行抗菌药物敏感性测试,根据CLSI 2014年版标准判断结果。药敏纸片均购自英国 OXOID公司。
1.2方法
1.2.1质粒抽提与耐药基因blaKPC-2的筛选 采用Qiagen Plasmid Midi kit (Qiagen公司, 德国)抽提野生株质粒,作为后续实验的DNA模板。Taq DNA聚合酶购自美国Promega公司。采用引物KPCFOR:5'-TCGCTAAACTCGAACAGG-3' 和 KPCREV:5'-TTACTGCCCGTTGACGCCCAATCC-3'[6],以抽提的质粒为模板,对42株细菌进行PCR,将PCR产物送上海迈浦生物科技有限公司纯化后正反双向测序。
1.2.2多为点序列分型(MLST) 扩增肺炎克雷伯菌基因组上的7个管家基因:gapA、 infB、 mdh、pgi、 phoE、 rpoB、 tonB,将PCR产物送上海迈浦生物科技有限公司纯化后测序。参照肺炎克雷伯菌MLST分型网站(http://www.pasteur.fr/recherche/ genopole/PF8/mlst/Kpneumoniae.html)提供的步骤进行分型。
1.2.3携带blaKPC-2的基因环境分析 根据pKP048和pKPHS2的序列,设计了一系列PCR引物(图 1和表1)进行扩增与测序,分别用于检测本批菌株中是否存在与上述已经报道的blaKPC-2基因环境相同的结构。Junction PCR的引物主要用于确定携带blaKPC-2基因结构的界限和区分pKP048和pKPHS2上结构差异的关键序列,Mapping PCR主要用于扩增携带blaKPC-2基因结构的主干部分,包括M1、M2、 M3、 M4、 M5和M6,见图1。结合后续的Crossing PCR可以覆盖整个基因环境的序列。PCR阳性产物纯化后测序。
根据Junction PCR和Mapping PCR的结果,当出现区别于以上两种结构的新结构时,拟采用代表性质粒测序及以纯质粒为模板进行 PCR 步移测序等,获得整个新结构的完整基因环境的序列。引物均由北京六合华大基因科技有限公司(上海分公司)合成。
2 结果
2.1菌株情况和药敏结果
共收集到42株不重复的碳青霉烯类耐药肺炎克雷伯菌:2006年1株,2007年3株,2008年6株,2009年32株。其中13株来源于尿液,27株来源于痰液,2株来源于血液。所有菌株均对亚胺培南和美罗培南等碳青霉烯类药物耐药。
2.2blablaKPC-2KPC-2的筛选和MLST分型LST
对抽提质粒,采用blaKPC特异性引物进行PCR及测序,结果显示质粒上均携带有blaKPC-2基因。42株耐药肺炎克雷伯菌 (MLST)分型显示:34株为ST11型,5株属于ST423,2株为ST65,1株为ST977。主要的流行型别为ST11型,这与QI等[7]报道一致。
2.3携带 blablaKPC-2KPC-2的基因环境分析
Junction PCR和Mapping PCR结果见表2,图2展示了所有菌株携带blaKPC-2的基因结构及该结构两侧的序列。本批菌株检测到了与pKP048和pKPHS2不同的blaKPC-2的基因环境,存在多态性。
A型为Tn1721-blaKPC-2-Tn3样结构,显示具备与pKP048相似的blaKPC-2的基因环境,但仍存在差异,表现为Tn1721的IRR上游序列为parA基因,而不是pKP048的ΔIS26;Tn3的IRL后直接连接一个DNA distortion protein 3-like基因,而不是Tn1721的IRL2。代表性质粒为pHS062105-3(GenBank accession no: KF623109.1)。A型结构,分别位于ST423、ST65、ST97型菌株中。

图1 Junction PCR和 Mapping PCR引物设计示意图Figure 1 Schematic map of primer annealing sites for Junction PCR and Mapping PCR
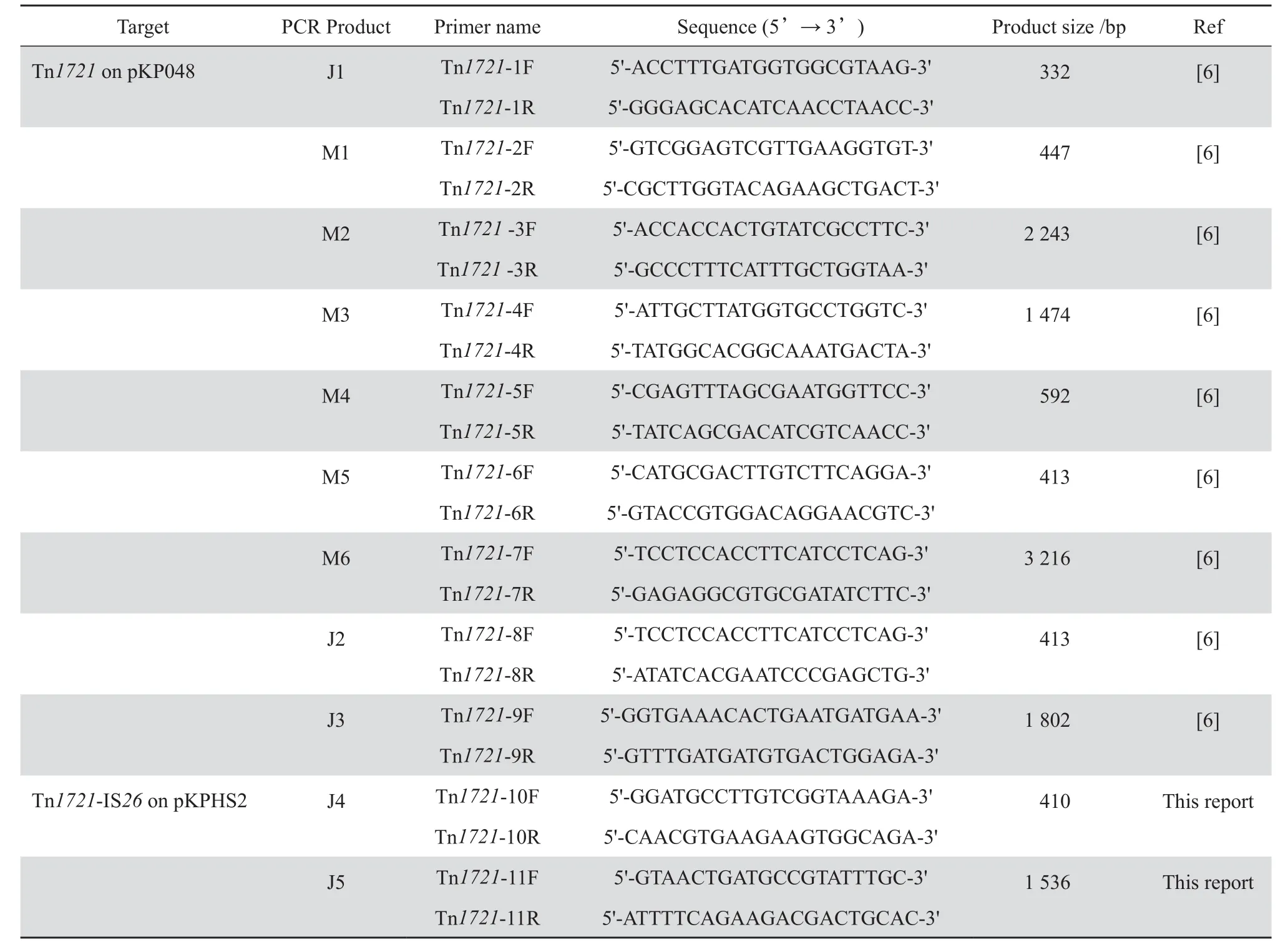
表1 用于本研究PCR的引物Table 1 Primers used in this study

表2 blaKPC-2阳性肺炎克雷伯菌MLST分型与Junction和Mapping PCR结果Table 2 MLST genotyping of blaKPC-2-carrying K. pneumoniae isolates and the result of Junction PCR and Mapping PCR

图2 A型与B1、B2结构携带blaKPC-2基因结构示意图Figure 2 Schematic representations of blaKPC-2-bearing genetic elements
Genes are depicted as arrows according to the direction of transcription. blaKPC-2is indicated in dark gray. Inverted repeats are indicated by colorvariable, vertical bars shown in corresponding matching colors: Tn1721 (black), Tn3 (white) and IS26 (gray). Regions sharing identical or nearidentical sequence across plasmids are indicated by the gray shading between the representations of different plasmids.
B1型显示具有与pKPHS2相同的blaKPC-2的基因环境,即Tn1721- blaKPC-2-ΔTn3-IS26。且该结构外围也具备相同的基因结构,即Tn1721的IRR上游连接ΔIS26,IS26下游连接TniA基因。代表质粒为pHS082416 (GenBank accession no:KF724507.1)。
B2型与B1型唯一的区别是J1阴性,以纯质粒为模板,应用引物2R向一侧步移测序。该型Tn1721的IRR上游序列与肺炎克雷伯菌JM45 plasmid p2 (GenBank accession no:CP006658.1)上的一个功能未知的蛋白序列高度一致,与ΔIS26无相似性。代表型质粒为pHS092753 (GenBank accession no: KF826293.1)。B2和B1结构中IS26的下游均连接着TniA基因。综上,B1和B2存在相同的骨干结构,且均位于ST11型菌株中。
3 讨论
华山医院分离的第1株碳青霉烯类耐药肺炎克雷伯菌为ST423型,而广泛播散时流行的菌株型别为ST11型,区别于国外流行的ST258型[8],同时也提示碳青霉烯类耐药性存在菌株间水平传播。
华山医院自碳青霉烯类耐药肺炎克雷伯菌出现初期,即存在blaKPC-2基因环境的多态性,提示其blaKPC-2播散可能存在不同的来源,为blaKPC-2播散的流行病学调查提供了研究依据。A型首先被发现,并可以稳定存在于不同ST型别的肺炎克雷伯菌中;B型出现晚于A型,推测可能是A型在结构上发生变化而形成,也有可能是外源性侵入而在医院内广泛播散。尤其B1与B2存在不同的侧翼序列,推测该结构是一种新型的嵌合型可移动元件,可能具备水平转移的能力,转移过程中发生了不同位置的插入。
在欧美,导致KPC-2广泛播散的原因是编码该酶的基因blaKPC-2主要位于活跃的转座子(Tn4401)上[9]。中国大陆地区至今还没有一例关于Tn4401携带blaKPC-2的报道。与欧美截然不同的基因环境,提示该耐药基因在中国可能存在不同的水平转移机制。
本研究在方法上也有一定的创新:克服了采用全质粒测序后获得携带blaKPC-2基因环境费时费力及难以进行大样本分析的弊端;同时以往的研究中,通常只是分析blaKPC-2上下游的一小段序列[4,10-12],而本研究完整展示了携带blaKPC-2基因环境,为进一步分析该结构可能的移动模式和机制提供了有价值的基因背景。PCR结合测序获取耐药基因周围环境的方法简单实用,可以推广至其他耐药基因周围环境序列的检测。
本研究的局限性在于只检测了碳青霉烯类耐药肺炎克雷伯菌广泛播散初期2006-2009年携带blaKPC-2的基因环境,同时鉴于碳青霉烯类耐药还广泛存在于其他肠杆菌科细菌中的事实,下一步研究将在菌株收集的时间和种类上进一步扩展,以获得华山医院耐碳青霉烯类肠杆菌科细菌携带blaKPC-2的基因环境,为控制医院感染提供有价值的信息。
研究显示,华山医院碳青霉烯类耐药肺炎克雷伯菌的主要流行株为ST11型,且临床数据显示携带耐药基因blaKPC-2的基因环境存在多态性,提出该耐药基因的水平播散可能存在不同的机制,为下一步研究该耐药基因的水平播散模式和机制奠定了研究基础。
[1] MUNOZ-PRICE LS, POIREL L, BONOMO RA, et al. Clinical epidemiology of the global expansion of Klebsiella pneumoniae carbapenemases[J]. Lancet Infect Dis, 2013,13(9):785-796.
[2] ZHANG Y, JIANG XF, WANG YY, et al. Contribution of beta-lactamases and porin proteins OmpK35 and OmpK36 to carbapenem resistance in clinical isolates of KPC-2-producing Klebsiella pneumoniae[J]. Antimicrob Agents Chemother, 2014,58(2):1214-1217.
[3] MUNOZ-PRICE LS, QUINN JP. The spread of Klebsiella pneumoniae carbapenemases: a tale of strains, plasmids, and transposons[J]. Clin Infect Dis, 2009,49(11):1739-1741.
[4] JIANG Y, YU DL, WEI ZQ, et al. Complete nucleotide sequence of Klebsiella pneumoniae multidrug resistance plasmid pKP048, carrying blaKPC-2, blaDHA-1, qnrB4, and armA[J]. Antimicrob Agents Chemother, 2010,54(9):3967-3969.
[5] LIU PL, LI P, JIANG XF, et al. Complete genome sequence of Klebsiella pneumoniae subsp. pneumoniae HS11286, a multidrug-resistant strain isolated from human sputum[J]. J Bacteriol, 2012,194(7):1841-1842.
[6] LI G, ZHANG Y, BI DX, et al. First report of a clinical,multidrug-resistant Enterobacteriaceae isolate coharboring fosfomycin resistance gene fosA3 and carbapenemase gene blaKPC-2on the same transposon, Tn1721[J]. Antimicrob Agents Chemother, 2015,59(1):338-343.
[7] QI Y, WEI Z, JI S, et al. ST11, the dominant clone of KPC-producing Klebsiella pneumoniae in China[J]. J Antimicrob Chemother, 2011,66(2):307-312.
[8] DELEO FR, CHEN L, PORCELLA SF, et al. Molecular dissection of the evolution of carbapenem-resistant multilocus sequence type 258 Klebsiella pneumoniae[J]. Proc Natl Acad Sci U S A, 2014,111(13):4988-4993.
[9] CUZON G, NAAS T, NORDMANN P. Functional characterization of Tn4401, a Tn3-based transposon involved in blaKPCgene mobilization[J]. Antimicrob Agents Chemother,2011,55(11):5370-5373.
[10] CHEN YT, LIN JC, FUNG CP, et al. KPC-2-encoding plasmids from Escherichia coli and Klebsiella pneumoniae in Taiwan[J]. J Antimicrob Chemother, 2014,69(3):628-631.
[11] SHEN P, WEI ZQ, JIANG Y, et al. Novel genetic environment of the carbapenem-hydrolyzing beta-lactamase KPC-2 among Enterobacteriaceae in China[J]. Antimicrob Agents Chemother,2009,53(10):4333-4338.
[12] LUO YP, YANG JY, YE LY, et al. Characterization of KPC-2-producing Escherichia coli, Citrobacter freundii, Enterobacter cloacae, Enterobacter aerogenes, and Klebsiella oxytoca isolates from a Chinese Hospital[J]. Microb Drug Resist, 2014,20(4):264-269.
Diversity of genetic environment of carbapenem-hydrolyzing β-lactamase KPC-2 among Klebsiella pneumoniae
SHEN Pinghua, ZHANG Ying, JIANG Xiaofei. (Department of Laboratory Medicine, Huashan Hospital, Fudan University, Shanghai 200040, China)
Objective This study was designed to determine the genetic structure bearing blaKPC-2, which prevailed in the clinical K. pneumoniae isolates recovered in Huashan Hospital, Fudan University, and examine the genetic background of blaKPC-2prevalence. Methods Forty-two consecutive and nonduplicate K. pneumoniae strains isolated during the period from August 2006 to December 2009 were included in this study. All strains were subjected to plasmid extraction, blaKPC-2detection, and multilocus sequence typing (MLST). A series of primers were designed to analyze the genetic environment of the blaKPC-2gene. Results MLST genotyping showed that 34 of the K. pneumoniae isolates belonged to ST11, five to ST423, two to ST65 and one to ST977. Two types of blaKPC-2-bearing genetic structures were identifi ed: type A (8/42): Tn1721-blaKPC-2-Tn3; type B (34/42): Tn1721-blaKPC-2-ΔTn3-IS26. The fl anking sequence of type B was distinct. Conclusions The genetic environment of blaKPC-2is diverse. Tn1721-blaKPC-2-ΔTn3-IS26 is the dominant chimeric structure bearing blaKPC-2in this set of clinical K. pneumoniae isolates. The complete genetic structure bearing blaKPC-2and the fl anking sequences characterized in this study will help to further understanding of the mode and mechanism of blaKPC-2dissemination.
Klebsiella pneumoniae; carbapenem resistance; KPC-2; genetic environment
R378.996
A
1009-7708 ( 2016 ) 06-0680-05
10.16718/j.1009-7708.2016.06.002
国家自然科学基金项目(81572031)。
1. 复旦大学附属华山医院检验医学科,上海 200040;2. 复旦大学附属妇产科医院检验医学科。
沈平华(1989—),女,硕士研究生,现在同济大学附属第一妇婴保健院检验科工作,主要从事肠杆菌科细菌耐药机制研究。
蒋晓飞,E-mail:jiangxi2154@aliyun.com。
2016-01-06
2016-02-17
