K 33位乙酰化修饰SUMO蛋白的化学合成
2016-11-23王业海孔一夫陈晨晨2李宜明
王业海,孔一夫,陈晨晨2,李宜明
(1.合肥工业大学生物与医学工程学院,合肥230009;2.中国科学院强磁场科学中心,合肥230031)
K 33位乙酰化修饰SUMO蛋白的化学合成
王业海1,孔一夫1,陈晨晨2,李宜明1
(1.合肥工业大学生物与医学工程学院,合肥230009;2.中国科学院强磁场科学中心,合肥230031)
采用高温辅助固相合成技术及基于多肽酰肼的自然化学连接技术,高效获得了小泛素相关修饰物(SUMO)蛋白及33位赖氨酸(K33)乙酰化SUMO蛋白.聚丙烯酰胺凝胶电泳(SDS⁃PAGE)和圆二色光谱分析结果表明,与生物表达蛋白相比,化学合成蛋白具有较好的纯度和类似的二级结构.
小泛素相关修饰物;乙酰化;高温辅助固相合成;多肽酰肼;自然化学连接
在真核细胞中小泛素相关修饰物(SUMO)修饰已经成为调控蛋白质功能的一种关键翻译后修饰[1~6].研究发现,数以百计的蛋白可以被 SUMO化[7~9],SUMO化蛋白进而参与到染色质的组装、DNA的修复、蛋白质的体内平衡和信号转导等生理过程中[10,11].与蛋白质泛素化类似,蛋白质的SUMO化同样需要E1激活酶、E2结合酶和E3连接酶等3种酶的参与[12].这3种酶的协同作用使底物蛋白SUMO化,而底物蛋白的SUMO化位点主要发生在一段收敛的ΨK x D/E(Ψ代表疏水性氨基酸;x代表任意氨基酸)序列的赖氨酸上[13].
SUMO化蛋白的识别主要是通过SUMO与其效应蛋白上SUMO相互作用基序(SIM域)的非共价相互作用来实现[14~16].SIM是一段少于10个氨基酸的多肽序列,包含1个以3~4个氨基酸为核心的疏水序列(主要是Val或者Ile)和1个邻近的酸性区域[17].SIM中的疏水残基会结合SUMO结构中的β2股,并且会使β折叠延伸.SIM中的酸性残基通过结合SUMO蛋白表面上的酸性区域来增加SIM与SUMO蛋白的结合力.SUMO化蛋白与效应蛋白SIM域的正确识别是SUMO化蛋白发挥正常功能的关键.
SIM中的一些Ser或Thr可以被磷酸化,磷酸化后的SIM则可通过电荷相互作用增强其与SUMO化蛋白的结合力[18~20];SUMO蛋白可以被乙酰化,乙酰化后的SUMO蛋白与SIM的结合力则大幅度降低[21].SUMO蛋白与SIM之间的这种精细化调控是SUMO化蛋白相关功能得以正常发挥的重要保证.
与磷酸化SIM的制备相比,制备大量、性质均一的乙酰化SUMO则要困难得多.目前,制备乙酰化SUMO蛋白的主要方法为通过非天然氨基酸定点嵌入方法将乙酰化Lys嵌入到SUMO蛋白中,或通过酶促反应,体外获得乙酰化SUMO蛋白[22].这2种方法都难以获得大量的乙酰化SUMO蛋白.近年来,由于多肽固相合成及片段连接技术的发展,使得蛋白质化学合成成为一种高效获得翻译后修饰蛋白的方法[23~34].尤其是高温辅助的Fmoc固相多肽合成技术有助于一次性合成序列较长的多肽片段[35].基于此,本文以高温辅助的Fmoc固相合成技术结合基于多肽酰肼的自然化学连接方法,将乙酰化SUMO蛋白分成两片段合成,然后将两片段肽连接获得全长蛋白,合成路线如Scheme 1所示.
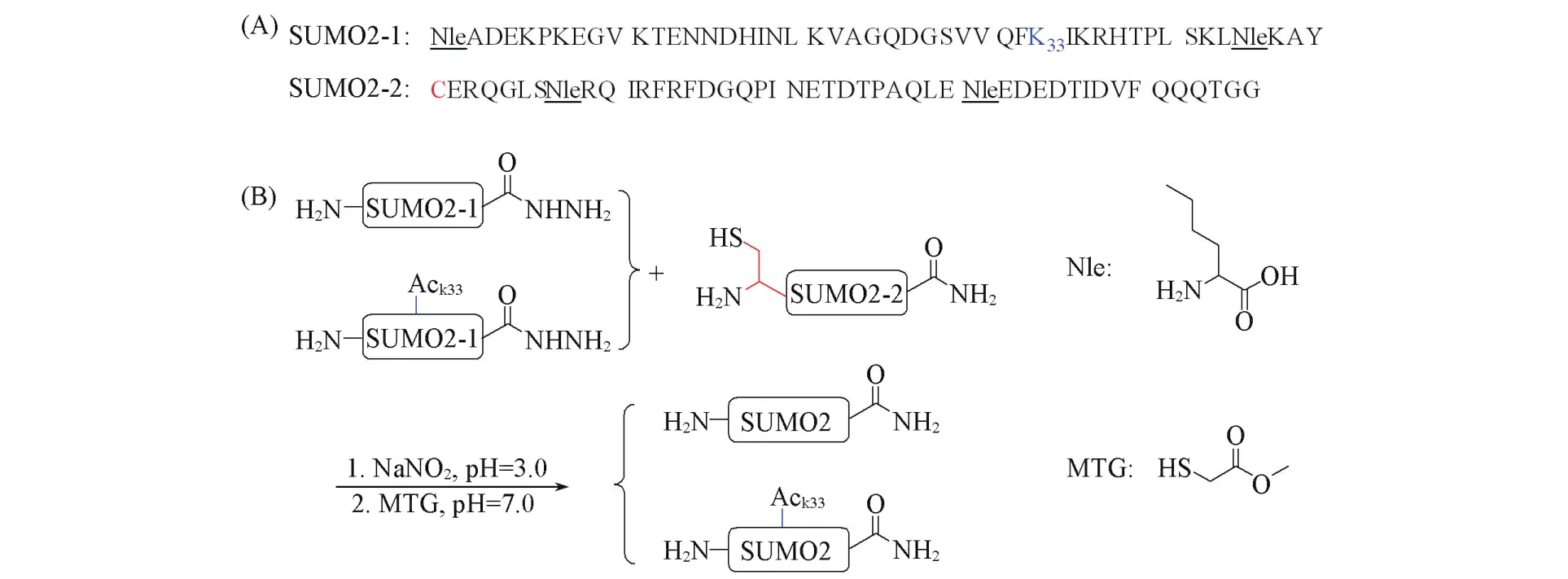
Scheme 1 Am ino acid sequence of SUMO2(A)and the synthesis strategy of SUMO2 and K 33⁃Ac⁃SUMO2(B)
1 实验部分
1.1 试剂与仪器
20种Fmoc氨基酸、Fmoc⁃Nle⁃OH和6⁃氯苯并三氮唑⁃1,1,3,3⁃四甲基脲六氟磷酸酯(HCTU)购自吉尔生化(上海)有限公司;N,N′⁃二甲基甲酰胺(DMF)、二氯甲烷(DCM)、N,N′⁃二异丙基乙胺(DIEA)、三氟乙酸(TFA)、甲醇、苯酚和乙腈均为分析纯,巯基乙酸甲酯(纯度为96%),购自上海阿拉丁生化科技股份有限公司;哌啶、乙醚和水合肼均为分析纯,购自国药集团化学试剂有限公司;三苯基甲基氯树脂(取代度为0.43mmol/g)和Rink酰胺树脂(取代度为0.34 mmol/g)购自天津南开和成科技有限公司;2⁃肟氰乙酸乙酯(Oxyma,纯度为98%)和N,N′⁃二异丙基碳二亚胺(DIC,纯度为99%)购自安耐吉化学公司.
Centrifuge 5424型离心机(德国艾本德股份公司);FE20⁃K⁃Plus型pH计(瑞士梅特勒⁃托利多公司);SHB⁃Ⅲ型台式循环水式多用真空泵(郑州长城仪器有限公司);FD⁃1A⁃50型冷冻干燥机(北京博医康实验仪器有限公司);IKA C⁃MAG SH7型磁力搅拌器[德国艾卡(广州)仪器设备有限公司];ME103E型分析天平(瑞士梅特勒⁃托利多公司);LC⁃20AT型高效液相色谱(HPLC)仪(日本岛津公司);Ultimate XB⁃C18型分析型色谱柱(φ250 mm×4.6 mm,美国格雷斯公司);Ultimate XB⁃C18型半制备型色谱柱(φ250 mm×10 mm,美国格雷斯公司);安捷伦1100型液相色谱⁃质谱联用仪(美国An⁃glient公司);J⁃1100型圆二色光谱(CD)仪(香港佰泰科技有限公司).
1.2 酰肼树脂的制备
将2 g三苯基甲基氯树脂(0.86 mmol)溶于30 mL DMF/DCM(体积比1∶1)混合溶剂中,加入8.6 mmol水合肼和17.2 mmol DIEA,冰浴条件下反应12 h;加入2 mL甲醇封闭未反应掉的三苯基甲基氯树脂;反应结束后,依次用DMF、水、甲醇和乙醚洗涤得到的酰肼树脂,风干并置于4℃冰箱中备用(经测定酰肼树脂的取代度为0.38 mmol/g).
1.3 小泛素相关修饰物第一片段(SUMO2⁃1)的制备
将263.1mg酰肼树脂置于固相合成管中,用15 mL DMF/DCM混合溶剂(体积比1∶1)溶胀15min后抽干,采用高温辅助的Fmoc固相合成法合成SUMO2⁃1(0.4 mmol氨基酸+0.4 mmol Oxyma+0.4 mmol DIC),反应时间为20~30 min,反应温度为75℃.其中,精氨酸(Arg)采用HCTU作为缩合剂,室温下反应40 min,反应2次;组氨酸(His)采用DIC作为缩合剂,室温下反应40 min;半胱氨酸(Cys)采用DIC作为缩合剂,50℃反应40 min.反应完毕,依次用DMF,DCM和DMF各洗涤5次,加入含有哌啶(体积分数20%)的DMF溶液反应2次(反应时间分别为2和10 min),脱除Fmoc保护基.组装完肽链后,向多肽合成管中加入8 mL切割试剂[V(TFA)∶V(苯酚)∶V(水)∶V(三异丙基硅烷,TIPS)=88∶5∶5∶2],反应2 h,将多肽从树脂上切下,并脱除侧链保护基团.将切割液置于离心管中,
用N2气鼓泡,浓缩溶液,最后用冰乙醚沉淀,离心,得到粗肽,经LC⁃MS分析确定是否为目标产物,使用半制备型HPLC分离得到SUMO2⁃1(110 mg,分离产率20.8%).
1.4 K 33乙酰化小泛素相关修饰物第一片段(K 33⁃Ac⁃SUMO2⁃1)的制备
将263.1mg酰肼树脂置于固相合成管中,用15mLDMF/DCM混合溶剂(体积比1∶1)溶胀15min后抽干;采用高温辅助的Fmoc固相合成法合成K33⁃Ac⁃SUMO2⁃1.每一种氨基酸耦合的合成步骤与SUMO2⁃1相同,不同之处在于第33位的Lys采用侧链氨基Alloc保护的Fmoc氨基酸进行耦合.当所有氨基酸耦合完成后,先用四(三苯基膦)钯(0.01mmol)脱除K33位赖氨酸上的Alloc保护基(反应2次,每次30 min);冲洗过后再加入乙酸酐试剂(1 mL乙酸酐+1 mL DIEA+8 mL DMF)对K33进行乙酰化修饰,反应10 min后,将肽链末端的Fmoc保护基用哌啶脱除掉.最后,将多肽从树脂上切割下来,采用相同的后处理过程得到粗肽,经LC⁃MS分析确定是否为目标产物,使用半制备型HPLC分离得到K33⁃Ac⁃SUMO2⁃1(106 mg,分离产率19.8%).
1.5 小泛素相关修饰物第二片段SUMO2⁃2的制备
将294 mg Rink酰胺树脂置于固相合成管中,用15 mL DMF/DCM混合溶剂(体积比1∶1)溶胀15 min后抽干;采用高温辅助的Fmoc固相合成法合成SUMO2⁃2.每一种氨基酸耦合的合成步骤与SUMO2⁃1相同.加完所有氨基酸后,将多肽从树脂上切割下来,采用相同的后处理过程得到粗肽,经LC⁃MS分析确定是否为目标产物,使用半制备型HPLC分离得到SUMO2⁃2(95 mg,分离产率17.9%).
1.6 SUMO2⁃1与SUMO2⁃2肽片段连接
参照文献[36~38]方法,将26.3 mg SUMO2⁃1用PBS缓冲液(6.0 mol/L Gn·HCl,0.2 mol/L Na2HPO4)溶解,在-10℃,pH=3.0条件下用亚硝酸钠氧化,得到酰基叠氮产物,再加入巯基乙酸甲酯(MTG),调节pH值至7.0,得到SUMO2⁃1硫酯;加入39.7 mg SUMO2⁃2,用分析型HPLC监测反应;待完全转化为SUMO2后,经LC⁃MS分析确定是否为目标产物,用半制备型HPLC分离,冻干,得到全长的SUMO2蛋白(24 mg,分离产率45.6%).
1.7 K 33⁃Ac⁃SUMO2⁃1与SUMO2⁃2肽片段连接
将26.5 mg K33⁃Ac⁃SUMO2⁃1用PBS缓冲液溶解,在-10℃,pH=3.0条件下用亚硝酸钠氧化,得到酰基叠氮产物;再加入巯基乙酸甲酯(MTG),调节pH值至7.0,得到K33⁃Ac⁃SUMO2⁃1的硫酯;加入39.7 mg SUMO2⁃2,用分析型HPLC监测反应;待完全转化为K33⁃Ac⁃SUMO2后,经LC⁃MS分析确定是否为目标产物,用半制备型HPLC分离,冻干,得到全长的K33⁃Ac⁃SUMO2蛋白(21mg,分离产率39.7%).
2 结果与讨论
2.1 SUMO2肽片段的合成与HPLC表征
实验中尝试采用标准的Fmoc固相合成方法合成SUMO2肽片段.将所需的氨基酸(0.4 mmol)、2⁃(7⁃偶氮苯并三氮唑)⁃N,N,N′,N′⁃四甲基脲六氟磷酸酯(0.36 mmol)和 N,N′⁃二异丙基乙胺(0.8 mmol)依次加入合成管中反应生成SUMO2肽片段,由于片段过长导致合成效果并不理想.在Fmoc固相合成方法中,高温可以提高氨基酸之间的耦合效率,进而提高长片段多肽合成产率,因此本文采用该方法合成SUMO的2个片段.通过这种方法制得了纯度较高的2个片段的粗肽,但质谱分析发现所得到的产物并不是SUMO2⁃1和SUMO2⁃2.经过分析发现,相比于正常肽片段每个产物的分子量都多32.由于甲硫氨酸(Met)易于氧化,且每条肽片段都含有2个Met,故推测是在合成过程中Met发生了氧化.因此,采用正亮氨酸(Nle)替代Met来获得正确的多肽片段[24].经过改进获得了纯度较高的粗肽产物,HPLC分析结果如图1所示.虽然乙酰化片段1的纯度比天然片段略有下降,但整体产率依然较好(SUMO2⁃1产率20.8%,K33⁃Ac⁃SUMO2⁃1产率19.8%,SUMO2⁃2产率17.9%).
2.2 SUMO2的获得与HPLC表征
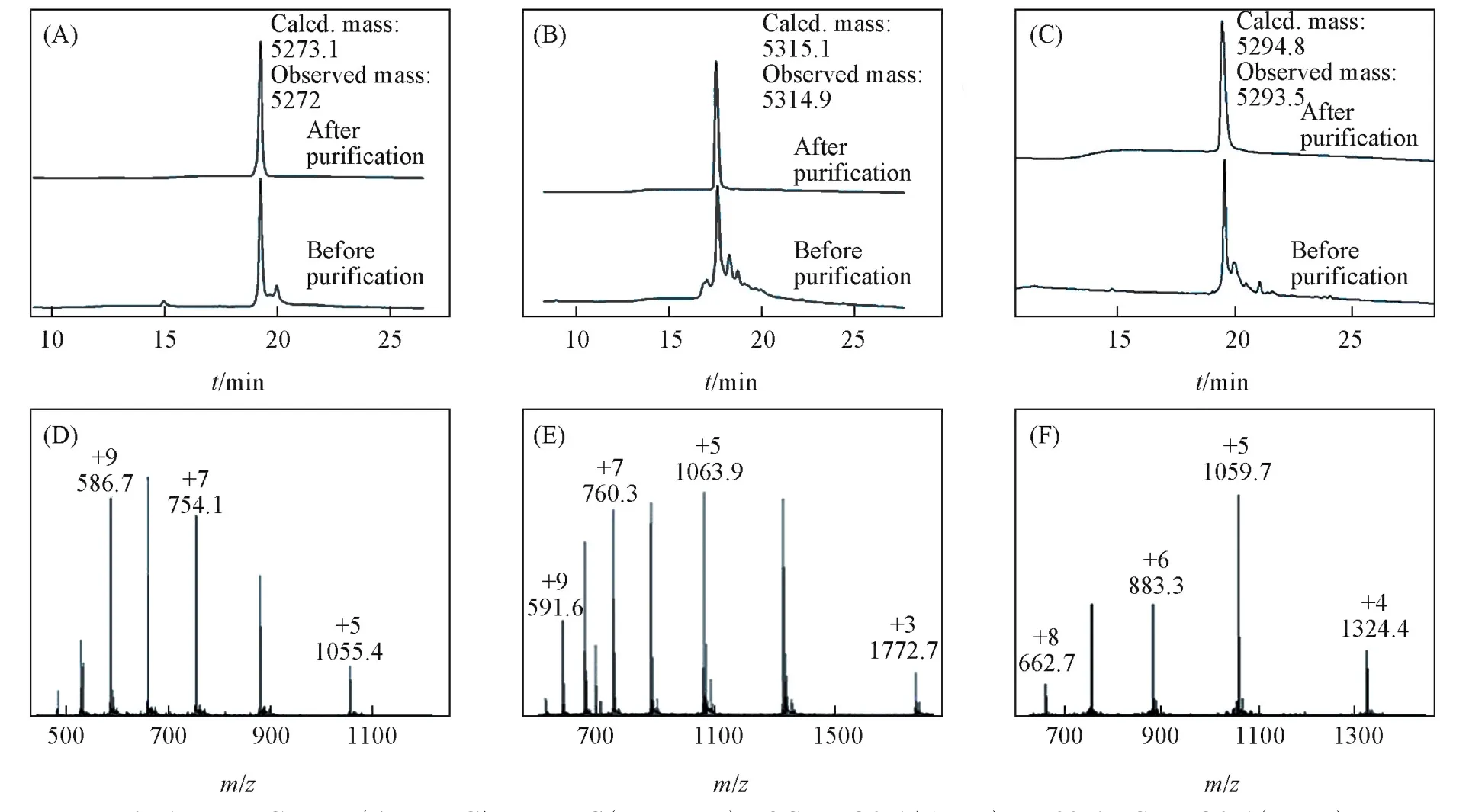
Fig.1 HPLC trace(A,B,C)and MS(D,E,F)of SUMO2⁃1(A,D),K 33⁃Ac⁃SUMO2⁃1(B,E)and SUMO2⁃2(C,F)
在获得SUMO2肽片段后,利用基于多肽酰肼的自然化学连接方法进行肽片段的连接.首先,使用MPAA作为硫醇试剂进行肽片段的连接,经过4~5 h的反应即可获得连接产物.但在随后的分离过程中发现,产物SUMO2的出峰位置与MPAA的出峰位置重合,调整分离梯度后依然无法将两者分开.为了解决这个问题,使用MTG作为反应的硫醇试剂进行肽片段的连接[24].经过5~6 h反应,获得了全长的SUMO2产物(产率45.6%),HPLC分析结果如图2所示,并且在后续的分离纯化过程中并未受到硫醇出峰的影响.
2.3 K 33⁃Ac⁃SUMO2的获得与HPLC表征
以MTG作为硫醇试剂,通过两片段多肽酰肼连接反应,获得了目标K33⁃Ac⁃SUMO2蛋白(产率39.7%),图3示出了HPLC分析结果.同样,使用MTG作为硫醇可使产物的分离较为顺利.

Fig.3 Ligation trace of K 33⁃Ac⁃SUMO2⁃1(A)and SUMO 2⁃2(B),and MS(C)of K 33⁃Ac⁃SUMO 2
2.4 SDS⁃PAGE分析
通过SDS⁃PAGE鉴定了SUMO和K33⁃Ac⁃SUMO蛋白是否具有正确的分子量.由图4可知,化学全合成的SUMO及K33⁃Ac⁃SUMO蛋白与生物表达的SUMO蛋白处在同一位置,且具有较好的纯度.
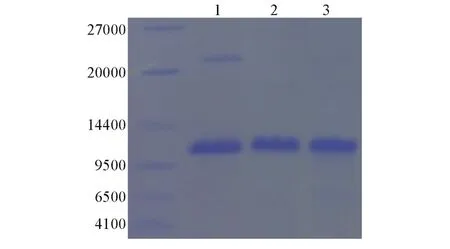
Fig.4 SDS⁃PAGE of expressed SUMO 2(1),syn⁃thesized SUMO2(2)and synthesized K 33⁃Ac⁃SUMO2(3)
2.5 圆二色光谱(CD)分析
利用圆二色光谱(CD)鉴定了SUMO蛋白和K33⁃Ac⁃SUMO蛋白是否具有正确的二级结构.将合成蛋白溶于水中使其自发折叠,调节浓度至0.5~1 mg/mL用于CD测试.由图5可知,化学全合成的SUMO蛋白和K33⁃Ac⁃SUMO蛋白与生物表达的SUMO蛋白在溶液中具有类似的二级结构.

Fig.5 CD spectra of exp ressed SUMO 2,synthe⁃sized SUMO2 and synthesized K 33⁃Ac⁃SU⁃MO2
3 结 论
采用高温辅助的Fmoc固相多肽合成方法,结合多肽酰肼连接技术以两片段高效合成了SUMO2蛋白与K33⁃Ac⁃SUMO2蛋白.通过与生物表达SUMO2的SDS⁃PAGE和CD数据进行分析比较,证明了合成蛋白具有较好的均一性与类似的二级结构.本文通过化学合成的策略获得了含有翻译后修饰的SUMO2蛋白,为研究乙酰化SUMO2与底物蛋白SIM的相互作用提供了重要的基础.除SUMO蛋白外,泛素蛋白也含有类似的乙酰化及磷酸化修饰[39,40],这些翻译后修饰发挥了重要的生理功能.该合成策略的成功应用也为高效获得含有类似翻译后修饰的泛素蛋白提供了有效的途径.
[1] Geoffroy M.C.,Hay R.T.,Nat.Rev.Mol.Cell Biol.,2009,10(8),564—568
[2] Flotho A.,Melchior F.,Annu.Rev.Biochem.,2013,82,357—385
[3] Bergink S.,Jentsch S.,Nature,2009,458(7237),461—467
[4] Van der Veen A.G.,Ploegh H.L.,Annu.Rev.Biochem.,2012,81,323—357
[5] Gartner A.,Muller S.,Mol Cell,2014,55(1),1—3
[6] Shen Q.Q.,Zeng M.Y.,Zhao Y.H.,Chem.J.Chinese Universities,2014,35(5),965—970(沈晴晴,曾明湧,赵元辉.高等学校化学学报,2014,35(5),965—970)
[7] Impens F.,Radoshevich L.,Cossart P.,Ribet D.,Proc.Natl.Acad.Sci.USA,2014,111(34),12432—12437
[8] Lamoliatte F.,Caron D.,Durette C.,Mahrouche L.,MarouiM.A.,Caron⁃Lizotte O.,Bonneil E.,Chelbi⁃Alix M.K.,Thibault P.,Nat.Commun.,2014,5,5409
[9] Hendriks I.A.,D′Souza R.C.,Chang J.G.,Mann M.,Vertegaal A.C.,Nat.Commun.,2015,6,7289
[10] Gareau J.R.,Lima C.D.,Nat.Rev.Mol.Cell Biol.,2010,11(12),861—871
[11] Wilkinson K.A.,Henley J.M.,Biochem.J.,2010,428(2),133—145
[12] Streich F.C.Jr.,Lima C.D.,Annu.Rev.Biophys.,2014,43,357—379
[13] Sriramachandran A.M.,Dohmen R.J.,Biochim.Biophys.Acta,2014,1843(1),75—85
[14] LiW.Z.,MengW.,Tian P.,Chem.Res.Chinese Universities,2015,31(1),149—155
[15] Kerscher O.,EMBO Rep.,2007,8(6),550—555
[16] Van Wijk S.J.,Muller S.,Dikic I.,Genes Dev.,2011,25(17),1763—1769
[17] Hickey C.M.,Wilson N.R.,Hochstrasser M.,Nat.Rev.Mol.Cell Biol.,2012,13(12),755—766
[18] Stehmeier P.,Muller S.,Mol.Cell,2009,33(3),400—409
[19] Chang C.C.,Naik M.T.,Huang Y.S.,Jeng J.C.,Liao P.H.,Kuo H.Y.,Ho C.C.,Hsieh Y.L.,Lin C.H.,Huang N.J.,Mol.Cell,2011,42(1),62—74
[20] Cappadocia L.,Mascle X.H.,Bourdeau V.,Tremblay⁃Belzile S.,Chaker⁃Margot M.,Lussier⁃Price M.,Wada J.,Sakaguchi K.,Aubry M.,Ferbeyre G.,Omichinski J.G.,Structure,2015,23(1),126—138
[21] Ullmann R.,Chien C.D.,Avantaggiati M.L.,Muller S.,Mol.Cell,2012,46(6),759—770
[22] Fang G.M.,Li Y.M.,Shen F.,Huang Y.C.,Li J.B.,Lin Y.,Cui H.K.,Liu L.,Angew.Chem.Int.Ed.,2011,50(33),7645—7649
[23] Xu B.F.,Yang S.,Zhu J.M.,Ma Y.D.,Zhao G.,Guo Y.,Xu L.,Chem.Res.Chinese Universities,2014,30(1),103—107
[24] Zheng J.S.,Tang S.,Huang Y.C.,Liu L.,Acc.Chem.Res.,2013,46(11),2475—1484
[25] Huang Y.C.,Fang G.M.,Liu L.,Natl.Sci.Rev.,2016,3(1),107—116
[26] Zheng J.S.,Tang S.,Qi Y.K.,Wang Z.P.,Liu L.,Nat.Protoc.,2013,8(12),2483—2495
[27] Huang Y.C.,Li Y.M.,Chen Y.,Pan M.,Li Y.T.,Yu L.,Guo Q.X.,Liu L.,Angew.Chem.Int.Ed.,2013,52(18),4858—4862
[28] Huang Y.C.,Chen C.C.,Gao S.,Wang Y.H.,Xiao H.,Wang F.,Tian C.L.,Li Y.M.,Chem.Eur.J.,2016,22(22),7623—7628
[29] Chen C.C.,Huang Y.C.,Xu L.,Zhen Y.,Xu H.J.,Guo Q.X.,Tian C.L.,Li Y.M.,Shi J.,Org.Biomol.Chem.,2014,12(46),9413—9418
[30] Chen C.C.,Li S.J.,Chen Y.Q.,Xu H.J.,Li Y.M.,Chin.J.Org.Chem.,2014,37(7),1452—1457
[31] Kent S.B.H.,Chem.Soc.Rev.,2009,38(2),338—351
[32] Huang Y.C.,Liu L.,Sci.China Chem.,2015,58(12),1779—1781
[33] Dawson P.E.,Muir T.W.,Clarklewis I.,Kent S.B.H.,Science,1994,266(5186),776—779
[34] Fang G.M.,Wang J.X.,Liu L.,Angew.Chem.Int.Ed.,2012,51(41),10347—10350
[35] Huang Y.C.,Guan C.J.,Tan X.L.,Chen C.C.,Guo Q.X.,Li Y.M.,Org.Biomol.Chem.,2015,13(5),1500—1506
[36] Tang S.,Si Y.Y.,Wang Z.P.,Mei K.R.,Chen X.,Cheng J.Y.,Zheng J.S.,Liu L.,Angew.Chem.Int.Ed.,2015,54(19),5713—5717
[37] Wang J.X.,Fang G.M.,He Y.,Qu D.L.,Yu M.,Hong Z.Y.,Liu L.,Angew.Chem.Int.Ed.,2015,54(7),2194—2198
[38] Pan M.,Gao S.,Zheng Y.,Tan X.L.,Lan H.,Tan X.D.,Sun D.,Lu L.,Wang T.,Zheng Q.,Huang Y.,Wang J.W.,Liu L.,J.Am.Chem.Soc.,2016,138(23),7429—7435
[39] Wauer T.,Simicek M.,Schubert A.,Kmomander D.,Nature,2015,524(7565),370—374
[40] Ohtake F.,Saeki Y.,Sakamoto K.,Ohtake K.,Nishikawa H.,Tsuchiya H.,Ohta T.,Tanaka K.,Kanno J.,EMBO Rep.,2015,16(2),192—201
Chem ical Synthesis of K33 Acetylated SUMO Protein†
WANG Yehai1,KONG Yifu1,CHEN Chenchen2,LIYiming1∗
(1.School ofBiological and Medical Engineering,Hefei University of Technology,Hefei230009,China;2.High Magnetic Field Laboratory of the Chinese Academy ofSciences,Hefei230031,China)
Sumoylated protein was found to reduce the protein binding force with the SUMO⁃interaction motif (SIM)of effector protein when K33 of SUMO was acetylated.To further study the structures and mechanism,a large amount of uniformity acetylated SUMO protein was greatly needed.In this work,SUMO protein and K33 acetylated SUMO protein was efficiently obtained using high temperature assisted solid⁃phase peptide syn⁃thesis(SPPS)technology combined with peptide hydrazide ligation.SUMO and K33 acetylated SUMO was identified with corrected molecularweightby the SDS⁃PAGE.Itwas also confirmed that the chemically synthe⁃tic protein and biologically expressed SUMO2 own similar homogeneity and secondary structure by the CD analysis.These results provided a foundation for the follow⁃up study of K33 acetylated SUMO protein.
Small ubquitin⁃related modifier;Acetylation;High temperature assisted solid⁃phase peptide syn⁃ thesis;Peptide hydrazide;Native chemical ligation
O629
A
10.7503/cjcu20160417
(Ed.:P,H,W,K)
†Supported by the National Natural Science Foundation of China(Nos.21372058,21572043).
2016⁃06⁃12.
日期:2016⁃10⁃19.
国家自然科学基金(批准号:21372058,21572043)资助.
联系人简介:李宜明,男,博士,副教授,主要从事翻译后修饰蛋白的化学合成、结构及功能方面的研究. E⁃mails:lym2007@mail.ustc.edu.cn;ymli@hfut.edu.cn
