Y220C突变体影响p53C蛋白质构象转换的分子动力学模拟
2016-11-22沈洪辰丁吉勇刘夫锋
沈洪辰 丁吉勇 李 丽 刘夫锋,3,*
(1天津大学化工学院生物工程系,天津300072;2天津科技大学海洋与环境学院,天津300457;3天津科技大学生物工程学院,天津300457)
Y220C突变体影响p53C蛋白质构象转换的分子动力学模拟
沈洪辰1丁吉勇1李丽2刘夫锋1,3,*
(1天津大学化工学院生物工程系,天津300072;2天津科技大学海洋与环境学院,天津300457;3天津科技大学生物工程学院,天津300457)
p53是迄今发现突变频率最高的一种肿瘤抑制蛋白质,突变会导致p53抑癌功能丧失并诱导癌症的发生。绝大多数的突变发生在p53的核心DNA结合区域(p53C),其中Y220C是研究较多的一种突变体。虽然已有研究表明该突变能够降低p53C的结构稳定性,但其影响p53C构象转换的分子机制尚不清晰。本文利用分子动力学(MD)模拟方法研究了p53C突变体Y220C(p53C-Y220C)的结构变化,发现Y220C突变主要影响Y220C cluster区域(包括残基138-164和215-238),且Y220C突变减少了Y220C cluster的β-折叠含量。进一步分析发现,Y220C突变不仅直接破坏突变氨基酸与周围氨基酸Leu145和Thr155之间的氢键,而且降低了Y220C cluster区域的折叠片S3和S8之间的氢键数量,使Y220C突变所形成的亲水性空腔变大,加速了水分子进入该蛋白质内部,并最终导致了p53C-Y220C变性。MD模拟结果揭示了Y220C突变影响p53C结构转换的分子机制,该研究对p53C-Y220C突变体高效稳定剂的筛选和设计具有重要意义。
癌症;p53;残基突变;构象转换;分子动力学模拟
1 引言
p53是一种重要的肿瘤抑制蛋白质。p53通过与DNA或其他蛋白质(如MDM2等1)发生相互作用从而在细胞生长周期的调控、细胞分化和细胞凋亡等生命过程中发挥着重要的作用2。因此p53具有阻滞肿瘤细胞周期,促进肿瘤细胞凋亡以及抑制肿瘤血管生成等多种抑癌功能。人类p53蛋白质含有393个氨基酸残基,其中94-292位氨基酸为该蛋白质的核心结构域,被定义为p53C3。据统计,约50%的人类癌症是由p53的残基突变引起的,其中90%以上的突变发生在p53C区域4。p53C的突变种类众多,基于尿素变性的折叠-去折叠平衡实验和低温条件下的DNA结合实验结果,Bullock等5将常见的p53突变体分成以下四类:DNA接触类(如R273H等),DNA结合区域(如R282W,G245S和R249S等),锌离子结合区域(如M237I,R175H和C242S等)和β-三明治区域(如V157F,F270C和Y220C等)。在最易引发癌症的p53突变中,Y220C突变排在第9位。另据世界卫生组织统计数据显示,由Y220C突变体所引发的癌症病例每年约7.5万。随着世界癌症发病率的增加,预计到2020年每年由Y220C突变所引发的病例将增加至每年10万以上6。图1所示为p53C-Y220C突变体的三维结构。从图1a可以看出,p53C包含10个β-折叠片(S1-S10)、5个环(L1-L3,S3/S4和S7/S8 loop)和2个螺旋结构(H1-H2)。位于S3/S4环和S7/ S8环之间的220位酪氨酸突变成半胱氨酸会在p53C表面形成一个空腔,如图1b所示。更重要的是Y220C突变会大大降低p53C的热稳定性(约16.75 kJ·mol-1)。但Y220C突变所引起的p53CY220C构象转换的作用机制尚不清晰,这严重影响了Y220C突变体稳定剂的虚拟筛选和设计。
由于蛋白质构象转换非常迅速,利用目前的实验技术很难从原子和分子角度检测蛋白质的构象转换,从而导致Y220C突变所引起的构象转换至今仍然无法用现有的实验方法进行研究。然而分子动力学(MD)模拟方法的出现弥补了现有实验研究的不足,并已广泛用于蛋白质折叠7、去折叠8和聚集9等领域的研究中。MD模拟方法也已用于p53构象转换的相关研究10。例如,Espinoza-Fonseca等11利用MD模拟方法研究了p53蛋白质N-末端的F19-L22螺旋区的结构稳定性。Calhoun和Daggett12利用MD模拟研究了L145Q、V157F和R282W突变对p53C结构的影响,发现L145Q和V157F会破坏β-折叠片的结构和环-折叠-螺旋模式,而R282W突变会使环-折叠-螺旋模式扭曲。且这些突变体均会使DNA结合区域的结构重排,从而影响p53结合DNA的活性,并最终降低了p53的抑癌功能。许朝莹等13利用全原子MD模拟研究了常见的3个残基突变R249S、R248W和G245S对p53的DNA结合域肽段230-258的结构影响。上述研究的突变体主要集中于DNA结合区域。与之相反,Y220C突变正好远离DNA结合区域。虽然已有研究结果表明Y220C突变能够降低该蛋白质稳定性并开发了多种能够稳定该突变体的稳定剂分子,如他克林14、PhiKan08315和PK51744等。但该突变影响p53C构象转换的分子机制尚不清晰,这严重阻碍了该突变体高效稳定剂的开发。因此利用MD模拟来研究Y220C突变所引起的蛋白质构象转换的分子机制对于进一步理性设计p53CY220C突变体的稳定剂具有非常重要的理论指导作用。
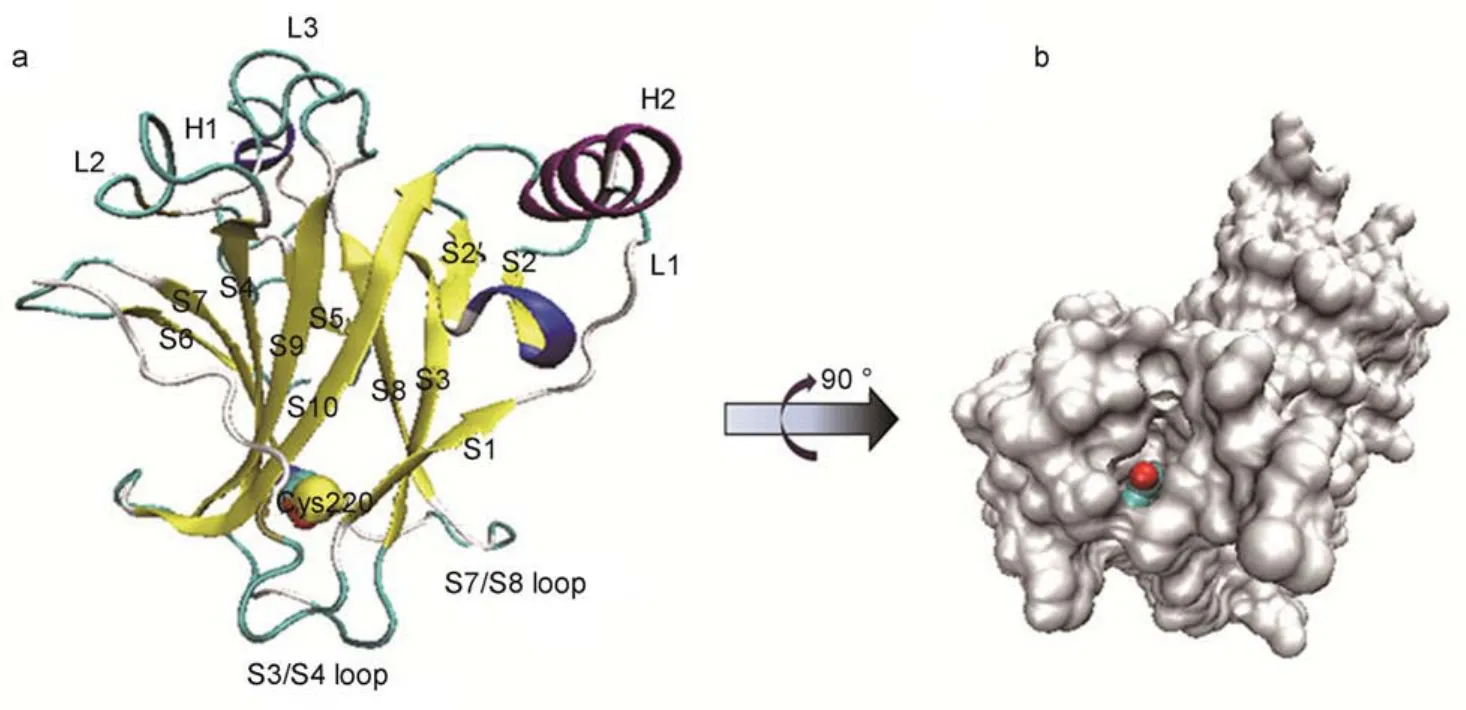
图1 p53C突变体Y220C的三维结构Fig.1 3D structure of p53C-Y220C
本研究利用全原子MD模拟方法研究了p53CY220C的构象转换。首先分析了Y220C突变对其周围区域结构的影响,并确定了Y220C突变所影响的结构区域;然后分析了Y220C突变对该区域二级结构和氢键作用力的影响;最后研究了Y220C突变对蛋白质表面疏水性的影响。该研究不仅解释了Y220C突变影响p53C构象转换的作用机理,而且为Y220C突变体稳定剂的设计和筛选奠定了坚实的理论基础。
2 实验部分
2.1模拟体系
蛋白质p53C-Y220C的晶体结构从蛋白质结构数据库(PDB)获得(http://www.rcsb.org/pdb/home/ home.do),它的PDB ID为2J1X16。本研究选取了该晶体结构中的B链作为p53C-Y220C的初始模拟结构。考虑到野生型p53C蛋白质的稳定性较差,从不同的溶液条件下获得的p53C的结构均有较大的差异。因此,选择不同实验条件下野生型p53C的结构作为野生型对照,可能会给后续分析引入较大的误差。因此,本研究在突变体p53C-Y220C结构基础上利用SYBYL 6.92软件将220位残基半胱氨酸突变成酪氨酸,然后利用全原子MD模拟优化后的稳定结构作为野生型p53C的初始结构。并以此为对照,以期从原子和分子角度解析Y220C突变对p53C结构造成的影响。
2.2分子动力学模拟
所用MD模拟均采用GROMACS 4.5软件进行17,水分子采用SPC模型18。首先将p53C-Y220C置于一个边长为8 nm的立方体盒子中心,并使蛋白质的每个原子与盒子之间的距离均大于1.0 nm。向盒子中随机加满水分子,并用5个Cl-离子替代5个水分子以使模拟体系为电中性。随后用两次1000步的能量最小化模拟优化该体系:首先固定蛋白质的结构不变,仅让水分子的结构和位置变化,随后使所有的分子都可以自由运动;然后利用限制性MD模拟分别在正则和等温等压系综下平衡该体系;这两步MD模拟的主要目的是优化目标蛋白质与溶剂和Cl-之间的相互作用,从而使该模拟体系达到最优值。最后将完全优化好的体系进行MD模拟。
本研究采用经典的Gromos96 53a6力场研究p53C-Y220C的构象转换19,采用Verlet蛙跳算法求解牛顿运动方程,积分步长设为2 fs。计算过程中利用Lennard-Jones函数计算范德华作用力,非键截断距离设为1.4 nm;用LINCS算法约束所有原子的键长20,利用particle mesh Ewald(PME)方法计算长程静电相互作用21,格点宽度设为0.12 nm。模拟过程中采用周期性边界条件。所有MD模拟均在等温等压系综下进行,温度为310 K,压力为1大气压,分别通过V-rescale22和Parrinello-Rahman23方法控制温度和压力,温度和压力耦合常数分别为0.1和0.5 ps。MD模拟共做三次平行实验,每次模拟时间为100 ns。所有MD模拟计算均在曙光TC2600刀片服务器(每刀片包括4路4核的AMD Opteron 8347HE CPU和8G内存)上完成(Dawning, Tianjin,China)。
2.3数据分析方法
p53C及其突变体中Cα原子的均方根偏差(Cα-RMSD)和均方根涨落(RMSF)以及整个蛋白质的溶剂可及表面积(SASA)由GROMACS软件自带的g_rms、g_rmsf和g_sas程序分别计算。采用DSSP软件分析p53C及其突变体在模拟过程中的二级结构变化,该过程使用GROMACS软件自带的do_dssp程序计算。蛋白质分子内的氢键数量利用GROMACS软件自带的g_hbond程序计算。氢供体和受体之间的距离截断值设定为0.35 nm,受体-供体-氢原子之间的角度截断值设定为30°。本文所有蛋白质结构的典型构象均使用视觉分子动力学(VMD)软件绘制(http://www.ks.uiuc.edu/Research/ vmd/)24。
3 结果与讨论
3.1Y220C突变对蛋白结构稳定性的影响
为了研究Y220C突变对p53C蛋白质结构稳定性的影响,首先计算了野生型p53C及其突变体p53C-Y220C的Cα-RMSD在100 ns模拟时间内的变化,结果如图2所示。RMSD数值常被用来表征模拟过程中某时刻的蛋白质结构与初始结构之间的差异。RMSD值越大,说明此模拟时刻蛋白质的结构与初始结构之间的偏差越大。因此,蛋白质的Cα-RMSD值是衡量模拟过程中蛋白质构象转换的重要依据。从图2可以看出,野生型p53C的Cα-RMSD数值在10 ns内急剧上升到0.3 nm,并经过缓慢的上升,在最后20 ns内趋于稳定。而p53CY220C的Cα-RMSD数值的变化趋势与野生型的基本一致。在10-45 ns内,p53C-Y220C的Cα-RMSD数值比野生型的还要小,仅在最后50 ns内突变体的Cα-RMSD数值比野生型的稍大(图2)。因此,仅采用Cα-RMSD的数值很难得出p53C的稳定性高于突变体p53C-Y220C的结论,这与实验结果是不一致的。大量的研究结果表明,Y220C会使p53C的稳定性大大降低16。究其原因,主要是野生型p53C的结构也不稳定,这导致在MD模拟过程中野生型的结构变化也较大,从而利用整个蛋白质的Cα-RMSD数值并不能准确反映蛋白质的稳定性。
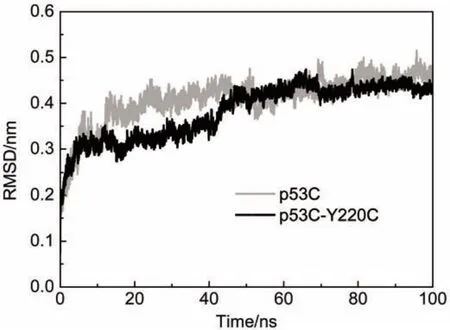
图2 p53C和p53C-Y220C的Cα原子均方根偏差随模拟时间的变化Fig.2 Root-mean-square deviations(RMSD)for Cαatoms of p53C and p53-Y220C as a function of time for the simulations
从前面的分析可知,利用整个蛋白质Cα-RMSD值很难表征野生型p53C和Y220C突变体的结构稳定性,因此推测该突变可能仅影响该蛋白质的部分结构区域。为了进一步确定Y220C突变所影响的区域,本研究计算了整个蛋白质Cα-RMSF值。该数值的大小代表蛋白质中Cα原子在模拟过程中构象变化的程度。Cα原子的RMSF数值大表示该原子在模拟过程中的构象变化较大;与之相反,则表示该原子所属结构在模拟过程中比较稳定。图3所示为两种蛋白质的Cα-RMSF数值。从图3可以看出,p53C-Y220C的S7和S8区域(分别对应残基215-220和233-238)的Cα-RMSF数值变化较大,明显高于野生型p53C。从图1可以看出S7和S8区域都临近突变点,因此可以推测这些结构变化均是由于Y220C突变所导致的。另外,从图1a可以看出残基Y220主要处于S3/S4环与S7/S8环之间。因此,本研究将S3、S4、S7、S8以及连接这4个β-折叠结构的环(S3/S4环和S7/S8环)定义为Y220C cluster,该结构域包括138-164和215-238位氨基酸残基。虽然H1/S5之间和S8/S9之间环结构的Cα-RMSF数值的变化也比较大,但这可能是因为这部分结构是无规卷曲,对结构变化更敏感从而导致该区域的Cα-RMSF数值发生较大改变的缘故,因此在后期的研究中不予考虑!
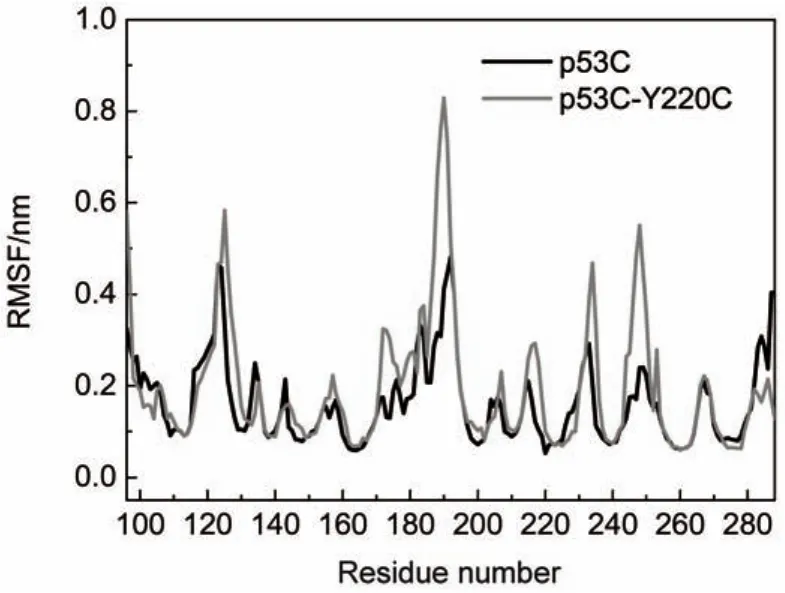
图3 分子动力学模拟过程中p53C和p53C-Y220C的Cα原子的均方根涨落Fig.3 Root mean square fluctuation(RMSF)for Cαatom per residue for p53C and p53C-Y220C during the MD simulations
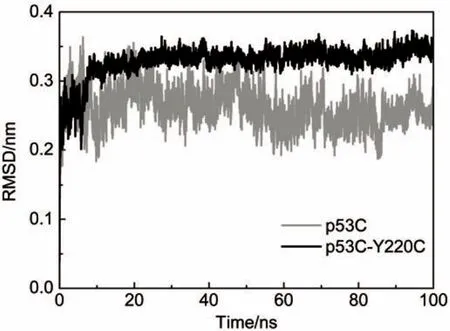
图4 p53C及其突变体Y220C中Y220C cluster的Cα原子的均方根偏差的数值随模拟时间的变化Fig.4 RMSD for Cαatom of Y220C cluster of p53C and p53C-Y220C as a function of time for the simulations
图4为野生型p53C及其Y220C突变体的Y220C cluster的Cα-RMSD数值随模拟时间的变化。从图4可以看出,在10 ns内野生型p53C和Y220C突变体中Y220C cluster的Cα-RMSD的增长趋势和大小基本相同。但在随后的90 ns内p53CY220C的Cα-RMSD数值均比野生型p53C高。这说明Y220C突变主要影响了Y220C cluster区域的稳定性,而对蛋白质其它结构的稳定性影响较小。因此,在下面分析中仅考虑了Y220C突变对Y220C cluster区域的二级结构、氢键和蛋白质表面疏水性的影响。
3.2Y220C突变对Y220C cluster区域二级结构的影响
为了进一步研究Y220C突变对Y220C cluster构象的影响,首先利用GROMACS软件中自带的do_dssp程序分析了Y220C cluster的二级结构变化,结果如图5和表1所示。图5a所示为野生型p53C的Y220C cluster的二级结构随模拟时间的变化。从图5a可以看出,Y220C cluster的二级结构在100 ns内的模拟中变化很小,其中S3、S4、S7和S8均保持为初始的β-折叠结构,基本没有变化。Y220C cluster的β-折叠含量在100 ns模拟后为42.9%,稍高于初始的40.8%(表1)。只有S3/S4和S7/S8连接部分的二级结构在模拟过程中发生了轻微的结构转换(图5a)。图5b所示为p53C-Y220C突变体的Y220C cluster的二级结构随模拟时间的变化。如图5b所示,当Tyr突变成Cys后,突变体中一些β-折叠结构遭到破坏,如S3、S4、S7和S8的β-折叠结构的含量随着模拟时间的延长逐渐降低。从表1可以看出,突变体p53C-Y220C中Y220C cluster的β-折叠含量从最初的40.8%降低到100 ns后的32.7%,而无规卷曲结构则从初始的42.9%增加到49%。因此,Y220C突变会破坏Y220C cluster中的β-折叠结构。众所周知,β-折叠是组成β-三明治的最主要结构,β-折叠的破坏是整个p53C构象转换的主要步骤之一。
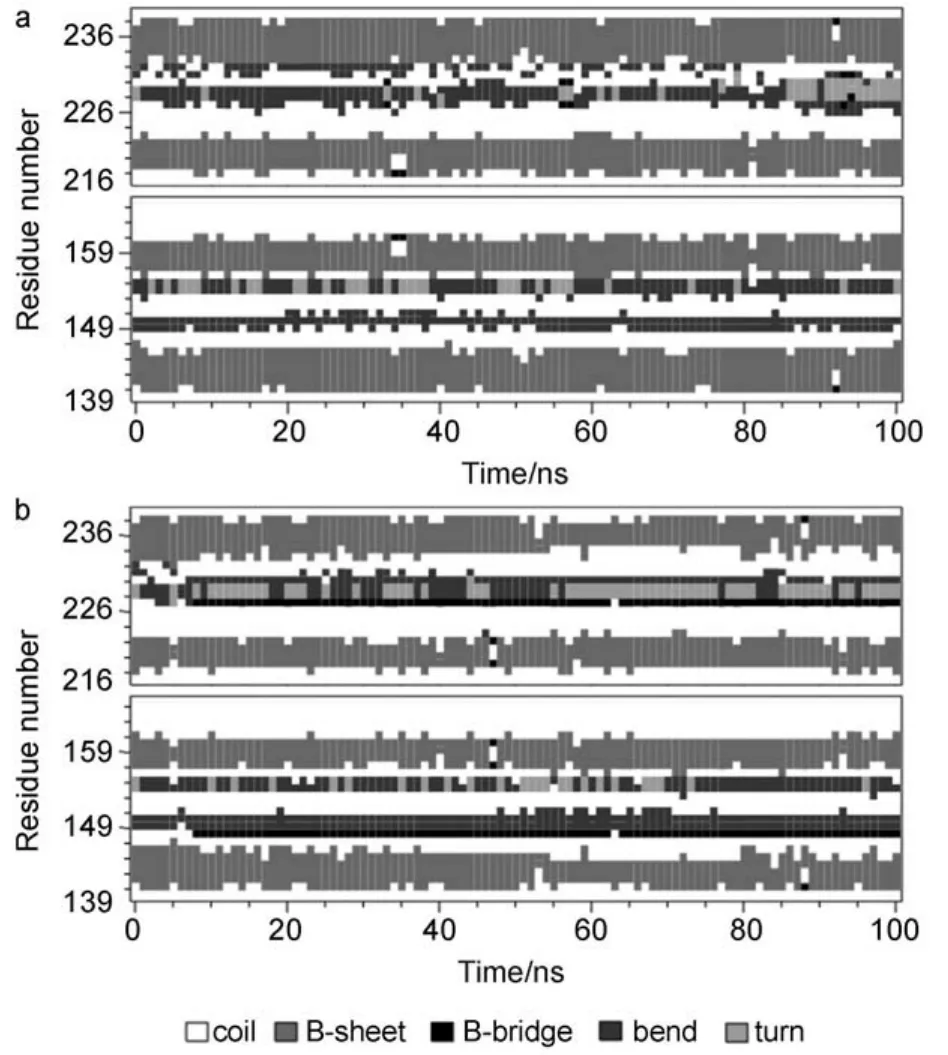
图5 p53C(a)和p53C-Y220C(b)中Y220C cluster二级结构随模拟时间的变化Fig.5 Secondary structure of Y220C cluster as a function of simulation time for p53C(a)and p53C-Y220C(b)
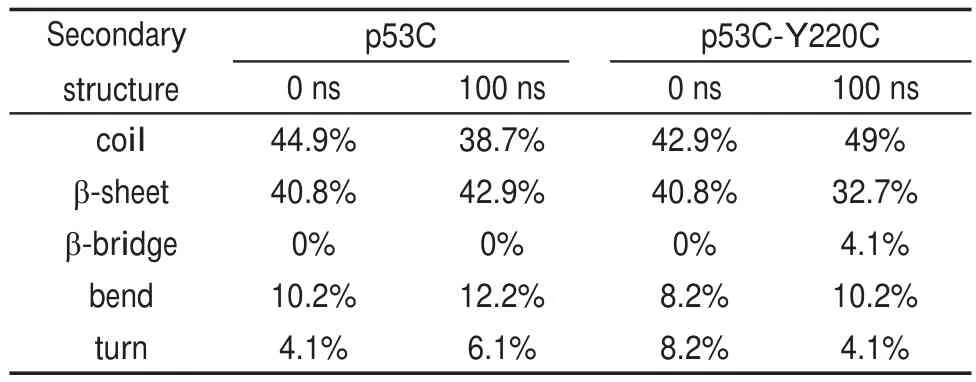
表1 p53和突变体p53-Y220C中Y220C cluster的二级结构含量Table 1 Content of secondary structures of Y220C cluster in p53C and p53C-Y220C
3.3Y220C突变对Y220C cluster区域中氢键作用力的影响
从前面的二级结构分析可以看出,Y220C cluster的二级结构主要是β-折叠片层结构。前期研究表明,氢键是β-折叠结构中主要的作用力之一,它在β-折叠的结构稳定过程中发挥了重要作用。此外,野生型p53C中的220位酪氨酸残基含有一个酚羟基,它可以和周围的基团形成氢键。而突变后的半胱氨酸残基的侧链仅含有一个亚甲基和一个巯基,这些基团均不能形成氢键作用。图6为100 ns模拟时间内野生型p53C的220位残基突变前后与其它残基之间形成的氢键随模拟时间的变化。从图6可以看出野生型p53C的Tyr220可以与附近的6个氨基酸残基形成10个不同的氢键。如图6a所示,Tyr220-Leu145和Tyr220-Thr155之间形成的两个氢键稳定性均较高,在模拟过程中90%以上的时间内均能够形成氢键。另外,Tyr220-Thr230在34%模拟时间内能够形成氢键。而另外7个氢键的稳定性均较差。残基Leu145和Thr155分别位于S3和S4上,因此这两个氢键可以使S7/S8环和S3/S4环之间发生较强的相互作用。从图7a可以看出Tyr220残基正好位于S3和S4之间,Tyr220能够和Thr155和Leu145分别形成1个氢键从而稳定S3和S4的二级结构。因此,Tyr220对整个Y220C cluster的稳定性有非常重要的作用。当Tyr220突变成Cys后,该残基仅能与周围的残基形成4个氢键(图7b),远小于Tyr220与周围残基形成的10个氢键(图7a)。从图7b可以看出Cys220仅能与Thr155形成1个稳定的氢键。由于Tyr220突变成Cys后,极大降低了该残基与Y220C cluster周围残基之间的氢键作用力,从而使Y220C cluster的稳定性急剧下降。

图6 野生型p53C(a)和p53C-Y220C(b)中220位氨基酸残基在模拟100 ns内形成的氢键Fig.6 Hydrogen bonds interaction of Tyr220 with surrounding residues in p53C(a)and Cys220 with surrounding residues in p53C-Y220C(b)in the 100 ns MD simulations
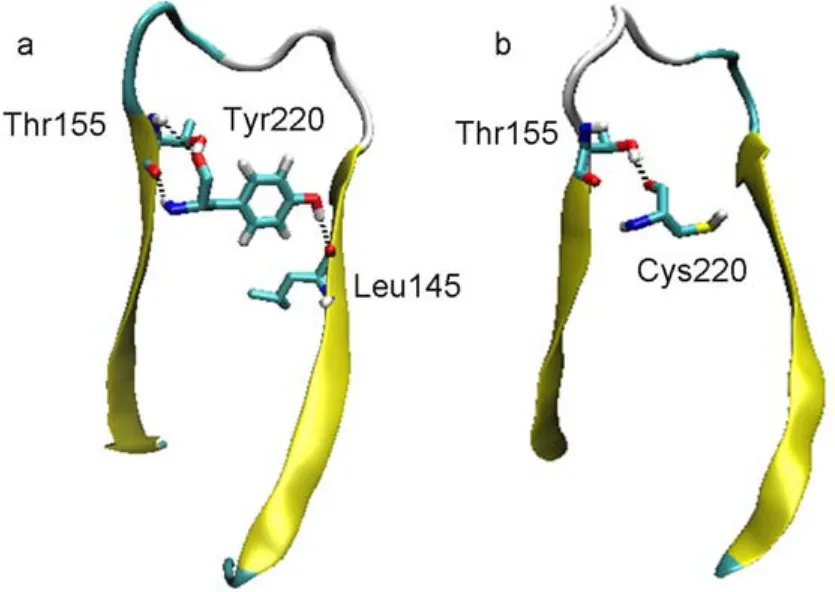
图7 (a)p53C的Tyr220和(b)p53C-Y220C的Cys220氨基酸和它们周围氨基酸之间的氢键分析Fig.7 Hydrogen bond analyses of Tyr220 with surrounding residues in p53C(a)and Cys220 with surrounding residues in p53C-Y220C(b)
从上面的分析可以得出,由于Y220C突变使S7/S8环稳定性降低,所以当S7/S8环位置发生变化时也会影响与之连接的S8结构,从而导致与S8相邻的S3之间的氢键作用遭到部分破坏。图8所示为100 ns内S3与S8之间形成的氢键数量随模拟时间的变化。从图8可以看出,野生型p53C中S3与S8在60-100 ns之间均有氢键,形成的氢键数量一直在4-8左右波动。然而,在Y220C突变体中S3与S8形成的氢键数随着时间的变化呈现下降趋势,在最后的40 ns内形成的氢键数量在2-5之间波动。图9为100 ns时p53C和p53C-Y220C中S3与S8形成氢键的典型构象。从图9a可以看出,野生型p53C中S3与S8之间形成7个氢键,主要是S3和S8中的主链氨基上的氢原子供给羧基上的氧原子而形成的氢键,这是β-折叠结构中最为常见的氢键形式25。当220位残基Y突变成C后,Y220C突变体的S3与S8之间的氢键数仅剩下中间的4个,而N235与L139、T230与L145之间的3个氢键在模拟过程中消失(图9),从而破坏了S3与S8之间的氢键作用力并降低了Y220C cluster的β-折叠的含量(表1)。上述结果说明了S7/S8环结构的不稳定会进一步影响与之相连的区域,这也证明了Y220C突变会使Y220C cluster整个结构受到破坏。在其它导致p53C稳定性降低的突变体中,也存在这种β-折叠片层之间氢键遭到破坏的现象,如L145Q和V157F12。由于β-三明治结构是p53C的骨架结构,所以这种破坏是导致p53-Y220C不稳定的重要因素之一。
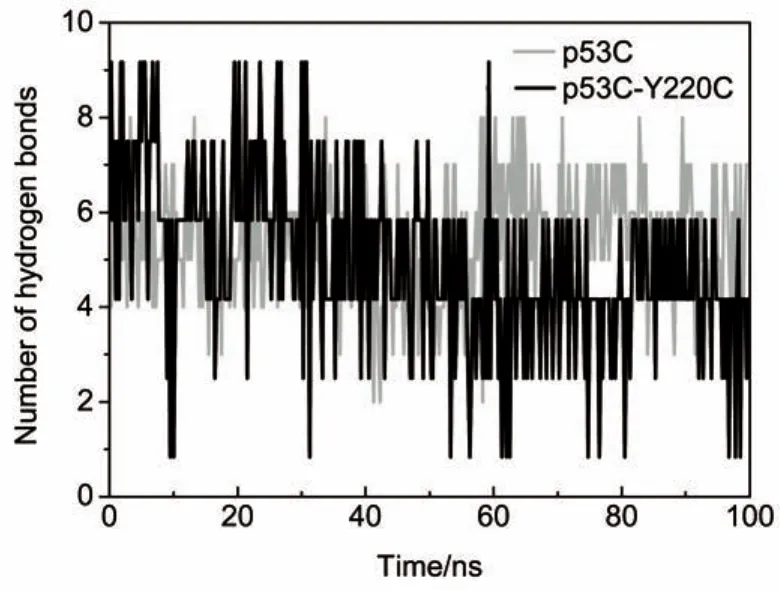
图8 野生型p53C和p53C-Y220C突变体中的S3与S8形成氢键个数随时间变化Fig.8 Numbers of H-bonds between S3 and S8 of p53C and p53C-Y220C as functions of simulation time

图9 模拟100 ns过程中p53C(a)和p53C-Y220C(b)中的S3与S8的主链之间的氢键示意图Fig.9 Scheme of hydrogen bonds between backbones of S3 and S8 for p53C and p53C-Y220C during the 100 ns MD simulation
3.4Y220C突变对蛋白表面疏水性的影响
野生型p53C的Tyr220残基的苯环位于蛋白质的疏水表面,而突变后的Cys是一个亲水型残基,且Cys的体积比Tyr的要小,因此Y220C突变会在蛋白质表面形成一个亲水空腔。为了进一步分析Y220C突变对蛋白质表面性质的影响,我们又计算了Y220C cluster的溶剂可及表面积。SASA是指使用半径为0.14 nm的溶剂分子在蛋白质的范德华表面滚动时其球心所经过的曲面面积。蛋白质在折叠过程中疏水基团倾向于远离溶剂而埋藏在蛋白质内部,因此通过对蛋白质的疏水性和亲水性SASA的计算可以获得蛋白质表面水分子的分布情况。若亲水性可及表面积随着模拟时间的增加而增加就表示蛋白质表面的亲水性空腔面积增加而更使空腔中的水分子的数量增加。否则,就表示蛋白质将空腔中的水分子外排而发生了塌缩。从图10可以看出,在模拟100 ns内,p53C-Y220C的Y220C cluster的亲水区域SASA要高于野生型p53C,疏水区域的SASA则恰好相反。位于p53C表面的Tyr220残基上的苯环基团是疏水的,其可以阻止水分子进入蛋白质内部;而当Tyr220突变成Cys后,Y220C cluster的亲水SASA大于野生型的。因此由于Y220C突变在蛋白表面形成的亲水性空腔容易结合水分子,使水分子能够进入突变所形成的亲水空腔内部。而且Y220C突变降低了Y220C cluster之内的氢键,从而使整个蛋白结构变的松散,暴露出了更多内部的亲水基团。最终导致p53C-Y220C构象转换和变性。

图10 p53C和p53C-Y220C中Y220C cluster在模拟100 ns内亲水和疏水SASA随模拟时间的变化Fig.10 Values of hydrophilic and hydrophobic solvent accessible surface area(SASA)of Y220C cluster as a function of simulation time for p53C and p53C-Y220C during the 100 ns MD simulation
上述研究结果表明Y220C突变会使Y220C Cluster的β-折叠含量和氢键减少以及亲水性空腔变大。这些研究结果均可为后续的p53C-Y220C稳定剂的开发和设计提供一定的理论基础。在设计稳定剂过程中应该考虑使Y220C稳定剂分子和220位氨基酸残基周围氨基酸(如,L145、V147、T150、D228、L257等)之间形成较强的作用力(如氢键等),从而弥补Y220C突变对p53C-Y220C作用力的减弱。例如,Basse等26设计得到的Phikan083能够和D228形成一个氢键,同时能够结合到Y220C突变所形成的空腔中从而抑制该突变体构象转换。另外,在设计和开发p53C-Y220C稳定剂时,也要考虑突变体和稳定剂结合后应该能够降低Y220C突变所引起的亲水性空腔的体积。这些均为后期Y220C突变体稳定剂的开发奠定了坚实的理论基础。
根据上述研究结果,可以推断上述构象和作用力变化的主要原因为Y220C突变破坏了220位氨基酸与它附近残基之间的作用力,尤其是破坏了220位残基与它周围氨基酸残基之间的氢键相互作用,从而引起Y220C cluster以及整个蛋白质的构象转换。因此,将Y220突变成其它氨基酸是否同样会引起上述构象转换尚不清楚。这可能主要取决于突变后的氨基酸能否替代酪氨酸继续与周围的氨基酸形成较强的相互作用。若突变引起220位氨基酸残基与其周围的残基之间的作用力明显减弱可能就会引起p53C的结构发生改变,否则就可能对突变体的三维结构影响不大。后续研究应该继续利用全原子MD模拟方法系统研究Y220突变成其它氨基酸对p53C构象的影响,从而为详细了解Y220C影响p53C构象转换提供更多的信息。
4 结论
利用全原子MD模拟研究了野生型p53C和p53C-Y220C的构象转换,Cα-RMSD结果表明Y220C突变对于整个蛋白质的稳定性影响不大。进一步利用Cα-RMSF分析发现,Y220C突变主要影响其突变位点附近的区域,主要包含138-164和215-238位氨基酸残基,将其命名为Y220C cluster。二级结构分析表明Y220C突变会降低Y220C cluster的β-折叠含量。Y220C突变不仅降低了该残基与周围残基所形成的氢键相互作用,而且降低了Y220C cluster的折叠片S3和S8之间的氢键数量。Y220C突变破坏了Y220C cluster的β-折叠结构,最终使整个β-三明治结构瓦解。并进一步使Y220C突变所形成的亲水空腔变大,加速了水分子进入该蛋白质内部,最终加速了p53C的变性和聚集。
References
(1)Sheng,R.;Hu,C.Q.;Huang,W.H.;Hu,Y.Z.Acta Phys.-Chim. Sin.2007,23,1815.[盛荣,胡纯琦,黄文海,胡永洲.物理化学学报,2007,23,1815.]doi:10.3866/PKU.WHXB20071131
(2)Duffy,M.J.;Synnott,N.C.;McGowan,P.M.;Crown,J.; O′Connor,D.;Gallagher,W.M.Cancer Treat.Rev.2014,40 (10),1153.doi:10.1016/j.ctrv.2014.10.004
(3) Okorokov,A.L.;Orlova,E.V.Curr.Opin.Struct.Biol.2009, 19(2),197.doi:10.1016/j.sbi.2009.02.003
(4)Bykov,V.J.;Wiman,K.G.FEBS Lett.2014,588(16),2622. doi:10.1016/j.febslet.2014.04.017
(5) Bullock,A.N.;Henckel,J.;Fersht,A.R.Oncogene 2000,19 (10),1245.doi:10.1038/sj.onc.1203434
(6) Petitjean,A.;Mathe,E.;Kato,S.;Ishioka,C.;Tavtigian,S.V.; Hainaut,P.;Olivier,M.Hum.Mutat.2007,28(6),622. doi:10.1002/humu.20495
(7) Liu,F.F.;Dong,X.Y.;Sun,Y.Acta Phys.-Chim.Sin.2010,26, 1643.[刘夫锋,董晓燕,孙彦.物理化学学报,2010,26, 1643.]doi:10.3866/PKU.WHXB20100613
(8) Zhang,N.;Liu,F.F.;Dong,X.Y.;Sun,Y.J.Phys.Chem.B 2012,116(24),7040.doi:10.1021/jp300171h
(9) Liu,F.F.;Ji,L.;Dong,X.Y.;Sun,Y.J.Phys.Chem.B 2009, 113(32),11320.doi:10.1021/Jp905580j
(10)Ng,J.W.K.;Lama,D.;Lukman,S.;Lane,D.P.;Verma,C.S.; Sim,A.Y.L.Proteins 2015,83(12),2240.doi:10.1002/ prot.24940
(11) Espinoza-Fonseca,L.M.;Trujillo-Ferrara,J.G.Biochem. Biophys.Res.Commun.2006,343(1),110.doi:10.1016/j. bbrc.2006.02.129
(12) Calhoun,S.;Daggett,V.Biochemistry 2011,50(23),5345. doi:10.1021/bi200192j
(13) Xu,Z.Y.;Zhao,L.L.;Cao,Z.X.;Wang,J.H.Acta Phys.-Chim. Sin.2012,28,1665.[许朝莹,赵立岭,曹赞霞,王吉华.物理化学学报,2012,28,1665.]doi:10.3866/PKU.WHXB201204182
(14) Ding,J.Y.;Shen,H.C.;Liu,F.F.Chem.J.Chin.Univ.2016,37 (4),706.[丁吉勇,沈洪辰,刘夫锋.高等学校化学学报,2016, 37(4),706.]doi:10.7503/cjcu20150790
(15) Boeckler,F.M.;Joerger,A.C.;Jaggi,G.;Rutherford,T.J.; Veprintsev,D.B.;Fersht,A.R.Proc.Natl.Acad.Sci.U.S.A. 2008,105(30),10360.doi:10.1073/pnas.0805326105
(16) Joerger,A.C.;Ang,H.C.;Fersht,A.R.Proc.Natl.Acad.Sci. U.S.A.2006,103(41),15056.doi:10.1073/pnas.0607286103
(17) Pronk,S.;Pall,S.;Schulz,R.;Larsson,P.;Bjelkmar,P.; Apostolov,R.;Shirts,M.R.;Smith,J.C.;Kasson,P.M.;van der Spoel,D.;Hess,B.;Lindahl,E.Bioinformatics 2013,29(7), 845.doi:10.1093/bioinformatics/btt055
(18) Berendsen,H.J.C.;Postma,J.P.M.;van Gunsteren,W.F.; Hermans,J.Intermolecular Forces;Pullman,B.Ed.;Reidel: Dordecht,Holland,1981;p 331.
(19) Van Gunsteren,W.F.;Billeter,S.R.;Eising,A.A.; Hünenberger,P.H.;Krüger,P.;Mark,A.E.;Scott,W.R.P.; Tironi,I.G.The GROMOS96 Manual and User Guide.In Biomolecular Simulation;Vdf HochschulverlagAG an der ETH:Zürich,Groninigen,1996.
(20) Hess,B.J.Chem.Theory Comput.2008,4(1),116. doi:10.1021/ct700200b
(21) Darden,T.;York,D.;Pedersen,L.J.Chem.Phys.1993,98, 10089.doi:10.1063/1.464397
(22) Bussi,G.;Donadio,D.;Parrinello,M.J.Chem.Phys.2007,126 (1),014101.doi:10.1063/1.2408420
(23) Parrinello,M.;Rahman,A.J.Appl.Phys.1981,52,7182. doi:10.1063/1.328693
(24) Humphrey,W.;Dalke,A.;Schulten,K.J.Mol.Graph.Model. 1996,14(1),33.doi:10.1016/0263-7855(96)00018-5
(25) Liu,F.F.;Liu,Z.;Bai,S.;Dong,X.Y.;Sun,Y.J.Chem.Phys. 2012,136(14),145101.doi:10.1063/1.3702195
(26) Basse,N.;Kaar,J.L.;Settanni,G.;Joerger,A.C.;Rutherford, T.J.;Fersht,A.R.Chem.Biol.2010,17(1),46.doi:10.1016/j. chembiol.2009.12.011
Effect of Y220C Mutant on the Conformational Transition of p53C Probed by Molecular Dynamics Simulation
SHEN Hong-Chen1DING Ji-Yong1LI Li2LIU Fu-Feng1,3,*
(1Department of Biochemical Engineering,School of Chemical Engineering and Technology,Tianjin University,Tianjin 300072, P.R.China;2College of Marine and Environmental Sciences,Tianjin University of Science&Technology,Tianjin 300457;3College of Biotechnology,Tianjin University of Science&Technology,Tianjin 300457,P.R.China)
At present,p53 is the tumor suppressor protein with the highest known frequency of mutation. Mutations in p53 will lead to the loss of its anti-cancer function and initiate cancers.The majority of the mutations in p53 are located in its core DNAbinding domain(p53C).One of the most frequent mutation in p53C is Y220C. However,the molecular mechanism of the conformational transition of the Y220C mutant of p53C remains unclear,although it is known that the Y220C mutant greatly decreases the stability of p53C.In this study, molecular dynamics(MD)simulations are used to probe the conformational transition of the Y220C mutant of p53C.The Y220C cluster including residues 138-164 and 215-238,which are strongly affected by the mutant, is identified.The Y220C mutant decreases the content of β-sheets in the Y220C cluster.The Y220C mutation not only disrupts the hydrogen bonds between the mutated residue and surrounding residues such as Leu145 and Thr155,but also weakens the hydrogen bonds between S3 and S8 of the Y220C cluster.This causes the volume of the hydrophilic cavity to increase,accelerating water molecule entry into the cavity,which eventually unfolds the protein.The above MD results explain the molecular mechanism of the Y220C mutant in the conformational transition of p53C.These findings will benefit virtual screening and design of novel stabilizers of the mutant Y220C of p53C.
April 6,2016;Revised:June 21,2016;Published online:June 22,2016.
.Email:fufengliu@tju.edu.cn;Tel:+86-22-60602717.
Cancer;p53;Residue mutation;Conformational transition;Molecular dynamics simulation
O641
10.3866/PKU.WHXB201606224
The project was supported by the National Natural Science Foundation of China(21576199).
国家自然科学基金(21576199)资助项目©Editorial office ofActa Physico-Chimica Sinica
