PCR-DGGE分析甘薯酸浆自然发酵过程中细菌多样性
2016-11-14郑艳,姚婷
郑 艳,姚 婷
(沈阳农业大学食品学院,辽宁 沈阳 110866)
PCR-DGGE分析甘薯酸浆自然发酵过程中细菌多样性
郑 艳,姚 婷
(沈阳农业大学食品学院,辽宁 沈阳 110866)
以不同发酵阶段的甘薯酸浆为研究对象,采用变性梯度凝胶电泳技术(denaturing gradient gel electrophoresis,DGGE)分析甘薯酸浆发酵过程中细菌的多样性。提取样品总DNA,聚合酶链式反应(polymerase chain reaction,PCR)扩增16S rDNA的高变区序列,再通过变性梯度凝胶电泳得到特异性条带并对其进行序列分析及比对。结果表明:甘薯酸浆自然发酵过程中,主要以厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)的明串珠菌属(Leuconostoc)、不动杆菌属(Acinetobacter)、Reyranella属、泛菌属(Pantoea)、克罗诺杆菌属(Cronobacter)以及螺菌属(Spiriiium)的细菌为主;此外,还有拟杆菌门中黄杆菌属(Flavobacterium)以及蓝细菌中的色球藻属(Chroococci)的细菌参与其中。相似性分析结果表明,自然发酵甘薯酸浆中的菌群结构有很大相似性,其中最大相似度为88.5%,最小相似度为71.4%。
甘薯酸浆;变性梯度凝胶电泳;优势菌种;系统进化树;絮凝微生物
作为淀粉浆自然发酵产物,甘薯酸浆因具有促进淀粉颗粒沉降,提高淀粉纯度的功能,而被我国的淀粉生产企业广泛采用[1]。大量研究表明,酸浆之所以具有絮凝淀粉的能力,主要是由于微生物的作用,但具体是哪些种属的微生物目前尚不明确[2]。因此,了解甘薯酸浆自然发酵过程中的菌群结构,对控制酸浆质量以及开发微生物絮凝剂具有重要的意义。
聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCRDGGE)常用于研究微生物群落结构,起初是用来检测DNA片段的点突变,1993年,Muyzer[3]、Leman[4]等首次将这一技术应用于微生物群落结构的研究,并取得了显著成效[5]。运用PCR-DGGE技术可通过免培养技术再现原始群落结构,从而避免分离培养方法造成的分析误差[6-9]。采用PCR-DGGE分析甘薯酸浆的细菌多样性的相关报道尚未见到。本实验以不同发酵周期的甘薯酸浆为研究对象,利用PCR-DGGE技术,分析甘薯酸浆发酵过程中细菌群落变化,并结合Quantity One分析软件以及16S rDNA序列测定等分子鉴定技术对样品中菌群组成进行分析,确定发酵过程中的优势菌种及具絮凝作用菌,并通过构建系统发育树,分析样品间菌群的亲缘关系[6]。
1 材料与方法
1.1 材料与试剂
新鲜甘薯购自沈阳农业大学马官桥农贸市场。
Taq DNA聚合酶 宝生物工程(大连)有限公司;去离子甲酰胺 上海酶联生物科技有限公司;四甲基二乙胺(tetramethylethylenediamine,TEMED)、双丙烯酰胺 美国Sigma公司;过硫酸铵 生工生物工程(上海)股份有限公司。
1.2 仪器与设备
PCR仪 德国Biometra公司;DGGE仪、凝胶成像系统 美国Bio-Rad公司。
1.3 方法
1.3.1 甘薯酸浆的制备
参照邓福明[10]的方法,将甘薯洗净去皮后切成小块,按m(甘薯)∶V(水)=1∶3粉碎,浆液过80目筛,除去细小薯渣。在过滤后的淀粉乳中,加入1/3淀粉乳(V/V)的酸浆后立即搅拌、静置,待淀粉沉淀完全后将黑粉层以上的浑浊废渣倒掉。乳液在25 ℃条件下发酵,每12 h搅拌2 min。每隔24 h取样一次,每次2 个重复,连续取样5 d,样品编号分别为1、2、3、4、5。
1.3.2 总DNA的提取
采用Mobil DNA Isolation Kit提取样品总DNA[11],在2 mL离心管中加入1 mL甘薯酸浆发酵液,1 000hg离心2 min,弃去上清液;检查S1溶液是否有沉淀,若有,使用前加热到60 ℃使之溶解。向上述离心管中的沉淀加入60 μL S1溶液,漩涡混合;再加入200 μL IRS溶液充分漩涡10 min,将离心管在1 000hg下离心30 s,将上清液转移至另一干净的收集管中;向收集管中加入250 μL S2溶液,漩涡振荡5 s。4 ℃条件下静置5 min后1 000hg离心1 min,将上清转移到新的收集管中;向收集管中加1.3 mL摇匀的S3溶液,漩涡5 s;将溶液转移至Spin Filter 1 000hg离心1 min,将残留的清液再次加入到Spin Filter中1 000hg离心1 min。重复这一过程,直至所有的上清液都通过Spin Filter。向清液中加入300 μL S4溶液,1 000hg离心30 s;弃掉上层洗涤流出液,再1 000hg离心1 min,小心地将Spin Filter放入新的2 mL收集管中;向收集管中加入500 μL S5溶液至Spin Filter的中央,1 000hg离心30 s,弃掉Spin Filter,试管中的DNA在-20 ℃条件下保存。
1.3.3 16S rDNA的PCR扩增
采用细菌通用引物GC-338F和518R扩增样品16S rDNA高变区序列。引物序列[12]见表1。

表1 引物信息Table 1 Primer information
PCR扩增体系(50 μL):10hPCR Buffer 5 μL;dNTP(2.5 mmol/L)3.2 μL;rTaq(5 U/μL)0.4 μL;GC-338F(20 μmol/L)1 μL;518R(20 μmol/L)1 μL;模板DNA 50 ng;补ddH2O至50 μL。
PCR扩增程序:94 ℃预变性5 min;94 ℃变性1 min,55 ℃复性45 s,72 ℃延伸1 min,30 个循环;最终72 ℃延伸10 min。
PCR产物采用DNA凝胶提取试剂盒纯化回收[13]。
1.3.4 扩增片段的变性梯度凝胶电泳
取10 μL PCR的产物进行DGGE分析。采用变性梯度为35%~55%、质量分数8%的聚丙烯酰胺凝胶在1hTAE缓冲液中150 V、60 ℃条件下电泳5 h。
DGGE完毕后,采用银染法染色[14-15]:倒掉去离子水,加入固定液固定15 min后用超纯水清洗凝胶2 次。再加入500 mL银染液染色15 min。然后用超纯水清洗2 次。之后加入500 mL显色液对凝胶显色5~7 min。显色完成后用终止液停止反应。
1.3.5 DGGE图谱中优势条带的回收与测序
用灭菌的手术刀切下待回收DGGE条带,采用多凝胶DNA提取试剂盒回收目的条带[15]。以2 μL回收产物为模板,338F/518R为引物进行PCR扩增。
PCR扩增体系和PCR扩增程序与1.3.3节相同。将重新扩增的DNA片段切胶回收、纯化后,连接到pMD18-T载体上,并转化至DH5α感受态细胞中,筛选阳性克隆,进行序列测定。
1.4 数据分析
细菌多样性指数是研究群落物种数和个体数以及均匀度的综合指标,其中香农指数(H)、丰度(S)和均匀度(E)被用来比较不同样品的多样性情况。采用Quantity One软件,根据电泳图谱中样品条带数目及每个条带的强度(灰度),对H、E和S进行分析[16],来比较不同样品的多样性情况。H、E按下列公式计算。

式中:pi为样品中每一条带在该样品所有条带中的强度比;N为DGGE图谱单一泳道上所有条带的丰富度;Ni为第i条带的丰度;S为该样品中所有条带数目总和。
测序结果采用DNAstar和Cluster软件进行序列分析,下载最相似的菌株序列作为系统发育树的参考序列。然后采用MEGA软件,Neighbor-Joining法构建系统发育树,自展数(bootstrap)为1 000。
2 结果与分析
2.1 DNA提取及PCR扩增结果

图1 发酵1~5 d样品DNA凝胶电泳图Fig.1 Agarose gel electrophoresis of genomic DNA from samples fermented for 1-5 days
采用试剂盒法提取发酵1~5 d的酸浆样品DNA后,经2%琼脂凝胶电泳得到电泳图。由图1可知,提取的DNA条带较亮,提取效果较好,可用于后续实验。
2.2 细菌16S rDNA的PCR扩增结果
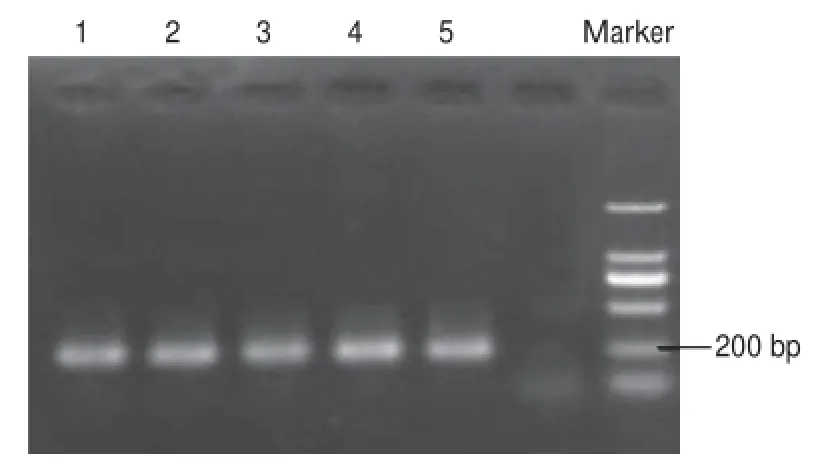
图2 16S rDNA片段PCR扩增电泳图Fig.2 Agarose gel electrophoresis of 16S rDNA PCR products
由图2可知,发酵酸浆细菌总DNA进行16S rDNA序列扩增后得到约200 bp的DNA片段,可用于DGGE分析。
2.3 PCR产物的DGGE结果

图3 PCR产物的DGGE图Fig.3 DGGE electrophoresis
由图3可知,不同发酵周期的甘薯酸浆经过PCRDGGE电泳分析后,分离出数目不等的电泳条带,且各条带的强度和迁移率各不相同。
2.4 Quantity One 分析

图4 量化分析胶图Fig.4 Quantitative analysis of gels
采用Quantity One软件对2.3节中的电泳条带进行匹配分析。由图4可知共获得16 类条带。在获得的16 类条带中,5、6、10、15、16号均显示为优势条带(DGGE图谱量化后较宽较深),9、12、13号被视为特征条带(某个样品中特有),将这些条带均进行割胶回收分析。
2.5 不同发酵时间甘薯酸浆PCR-DGGE图谱相似度分析
对照条带的强度与迁徙程度,根据戴斯系数(dicecoefficient,Cs)计算出样品间相似度的矩阵图。由表2可知,样品间的最大相似度为88.5%,最小相似度为71.4%。说明发酵期间的菌群结构相似度较高。
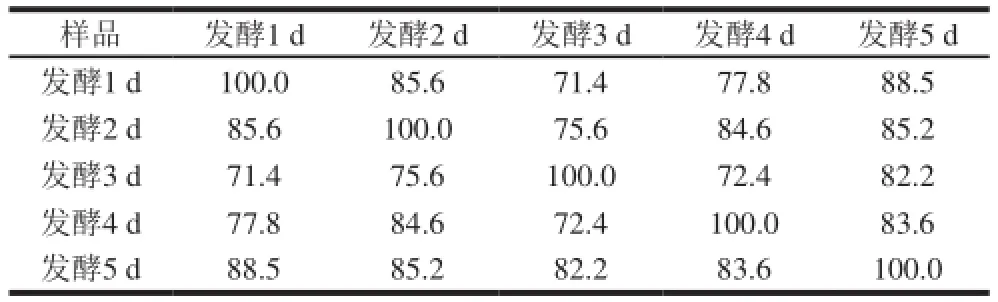
表2 PCR-DGGE图谱的相似性比较结果Table 2 Comparison of PCR-DGGE profiles for similarity with dice coefficient %
利用相似性矩阵数据,通过UPGAMA(the unweighted pair group method with arithmetic averages)算法可实现聚类分析,生成系统树,如图5所示。

图5 样品聚类分析图Fig.5 Dendrogram (UPGMA) among 1-5 samples
2.6 不同发酵时间甘薯酸浆细菌多样性
由Quantity One软件输出各样品各条带的灰度值,利用Biodap计算出样品间的香农指数、均匀度和丰度。结果如图6所示。

图6 细菌多样性指数柱状图Fig.6 Diversity analysis of bacteria from sweet potato sour liquid during the fermentation period
分析结果表明,甘薯酸浆自然发酵过程中细菌多样性显著,分布较为均匀。其中,发酵3 d的甘薯酸浆细菌香农指数可达2.53,丰度指数可达14,均匀度指数可达0.96,这与DGGE图谱(图3)的结果一致。
2.7 主要电泳条带的序列鉴定和同源性分析
对DGGE电泳所得16 条特异性条带切胶回收,以338F/518R为引物进行PCR扩增,获得约200 bp的DNA片段。PCR产物纯化后连接到pMD18-T载体上,转化至DH5α感受态细胞中,筛选阳性克隆测序。
登陆美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)网站,测序结果与GenBank中的序列进行比对,得到条带所代表的细菌类型。每个回收条带选取3 个克隆进行了序列测定,比对结果见表3。

表3 序列鉴定和同源性分析结果Table 3 Sequence identification and homology analysis

图7 系统发育树Fig.7 Phylogenetic tree
结果表明,所有序列与数据库中16S rDNA序列的相似度在91%~100%之间。采用MEGA5软件,Neighbor-Joining法构建系统发育树如图7所示,进行同源性分析,判断它们的亲缘关系[18-20],自展数(bootstrap)为1 000。
由图7可知,根据进化上亲缘关系的相似度,条带1、2、5、6、7、8、9、15序列形成了第1类群;条带3、4、10、11、13、14、16形成了第2类群;条带12为第3类群。
3 结 论
甘薯酸浆是酸浆法生产淀粉、粉丝过程中促使淀粉颗粒聚集成团,促进淀粉絮凝,并且能够提高淀粉纯度的一种生物絮凝剂[2],是由新鲜的甘薯淀粉乳经自然发酵后形成的一种具有微酸性的乳白色液体。我国对甘薯酸浆的研究相对较多,主要集中在甘薯酸浆工艺优化,对甘薯酸浆中的微生物计数、分离提取。利用PCR-DGGE分析甘薯酸浆发酵过程中的细菌多样性还未见报道[11]。
PCR-DGGE是研究微生物菌群结构的主要方法之一[21-23]。本研究采用PCR-DGGE法对自然发酵过程中甘薯酸浆的细菌多样性进行分析,根据得到的16 条条带将甘薯酸浆中的微生物进行归类。结果表明:在甘薯酸浆发酵过程中共鉴定出7 个细菌菌属,主要以厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)的细菌为主;厚壁菌门以Leuconostoc为主,而变形菌门则以Acinetobacter、Reyranella、Pantoea、Cronobacter以及Spiriiium为主;此外,还有拟杆菌门中Flavobacterium以及蓝细菌中的Chroococci。这与洪安安[24]于2009年研究活性污泥中絮凝菌群的结果一致。
在甘薯酸浆自然发酵过程中,细菌菌群群落结构也有变化,Leuconostoccitreum KM20、Leuconostoc mesenteroides、Acinetobacter lwoffii、Reyranella graminifolii、Pantoea eucalypti、Cronobacter dublinensis、Chroococci diopsisthermalis、Niveispirillum irakense在整个发酵过程中都有出现,Niveispirillum irakense数量在整个发酵过程中先随着发酵时间的延长增多,发酵中期过后又逐渐减少,可以推测它对絮凝起到重要作用。F. oncorhynchi只出现在发酵中期,此时甘薯酸浆絮凝性最高,可以判断为具有絮凝作用菌。这种细菌具有良好的絮凝效果,这在王兆慧[25]产絮凝剂黄杆菌的筛选及絮凝特性研究中得到了验证。
[1] 曹宗巽, 卢光莹. 乳酸链球菌凝集淀粉粒机理的进一步研究[J].微生物学报, 1980, 20(3): 271-275. DOI:10.13343/j.cnki. wsxb.1980.03.008.
[2] 张莉力, 李新华. 甘薯酸浆絮凝淀粉机理及人工发酵剂制备酸浆的工艺研究[D]. 沈阳: 沈阳农业大学, 2010: 18-21.
[3] MUYZER G, WAAL E C, UITTERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700.
[4] LEMAN L, FISCHER S, HURLEYI, et al. Sequence-determined DNA separations[J]. Annual Review of Biophysics and Bioengineering, 1984, 13(1): 399-423. DOI:10.1146/annurev.bb.13.060184.002151.
[5] ABRIOUELH, BENOMAR N, LUCAS R, et al. Culture-independent study of the diversity of microbial populations in brines during fermentation of naturally-fermented Aloreña green table olives[J]. International Journal of Food Microbiology, 2011, 144(3): 487-496. DOI:10.1016/j.ijfoodmicro.2010.11.006.
[6] 武俊瑞. 东北传统发酵特色食品中主要微生物多样性研究[D]. 沈阳: 沈阳农业大学, 2013: 25-30.
[7] 许文涛, 郭星, 罗云波, 等. 微生物菌群多样性分析方法的研究进展[J]. 食品科学, 2009, 30(7): 258-265. DOI:10.3321/ j.issn:1002-6630.2009.07.060.
[8] 朱扬玲. 采用PCR-DGGE方法研究浙江玫瑰醋酿造过程中的微生物多样性[D]. 杭州: 浙江工商大学, 2009: 41-43.
[9] LÜ X C, WENG X, ZHANG W, et al. Microbial diversity of traditional fermentation starters for Hong Qu glutinous rice wine as determined by PCR-mediated DGGE[J]. Food Control, 2012, 28(2): 426-434. DOI:10.1016/j.foodcont.2012.05.025.
[10] 邓福明. 酸浆法与旋流分离法制备甘薯淀粉的物化特性及粉条品质比较研究[D]. 北京: 中国农业科学院, 2012: 15-18.
[11] SADET S, MARTIN C, MEUNIER B, et al. PCR-DGGE analysis reveals a distinct diversity in the bacterial population attached to the rumen epithelium[J]. Animal, 2007, 51(1): 939-944. DOI:10.1017/ S1751731107000304.
[12] FAHLE G A, FISCHER S H. Comparison of six commercial DNA extraction kits for recovery of cytomegalovirus DNA from spiked human specimens[J]. Journal of Clinical Microbiology, 2000, 38(10): 3860-3863.
[13] HARUTA S, UENO S, EGAWA I, et al. Succession of bacterial and fungal communities during a traditional pot fermentation of rice viengar assessed by PCR-DGGE[J]. International Journal of Food Microbiology, 2006, 109(1): 79-87. DOI:10.1016/ j.ijfoodmicro.2006.01.015.
[14] 姚粟. 芝麻香型白酒高温大曲细菌群落多样性研究[D]. 北京: 北京林业大学, 2013: 34-37.
[15] NIEMI R M, HEISKANMEN I, WALLENIUS K, et al. Extraction and purification of DNA in rhizosphere soil samples for PCR-DGGE analysis of bacterial consortia[J]. Journal of Microbiological Methods, 2001, 45(3): 155-165. DOI:10.1016/S0167-7012(01)00253-6.
[16] KUANG Y, TANI K, AIDAN J, et a1. Characterization of bacterial population of raw milk from bovine mastits by culture-independent PCR-DGGE method[J]. Biochemical Engineering Journal, 2009, 45(4): 76-81. DOI:10.1016/j.bej.2009.02.010.
[17] 马俊孝, 季明杰, 孔健. PCR-DGGE技术在微生物物种多样性研究中的局限性及其解决措施[J]. 食品科学, 2008, 29(5): 493-497. DOI:10.3321/j.issn:1002-6630.2008.05.112.
[18] YU Z T, MORRISON M. Comparisons of different hypervariable regions of rrs genes for use in fingerprinting of microbial communities by PCR-DGGE[J]. Applied and Environmental Microbiology, 2004, 70(8): 4800-4806. DOI:10.1128/AEM.70.8.4800-4806.2004.
[19] 李正国, 付晓红, 邓伟, 等. 传统分离培养结合DGGE法检测榨菜腌制过程的细菌多样性[J]. 微生物学通报, 2009(3): 371-376.
[20] 杨代永, 范光先, 汪地强, 等. 高温大曲中的微生物研究[J]. 酿酒科技, 2007(5): 37-38. DOI:10.3969/j.issn.1001-9286.2007.05.006.
[21] ERCOLINI D. PCR-DGGE fingerprinting: novel strategies for detection of microbes in food[J]. Journal of Microbiological Methods, 2004, 56(3): 297-314. DOI:10.1016/j.mimet.2003.11.006.
[22] 张春林. 内蒙古传统发酵酸粥中微生物多样性分析[D]. 呼和浩特:内蒙古农业大学, 2010: 54-57.
[23] WANG H Y, ZHANG X J, ZHAO L P, et al. Analysis and comparison of the bacterial community in fermented grains during the fermentation for two different styles of Chinese liquor[J]. Journal of Industrial Microbiology and Biotechnology, 2008, 35(5): 603-609. DOI:10.1007/ S10295-008-0323-z.
[24] 洪安安. 活性污泥的主要微生物菌群及研究方法[J]. 工业水处理, 2009, 29(2): 10-14. DOI:10.11894/1005-829x.
[25] 王兆慧. 产絮凝剂黄杆菌的筛选及其絮凝特性研究[J]. 安徽农业科学, 2009, 37(35): 17327-17332. DOI:10.3969/ j.issn.0517-6611.2009.35.013.
Analysis of Bacterial Diversity during Natural Fermenation of Sweet Potato Sour Liquid by PCR-DGGE
ZHENG Yan, YAO Ting
(Food Science College, Shenyang Agricultural University, Shenyang 110866, China)
In the present study, changes in the bacteria composition of sweet potato sour liquid during the natural fermentation process were investigated by polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE). Total bacterial DNA was extracted from the samples, and the touch down PCR was applied to amplify the V3 region of 16S rDNA for identification based on DGGE fingerprints. The specific gel bands were excised, then sequenced and analyzed by comparison with the GenBank database. Results showed that the representative bacteria during the fermentation process of sour liquid were identified as Leuconostoc, Acinetobacter, Reyranella, Pantoea, Cronobacter, Spiriiium which belong to Firmicutes and Proteobacteria, respectively. Flavobacterium belonging to the phylum Bacteroidetes and Chroococci belonging to the phylum Cyanobacteria were also involved. The coefficient of similarity showed that the similarity of bacterial community in sweet potato sour liquid ranged from 88.5% to 71.4%.
sweet potato sour liquid; denaturing gradient gel electrophoresis (DGGE); dominant bacteria; phylogenetic tree; flocculent microorganisms
10.7506/spkx1002-6630-201607019
TS201.3
A
1002-6630(2016)07-0099-05
郑艳, 姚婷. PCR-DGGE分析甘薯酸浆自然发酵过程中细菌多样性[J]. 食品科学, 2016, 37(7): 99-103. DOI:10.7506/ spkx1002-6630-201607019. http://www.spkx.net.cn
ZHENG Yan, YAO Ting. Analysis of bacterial diversity during natural fermenation of sweet potato sour liquid by PCRDGGE[J]. Food Science, 2016, 37(7): 99-103. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201607019. http://www.spkx.net.cn
2015-04-27
郑艳(1973—),女,副教授,博士,研究方向为食品微生物学及食品生物技术。E-mail:23574196@qq.com
