三角帆蚌短型肽聚糖识别蛋白S3基因克隆及表达分析
2016-11-12李海军张建强吴圣楠隗黎丽
李海军 张建强 吴圣楠 隗黎丽 刘 毅
(1. 江西师范大学生命科学学院,南昌 330022; 2. 江西农业大学动物科学学院,南昌 330045)
三角帆蚌短型肽聚糖识别蛋白S3基因克隆及表达分析
李海军1张建强1吴圣楠1隗黎丽2刘毅1
(1. 江西师范大学生命科学学院,南昌 330022; 2. 江西农业大学动物科学学院,南昌 330045)
肽聚糖识别蛋白(PGRPs)是机体先天免疫系统中的重要模式识别受体,研究对三角帆蚌的一种短型肽聚糖识别蛋白进行了克隆与表达分析。研究采用5′、3′ RACE技术,对高通量转录组测序所得三角帆蚌PGRPS3基因(hcPGRPS3)片段分别进行了5′,3′末端克隆,经拼接得到hcPGRPS3 cDNA全长序列。采用多种分子生物学软件对hcPGRPS3 cDNA全长序列进行了特征分析,实时荧光定量(qRT-PCR)检测了其组织分布,及经肽聚糖(Peptidoglycan,PGN)和脂多糖(Lipopolysaccharide,LPS)刺激后其在肝胰腺中的基因表达变化。hcPGRPS3 cDNA全长1438 bp,开放阅读框为858 bp,编码285个氨基酸,预测分子量大小为32.3 ku,pH 7.0时的理论等点为7.98。序列中不存在信号肽与跨膜结构。氨基酸序列保守性分析表明,其他物种短型PGRP中,以夏威夷短尾鱿PGRP4与hcPGRPS3同源性最高(51%)。qRT-PCR检测结果显示,hcPGRPS3在性腺及肝胰腺中表达水平较高。经PGN或LPS刺激后,肝胰腺中hcPGRPS3表达水平显著上调,表明hcPGRPS3可能在机体抗菌免疫反应中发挥重要作用。
肽聚糖识别蛋白;三角帆蚌;基因克隆;基因表达
肽聚糖识别蛋白(Peptidoglycan recognition proteins,PGRPs)是一类在先天免疫中发挥重要作用的模式识别受体(PRRs),因其对细菌细胞壁成分肽聚糖(PGN)的识别作用而命名为肽聚糖识别蛋白,从昆虫到哺乳动物其结构高度保守[1,2]。研究者最早于1996年在家蚕(Bombyx mori)发现PGRP,发现其能结合革兰氏阳性菌PGN,并激活酶原级联反应[3]。除了识别病原体,某些PGRPs还表现出显著的酰胺酶活性[4],及强力杀菌活性[5]。研究还发现,PGRPs不仅能结合PGN,部分PGRPs还能结合LPS,以及肽聚糖结合槽结合位点以外的其他病原体配体[6]。PGRPs在触发免疫信号转导通路进而激活抗菌肽、细胞的吞噬作用和PGN水解中发挥重要功能,在先天免疫中具有不可或缺的作用[7—9]。
PGRPs可分为短型(无脊椎动物PGRP-S与脊椎动物PGLYRP-1)、长型(无脊椎动物PGRP-L与脊椎动物PGLYRP-2)和中间型(无脊椎动物PGLYRP-3、PGLYRP-4),在大多数种类动物中都有发现,包括昆虫、软体动物、棘皮动物、鱼类、两栖动物、鸟类及哺乳动物[10]。对软体动物PGRPs的研究较晚,近年来陆续有研究报道,包括夏威夷短尾鱿(Euprymna scolopes)[11]、海湾扇贝(Argopecten irradians)PGRP[12]、栉孔扇贝(Chlamys farreri)[13]、盘鲍(Haliotis discus discus)[14]、长牡蛎(Crassostrea gigas)[15]、大竹蛏(Solen grandis)[16],在三角帆蚌(Hyriopsis cumingii)中也有关于PGRPS1基因的研究报道[17]。
三角帆蚌分布广泛,主要分布在鄱阳湖、洞庭湖、太湖、巢湖和洪泽湖五大湖,是目前应用最广的良种淡水育珠蚌,而细菌感染引起的疾病日益严重[18]。本研究克隆了hcPGRPS3 cDNA序列全长,对其基因序列特征进行了分析,并研究了其在PGN及LPS后刺激后的表达变化情况,为三角帆蚌细菌性疾病的免疫防治提供基础资料。
1 材料与方法
1.1材料
本研究所选用的三角帆蚌购自南昌市人民公园菜市场。研究选用的三角帆蚌体长约10—15 cm。购回实验室后,立即用自来水将蚌壳上的淤泥仔细刷洗干净,置于盛有曝气自来水的水族箱(100 cm× 60 cm×45 cm)中暂养,箱中水量不宜过多,以刚刚淹没蚌体为佳。每天用曝气自来水换水1—2次,充气泵充气饲养,暂养1周确认蚌体健康无病之后方开始实验。三角帆蚌的具体饲养条件同文献[19]。
本研究所用外源刺激物PGN和LPS购自Sigma-Aldrich公司,PGN源自枯草芽孢杆菌(Bacillus subtilis),LPS源自大肠杆菌(Escherichia coli,0111:B4),使用前分别溶解于PBS缓冲液中使最终浓度为1 mg/mL。
1.2核酸提取与cDNA模板合成
采用TaKaRa公司的RNAiso Plus提取组织总RNA,操作过程中(包括组织总RNA提取以及应用RNA的所有相关实验)琉璃器皿及金属器械经180℃高温烘烤6h以上使RNase彻底失活,使用Thermo公司QSP RNase & DNase Free离心管和枪头。RNA的具体提取参考提取试剂说明书进行。cDNA合成按Takara PrimeScriptTMReverse Transcriptase Kit操作手册进行。
1.3hcPGRPS3 cDNA全长获得
hcPGRPS3 cDNA 5′端片段克隆始自hcPGRPS3 cDNA的1条长段序列(1426 bp)由转录组高通量测序结果获得。依据此序列,采用Invitrogen公司的5′RACE System for Rapid Amplification of cDNA Ends,Version 2.0试剂盒获得该基因的5′端序列。扩增时共设计3条基因特异引物:GSP1、GSP2及GSP3。首先提取总RNA; 使用SUPERSCRIPT II RT 酶和引物GSP1 对总RNA进行目的基因第一链cDNA的合成,使用RNase Mix对合成的cDNA进行去RNA处理; 使用DNA 纯化试剂盒对经RNAase处理过的cDNA进行纯化; 使用TdT酶和dCTP对纯化后的cDNA进行末端加上多聚C; 使用引物GSP2和试剂盒里面带的桥连铆钉引物AAP对已经加dC尾的cDNA进行PCR第一轮扩增; 使用引物GSP3和试剂盒里面带的桥连通用扩增引物AUAP进行巢式PCR第二轮扩增; 将第二轮PCR产物进行电泳并对目的条带进行切胶回收纯化; 纯化后的PCR产物与pMD18-T进行连接,转化后对阳性克隆进行测序。
hcPGRPS3 cDNA 3′端片段克隆采用Clontech公司的SMARTerTMRACE cDNA Amplification Kit试剂盒获得该基因的3′端序列。扩增时共设计2条引物:PGRPS3-F1与PGRPS3-F2。首先使用逆转录酶SMARTScribeTMReverse Transcriptase和引物3′CDS primer A 对总RNA进行逆转录合成cDNA; 使用引物PGRPS3-F1和通用引物UPM(Long引物和Short引物按1∶3比例混匀所得),以上面合成的cDNA为模板进行第一轮PCR扩增; 将第一轮PCR扩增产物稀释50倍,然后用引物PGRPS3-F2和 UPM进行第二轮PCR扩增,将第二轮PCR产物进行电泳并对目的条带进行切胶回收纯化; 纯化后的PCR产物与pMD18-T进行连接,转化后挑取阳性克隆测序。将5′RACE、3′RACE序列及高通量测序片段拼接,从而得到hcPGRPS3基因全长cDNA序列。用于hcPGRPS3基因扩增与表达的引物见表1。

表1 hcPGRPS3基因扩增和表达所用引物Tab. 1 Primers used in the current study
1.4hcPGRPS3基因序列分析与系统进化树构建
利用NCBI ORF Finder在线软件寻找开放阅读框,并翻译出氨基酸序列全长; ProtParam tool预测蛋白质分子大小、理论等电点; NetNGlyc 1.0 Server分析潜在N-糖基化位点; Signalp 4.1 server预测信号肽; TMHMM在线预测跨膜螺旋; Pfam HMM分析蛋白质结构域; 使用NCBI上BLASTp软件进行同源性搜索和不同物种PGRPs氨基酸同一性比对;ClustalW 1.83软件进行氨基酸序列比对,Mega 5.0软件N-J法构建进化树。用于氨基酸比对和进化树构建的基因名称及其GenBank序列登录号见表2。
1.5hcPGRPS3在不同组织中的表达
本实验的总RNA提取每个样本挑选3只健康未经任何处理的三角帆蚌作为3个平行样,分别取鳃、性腺、心脏、肝胰腺和外套膜组织,用Trizol法提取总RNA,并经反转录得到cDNA。根据hcPGRPS3基因cDNA全长序列,使用Primer Premier 5.0设计hcPGRPS3基因qRT-PCR特异性引物(表1),根据三角帆蚌β-actin基因全长cDNA序列设计内参照基因引物(表1)。反应条件如下:(1) 95℃ 3min;95℃ 10s,55℃ 20s,72℃ 20s,40个循环; 72℃ 5s;(2) 从65℃开始,每5s升温0.5℃至95℃绘制溶解曲线。每1个平行样hcPGRPS3基因和β-actin基因做3次重复荧光定量,3次重复平均值为最终荧光定量值。

表2 用于氨基酸序列比对和构建进化树的基因登录号Tab. 2 GenBank accession number used in the alignment and phylogenetic tree construction
使用BIO-RAD CFX ConnectTM荧光定量 PCR检测系统内置软件进行溶解曲线分析,将心脏组织PGRPS3基因mRNA的表达量设为1,采用2-ΔΔCt法计算hcPGRPS3基因的相对表达值,使用SPSS 18.0计算标准误差。
1.6注射PGN或LPS后hcPGRPS3在肝胰腺中的表达变化
取三角帆蚌3只于闭合肌注射PBS缓冲液100 μL,作为对照组(0),注射后取肝胰腺组织。取三角帆蚌60只用作刺激实验,随机分为2组。分别于闭合肌注射100 μL PGN或 LPS进行免疫,并于注射后3h、6h、12h、24h和36h取蚌的肝胰腺组织(n=3)。Trizol法提取各组织的总RNA,利用qRT-PCR法检测经PGN和LPS刺激后hcPGRPS3在肝胰腺组织中的表达变化(扩增条件同1.5)。将对照组0 h肝胰腺组织hcPGRPS3基因mRNA表达量设为1,采用2-ΔΔCt法计算不同刺激组肝胰腺中hcPGRPS3基因的相对表达值,结果使用SPSS18.0单因素方差分析法(One-way ANOVA)进行统计分析(n=3),P<0.05为显著性差异。
2 结果
2.1hcPGRPS3基因cDNA序列全长及推导的氨基酸
5′ RACE扩增获得472 bp片段,3′ RACE扩增获得664 bp片段,序列拼接获得的hcPGRPS3基因cDNA序列全长1438 bp(GenBank登录号:KJ123767),其中5′端非翻译区72 bp和3′非翻译区508 bp,3′非翻译区,有2个mRNA不稳定信号(ATTTA)和多聚腺苷酸加尾信号(ATTAAA)。该基因开放阅读框为858 bp,编码285个氨基酸,预测蛋白分子量大小为32.3 ku,pH7.0时理论等电点为7.98,含2个潜在N-糖基化位点(N40PT和N92YS),有PGRP结构域(aa126—263)。多重比对结果证明hcPGRPS3符合PGRPs所有特征。hcPGRPS3无信号肽与跨膜螺旋结构。
2.2PGRPs同源性比较与系统进化关系
利用NCBI在线Blast对软体动物多个物种的多种PGRPs氨基酸序列进行了同源性比对,各物种间PGRPs的相似性见表2。由表可知,其他物种短型PGRP中,以夏威夷短尾鱿PGRP4与hcPGRPS3同源性最高(51%)。选取其他软体动物类PGRPs蛋白序列应用Mega 5.0软件按邻接法构建系统进化树。系统进化树表明:hcPGRPS3与夏威夷短尾鱿PGRP4(短型)及盘鲍短型PGRP最先聚为一枝,又与夏威夷短尾鱿其他短型PGRP聚为一枝,表明在所选物种中,它们之间的进化关系最为密切(图1)。
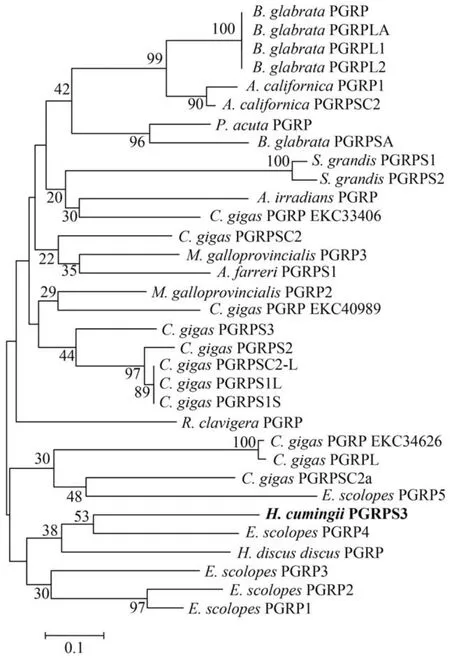
图1 hcPGRPS3的N-J系统进化树Fig. 1 Neighbor-joining phylogenetic tree analysis of hcPGRPS3使用Clustral X 1.83 软件进行序列比对,MEGA 5.0按邻接法构建系统进化树。结点处数值代表1000评估的自举检验置信度。PGRP氨基酸序列登录号详见表2Full-length amino acid sequences were aligned using Clustral X 1.83 and a phylogenetic tree was constructed using Neighbor-Joining algorithm with MEGA 5.0(1000 bootstrap). The GenBank accession numbers of PGRP are shown in Tab. 2
2.3hcPGRPS3组织表达
hcPGRPS3组织表达分布如图2所示。hcPGRPS3在所检测组织中均有表达,在性腺中的表达水平最高,其次为肝胰腺,而在鳃、心脏和外套膜表达量最低。
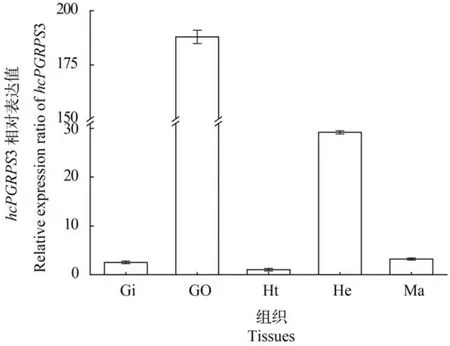
图2 不同组织中hcPGRPS3相对表达量Fig. 2 Expression of hcPGRPS3 in different tissues
2.4PGN或LPS刺激后肝胰腺中hcPGRPS3表达变化
经PGN刺激3—12h肝胰腺中hcPGRPS3表达水平显著性上调(P<0.05),3h时表达水平为最高,在24h后表达水平逐渐恢复为0h正常值(图3); 而经LPS刺激6—24h肝胰腺中hcPGRPS3表达水平显著上调(P<0.05),以6h表达水平为最高,且在36h后hcPGRPS3的表达水平逐渐恢复为0时正常值(图3)。

图3 PGN或LPS刺激后hcPGRPS3相对表达量Fig. 3 The role of PGN and LPS on the expression of hcPGRPS3
3 讨论
在进化过程中,先天免疫系统通过一系列高度保守的模式识别受体(PRRs)识别微生物[1]。肽聚糖识别蛋白(PGRPs)属于先天免疫PRRs家族,从昆虫到哺乳动物高度保守[2],所有的PGRPs都有一个长约160个氨基酸的PGRP结构域。短型PGRPs蛋白约为200个氨基酸长,分子量大小约为18—20 ku,大多数长型和中间型大小的PGRPs(无脊椎动物PGRP-L和脊椎动物PGLYRP-2)蛋白至少有400个氨基酸长[10]。大竹蛏(S. grandis) PGRP-S1、PGRP-S2分别编码270和141个氨基酸[16]。许氏平鲉(Sebastes schlegeli)PGRP-L1、PGRP-L2分别编码466和482个氨基酸[20]。本研究中所克隆的hcPGRPS3开放阅读框编码285个氨基酸,预测蛋白质分子量为32.3ku,等点为7.98,有PGRP结构域(aa126—263)长度为138个氨基酸。氨基酸序列同源比对显示hcPGRPS3与夏威夷短尾鱿PGRP4(短型)相似性最高,达51%,系统进化树表明hcPGRPS3与夏威夷短尾鱿和盘鲍短型PGRP聚为一枝。以上结果表明:本研究克隆的hcPGRPS3确为PGRPs家族成员,是一种短型PGRP。
PGRPs主要功能是识别和结合PGN及激活下游免疫应答,当与细菌PGN结合后,PGRPs可以抑制或激活下游免疫信号通路如Toll和IMD途径[21,22]或JNK信号通路[23]。但一些PGRPs也表现出显著酰胺酶活性和强力杀菌活性[4,5]。PGRPs分子正面是肽聚糖结合槽,背面为PGRP特异性区域[24]。酰胺酶可以通过水解细菌PGN的酰胺键起到杀菌功能,所有具有酰胺酶活性的PGRPs(无脊椎动物和脊椎动物)PGN结合位点,由两个组氨酸(His)、一个酪氨酸(Tyr)及一个半胱氨酸(Cys)组成。在无酰胺酶活性的PGRPs PGN结合位点中,半胱氨酸(Cys)被丝氨酸(Ser)取代,可以由此来推测PGRPs有无酰胺酶活性[25]。与其他物种PGRPs的序列比对发现,hcPGRPS3肽聚糖结合槽处高度保守的四个氨基酸残基为His141、Tyr176、His250和Ser259,hcPGRPS3酰胺酶活性位点中的半胱氨酸(Cys)被丝氨酸(Ser)取代而不具有酰胺酶活性,可能不具有水解细菌PGN的酰胺键功能。类似的,夏威夷短尾鱿PGRP4(esPGRP4)的酰胺酶活性位点C160也被丝氨酸(Ser)取代而不具有酰胺酶活性,esPGRP4功能可能仅限于:识别PGN,或者诱导触发Toll/NF-κB磷酸化催化级联反应的信号转导功能[11]。这种功能也与果蝇(Drosophila melanogaster) PGRP-SA类似[26],hcPGRPS3可能也具有类似功能。通过预测发现hcPGRPS3不存在信号肽和跨膜螺旋,可能为胞内蛋白。esPGRP1也无信号肽和跨膜螺旋,通过蛋白结构分析认为是一种胞内定位蛋白[11]。hcPGRPS3是一种非典型的软体动物PGRP,蛋白无酰胺酶活性,很可能为胞内蛋白。
PGRPs在动物组织中表达广泛,而不同生物体PGRPs在不同组织中的表达水平各异。美国红鱼(Sciaenops ocellatus) PGRP在脑、肾、肌肉、鳃、血液、脾脏、心脏及肝脏均有表达,以肝脏的表达量为最高[27]。海湾扇贝(A. irradians)PGRP在性腺、鳃、血细胞、肾、外套膜及闭合中均有表达,以血细胞中的表达量最高[12]。三角帆蚌PGRPS1基因在肝胰腺中表达水平最高,其次为性腺[17]。而本研究则发现hcPGRPS3在三角帆蚌的鳃、性腺、心脏、肝胰腺和外套膜组织均有表达,而以性腺的表达水平最高,其次为肝胰腺。hcPGRPS3在不同组织中的表达模式表明:性腺和肝胰腺可能是主要的效应器官。
在一些细菌或病原相关分子模式(PAMPs)刺激下PGRP表达水平会发生上调或者下降,或者无显著变化。在细菌刺激下栉孔扇贝(C. farreri) 血细胞中肽聚糖识别蛋白S1(cfPGRP-S1)的表达水平显著上升,鳗弧菌(革兰氏阴性菌)刺激下24h后cfPGRP-S1表达量上升到显著水平(持续到48h),溶壁微球菌(革兰氏阳性菌)刺激下12h后cfPGRP-S1表达量上升到显著水平(持续到48h)[13]。盘鲍(H. discus discus)鳃中肽聚糖识别蛋白(hdPGRP)在PGN(源自金黄色葡萄球菌)刺激下,早期阶段(3—6h)表达水平下调,后期阶段(24—48h) hdPGRP表达水平显著上调; 在LPS(源自大肠杆菌)刺激下,早期阶段(6—12h) hdPGRP表达水平上调,后期阶段(24—48h)恢复为正常水平,但在3h暂时出现表达水平下调现象[14]; PGN(3—6h)和LPS(3h)刺激下hdPGRP表达水平的下降,可能是由于鳃组织免疫细胞与外部环境微生物频繁接触产生了耐受性内毒素造成的[14]。三角帆蚌肝胰腺中PGRPS1 基因在LPS或PGN刺激后表达水平无显著性变化[17]。而本研究结果表明,三角帆蚌在经PGN刺激后3—12h,肝胰腺中hcPGRPS3表达水平显著性上调,在3h达到最大值,后期恢复(24h后)为正常水平; 经LPS刺激后6—24h,肝胰腺中hcPGRPS3表达水平显著上调,在6h达到最大值,而36h后表达恢复为正常水平。经PGN和LPS诱导后hcPGRPS3表达上调,这与aiPGRP、sgPGRPS1、sgPGRP-S2的诱导结果类似; PGN及LPS对hcPGRPS3的诱导结果非常相似,表明hcPGRPS3在对细菌PGN和LPS的识别或结合上具有重要的作用。本研究结果将为开展更准确的hcPGRPS3基因研究提供一定的参考理论基础。
[1]Dziarski R. Review:Recognition of bacterial peptidoglycan by the innate immune system [J]. Cellular and Molecular Life Sciences,2003,60(9):1793—1804
[2]Liu C,Xu Z,Gupta D,et al. Peptidoglycan recognition proteins:a novel family of four human innate immunity pattern recognition molecules [J]. The Journal of Biological Chemistry,2001,276(37):34686—34694
[3]Yoshida H,Kinoshita K,Ashida M. Purification of peptidoglycan recognition protein from hemolymph of the silkworm,Bombyx mori [J]. The Journal of Biological Chemistry,1996,271(23):13854—13860
[4]Mellroth P,Steiner H. PGRP-SB1:an N-acetylmuramoyl L-alanine amidase with antibacterial activity [J]. Biochemical and Biophysical Research Communications,2006,350(4):994—999
[5]Mellroth P,Karlsson J,Steiner H. A scavenger function for a Drosophila peptidoglycan recognition protein [J]. The Journal of Biological Chemistry,2003,278(3):7059—7064
[6]Liu C,Gelius E,Liu G,et al. Mammalian peptidoglycan recognition protein binds peptidoglycan with high affinity,is expressed in neutrophils,and inhibits bacterial growth [J]. The Journal of Biological Chemistry,2000,275(32):24490—24499
[7]Lemaitre B,Nicolas E,Michaut L,et al. The dorsoventral regulatory gene cassette spätzle/Toll/cactus controls the potent antifungal response in Drosophila adults [J]. Cell,1996,86(6):973—983
[8]Lemaitre B,Kromer-Metzger E,Michaut L,et al. A recessive mutation,immune deficiency(imd),defines two distinct control pathways in the Drosophila host defense [J]. Proceedings of the National Academy of Sciences of the United States of America,1995,92(21):9465—9469
[9]Werner T,Liu G,Kang D,Hultmark D,et al. A family of peptidoglycan recognition proteins in the fruit fly Drosophila melanogaster [J]. Proceedings of the National Academy of Sciences of the United States of America,2000,97(25):13772—13777
[10]Dziarski R,Gupta D. The peptidoglycan recognition proteins(PGRPs) [J]. Genome Biology,2006,7(8):232—245
[11]Goodson M,Kojadinovic M,Troll J,et al. Identifying components of the NF-κB pathway in the beneficial euprymna scolopes-vibrio fischeri light organ symbiosis [J]. Applied and Environmental Microbiology,2005,71(11):6934—6946
[12]Ni D J,Song L S,Wu L T,et al. Molecular cloning and mRNA expression of peptidoglycan recognition protein(PGRP) gene in bay scallop(Argopecten irradiant,Lamarck 1819) [J]. Developmental and Comparative Immunology,2007,31(6):548—558
[13]Su J G,Nia D J,Song L S,et al. Molecular cloning and characterization of a short type peptidoglycan recognitionprotein(CfPGRP-S1) cDNA from Zhikong scallop Chlamys farreri [J]. Fish & Shellfish Immunology,2007,23(3):646—656
[14]Premachandra H K A,Don A S E,Whang I,et al. Identification of a novel molluscan short-type peptidoglycan recognition protein in disk abalone(Haliotis discus) involved in host antibacterial defense [J]. Fish & Shellfish Immunology,2014,39(1):99—107
[15]Zhang Y,Yu Z N. The first evidence of positive selection in peptidoglycan recognition protein(PGRP) genes of Crassostrea gigas [J]. Fish & Shellfish Immunology,2013,34(5):1352—1355
[16]Wei X M,Yang J M,Yang D L,et al. Molecular cloning and mRNA expression of two peptidoglycan recognition protein(PGRP) genes from mollusk Solen grandis [J]. Fish & Shellfish Immunology,2012,32(1):178—185
[17]Yang Z Y,Li J H,Li Y,et al. Molecular cloning and functional characterization of a short peptidoglycan recognition protein(HcPGRPS1) from the freshwater mussel,Hyriopsis cumingi [J]. Molecular Immunology,2013,56(4):729—738
[18]Qian R H,Li J L,Dong Z G,et al. Morphological variations analysis among populations of Hyriopsis cumingii in five large lakes of China [J]. Oceanologia et Limnologia Sinica,2003,34(4):436—442 [钱荣华,李家乐,董志国,等. 中国五大湖三角帆蚌形态差异分析. 海洋与湖沼,2003,34(4):436—442]
[19]Wu S N,Liu D W,Liu Y,et al. Cloning and expression analysis of heat shock protein 60 gene from Hyriopsis cumingii [J]. Acta Hydrobiologica Sinica,2014,38(5):897—902 [吴圣楠,刘大伟,刘毅,等. 三角帆蚌热休克蛋白60基因克隆及其表达分析. 水生生物学报,2014,38(5):897—902]
[20]Kima M Y,Jang J H,Lee J W,et al. Molecular cloning and characterization of peptidoglycan recognition proteins from the rockfish,Sebastes schlegeli [J]. Fish & Shellfish Immunology,2010,28(4):632—639
[21]Choe K M,Werner T,Stoven S,et al. Requirement for a peptidoglycan recognition protein(PGRP) in relish activation and antibacterial immune responses in Drosophila [J]. Science,2002,296(5566):359—362
[22]Michel T,Reichhart J M,Hoffmann J A,et al. Drosophila Toll is activated by Gram-positive bacteria through a circulating peptidoglycan recognition protein [J]. Nature,2001,414(6865):756—759
[23]Maillet F,Bischoff V,Vignal C,et al. The Drosophila peptidoglycan recognition protein PGRP-LF blocks PGRP-LC and IMD/JNK pathway activation [J]. Cell Host Microbe,2008,3(5):293—303
[24]Guan R,Wang Q,Sundberg E J,et al. Crystal structure of human peptidoglycan recognition protein S(PGRP-S) at 1.70 Å resolution [J]. Journal of Molecular Biology,2005,347(4):683—691
[25]Gelius E,Persson C,Karlsson J,et al. A mammalian peptidoglycan recognition protein with N-acetylmuramoyl-Lalanine amidase activity [J]. Biochemical & Biophysical Research Communications,2003,306(4):988—994
[26]Reiser J B,Teyton L,Wilson I A. Crystal structure of the Drosophila peptidoglycan recognition protein(PGRP)-SA at 1.56 Å resolution [J]. Journal of Molecular Biology,2004,340(4):909—917
[27]Li M F,Zhang M,Wang C L,et al. A peptidoglycan recognition protein from Sciaenops ocellatus is a zinc amidase and a bactericide with a substrate range limited to Gram-positive bacteria [J]. Fish & Shellfish Immunology,2012,32(2):322—330
CLONING AND EXPRESSION ANALYSIS OF SHORT TYPE PEPTIDOGLYCAN RECOGNITION PROTEIN S3 GENE FROM HYRIOPSIS CUMINGII
LI Hai-Jun1,ZHANG Jian-Qiang1,WU Sheng-Nan1,WEI Li-Li2and LIU Yi1
(1. College of Life Sciences,Jiangxi Normal University,Nanchang 330022,China; 2. College of Animal Science and Technology,Jiangxi Agricultural University,Nanchang 330045,China)
The present study cloned and sequenced Hyriopsis cumingii PGRPS3(hcPGRPS3) and measured its expreesion in multiple tissues. Results showed that the hcPGRPS3 cDNA sequence was 1438 bp in length,consisting of a 858 bp open reading frame(ORF) encoding 285 amino acid residues with 32.3 ku of predicted molecular weight and 7.98 of the theoretical isoelectric point,which have no signal petptide and transmembrane helices but have two latent N-glycosylation sites N40PT and N92YS. Multiple alignment analysis revealed that hcPGRPS3 shareed the highest identity with Euprymna scolopes PGRP4(51%). hcPGRPS3 expressed in all examined organs of healthy H. cumingii with the low level in gill,heart and mantle and high level in gonad and hepatopancreas. Lipopolysaccharide and Peptidoglycan obviously up-regulated the expression of hcPGRPS3 in hepatopancreas,suggesting that hcPGRPS3 may play an important role in H. cumingii hepatopancreas innate immunity system against bacterial challenged.
Peptidoglycan recognition proteins S3; Hyriopsis cumingii; Clone; Expression
Q344+.1
A
1000-3207(2016)05-0921-07
10.7541/2016.119
2015-08-25;
2015-12-25
国家自然科学基金项目(31060359); 江西省自然科学基金(20122BAB204017)资助 [Supported by the National Natural Science Foundation of China(31060359); the Natural Science Foundation of Jiangxi Province(20122BAB204017)]
李海军(1988—),男,河南信阳人; 硕士研究生; 主要研究方向为水产动物分子免疫。E-mail:haijunlisc@gmail.com
刘毅,男,博士; E-mail:yiliusan@jxnu.edu.cn
