斑点叉尾鮰GHRH基因3个SNPs位点及其单倍型组合与生长性状的关联分析
2016-11-12张世勇钟立强王明华潘建林陈校辉边文冀
张世勇 钟立强 秦 钦 王明华 潘建林 陈校辉 边文冀
(1. 江苏省淡水水产研究所,南京 210017; 2. 江苏省农业种质资源保护与利用平台,南京 210014)
斑点叉尾鮰GHRH基因3个SNPs位点及其单倍型组合与生长性状的关联分析
张世勇1,2钟立强1,2秦钦1,2王明华1,2潘建林1,2陈校辉1,2边文冀1,2
(1. 江苏省淡水水产研究所,南京 210017; 2. 江苏省农业种质资源保护与利用平台,南京 210014)
研究旨在探讨生长激素释放激素基因(Growth hormone-releasing hormone,GHRH)对斑点叉尾鮰(Ictalurus punctatus)生长性状的影响。采用DNA混池测序法筛选GHRH基因的单核苷酸多态性(Single nucleotide polymorphisms,SNPs)位点,使用SNaPshot法将筛选到的SNPs多态性位点进行分型,并对这些位点进行连锁不平衡和单倍型分析。结果表明,在GHRH基因内含子区域共检测到4个SNPs位点,并成功地对3个位点进行了分型,3个位点间均不存在强连锁不平衡; 3个SNPs位点在176尾斑点叉尾鮰中形成了6种有效单倍型。关联分析表明SNP位点g.6301 G>A的AA基因型的体质量显著性地高于AG和GG型(P<0.05),比群体的平均体质量高14%。单倍型组合H1/H4和H1/H5个体的体质量和体长极显著性地高于其他单倍型组合(P<0.01),体质量比群体平均体质量分别高30%和15%,体长比群体平均体长分别高7%和6%。研究为斑点叉尾鮰生长性状分子标记辅助选育和QTL定位提供了参考依据。
斑点叉尾鮰;GHRH;SNP;SNaPshot;生长性状;单倍型分析;关联分析
生长激素释放激素(Growth hormone-releasing hormone,GHRH)又被称为生长激素释放因子,1981年首次从肢端肥大症病人的胰腺肿瘤组织中分离出来[1]。在哺乳动物中,GHRH最基本的功能是促进生长激素(Growth hormone,GH)的合成和分泌[2],除此之外还具有促进细胞的增殖[3]、细胞分化[4]、免疫调控[5]、睡眠调控[6]等功能。鱼类GHRH基因最早于2000年从斑马鱼(Danio rerio)中克隆鉴定出来[7],与大多数哺乳动物相似,鱼类GHRH基因具有5个外显子。鱼类GHRH成熟多肽由27个氨基酸组成,具有高度的保守性[8],与人类GHRH同源性高达81.5%。鱼类GHRH由下丘脑分泌,然后与垂体GH分泌细胞表面的生长激素释放激素受体(Growth hormone-releasing hormone receptor,GHRH-R)结合,并通过腺甘酸环化酶/ cAMP/蛋白激酶信号通路和NO/NO合酶信号通路介导,从而促使GH分泌细胞分泌GH[9]。GHRH/ GHRH-R体系已经陆续在一批硬骨鱼类中得到证实,包括斑马鱼(Danio rerio)[10]、金鱼(Carassius auratus)[10]、石斑鱼(Epinephelus coioides)[11]、牙鲆(Paralichthys olivaceus)[12]和半滑舌鳎(Cynoglossus semilaevis)[13]等。
GHRH基因的SNPs多态性与生长性状的关联分析在畜牧动物中已经有较多的报道。Baile和Buonomo[14]在奶牛GHRH基因中发现一种低频率基因型(AA型)个体比其他基因型产奶率高30%。Cheong等[15]在朝鲜牛GHRH基因5′端调控区发现SNP位点-4241A> T与屠宰率显著性相关,类似的现象也在猪中发现[16]。Franco等[17]检测出大量SNPs位点与长白猪日增重、脂肪厚度等性状相关联。鱼类其他生长相关基因的SNP多态性与生长性状的关联分析在大西洋鲑(Salmo salar)[18]、亚洲鲈(Lates calcarifer)[19]、鲤(Cyprinus carpio)[20—22]等鱼类中已进行过大量的研究。然而GHRH基因仅在北极红点鲑(Salvelinus alpinus)和半滑舌鳎(Cynoglossus semilaevis)中做过此方面研究,2003年Tao和Boulding[23]在北极红点鲑GHRH基因第四内含子上筛选到1个SNP位点与早期生长的生长率显著相关; 2015年Guo等[24]利用高通量测序技术在半滑舌鳎GHRH基因的第二内含子上发现一个突变位点与多种生长参数显著相关。然而,其他水产动物至今还未见相关研究报道。
1 材料与方法
1.1试验动物
本研究所用斑点叉尾鮰样本均来自于江苏省淡水水产研究所禄口试验基地。1997—2004 年从美国引进德克萨斯(1997)群体、阿肯色(1999)群体、密西西比(2001)群体、阿肯色(2003)群体和阿肯色(2004)群体共405尾建立育种基础群体,利用基础群体进行G0代家系构建。2013年6月利用G0代家系构建G1代家系,为了减小环境对斑点叉尾鮰生长的影响,苗种培育按照同一标准进行,即均一的换水速率、投喂量、养殖密度、充氧量以及水温。待到家系平均日龄为520d时扫描记录存活个体编号,测量个体的体质量和体长等信息。同时采集每尾鱼的尾鳍组织,95%酒精浸泡,-20℃保存。
1.2基因组DNA提取
斑点叉尾鮰尾鳍DNA的提取使用UNIQ-10柱式动物基因组DNA抽提试剂盒(上海生工生物工程股份有限公司),操作过程按照试剂盒说明进行。1%琼脂糖凝胶电泳检测DNA提取效果,紫外分光光度计(Eppendorf,德国)测定DNA样品浓度。
1.3引物设计
根据GenBank数据库公布的斑点叉尾鮰GHRH全基因序列(FJ882999),使用Primer Premier 5软件设计两对引物P1和P2。引物P1(F:AACACCGAG CTGCGATTACTC; R:GCCTCGTGACTGTC TGATTGG)主要用于扩增第一内含子部分序列、第二外显子、第二内含子、第三外显子、第三内含子部分序列; 引物P2(F:AACCAGGCCAGGA TCTGAACAC; R:ACAGTTTGGCTCAGCATT TCTG)主要用于扩增第四外显子、第四内含子、第五外显子。
1.4PCR扩增及测序
超临界流体萃取技术是现代化工分离中出现的新技术,是现在国家应用比较广泛的技术,超临界流体萃取技术具有工艺简单,无有机溶剂残留,操作条件温和等传统工艺不可比拟的优点,在油脂生产中,避免油脂氧化酸败,而且不存在溶剂残留,有效的克服压榨法产率低、精制工艺繁琐,产品色泽不理想等缺点。
随机抽取40尾斑点叉尾鮰DNA样品,调整每个样品浓度至100 ng/μL,各取1 μL混合构建DNA池。以DNA池和20尾个体基因组DNA为模板,根据设计好的两对引物进行PCR扩增。PCR扩增使用即用型UtraTaq 酶PCR试剂盒(上海捷瑞生物工程有限公司)。PCR反应体系为40 μL:2×UltraTaq Master Mix试剂20 μL,基因组DNA 2 μL,上下游引物(浓度为10 pmol/μL)各2 μL,ddH2O 14 μL。PCR扩增条件:94℃预变性5min; 94℃变性30s,退火30s,72℃延伸60s,30个循环; 72℃延伸10min。1%琼脂糖凝胶电泳检测PCR产物,凝胶成像仪观察电泳结果。PCR产物纯化以后送至上海生工生物工程股份有限公司,直接使用ABI 3730XL测序仪(ABI,美国)测序。使用chromas软件观察测序峰图,并结合ClustalX软件进行DNA序列比对来判断SNP位点的碱基类型。
1.5SNPs分型
使用SNaPshot法对选自斑点叉尾鮰核心选育群体的176尾鱼进行SNPs位点分型。根据SNPs位点侧翼序列设计扩增引物,使扩增片段长度为200—500 bp。延伸引物3′端第一个碱基紧邻待测SNPs位点,Tm值为50℃以上,并且在引物的5′末端加上不同长度的Poly C或Poly T(表1)。
使用Touchdown PCR程序进行多重PCR扩增,反应体系为20 μL:0.8 μL MgCl2(50 mmol/L)、2 μL 10×PCR Buffer(Mg2+free)、0.5 μL Dntp(10 mmol/ L)、混合引物0.5 μL、模板DNA 1 μL、0.5 μL Platinum Taq(5 U)(Invitrogen,英国)和14.2 μL ddH2O。PCR反应程序为:95℃预变性5min;94℃变性15s,60℃退火15s,72℃延伸30s,共11个循环,每个循环退火温度降低0.5℃; 94℃变性15s,54℃退火15s,72℃延伸30s,共24个循环; 72℃延伸5min。反应后进行PCR产物纯化,反应体系为10 μL:FastAP(Fermentas,美国)0.8 μL,Exo I(Fermentas,美国) 0.2 μL,Exo I buffer 0.7 μL,PCR产物3 μL,H2O 5.3 μL。37℃ 15min,80℃ 15min,纯化后进行延伸反应,并预先混好延伸引物。

表1 用于SNPs分型的扩增引物和延伸引物Tab. 1 Amplification primers and extension primers used for Multiplex SNaPshot genotyping
SNPs分型使用ABI SNaPshot Multiplex PCR试剂盒(ABI,美国),反应体系为6 μL:Snapshot Mix 1 μL,延伸引物0.1 μL,多重PCR产物2 μL,ddH2O 2.9 μL。反应条件为:96℃预变性1min; 96℃变性10s,52℃退火5s,60℃延伸30s,共30个循环。反应结束后加入1U FastAP,37℃ 60min,85℃ 15min进行纯化。以GeneScan-120Liz Size Standard为内标,取1 μL 产物与9 μL含有Liz的Hi-Di(GS-120Liz∶Hi-Di=1∶200)混合,95℃变性3min,立即冰浴3min 后上测序仪。检测及分析采用ABI PRISM 3730 XI型自动遗传分析系统(ABI,美国),并利用Genemapper v4.1软件进行分型。
1.6数据分析
使用SHEsis软件分析GHRH基因SNPs位点的单倍型以及连锁不平衡。利用R(3.2.1)软件对试验斑点叉尾鮰SNPs位点的各基因型体质量和体长进行方差齐性分析(Homogeneity of variance test)和单因素ANOVA(One-way ANOVA)分析。采用均值多重比较(Multiple comparisons of means)方法分析SNPs位点各基因型及其单倍型组合与生长性状关联程度。统计分析模型:

式中:Yij为某个性状第i个标记第j个个体观测值;μ为实验观测所有个体的平均值(即总体平均值);Gi为第i个标记的效应值; eij为对应于观察值的随机残差效应。
2 结果
2.1GHRH基因SNPs位点筛选
利用设计的两对引物对 40尾斑点叉尾鮰构建的DNA池以及20尾个体进行PCR扩增和测序分析。共筛选出4个SNPs位点,即g.4333 A>G、g.4616 A>C、g.4632 A>T及g.6301 G>A。4个SNPs位点全部位于内含子区域,其中g.4333 A>G位于第二内含子; g.4616 A>C、g.4632 A>T位于第三内含子; g.6301 G>A位于第四内含子。4个位点均是双等位多态性(Biallelic polymorphisms),转换和颠换各两个。
成功分型的3个SNPs位点遗传多样性参数见表2,观测杂合度(Observed heterozygosity,Ho)的分布范围为0.4148—0.6136; 期望杂合度(Expected heterozygosity,He)的分布范围为0.3674—0.4949,有效等位基因数(Effective number of alleles,Ne)的分布范围为1.5781—1.9745。多态性信息容量(Polymorphism information content,PIC)的分布范围为0.2992—0.3717,均大于0.25小于0.5,表明3个SNPs位点具有中等遗传多样性。g.4333 A>G和g.6301 G>A显著地偏离哈迪-温伯格平衡(Hardy-Weinberg equilibrium,HWE),表明这两个位点在群体中具有较高的遗传变异和选择压力。
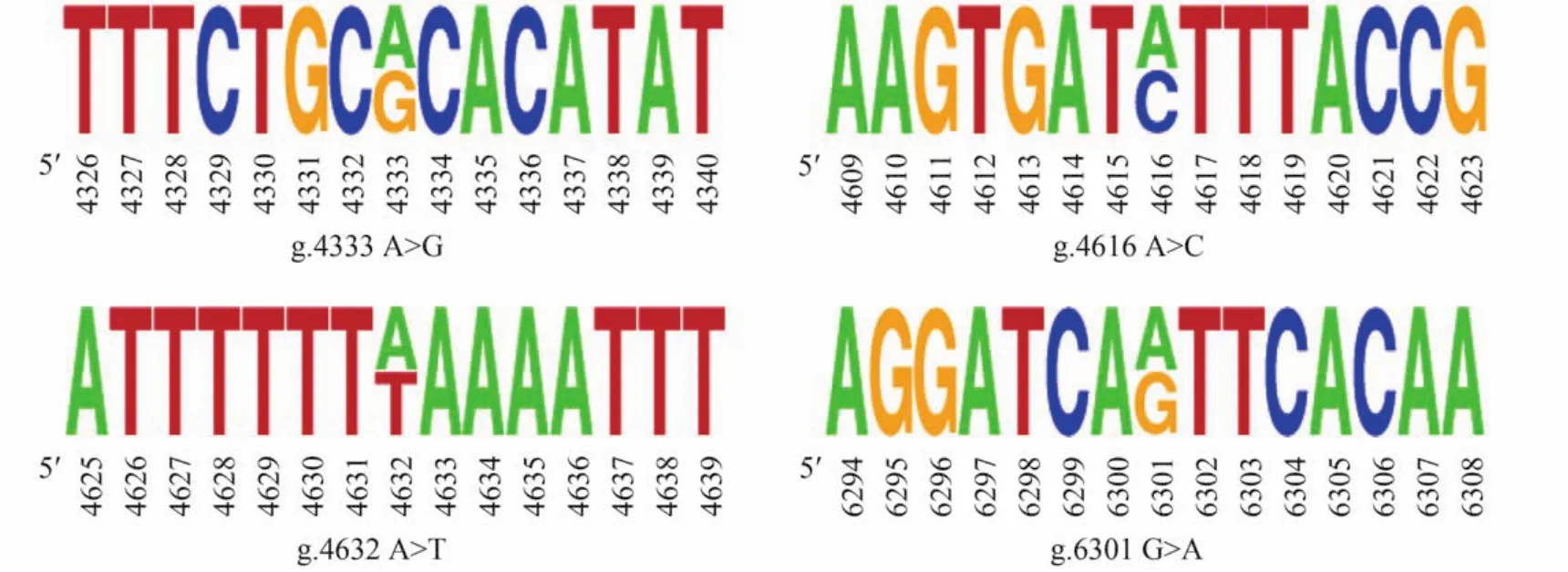
图1 GHRH基因4个SNPs位点所处位置Fig. 1 The localization of four identified SNPs of GHRH gene
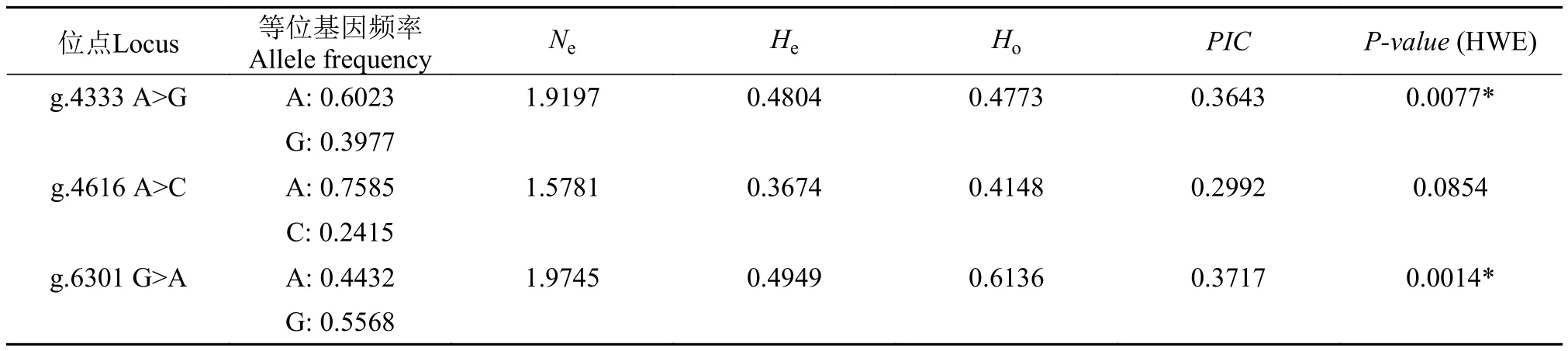
表2 斑点叉尾鮰GHRH基因SNPs遗传多样性参数Tab. 2 Diversity parameters of SNPs of GHRH gene in channel catfish
2.2连锁不平衡和单倍型分析
GHRH基因3个SNPs位点的连锁不平衡分析如图2所示,g.4616 A>C与g.6301 G>A的D′值和r2值显著性地高于其他两个位点之间的D′和r2值:g.4333 A>G与g.4616 A>C的D′值和r2值为0.19和0.01,g.4616 A>C与g.6301 G>A的D′值和r2值为0.99和0.25,g.4333 A>G与g.6301 G>A的D′值和r2值为0.29和0.04。所以g.4333 A>G位点与其他2个位点间均不存在连锁不平衡(r2=0或r2≈0),而g.4616 A>C与g.6301 G>A不能满足D′>0.8且r2>0.33,说明这两个SNPs位点间不存在强连锁不平衡(D′<0.8或r2<0.33)。同时,3个位点的单倍型分析在表3中列出,本文检测到GHRH基因的3个突变位点在176尾斑点叉尾鮰选育群体中形成7种单倍型(理论单倍型数为8种)。H1(AAA)的频率最高(32%),H3(ACA)的频率最低(近似为0)。

图2 GHRH基因3个SNPs位点的连锁不平衡分析(左图是D′值,右图是r2值)Fig. 2 D′ values(left) and r2values(right) of pairwise linkage disequilibrium analysis of the GHRH gene
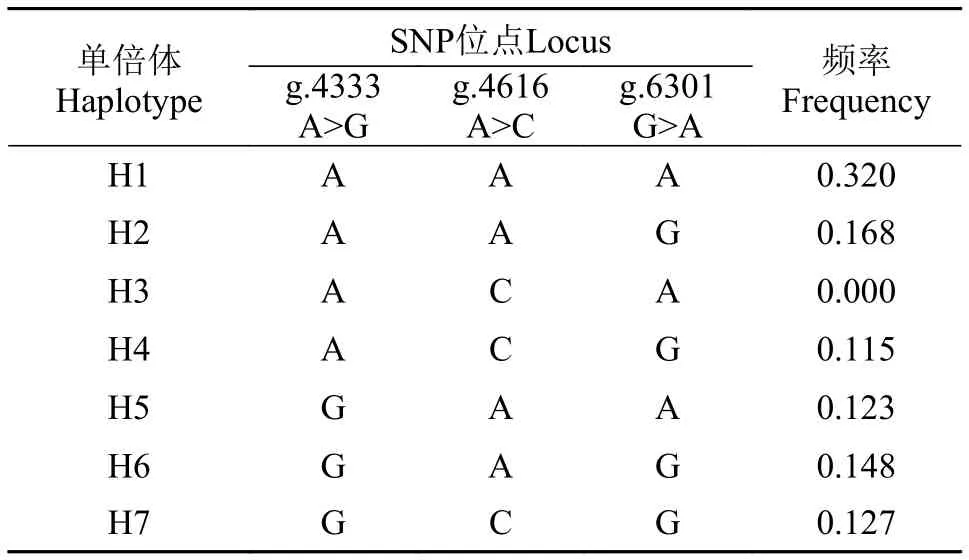
表3 GHRH基因的单倍型分析Tab. 3 Haplotype frequencies in the GHRH gene
2.3SNPs位点及单倍型组合与生长性状的关联分析
对176尾斑点叉尾鮰的体质量和体长进行统计分析,数据以“平均数±标准误”来表示。体质量和体长分别为(280.63±13.21) g和(31.26±0.56) cm。利用R软件将筛选得到的3个SNPs位点与测量的斑点叉尾鮰体质量和体长进行单因素ANOVA 分析并结合均值多重比较分析得出各有一个位点与斑点叉尾鮰的体质量呈显著性正相关和负相关(P<0.05)。在位点g.4616 A>C,具有基因型CC型个体的体质量和体长显著性低于那些具有AA型和AC型个体(P<0.05),比群体的平均体质量低25%;而g.6301 G>A位点的AA基因型的体质量显著性地高于AG和GG型(P<0.05),比群体的平均体质量高14%。
GHRH单倍型组合与斑点叉尾鮰生长性状的关联分析如表4所示,在所有构建的单倍型组合中,个体数小于10尾的单倍型组合不参与统计分析和多重比较。分析结果表明,单倍型组合H1/H4和H1/ H5个体的体质量和体长极显著性地高于其他单倍型组合(P<0.01),体质量比群体平均体质量分别高30%和15%,体长比群体平均体长分别高7%和6%。

表4 斑点叉尾鮰GHRH基因SNPs位点与生长性状的关联分析Tab. 4 Association between SNPs of GHRH and growth traits in channel catfish
3 讨论
He、Ho、Ne和PIC等遗传参数常用来评估群体的遗传多样性。GHRH基因中的3个SNPs位点均处于中等多态性水平,这表明该斑点叉尾鮰核心选育群体保持了较高的遗传多样性。期望杂合度(He)反映了基因的一致性,当He较低时,基因的多样性较低; 当He>0.5时,表明这个群体没有经历选择压力,而保持较高的多样性。在本研究中g.4333 A>G和g.6301 G>A两个位点的He值分别为0.4804和0.4949,仍然处于0.5附近且所选群体较小。PIC用来描述种群内个体间的遗传多性,当PIC值大于0.5时具有高遗传多样性,当0.25<PIC<0.5时具有中等的遗传多样性,当PIC小于0.25时具有低遗传多态性[26]。本文3个SNPs的PIC值均大于0.25小于0.5,说明该斑点叉尾鮰核心选育群体具有中等遗传多样性。g.4333 A>G和g.6301 G>A显著地偏离哈迪-温伯格平衡(P<0.01),表明这两个位点在群体中具有较高的遗传变异和选择压力。综上分析,该核心选育群体仍具有较高遗传多样性,可以用于后续的品种选育。
连锁不平衡常使用参数D′和r2来评估,D′ >0.8表示两个位点处于强连锁不平衡状态[27,28],r2>0.33则可以认为这两个SNPs是紧密连锁,作为一个整体遗传的[29]。本文中各SNPs位点间均不能满足D′ >0.8且r2>0.33,所以这些SNPs位点间均不存在强连锁不平衡(D′<0.8或r2<0.33)或者连锁不平衡(r2=0或r2≈0),表明它们趋向于相互独立遗传。单倍型分析相较于单个SNP位点分析,能提供更多的遗传信息,并且可以反映祖先基因的基因结构[30]。本研究应用SHEsis软件在3个SNPs位点中,发现了7种单倍型,其中最高频率的单倍型是H1(AAA),最低频率的单倍型H3(ACA)的频率近似为0,其他5种单倍型频率均位于10%—20%,因此有效单倍型组合为6种。单倍型AAA可能最早出现在原始的祖先基因中,在进化过程中再产生SNP位点突变从而导致其他类型单倍型出现。
本研究在斑点叉尾鮰GHRH基因中共筛选到4个SNPs位点,均位于内含子区域。将筛选到的SNP位点与生长性状进行关联分析,结果表明在位点g.4616 A>C,具有基因型CC型个体的体质量和体长显著性低于那些具有AA型和AC型个体(P<0.05)。鉴于CC型个体的数量太少(6尾),因此该位点与生长性状的关联还有待于后期进一步验证。g.6301 G>A位点的AA基因型的体质量显著性地高于AG和GG型(P<0.05)。数量性状多是由多种微效基因或是某一基因的多个位点共同调控的,所以仅仅分析某一基因中单个位点的多态性往往不能准确得出基因或等位基因与性状的真实相关性,需要同时对多个位点的综合效应进行探讨[31]。因此本研究对GHRH基因3个SNPs位点进行单倍型联合分析,除去那些个体数少于10尾的单倍型组合,共获得6组有效的单倍型组合。将单倍型组合与生长性状进行关联分析,结果表明单倍型组合H1/H4和H1/H5是具有最高均值(无论是体质量还是体长)的优势单倍型组合,与其他大部分单倍型组合差异极显著(P<0.01)。H1/H2和H2/H7是均值最低的单倍型组合,极显著低于其他大部分单倍型组合(P<0.01)。
本研究中获得的与生长性状显著关联的SNPs位点位于内含子区域。这主要是因为内含子序列不具有编码蛋白质的能力,因此受到的选择压力较小,相应地,突变频率要高于外显子,所以容易积累更多的变异。基因组中的内含子,特别是内含子与外显子相衔接的部分序列在调控mRNA剪切、转录和基因表达方面起着重要的作用[32],有些内含子可以参与形成长链非编码RNA(lncRNA)和小RNA(miRNA)[33,34]来调控基因的表达。然而本文中的SNP位点具体通过什么机制参与调控GHRH基因还有待于进一步的研究。
4 结论
本研究针对GHRH基因的外显子和部分内含子序列在斑点叉尾鮰核心选育群体中检测到4个SNPs位点,使用SNaPshot法成功对3个位点在176尾斑点叉尾鮰个体中成功地进行了分型。单倍型分析得到6种有效单倍型以及6种单倍型组合。对SNPs位点及单倍型组合与生长性状进行关联分析表明,SNP位点g.6301 G>A的AA基因型体质量显著性地高于AG和GG型(P<0.05),单倍型组合H1/H4和H1/H5个体的体质量和体长极显著性地高于其他单倍型组合(P<0.01); H1/H2和H2/H7为生长性状劣势单倍型组合,极显著低于其他大部分单倍型组合(P<0.01)。因此在后续斑点叉尾鮰选育过程中可以适当考虑选留单倍型组合为H1/H4和H1/H5的个体,淘汰单倍型组合为H1/H2和H2/H7的个体。本研究为斑点叉尾鮰生长性状分子标记辅助选育和QTL定位提供了参考依据。
[1]Guillemin R,Brazeau P,Bohlen P,et al. Growth hormone-releasing factor from a human pancreatic tumor that caused acromegaly [J]. Science,1982,218(4572):585—587
[2]Bloch B,Brazeau P,Ling N,et al. Immunohistochemical detection of growth hormone-releasing factor in brain [J]. Nature,1983,301(2):607—608
[3]Billestrup N,Swanson L W,Vale W. Growth hormonereleasing factor stimulates proliferation of somatotrophs in vitro [J]. Proceedings of the National Academy of Sciences,1986,83(18):6854—6857
[4]Dean C E,Porter T E. Regulation of somatotroph differentiation and growth hormone(GH) secretion by corticosterone and gh-releasing hormone during embryonic development [J]. Endocrinology,1999,140(3):1104—1110
[5]Csaba G. Hormones in the immune system and their possible role. A critical review [J]. Acta Microbiologica et Immunologica Hungarica,2014,61(3):241—260
[6]Liao F,Zhang T J,Mahan T E,et al. Effects of growth hormone-releasing hormone on sleep and brain interstitial fluid amyloid-β in an APP transgenic mouse model[J]. Brain,Behavior,and Immunity,2014,47(7):163—171
[7]Fradinger E A,Sherwood N M. Characterization of the gene encoding both growth hormone-releasing hormone(GRF) and pituitary adenylate cyclase-activating polypeptide(PACAP) in the zebrafish [J]. Molecular and Cellular Endocrinology,2000,165(1):211—219
[8]Han L Q,Bai J J,Li S J. Comparison of gene structure,sequence homology and expression pattern of largemouth bass GHRH-LP and GHRH [J]. Acta Hydrobiologica Sinica,2011,35(3):473—481 [韩林强,白俊杰,李胜杰.大口黑鲈GHRH-LP和GHRH基因序列同源性、基因结构和时序表达研究. 水生生物学报,2011,35(3):473—481]
[9]Wang B,Qin C,Zhang C,et al. Differential involvement of signaling pathways in the regulation of growth hormone release by somatostatin and growth hormone-releasing hormone in orange-spotted grouper(Epinephelus coioides) [J]. Molecular and Cellular Endocrinology,2014,382(2):851—859
[10]Lee L T O,Siu F K Y,Tam J K V,et al. Discovery of growth hormone-releasing hormones and receptors in nonmammalian vertebrates [J]. Proceedings of the National Academy of Sciences,2007,104(7):2133—2138
[11]Qian Y,Yan A,Lin H,et al. Molecular characterization of the GHRH/GHRH-R and its effect on GH synthesis and release in orange-spotted grouper(Epinephelus coioides) [J]. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,2012,163(2):229—237
[12]Nam B H,Moon J Y,Kim Y O,et al. Molecular and functional analyses of growth hormone-releasing hormone(GHRH) from olive flounder(Paralichthys olivaceus) [J]. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,2011,159(2):84—91
[13]Ji X S,Chen S L,Jiang Y L,et al. Growth differences and differential expression analysis of pituitary adenylate cyclase activating polypeptide(PACAP) and growth hormone-releasing hormone(GHRH) between the sexes in half-smooth tongue sole Cynoglossus semilaevis [J]. General and Comparative Endocrinology,2011,170(1):99—109
[14]Baile C A,Buonomo F C. Growth hormone-releasing factor effects on pituitary function,growth,and lactation[J]. Journal of Dairy Science,1987,70(2):467—473
[15]Cheong H S,Yoon D H,Kim L H,et al. Growth hormone-releasing hormone(GHRH) polymorphisms associated with carcass traits of meat in Korean cattle [J]. BMC Genetics,2006,7(1):35
[16]Pierzchala M,Blicharski T,Kuryl J. Growth rate and carcass quality in pigs as related to genotype at loci POU1F1/ RsaI(Pit1/RsaI) and GHRH/AluI [J]. Animal Science Papers and Reports,2003,21(3):159—166
[17]Franco M M,Antunes R C,Silva H D,et al. Association of PIT1,GH and GHRH polymorphisms with performance and carcass traits in Landrace pigs [J]. Journal of Applied Genetics,2005,46(2):195—200
[18]Tsai H Y,Hamilton A,Guy D R,et al. Single nucleotide polymorphisms in the insulin-like growth factor 1(IGF1)gene are associated with growth-related traits in farmed Atlantic salmon [J]. Animal Genetics,2014,45(5):709—715
[19]He X P,Xia J H,Wang C M,et al. Significant associations of polymorphisms in the prolactin gene with growth traits in Asian seabass(Lates calcarifer) [J]. Animal Genetics,2012,43(2):233—236
[20]Feng X,Yu X,Pang M,et al. Molecular characterization and expression of three preprosomatostatin genes and their association with growth in common carp(Cyprinus carpio) [J]. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,2015,182(4):37—46
[21]Tao W J,Ma L J,Yu J H,et al. SNP loci associated with weight gain on growth hormone receptor genes in Cyprinus carpio var. jian [J]. Acta Hydrobiologica Sinica,2011,35(4):622—629 [陶文静,马龙俊,俞菊华,等. 建鲤GHR基因多态性及与增重相关的SNP位点的筛选.水生生物学报,2011,35(4):622—629]
[22]Li H X,Li J L,Tang Y K,et al. Correlation analysis between body weight gain and ODC1 genotypes in Cyprinus carpio var. jian [J]. Acta Hydrobiologica Sinica,2014,38(3):414—421 [李红霞,李建林,唐永凯,等. 建鲤ODC1基因型与增重的相关性分析. 水生生物学报,2014,38(3):414—421]
[23]Tao W J,Boulding E G. Associations between single nucleotide polymorphisms in candidate genes and growth rate in Arctic charr(Salvelinus alpinus L.) [J]. Heredity,2003,91(1):60—69
[24]Guo L,Xia J,Yang S,et al. GHRH,PRP-PACAP and GHRHR target sequencing via an ion torrent personal genome machine reveals an association with growth in orange-spotted grouper(Epinephelus coioides) [J]. International Journal of Molecular Sciences,2015,16(11):26137—26150
[25]Luan S,Bian W J,Deng W,et al. Genetic parameters for the growth and survival of the base population in channel catfish(Ictalures punctatus) [J]. Journal of Fisheries of China,2012,36(9):1313—1321 [栾生,边文冀,邓伟,等. 斑点叉尾鮰基础群体生长和存活性状遗传参数估计. 水产学报,2012,36(9):1313—1321]
[26]Yuan Z,Li J,Li J,et al. SNPs identification and its correlation analysis with milk somatic cell score in bovine MBL1 gene [J]. Molecular Biology Reports,2013,40(1):7—12
[27]Hohenlohe P A,Bassham S,Currey M,et al. Extensive linkage disequilibrium and parallel adaptive divergence across threespine stickleback genomes [J]. Philosophical Transactions of the Royal Society of London B:Biological Sciences,2012,367(1587):395—408
[28]Slatkin M. Linkage disequilibrium-understanding the evolutionary past and mapping the medical future [J]. Nature Reviews Genetics,2008,9(6):477—485
[29]de Camargo G M F,Costa R B,Lucia G,et al. Polymorphisms in TOX and NCOA2 genes and their associations with reproductive traits in cattle [J]. Reproduction,Fertility and Development,2015,27(3):523—528
[30]Akey J,Jin L,Xiong M. Haplotypes vs single marker linkage disequilibrium tests:what do we gain [J]?European Journal of Human Genetics,2001,9(4):291—300
[31]Xue Q,Wang J Y,Zhang G X,et al. Polymorphism of melanocortin 3 receptor gene(MC3R) and association analysis between the diplotypes and the carcass traits in Jinghai Yellow chicken [J]. Journal of Agricultural Biotechnology,2015,23(3):344—351 [薛倩,王金玉,张跟喜,等. 黑素皮质素受3基因(MC3R) 多态性及其单倍型组合与京海黄鸡屠体性状的关联分析. 农业生物技术学报,2015,23(3):344—351]
[32]Nott A,Meislin S H,Moore M J. A quantitative analysis of intron effects on mammalian gene expression [J]. RNA,2003,9(5):607—617
[33]Shi J,Chu W Y,Zhang J S. The functional studies of muscle-specigic microRNAs [J]. Acta Hydrobiologica Sinica,2015,39(6):1224—1230 [石军,褚武英,张建社.肌肉特异表达microRNA的功能研究. 水生生物学报,2015,39(6):1224—1230]
[34]Zhu X,Hu Y,Wang K Z,et al. The expressional characterization of miR-222 in mandarin fish(Siniperca chuatsi)[J]. Acta Hydrobiologica Sinica,2015,39(2):315—320[朱鑫,胡毅,王开卓,等. 翘嘴鳜miR-222的表达特征.水生生物学报,2015,39(2):315—320]
THREE SNPS POLYMORPHISM OF GROWTH HORMONE-RELEASING HORMONE GENE(GHRH) AND ASSOCIATION ANALYSIS WITH GROWTH TRAITS IN CHANNEL CATFISH
ZHANG Shi-Yong1,2,ZHONG Li-Qiang1,2,QIN Qin1,2,WANG Ming-Hua1,2,PAN Jian-Lin1,2,CHEN Xiao-Hui1,2and BIAN Wen-Ji1,2
(1. Freshwater Fisheries Research Institute of Jiangsu Province,Nanjing 210017,China; 2. The Jiangsu Provincial Platform for Conservation and Utilization of Agricultural Germplasm,Nanjing 210014,China)
This study assessed the effects of growth hormone-releasing hormone(GHRH) gene on channel catfish(Ictalurus punctatus) growth traits by identifying SNPs of GHRH gene by The Pooled DNA sequencing using 176 individuals from core breeding population of channel catfish by multiplex SNaPshot genotyping method and analyzing the associations between growth traits with genotypes and diplotypes of GHRH SNPs. Four SNPs(g.4333 A>G,g.4616 A>C,g.4632A>T and g.6301 G>A) were identified in intron,and three of them were genotyped successfully,and there were no strong LD between the SNPs. Six haplotypes and six diplotypes were found in these 176 channel catfish. Association analysis between these SNPs and growth traits showed that the individual with genotype AA of the mutation g.6301 G>A had significantly higher body mass and body length than those with genotype GG and AG(P<0.05);H1/H4 and H1/H5 of six diplotypes had the highest body mass and body length,which were significantly(P<0.01)higher than those of the other diplotypes. This study provide basic knowledge for marker-assisted breeding and QTL of growth traits of channel catfish.
Ictalures punctatus; GHRH; SNP; SNaPshot; Growth traits; Haplotype; Association
Q953
A
1000-3207(2016)05-0886-08
10.7541/2016.114
2015-10-29;
2016-04-15
国家科技支撑计划(2012BAD26B03-04); 江苏省科技支撑计划(BE2013445); 江苏省农业科技自主创新基金[CX(15)1013]; 江苏省水产三新工程项目(Y2014-25); 江苏省六大人才高峰项目(2014-NY-008)资助 [Supported by the National Key Technology R & D Program of China(2012BAD26B03-04); Science and Technology Support Program of Jiangsu Province(BE2013445);Independent Innovation on Agriculture Science and Technology of Jiangsu Province [CX(15)1013]; the Project of Human Resources and Social Security of Jiangsu Province(2014-NY-008)]
张世勇(1987—),男,安徽六安人; 硕士; 研究方向为水产动物遗传育种。E-mail:shiyongzhang@hotmail.com
陈校辉,副研究员,E-mail:cxiaohui416@hotmail.com; 边文冀,研究员,E-mail:js6060@sina.com
