水分胁迫下小麦TaWSI18基因的电子克隆及生物信息学分析
2016-10-18张艳娥张林生朱维宁张大鹏
樊 磊,陈 娟,张艳娥,张林生,朱维宁,张大鹏
(西北农林科技大学生命科学学院 旱区作物逆境生物学国家重点实验室, 陕西 杨凌 712100)
水分胁迫下小麦TaWSI18基因的电子克隆及生物信息学分析
樊磊,陈娟,张艳娥,张林生,朱维宁,张大鹏
(西北农林科技大学生命科学学院 旱区作物逆境生物学国家重点实验室, 陕西 杨凌 712100)

小麦;水分胁迫;TaWSI18基因;电子克隆;生物信息学
小麦是我国主要的粮食作物,非生物胁迫会导致细胞缺水,影响植物的生长发育,诱导多种基因表达[1],产生一系列重要的功能蛋白和调控蛋白,减轻水分胁迫对作物的损害。调控蛋白如转录因子、蛋白激酶等参与信号转导和基因表达调控,调节细胞的新陈代谢、转录并对各种刺激产生应答反应[2-3]。胚胎发育晚期丰富蛋白(Late embryogenesis abundant protein,LEA)自1981年首次在棉花中发现以来[4-5],人们在许多植物中都发现了此类蛋白的存在,尤其是在干旱等逆境胁迫下,发现LEA蛋白表达量升高[6-8]。在水分胁迫条件下,LEA蛋白对酶(如乳酸脱氢酶,苹果酸脱氢酶等)[9-10]和膜结构具有保护作用,增强植物的抗性。张瑞越[11]等从小麦中克隆的水分胁迫诱导基因W89受干旱、低温和ABA的诱导表达。从水稻中分离到水分胁迫诱导基因wsi18,wsi76,wsi724,其中wsi18在水分胁迫时表达量增加[12-13]。植物的水分胁迫应答涉及复杂的调控网络,胁迫机制尚有待进一步研究。本研究运用电子克隆的方法对小麦EST序列数据库进行比对分析,拼接克隆得到WSI18的cDNA序列,利用生物信息学分析其结构与功能,并进行同源性比对,为深入研究小麦水分胁迫诱导基因的功能具有重要意义。
1 材料与方法
1.1材料
供试小麦品种为陕合6号,由西北农林科技大学生命科学学院中心实验室提供。
试剂:大肠杆菌JM109,由西北农林科技大学生命科学学院中心实验室保存;PrimeSTARHS DNA,pMD18-T载体,DNA Marker均购自TaKaRa公司;凝胶回收试剂盒及小量质粒提取试剂盒,均购自天根公司;RNA提取试剂Trizol购自Invitrogen公司。序列测定由华大基因完成。
1.2引物的设计合成
以电子克隆序列为基础,采用OLIGO7软件设计RT-PCR扩增的1对引物,上游引物F:5 ‘ACTTTTCGAGCCCTATCCAGT 3’,下游引物R:5‘AAGATACATCAGGGTCGCCTT 3’。由华大基因合成。
1.3电子克隆的技术路线和方法
目前电子克隆最常用的方法是利用EST信息进行同源性检索、聚类、序列拼装等获得部分乃至全长cDNA序列。本研究参考张德礼等的方法[14],略有改动。在NCBI网站的GenBank数据库中检索小麦或与小麦亲缘关系较近的cDNA序列,以该序列作为种子序列,对小麦EST数据库进行Blast检索,提取高同源性WSI18基因的EST序列,利用CExpress软件进行拼接。再以拼接后的EST作为新的查询探针,重复以上步骤,继续搜索小麦EST数据库,直到没有新的WSI18基因的EST序列可供拼接为止。对拼接后的EST进行ORF预测、Blast及完整性分析。
1.4RNA提取及RT-PCR扩增
使用Trizol试剂盒提取干旱处理24 h的小麦幼苗的总RNA,置于-80℃冰箱中备用。按照TaKaRa公司PrimeScriptRT reagent Kit Perfect Real Time的操作,将2 μl总RNA反转录成cDNA,作为PCR反应的模板。利用PrimeSTARHS DNA高保真酶进行PCR反应,体系为20 μl,程序为98℃ 3 min,98℃ 10 s,56℃ 20 s,72℃ 40 s,35个循环;72℃延伸10 min。最后1.2%琼脂糖凝胶电泳检测RT-PCR扩增结果。
1.5RT-PCR产物胶回收、基因克隆及测序分析
按照胶回收和基因克隆试剂盒说明,进行RT-PCR产物胶回收和基因克隆,获得的目的片段,通过加“A”反应与pMD18-T载体连接,并转化大肠杆菌JM109,在含有氨苄青霉素(Amp)、异丙基-β-D硫代半乳糖苷(IPTG)和5-溴-4-氯-3-吲哚-β-D-半乳糖苷(X-Gal)的LB平板上筛选阳性克隆,对菌落进行PCR鉴定。将鉴定的阳性菌在37℃ 220 r/min 培养过夜,收菌提取质粒,PCR鉴定,然后进行基因测序,将测序结果与电子克隆序列进行比对分析。
1.6小麦TaWSI18基因的生物信息学分析
利用ORF Finder软件对基因ORF进行预测;使用ExPASy在线程序ProtParam对蛋白序列的分子量、等电点、氨基酸组成等基本性质进行预测;ProtScale进行亲/疏水性预测;NetPhos 2.0在线软件对磷酸化位点进行分析;应用SignalP 3.0 Server工具进行信号肽预测;TargetP和PSORT在线分析亚细胞定位;TMHMM工具预测跨膜结构域;并利用神经网络HNN进行二级结构分析。使用ClustalX软件进行氨基酸序列比对,并结合MEGA 5.0软件,基于ML方法及500次bootstrap检测构建系统进化树。
2 结果与分析
2.1小麦TaWSI18基因的电子克隆
以无芒雀麦WSI18基因(登录号AB813835)为探针,从NCBI的小麦EST库中,筛选到两条序列,其登录号分别为BQ166490和GH727079。利用CExpress软件拼接,再以该EST为探针,在小麦EST数据库中Blast检索,重复以上过程,直至没有更多的重叠EST检出。通过ORF Finder软件预测,被筛选而拼接的序列具有完整的ORF(90-767 bp),编码225个氨基酸。以起始密码子的碱基为第1位(标记为+1),在+4位核苷酸为G,-3和-6位核苷酸为G,符合kozak原则(ATG附近的核苷酸序列以ANNATGG和GNNATGG利用率较高)。在3’UTR区有典型的poly A,表明拼接获得了1条完整的cDNA序列,命名为TaWSI18-EST(图1)。

图1电子克隆序列TaWSI18-EST及预测的氨基酸序列
Fig.1Electronic cloning sequence ofTaWSI18-ESTand the prediction of its amino acid sequence
2.2扩增产物序列与电子克隆序列比对分析
以干旱处理的小麦叶片的cDNA为模板,采用基因特异性引物F和R扩增目的片段,此目的片段长度为764 bp(图2),测序获得4条高度同源的TaWSI18基因,分别命名为TaWSI18-1、TaWSI18-2、TaWSI18-3和TaWSI18-4,Blast分析表明,克隆的4条序列与TaWSI-EST序列相似性分别为99%、99%、99%和98%。ClutalX比对分析与TaWSI18-EST序列的差异,以克隆序列的第一个碱基为第一位,TaWSI18-1基因在539、599、647和699位点与其存在碱基差异,752和753位存在两个碱基缺失差异。TaWSI18-2基因在289、290、291、293、539、599、647和699位点存在碱基差异,752和753位点两个碱基缺失差异。TaWSI18-3基因碱基差异位点与TaWSI-like2基因的差异位点相同,但752和753位点与TaWSI18-EST序列相同。TaWSI18-4基因与TaWSI18-EST序列的差异较其他3个基因的大,在25~39、209、288~296、346、362、365、370、499、536和566位点存在差异,40~57位点缺失差异(表1)。利用ORF Finder软件分析,结果表明TaWSI18-1、TaWSI18-2、TaWSI18-3和TaWSI18-4基因ORF框均为678 bp,编码225个氨基酸。Blast分析表明,克隆的基因与TaWSI18-EST蛋白序列相似性分别为99%,99%,99%及97%。ClustalX比对分析,TaWSI18-2和TaWSI18-3基因ORF框一致,编码相同的蛋白。小麦中的WSI18蛋白与TaWSI18-EST氨基酸序列的差异性如表2所示。

注 Note: M—Marker; I—PT-PCR产物 PT-PCR product
图2干旱条件下小麦叶片中TsWS118基因的RT-PCR电泳结果
Fig.2The RT-PCR ofTsWS118 from wheat
leaves under drought conditions
在NCBI网站通过Blast分析,本文克隆的基因与已公布的无芒雀麦的BiWSI18基因(AB813835)、二穗短柄草的lea3基因(XM_003569593.1)、野生水稻(Oryzabrachyacha)的lea3基因(XM_006644519)和日本水稻(OryzasativaJaponica)的WSI18基因(D26536)相似性分别约为94%、90%、90%、90%和90%。并与其他植物的水分胁迫相关的基因具有一定的相似性,证实所得到的基因为水分胁迫诱导基因。
表1小麦的TaWSI18-1、TaWSI18-2、TaWSI18-3、TaWSI18-4基因与电子克隆序列TaWSI18-EST的差异碱基
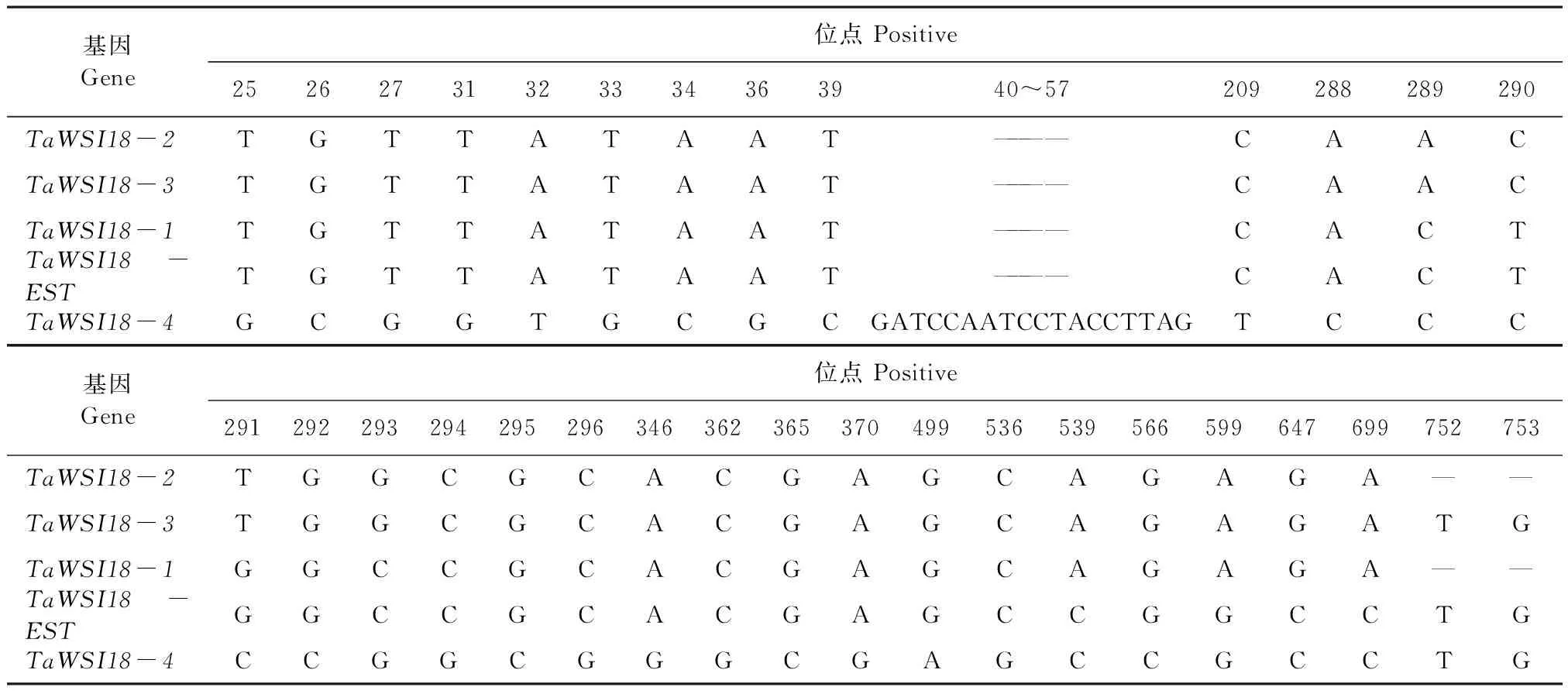
Table 1 The sequence differences between TaWSI18-1, TaWSI18-2, TaWSI18-3,TaWSI18-4 and TaWSI18-EST in wheat

表2 小麦的TaWSI18-1, TaWSI18-2/TaWSI18-3, TaWSI18-4蛋白与电子克隆序列TaWSI18-EST的编码蛋白的氨基酸差异
2.3小麦TaWSI18蛋白特性分析
利用ProtParam在线分析,结果表明TaWSI18-1、TaWSI18-2/TaWSI18-3和TaWSI18-4蛋白的相对分子质量分别为22.75 kD、22.90 kD和22.67 kD,等电点均为6.64。正(Arg+Lys)、负(Asp+Glu)电荷氨基酸残基数目均为31个;不稳定指数分别为4.45、4.37和13.3,表明蛋白均具有稳定性;用Prot Scale预测亲疏水性,结果显示4个蛋白多肽链的第35位表现为最低值-2.522,亲水性最强,第179位表现为最高值1.078,疏水性最强,多肽链的多数位点值均小于0,表现为亲水性(图3);GRAVY值分别为-0.771、-0.786和-0.741,表明蛋白为亲水性。这些性质与水分胁迫诱导性蛋白的稳定性、亲水性的特点相一致,有利于植物在受到胁迫时对细胞的保护作用。
2.4小麦TaWSI18蛋白质的磷酸化位点及信号肽预测分析
运用NetPhos对该蛋白质序列进行潜在磷酸化位点预测分析,结果表明,TaWSI18-1、TaWSI18-2和TaWSI18-3具有5个丝氨酸磷酸化位点、10个苏氨酸磷酸化位点和3个酪氨酸磷酸化位点(图4a)。TaWSI18-4存在4个丝氨酸磷酸化位点、9个苏氨酸磷酸化位点以及2个酪氨酸磷酸化位点(图4b)。同时利用SignalP预测蛋白信号肽结果显示,克隆的这4个蛋白均无信号肽结构域及剪切位点。
2.5小麦TaWSI18蛋白质亚细胞定位及跨膜结构域的预测分析
利用TargetP 1.1 Server和PSORT在线分析小麦TaWSI18蛋白,结果表明这4个蛋白可能定位于细胞质中,通过TMHMM工具预测结果表明,这4个蛋白均无跨膜螺旋区,为非膜蛋白。
2.6小麦TaWSI18蛋白质的二级结构分析
神经网络HNN对TaWSI18-1、TaWSI18-2/TaWSI18-3和TaWSI18-4蛋白二级结构的预测表明,TaWSI18-1和TaWSI18-2/TaWSI18-3蛋白的α螺旋、延伸链和无规则卷曲的比例均为56%、7.11%及36.89%(图5a),TaWSI18-4蛋白α螺旋的比例为50.67%,延伸链为6.67%,无规则卷曲为42.67%(图5b)。

图3小麦TaWSI18蛋白的亲水/疏水性预测
Fig.3Hydrophilic/hydrophobicity analysis of TaWSI18 protein inTriticumaestivum
2.7TaWSI18蛋白序列比对及聚类分析

利用MEGA 5.0软件进行聚类分析表明(图7),小麦中的WSI18蛋白同源性最高,在不同物种间,与禾本科植物的相似性也颇高,其结果与小麦和这些物种同属于禾木科植物的传统分类相一致。其中与无芒雀麦相似性最高,与二穗短柄草次之。芜菁与拟南芥这两个双子叶十字花科则形成了另外的独立分支。聚类分析基本上代表了它们在经典分类上的地位。

图4小麦TaWSI18蛋白的磷酸化位点预测
Fig.4Phosphorylation sites prediction of TaWSI18 protein inTriticumaestivum
3 讨 论
小麦是异源六倍体作物,含有42条染色体,其DNA含有大量的核苷酸重复,本实验以1条电子克隆序列为基础,设计1对特异引物,克隆得到4条高度同源的WSI18基因,这与小麦庞大的基因组及其异源六倍体有很多相关性。


图5小麦TaWSI18蛋白质的二级结构分析
Fig.5Secondary structure of TaWSI18 protein inTriticumaestivum
通过生物信息学分析,小麦TaWSI18蛋白相对分子量较小,蛋白序列缺少Cys、Trp和Phe,富含Ala、Gly、Lys和Thr,具有稳定性和亲水性,与已证实的LEA蛋白的序列特征极为相似[6,15]。Dure L对LEA3蛋白的11个氨基酸残基进行生物信息学分析认为,该肽段以α螺旋形式存在[16],圆二色谱和红外光谱对LEA3蛋白的结构分析表明,该类蛋白在水溶液中以无序化结构存在,而在具有甲醇、蔗糖、甘油、乙烯、乙二醇或者脱水状态时,则形成α螺旋结构[17-20],TFE和SDS也能够促使LEA3蛋白螺旋化[20]。许多研究还发现,缺水和复水条件下,LEA3蛋白结构的螺旋化与无序化之间的转化有利于其在细胞中行使多种功能[21]。HNN和GOR对TaWSI18蛋白的二级结构分析表明该类蛋白主要以α螺旋和无规则卷曲形式存在。TaWSI18蛋白的基本特点和二级结构与LEA3蛋白类似,推测其在细胞当中与LEA3蛋白具有相同或相似的功能。
Takahashi R等[12]发现,水稻的WSI18基因其蛋白序列的N端区域与LEA3蛋白高度同源。对该基因的启动子分析表明,其含有ABA响应元件,利用瞬时表达研究WSI18启动子和缺失功能元件启动子,结果表明,水稻的WSI18基因受干旱和ABA胁迫响应[13,22]。推测小麦TaWSI18基因的表达可能受到干旱和ABA等非生物胁迫的诱导表达,目前对小麦的TaWSI18基因的功能研究甚少,小麦基因组的庞大与复杂使重要功能基因的克隆存在困难。但随着测序技术的发展,新型大容量基因组cDNA文库的构建以及各种分子标记技术的发展,基因组文库和EST库的建立,为基因的克隆及其重要功能研究提供便利。EST序列电子克隆基因为进一步深入研究WSI18基因提供参考和奠定基础。
图6ClustalX比对分析小麦TaWSI18蛋白与LEA蛋白氨基酸序列(黑色框内为N端保守区)
Fig.6Protein sequence multi-alignment of TaWSI18 proteins and LEA protein ofTriticumaestivum
using ClustalX (The black box for N-terminal conservative region)
[1]杨献光,梁卫红,齐志广,等.植物非生物胁迫应答的分子机制[J].麦类作物学报,2006,26(6):158-161.
[2]Kaur N, Gupta A K. Signal transduction pathways under abiotic stresses in plants[J]. Current Science, 2005,88(11):1771-1780.
[3]Shinozaki K, Yamaguchi Shinozaki K. Gene expression and signal transduction in water-stress response[J]. Plant Physiology, 1997,115(2):327-334.
[4]Dure L, Chlan C. Developmental biochemistry of cottonseed embryogenesis and germination: XII. Purification and properties of principal storage proteins[J]. Plant physiology, 1981,68(1):180-186.
[5]Dure L, Galau G A. Developmental biochemistry of cottonseed embryogenesis and germination: XIII. Regulation of biosynthesis of principal storage proteins[J]. Plant physiology, 1981,68(1):187-194.
[6]Battaglia M, Olvera-Carrillo Y, Garciarrubio A, et al. The enigmatic LEA proteins and other hydrophilins[J]. Plant Physiology, 2008,148(1):6-24.
[7]Dalal M, Kumar G S, Mayandi K. Identification and expression analysis of group 3 LEA family genes inSorghumbicolor(L.)[J]. Moench Acta Physiologiae Plantarum, 2013,35(3):979-984.
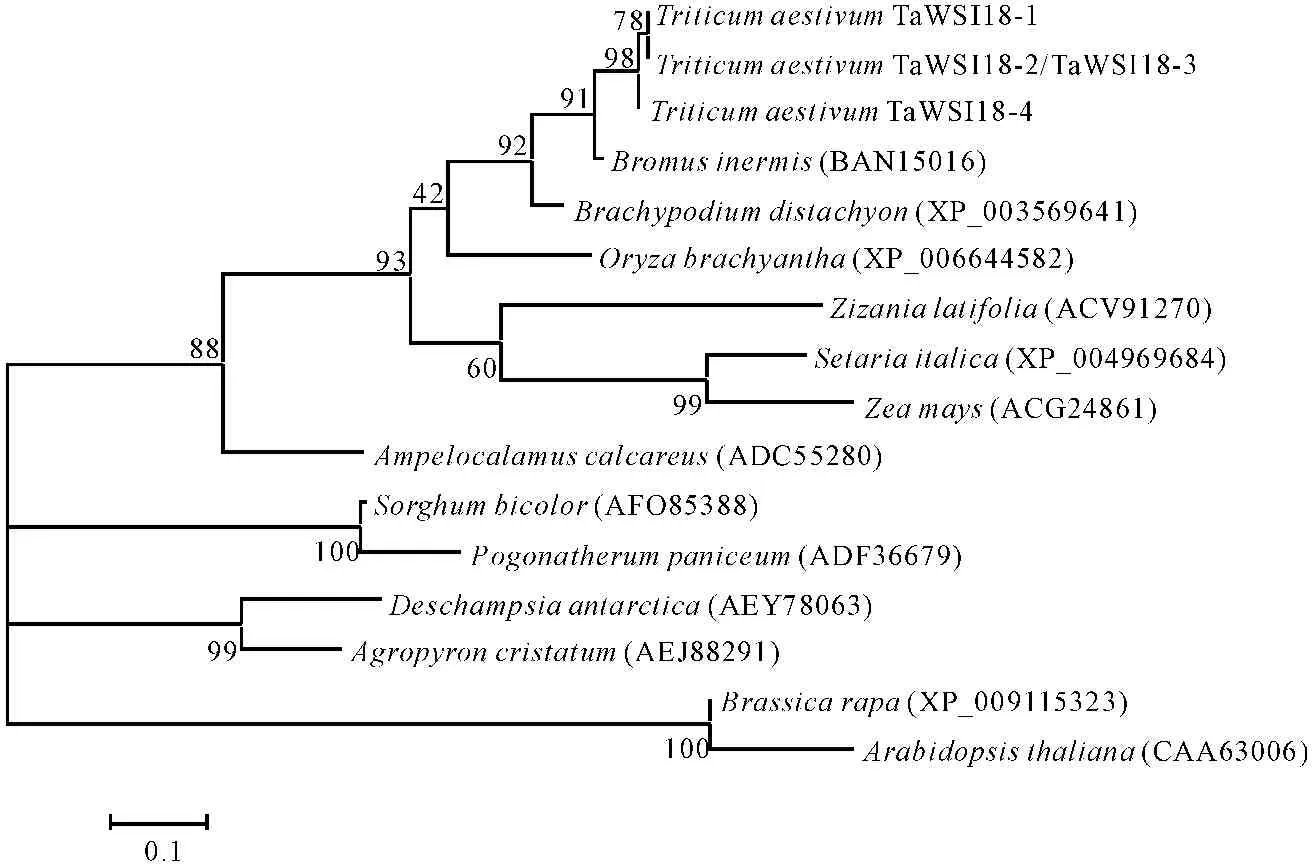
图7系统进化分析
Fig.7Phylogenetic tree of TaWSI18 and other homologous sequences
[8]Vaseva I I, Anders I, Feller U. Identification and expression of different dehydrin subclasses involved in the drought response of Trifolium repens[J]. Journal of Plant Physiology, 2014,171(3-4):213-224.
[9]Amara I, Odena A, Oliveira E, et al. Insights into maize LEA proteins: from proteomics to functional approaches[J]. Plant and Cell Physiology, 2012,53(2):312-329.
[10]Hatanaka R, Hagiwara-Komoda Y, Furuki T, et al. An abundant LEA protein in the anhydrobiotic midge, PvLEA4, acts as a molecular shield by limiting growth of aggregating protein particles[J]. Insect Biochemistry and Molecular Biology, 2013,43(11):1055-1067.
[11]张瑞越,徐兆师,李连城,等.一个新的小麦非生物胁迫诱导基因的克隆及表达特性[J].中国农业科学,2007,40(5):875-881.
[12]Takahashi R, Joshee N, Kitagawa Y. Induction of chilling resistance bywater-stress, and cdna sequence-analysis and expression of water stress-regulated genes in rice[J]. Plant Molecular Biology, 1994,26(1):339-352.
[13]Joshee N, Kisaka H, Kitagawa Y. Isolation and characterization of a water stress-specific genomic gene, pwsi 18, from rice[J]. Plant and Cell Physiology, 1998,39(1):64-72.
[14]张德礼,孙晓静,凌伦奖,等.人类SR蛋白超家族新成员—SFRS12(SRrp508)的基因克隆和特性分析[J].遗传学报,2002,29(5):377-383.
[15]Garay-Arroyo A, Colmenero-Flores J. M, Garciarrubio A, et al. Highly hydrophilic proteins in prokaryotes and eukaryotes are common during conditions of water deficit[J]. Journal of Biological Chemistry, 2000,275(8):5668-5674.
[16]Dure L. A repeating 11-MER Amino-acid motif and plant desiccation[J]. Plant Journal, 1993,3(3):363-369.
[17]Dure L. Occurrence of a repeating 11-mer amino acid sequence motif in diverse organisms[J]. Protein and Peptide Letters, 2001,8(2):115-122.
[18]Goyal K, Tisi L, Basran A, et al. Transition from natively unfolded to folded state induced by desiccation in an anhydrobiotic nematode protein[J]. Journal of Biological Chemistry, 2003,278(15):12977-12984.
[19]Tolleter D, Jaquinod M, Mangavel C, et al. Structure and function of a mitochondrial late embryogenesis abundant protein are revealed by desiccation[J]. Plant Cell, 2007,19(5):1580-1589.
[20]Wolkers W F, McCready S, Brandt W F, et al. Isolation and characterization of a D-7 LEA protein from pollen that stabilizes glasses in vitro[J]. Biochimica Et Biophysica Acta-Protein Structure and Molecular Enzymology, 2001,1544(1-2):196-206.
[21]Sun X, Rikkerink E H A, Jones W T. Multifarious roles of intrinsic disorder in proteins illustrate its broad impact on plant biology[J]. Plant Cell, 2013,25(1):38-55.
[22]Zhu W N, Zhang L S, Lv H, et al. The dehydrin wzy2 promoter from wheat defines its contribution to stress tolerance[J]. Functional & Integrative Genomics, 2014,14(1):111-125.
Identification and characterization of water stress-induced TaWSI18 gene in silico cloning fromTriticumaestivumand its bioinformatic analysis
FAN Lei, CHEN Juan, ZHANG Yan-e, ZHANG Lin-sheng, ZHU Wei-ning, ZHANG Da-peng
(StateKeyLaboratoryofCropStressBiologyinAridArea,CollegeofLifeSciences,NorthwestA&FUniversity,Yangling,Shaanxi712100,China)
WSI18, a water stress-induced genes, plays an important role in plants under water deficit. Silico cloning and RT-PCR were taken to isolate four new highly homologousWSI18 genes:TaWSI18-1,TaWSI18-2,TaWSI18-3 andTaWSI18-4 (GenBank accession number: KP226849, KP226850, KP226851and KP226852) in wheat, ORFs of which are all 678 bp encoding 225 amino acids. Bioinformatic analysis showed that they are highly hydrophilic and have multiple phosphorylation sites. They are located to cytoplasm and have no transmembrane domain signal peptides, as well as no splicing sites. The proportion ofαhelix and random coil are over 90% by a secondary structure prediction. Homology comparison and phylogenetic analysis showed that TaWSI18 proteins are highly homologous to WSI18 ofBromusinermisand LEA3Brachypodiumdistachyon, sharing 94% and 70% similarities respectively. In addition, TaWSI18 proteins have conserved amino acid sequences common to LEA protein in the N-terminal region. They also have a similar character to LEA3 protein by bioinformatic analysis that TaWSI18 proteins may have the same or similar function to LEA3 protein, which lies a foundation for the future study on its function under water-stress.
Triticumaestivum; water stress;TaWSI18; silico cloning; bioinformatics
1000-7601(2016)04-0069-08
10.7606/j.issn.1000-7601.2016.04.11
2015-05-22
国家自然科学基金资助项目(31071349);高等学校博士学科点专项基金(20120204110033)
樊磊(1989—),女,山西运城人,硕士研究生,研究方向为小麦抗旱相关基因的研究。 E-mail: echo18735439872@163.com。
张林生(1958—),教授,博士生导师,主要从事小麦抗旱方面的研究。 E-mail: linszhang@nwsuaf.edu.cn。
S512.1
A
