凡纳滨对虾选育系间杂交的生长性状及遗传多样性分析
2016-10-18吴怡迪杨章武黄永春游伟伟
吴怡迪,骆 轩,杨章武,黄永春,游伟伟*
(1.厦门大学海洋与地球学院,福建厦门361102;2.福建省水产研究所,福建厦门361013;3.集美大学水产学院,福建厦门361021)

凡纳滨对虾选育系间杂交的生长性状及遗传多样性分析
吴怡迪1,骆轩1,杨章武2,黄永春3,游伟伟1*
(1.厦门大学海洋与地球学院,福建厦门361102;2.福建省水产研究所,福建厦门361013;3.集美大学水产学院,福建厦门361021)
对2个凡纳滨对虾(Litopenaeusvannamei)选育系(A和B)的自繁与杂交后代的生长性状及遗传多样性进行了分析.养殖对比实验结果表明,杂交组合AB生长性状优于其他各组,表现出较好的杂种优势.使用11对荧光标记的微卫星引物对4个凡纳滨对虾自繁与杂交群体以及1个从美国引进的初代亲本SIS群体的基因组DNA进行扩增,结果显示除AA群体外,其余4个群体均表现出较丰富的遗传多样性,观测到的平均等位基因数(Na)为4.273~5.636,平均期望杂合度(He)为0.586~0.629,平均多态信息含量(PIC)为0.512~0.556.遗传距离分析结果显示,SIS群体和AA群体的遗传距离最远(0.670 4),而与AB群体的最近(0.131 4).研究结果表明,引自美国的凡纳滨对虾群体经多代自繁后,其遗传多样性水平降低;而选育系间杂交则可使杂交后代的生长性状和遗传多样性水平得到改善.
凡纳滨对虾;生长性状;遗传多样性;微卫星;杂交
凡纳滨对虾(Litopenaeusvannamei)又名南美白对虾,原产自南太平洋和美洲沿海水域,自1988年引入我国以来,已成为我国水产养殖的主要品种[1].作为水产经济虾类的重要养殖品种,我国每年的凡纳滨对虾种苗需求量高达4 000亿尾以上[2].然而近年来由于各种虾类疾病相继入侵及养殖过程中频繁近交导致的种质退化等原因,我国凡纳滨对虾在养殖过程中出现了个体小型化、生长速度慢、抗病性差等问题.因此,从分子水平上了解现有凡纳滨对虾群体的遗传变异情况,并在此基础上培育适合福建海域养殖的凡纳滨对虾新品种显得十分迫切.
微卫星标记技术作为第二代分子标记的代表,已被广泛应用于水产生物的遗传分析研究中[3].Maggioni等[4]用10个微卫星标记对巴西9个凡纳滨对虾养殖场的亲虾群体进行评估,结果显示9个亲虾群体的遗传多样性程度与来自中美洲的野生群体一致,处于较高水平.Artiles等[5]用4个微卫星标记对古巴1个凡纳滨对虾引进群体的不同世代进行遗传多样性分析,结果表明各后代群体的遗传多样性水平近似,但后代群体与基础群体间存在明显的遗传差异.此外,还有研究用微卫星标记对凡纳滨对虾的遗传多样性进行了分析,发现随繁育世代增加,养殖群体后代的遗传多样性呈下降趋势,并建议在后续繁育工作中通过增加杂交来改善或避免近交衰退的现象[6].
2008年起,厦门市厦兴龙水产种苗有限公司与福建省水产研究所等单位合作开展凡纳滨对虾良种选育工作.本研究针对该育种项目中的4个群体和1个美国引进群体,采用微卫星标记对其遗传结构和遗传多样性进行分析,从而监测该育种项目中各对虾群体的遗传变异情况.
1 材料与方法
1.1实验材料
实验所用的2个选育群体(A和B)以及它们的2个杂交群体取自厦门市厦兴龙水产种苗有限公司,初代亲虾群体则引自美国SIS公司(Shrimp Improvement System,USA),命名为SIS.选育系A来源于2008年由美国SIS公司引进亲虾的子一代,选育系B来源于2010年引自广东的另一个凡纳滨对虾繁育群体,随后针对生长与抗病性状对2个群体进行了连续多代的群体选育.2014年,用选育系A的F6代群体以及选育系B的F4代群体作为亲本,采用双列杂交获得了它们的自繁以及正、反杂交后代,并分别命名为AA(A♀×A♂)、BB(B♀×B♂)、AB(A♀×B♂,正交)和BA(B♀×A♂,反交).5个凡纳滨对虾群体分别于2014年7—9月随机取样30尾,活体运回实验室后,用无菌手术剪和镊子取对虾的尾节肌肉,保存于95%(体积分数)乙醇中备用.
表1 凡纳滨对虾11对微卫星引物的信息
Tab.1 The information for 11 microsatellite primer pairs of L. vannamei

位点荧光标记类型引物序列(5'→3')退火温度/℃片段大小/bpGenBank登录号TUMXLv7.121FAMF:GGCACACTGTTTAGTCCTCGR:CGAACAGAATGGCAGAGGAG56192~246AF360043TUMXLv7.56ROXF:CCATGGCTTTCCTCTTCTTTCR:AGGTAGGGAAGTCGTGAGGG62280~491AF360055TUMXLv7.97FAMF:TGTCGTTAGTGCAGCTCATTCR:GGGGAGGAATAAGAGGAAAGG52160~181AF360057TUMXLv8.25HEXF:ATTCTTTGTGTTTCTTCGCCR:CGTCCCTGAAACTTTATCTCC52104~111AF360075Pvan1815HEXF:GATCATTCGCCCCTCTTTTTR:ATCTACGGTTCGAGAGCAGA55126~139AY062925TUMXLv8.176HEXF:GCAACGCAATATAGCTCR:TCAAGGGAACAAAGTCAAG52162~166AF360063TUMXLv9.178ROXF:CATTGAAAACGGAATCCTCGR:GATATTCCCATCAACACAGCG57194~200AF360105TUMXLv10.207FAMF:GATCACTAGCCATATTTCATCCR:ATCGCATAATGAGCAAACTGG5681~107AF359963TUMXLv10.255HEXF:CTAAATAAATCACGGGTTGGGR:CCTTCTGGTTTACTGTTGAGGC57208~215AF359977TUMXLv10.312HEXF:ATACGAAACACCCCATCCCR:GTGGTCTTACCTCGTGGCTC59165~177AF359989TUMXLv10.481FAMF:CATAAGACTGCACACGTAGCGR:TTTAAAACGTGGTGTTCTGT-GG57207~213AF360009
1.2实验方法
1.2.14个群体生长指标的跟踪测量
4个群体的养殖对比实验于2014年7—11月在厦兴龙水产种苗有限公司的晋江基地进行.2014年7月31日,将每个交配组合的仔虾分别养殖在室外水泥池,每池6 m2,投苗密度100尾/m2,每组设置2个对照池.养殖期间随对虾生长阶段进行定期投饵,并保持不间断充气.在养殖40,60,80和100 d时对各群体对虾进行取样测量,每池取30尾,用游标卡尺(精确度0.01 mm)和电子天平(精确度0.01 g)对其体长、头胸甲长和体质量3个指标进行测量并记录.
1.2.2微卫星分析
基因组DNA的提取:实验所用凡纳滨对虾的基因组DNA采用北京天根生化科技有限公司的海洋动物组织基因组DNA提取试剂盒进行提取,并用1.2%(质量分数)琼脂糖凝胶电泳和NanoDrop2000分光光度计分别检测所提取基因组DNA的质量和浓度,之后于-20 ℃保存备用.
微卫星引物的筛选及PCR扩增:实验所用微卫星引物参考文献[7],选取并合成了44对引物进行PCR扩增,从中挑选出11对特异性强、多态性较高且重复性好的微卫星引物(表1),并在每对引物的5′端设计添加不同类型的特异性荧光标记(FAM、HEX或ROX)用于后期混样检测目的条带,再交由上海生工生物工程股份有限公司合成.
PCR反应体系(25 μL):10×PCR Buffer (含Mg2+) 2.5 μL,dNTPs(2.5 mmol/L) 2 μL,正、反向引物(10 mmol/L)各1 μL,TaqDNA聚合酶(5 U/μL) 0.25 μL,模板DNA(80~100 ng/μL) 1 μL,加ddH2O补足至25 μL.PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,相应退火温度退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸7 min.PCR产物经电泳检测和筛选后,委托上海生工生物工程股份有限公司进行短串联重复序列(STR)分型分析.
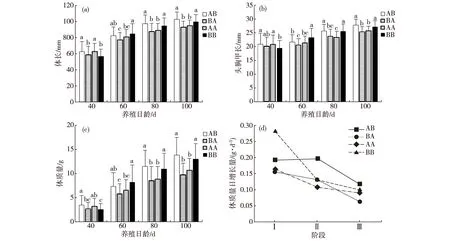
不同小写字母表示差异显著(p<0.05).Ⅰ.09-10—09-30;Ⅱ.09-31—10-21;Ⅲ.10-22—11-10.图1 4个群体的体长(a)、头胸甲长(b)、体质量(c)和体质量日增长量(d)Fig.1Body length(a),carapace length(b),body weight(c) and daily growth of body weight(d) for the four populations
1.2.3数据统计与分析
4个群体的生长数据用PASWStatistics18软件进行比较分析,群体间各指标的比较采用单因素方差分析进行,差异显著性水平为p<0.05.根据Falconer[8]的方法,对2个杂交组合AB和BA的中亲杂种优势率HMP及单亲杂种优势率HA、HB进行计算.
微卫星数据用PopGene 3.2软件进行统计分析,计算5个群体各微卫星位点的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)和期望杂合度(He),用以评估各群体的遗传多样性;Hardy-Weinberg(H-W)平衡检验和固定指数(Fis)的计算则分别用以检测Ho和He之间的差异和评估杂合子缺失情况;计算两两群体间的Nei′s无偏遗传距离(Ds)用以评估群体间的遗传差异.根据Botstein等[9]的方法,采用PIC-CALC 0.6软件,依据等位基因频率计算各个位点的多态信息含量(PIC).群体间遗传分化指数(FST)、群体内和群体间的分子变异分析(AMOVA)均采用Arlequin 3.5软件进行.
2 结果与分析
2.1自繁及杂交群体的生长及杂种优势分析
各取样时期内4个选育群体的体长、头胸甲长和体质量数据如图1(a)、(b) 和(c)所示.由图中可见,4个群体的各指标都随生长天数的增加而增加.对群体间各指标进行单因素方差分析,结果显示:在养殖40 d时,AB和AA群体的各项指标均高于另外2个群体(BA和BB),且AB群体与这2个群体差异显著;而至80 d后AB和BB群体的各指标显著高于BA和AA群体.图1(d)中体质量日增长量的统计分析结果表明:AA、BB和BA 3个群体的体质量日增长量随生长天数的增加而下降;自繁子代群体AA和BB的体质量日增长量在Ⅰ至Ⅱ阶段明显下降,其中BB群体的体质量日增长量下降了53.7%;杂交子代群体AB和BA的下降幅度较小,其中AB群体的体质量日增长量在Ⅰ至Ⅱ阶段基本保持不变.
杂交群体杂种优势率的分析结果表明,40~80 d各生长指标的HMP都随生长天数的增加而呈先降低后上升的趋势(表2).2个杂交群体在60 d时都表现出负向的中亲杂种优势,且整个过程中BA群体的HMP几乎全部为负值,即表现出杂种劣势.另外,2个杂交群体子代的HMP都介于HA和HB之间,且HA和HB之间存在明显差异.
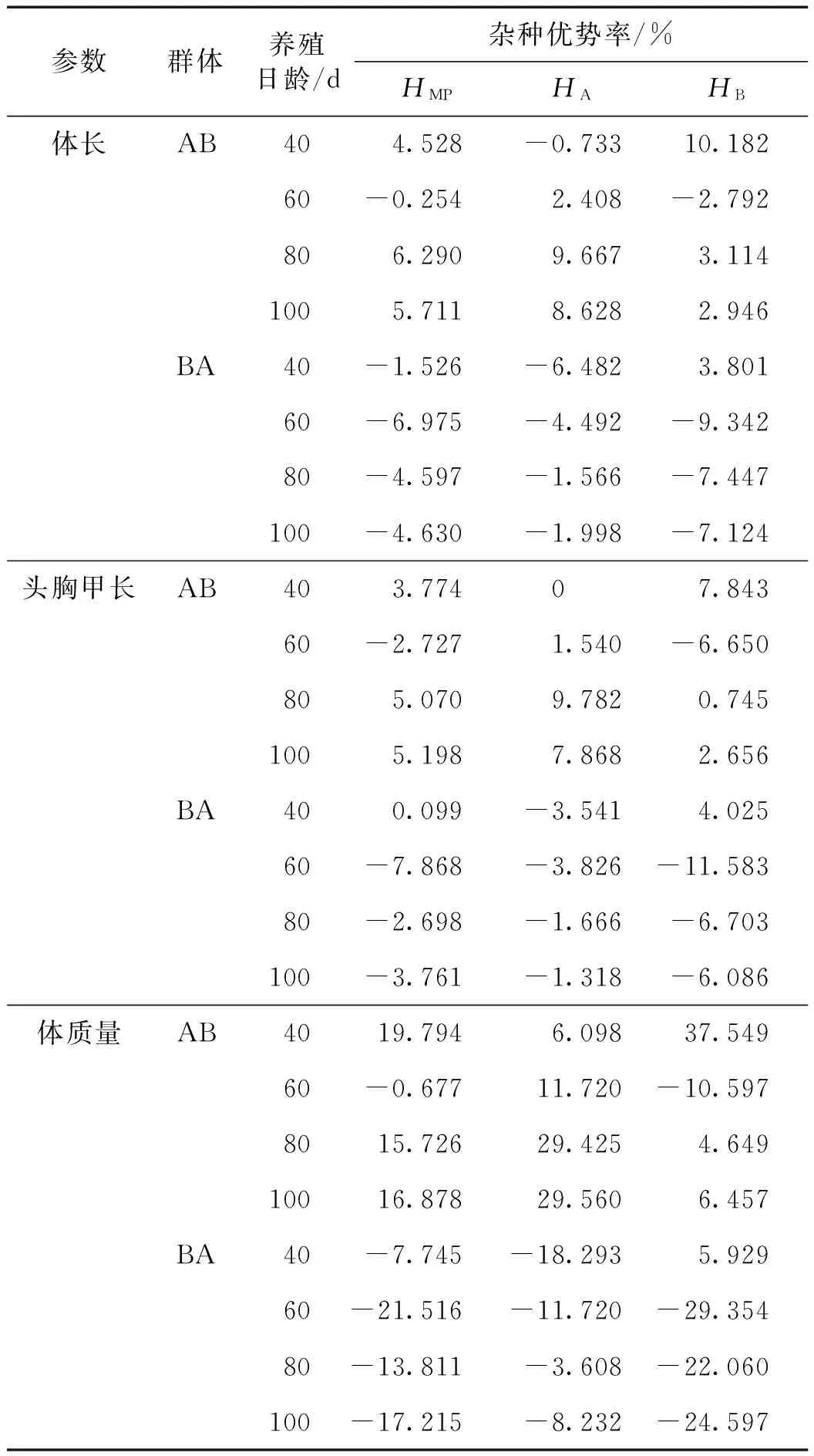
表2 2个杂交群体体长、头胸甲长和体质量的杂种优势率值
2.2凡纳滨对虾群体的遗传多样性
11对微卫星引物在5个凡纳滨对虾群体中均能扩增出目的条带.表3为5个群体在各微卫星位点上的遗传多样性信息,结果显示:11个微卫星位点上共检测到81个等位基因,在5个群体中,位点TUMXLv7.121、TUMXLv7.56、Pvan1815和TUMXLv10.312的PIC均高于0.5,位点TUMXLv10.207和TUMXLv9.178的PIC分别在AA和SIS群体中小于0.5,其余位点的PIC均介于0.25~0.5之间.5个群体的平均Fis均为正值,且在位点TUMXLv9.178的Fis也均为正值;5个群体在11个微卫星位点的平均Na为3.909~5.636,平均He为0.561~0.629,平均PIC为0.493~0.556;其中AB群体平均Na和Ho最高(Na=5.636,Ho=0.600),BB群体平均He和PIC最高(He=0.629,PIC为0.556),AA群体除平均Fis值外,各项遗传多样性指标最低,SIS群体各项遗传多样性指标都低于AB和BB群体并高于BA和AA群体;5个群体在11个微卫星位点上的H-W平衡遗传偏离指数(HWD)显示,2个自繁子代群体AA和BB在多数微卫星位点上都显著偏离平衡(p<0.05).
2.3群体遗传分化分析
5个凡纳滨对虾群体两两对比的FST和基于等位基因频率计算得到的各群体间的Ds见表4:各群体间FST和Ds分别介于0.037 5~0.254 8和0.064 0~0.670 4之间.5个群体中FST且Ds最大的是AA与SIS群体(FST=0.254 8,Ds=0.670 4),AB与BA群体之间的遗传分化最小(FST=0.037 5).AMOVA结果显示,81.62%的遗传变异源于群体内,18.38%来自群体间(表5).
3 讨 论
3.14个交配组合群体的生长及杂种优势情况
生长性状对比结果表明,整个生长过程中正交子代AB群体的生长相比其他3个群体具有明显的优势, 而AA群体和反交子代BA群体的各项生长指标都较低.不同亲本所获子代的性状表现有显著差异,这种正反交性状的不对称性现象在水产生物的育种中已有不少报道[10-11].此外,在养殖的前2个阶段,2个自繁群体的体质量日增长量随日龄的增加呈明显的下降趋势,而2个杂交群体的下降值低于2个自繁群体,这在一定程度上说明杂交子代在养殖早期的生长较稳定.
杂种优势分析结果表明,AB群体获得了较高的与生长性状相关的杂种优势,说明选育系间杂交是提高凡纳滨对虾养殖性能的一个有效途径,这与林红军等[12]的研究结果一致.2个杂交子代的HMP在40~80 d内呈现先降低后上升的趋势,可见杂种优势在早期并不稳定,而到了后期又逐渐恢复并保持稳定状态,这种早期不稳定的现象可能与杂种优势在早期被母性效应掩盖有关[13].BA群体表现出杂种劣势的现象则进一步说明杂交过程中母本的选择以及杂交配对起着非常重要的作用.

表3 5个凡纳滨对虾群体在11个微卫星位点上的遗传多样性信息
注:*p<0.05,**p<0.01.

表4 5个凡纳滨对虾群体的FST及Ds

表5 5个凡纳滨对虾群体的AMOVA
3.25个凡纳滨对虾群体的遗传多样性
微卫星标记是分析生物遗传多样性和遗传差异的有效工具,衡量微卫星位点多态性的一个重要指标是PIC.本研究中的11个微卫星位点在5个凡纳滨对虾群体中均表现出多态性,根据Botstein等[9]的划分标准,位点TUMXLv7.121、TUMXLv7.56、Pvan1815和TUMXLv10.312表现出较高的多态性(PIC>0.5),TUMXLv10.207和TUMXLv9.178分别在AA和SIS群体中表现出较低的多态性(PIC<0.25),其余位点均为中度多态性.5个群体在这11个微卫星位点上的平均PIC介于0.512~0.556之间,具有较高的多态性.
在遗传育种工作中,明确基础群体的遗传多样性情况,并采用遗传多样性水平较高的群体作为亲代选育,将增强后代对环境的适应性.Na和He是评价群体遗传多样性的重要指标,杂合度的评估中Ho易受样本大小的影响,而He更能反映群体的遗传多样性[14].本研究中,平均Na最高的是杂交子代AB群体(Na=5.636,He=0.606),平均He最高的是BB群体(Na=4.454,He=0.629),这2个群体的遗传多样性参数水平略高于冯娜娜等[15]和Vela Avitúa等[16]所研究的国内及墨西哥的凡纳滨对虾养殖群体,而低于Lima等[17]研究的巴西养殖群体.
对各群体H-W平衡检验结果表明,AA和BB群体在多数位点上都偏离了平衡.群体的Fis也代表其基因纯合度[18],5个凡纳滨对虾群体的平均Fis均为正值表明各群体均存在不同程度的近交及杂合子缺失现象,其中AB群体的近交程度最低(Fis=0.017),SIS群体次之(Fis=0.057),AA群体近交程度最高(Fis=0.151),说明AA群体的基因型已经达到了相对较纯合的状态,这与Knibb等[19]的研究结果一致.另外,位点TUMXLv9.178的Fis在所有群体中均为正值,表明该位点存在部分纯合子过剩,这可能是由于无效等位基因的存在所致[20].
正交子代AB群体遗传多样性水平在各群体中最高,而反交子代BA群体相对较低,这可能与亲本对子代基因库的贡献率不同相关[21],而亲本遗传贡献率的差异则是受精率、成活率等多种因素综合影响的结果.2个选育系杂交子代与自繁子代遗传多样性水平上的差异表明,选育系间杂交可使后代的遗传多样性水平得到明显改良.另外,SIS群体的遗传多样性低于AB和BB群体的现象,从一定程度上证实了目前国内凡纳滨对虾亲虾市场上关于近年来SIS公司亲虾质量下降的报道[22].
3.35个凡纳滨对虾群体的遗传差异
在水产生物育种过程中,亲本的选择除了要保证高的遗传多样性水平之外,还应考虑遗传距离及遗传分化程度的大小.选择遗传距离远且具有良好性状的凡纳滨对虾进行杂交试验,并评估最优组合,是培育优良新品种的有效途径[23].
本研究对5个凡纳滨对虾群体FST和Ds的分析显示,选育系A虽然也源自于美国,但经过多代的自繁选育后已与新引进的美国SIS亲代群体之间发生了明显的遗传分化(FST=0.254 8,Ds=0.670 4),这表明长期的自繁会导致同一地区来源的凡纳滨对虾后代之间发生明显的遗传分化,这种情况在罗氏沼虾(Macrobrachiumrosenbergii)[24]中也有发现.另外,2个杂交子代群体(AB和BA)和2个自交子代群体(AA和BB)之间出现了高度遗传分化(FST>0.20,Ds>0.4),而2个杂交子代群体与SIS群体之间则为中度遗传分化(0.05 综上可见,引进的亲虾群体经多代自繁后,其后代的遗传多样性有所下降,但基因型的纯合度得到提高.采用多代自繁的凡纳滨对虾纯合选育系进行选育系间杂交,其杂交后代能够表现出一定的杂种优势,同时遗传多样性水平也得到一定程度的提高,这有望成为培育凡纳滨对虾新品种(系)的一个有效途径. [1]于洋.凡纳滨对虾分子标记的开发及其在遗传育种中的应用[D].青岛:中国科学院研究生院 (海洋研究所),2014:1-6. [2]刘建勇.南美白对虾优质种苗培育技术(上)[J].当代水产,2014(1):1-3. [3]VASEEHARAN B,RAJAKAMARAN P,JAYASEELAN D,et al.Molecular markers and their application in genetic diversity of penaeid shrimp[J].Aquaculture International,2013,21(2):219-241. [4]MAGGIONI R,COIMBRA M R M,COSTA R B,et al.Genetic variability of marine shrimp in the Brazilian industry[J].Pesquisa Agropecuria Brasileira,2013,48(8):968-974. [5]ARTILES A,COBO R,BENTEZ L,et al.Assessment of genetic variation and productive markers through four progenies of the first introduced stock of cultured shrimpPenaeus(Litopenaeus)vannameiin Cuba[J].International Journal of Aquaculture,2015,5(23):1-12. [6]LUVESUTO E,FREITAS P D,GALETTI JUNIOR P M.Genetic variation in a closed line of the white shrimpLitopenaeusvannamei(Penaeidae)[J].Genetics and Molecular Biology,2007,30(4):1156-1160. [7]MEEHAN D,XU Z,ZUNIGA G,et al.High frequency and large number of polymorphic microsatellites in cultured shrimp,Penaeus(Litopenaeus)vannamei[Crustacea:Decapoda][J].Marine Biotechnology,2003,5(4):311-330. [8]FALCONER D S.Introduction to quantitative genetics[M].New York:The Ronald Press Company,1960:365. [9]BOTSTEIN D,WHITE R L,SKOLNICK M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314. [10]秦艳杰,刘晓,张海滨,等.海湾扇贝正反交两个家系形态学指标比较分析[J].海洋科学,2007,31(3):22-27. [11]李莉好,喻达辉,黄桂菊,等.尼罗罗非鱼、奥利亚罗非鱼及其正、反杂交群体的遗传多样性[J].中国水产科学,2008,15(4):585-592. [12]林红军,沈琪,张吕平,等.凡纳滨对虾生长性状的双列杂交分析[J].热带海洋学报,2010,29(6):51-56. [13]YOU W W,KE C H,LUO X,et al.Growth and survival of three small abalone Haliotis diversicolor populations and their reciprocal crosses[J].Aquaculture Research,2009,40(13):1474-1480. [14]包文斌,束婧婷,许盛海,等.样本量和性比对微卫星分析中群体遗传多样性指标的影响[J].中国畜牧杂志,2007,43(1):6-9. [15]冯娜娜,徐真,马洪雨,等.凡纳滨对虾7个不同家系遗传差异的微卫星标记分析[J].生物技术通报,2012,11:133-138. [17]LIMA A P S,SILVA S M B C,OLIVEIRA K K C,et al.Genetics of two marine shrimp hatcheries of the Pacific white shrimpLitopenaeusvannamei(Boone,1931) in Pernambuco,Brazil[J].Ciência Rural,2010,40(2):295-301. [18]陈晓汉,曾地刚,李咏梅,等.凡纳滨对虾遗传多样性的微卫星 DNA 分析[J].广西农业科学,2006,37(5):579-583. [19]KNIBB W,WHATMORE P,LAMONT R,et al.Can genetic diversity be maintained in long term mass selected populations without pedigree information?A case study using banana shrimpFenneropenaeusmerguiensis[J].Aquaculture,2014,428:71-78. [20]PEREZ-ENRIQUEZ R,HERNNDEZ-MARTNEZ F,CRUZ P.Genetic diversity status of white shrimpPenaeus(Litopenaeus)vannameibroodstock in Mexico[J].Aquaculture,2009,297(1):44-50. [21]王鸿霞,吴长功,相建海.凡纳滨对虾繁殖中不同亲本对子代遗传贡献率的差异[J].动物学报,2006,52(1):175-181. [22]吴群风.叶富良坦言:SIS默认选育路线失当,协会不减进口量是实事求是[J].当代水产,2013(10):30-31. [23]孙少华,师守坤.肉牛杂交优势预测,评估及其应用研究[J].遗传学报,2000,27(7):580-589. [24]高强,罗坤,宫金华,等.人工养殖与选育对罗氏沼虾遗传多样性的影响[J].水生生物学报,2012,36(5):866-873. Growth Performance and Genetic Diversity Analysis of Hybrids Between Selective Lines of Litopenaeus vannamei WU Yidi1,LUO Xuan1,YANG Zhangwu2,HUANG Yongchun3,YOU Weiwei1* (1.College of Ocean & Earth Sciences,Xiamen University,Xiamen 361102,China;2.Fisheries Research Institute of Fujian,Xiamen 361013,China;3.College of Fisheries,Jimei University,Xiamen 361021,China) The growth performance and genetic diversity of the inbred and crossbred progenies of twoLitopenaeusvannameiselective lines (A and B) were analyzed in this study.The results indicated that AB group showed the best growth performance and heterosis.Eleven fluorescently labeled microsatellite markers were used to amplify microsatellite DNA from fiveL.vannameipopulations.The results showed that four populations (except for AA) displayed higher genetic diversity with average numbers of alleles (Na=4.273-5.636),average expected heterozygosity (He=0.586-0.629),and average polymorphism information content (PIC=0.512-0.556). Nei′s unbiased genetic distance (Ds) indicated a close genetic relationship between populations SIS and AB (Ds=0.131 4),while populations SIS and AA showed the farthest genetic relationship (Ds=0.670 4).The results demonstrated that the genetic diversity decreased after several generations of selection,while hybridization between different selective lines could improve the growth performance and genetic diversity ofL.vannamei. Litopenaeusvannamei;growth performance;genetic diversity;microsatellite;hybridization 10.6043/j.issn.0438-0479.201511010农业生产专题 2015-11-09录用日期:2016-02-23 厦门市海洋经济创新发展区域示范项目(13PZY001SF25) wwyou@xmu.edu.cn 吴怡迪,骆轩,杨章武,等.凡纳滨对虾选育系间杂交的生长性状及遗传多样性分析[J].厦门大学学报(自然科学版),2016,55(5):646-653. WU Y D,LUO X,YANG Z W,et al.Growth performance and genetic diversity analysis of hybrids between selective lines ofLitopenaeusvannamei[J].Journal of Xiamen University(Natural Science),2016,55(5):646-653.(in Chinese) S 917.4 A 0438-0479(2016)05-0646-08
