三角帆蚌微卫星多重PCR体系的建立及其应用
2016-10-11殷浩白志毅韩学凯李家乐
殷浩白志毅韩学凯李家乐
(1. 上海海洋大学农业部淡水水产种质资源重点实验室,上海 201306;2. 上海市水产养殖工程技术研究中心,上海 201306)
三角帆蚌微卫星多重PCR体系的建立及其应用
殷浩1,2白志毅1,2韩学凯1,2李家乐1,2
(1. 上海海洋大学农业部淡水水产种质资源重点实验室,上海 201306;2. 上海市水产养殖工程技术研究中心,上海 201306)
为了提高三角帆蚌微卫星分析效率,从已开发的微卫星标记中构建了三组四重PCR,并将稳定的多重PCR体系用于56个三角帆蚌紫色选育系的遗传多样性研究。结果表明该群体的平均等位基因数18.75,平均有效等位基因数为9.010,平均多态信息含量为0.857,平均观测杂合度和期望杂合度分别为0.809和0.876,香农多样性指数为2.422。运用Cervus v3.0 软件对3个三角帆蚌全同胞家系共114个个体进行亲子鉴定,结果显示,使用该3组微卫星多重PCR体系进行亲子鉴定准确率为100%。该三角帆蚌微卫星多重PCR应用于三角帆蚌群体遗传多样性分析、亲子鉴定和家系管理等,可提高工作效率,降低实验成本。
三角帆蚌;微卫星;多重PCR;遗传多样性;亲子鉴定
三角帆蚌(Hyriopsis cumingii)作为我国特有的淡水珍珠蚌,孕育出世界上近80%的淡水珍珠[1]。我国三角帆蚌种质资源丰富,尤以五大湖区的野生种群分布广泛,其中鄱阳湖和洞庭湖种群生长和育珠性能最为优良[2]。自从20世纪70年代三角帆蚌的人工繁殖实验成功之后,珍珠蚌养殖和生产出现大幅增长,但是育苗场人工育苗往往以利益为先,使得养殖品种质量退化,近交严重,所产珍珠品质低下。加之环境污染和过度捕捞野生资源遭到破坏,部分地区甚至出现灭绝。为了解决这些问题,展开了大量关于三角帆蚌种质开发和保护的研究,包括五大湖区野生资源调查[3,4]、不同种质生长性状比较[2,5]、遗传参数估计[6-8],以及遗传图谱的绘制[9]。
微卫星标记具有高度多态性,在基因组广泛分布,共显性遗传,以及符合孟德尔遗传定律等优点,是育种中理想的分子标记。在三角帆蚌研究中,从第一批微卫星标记开发以来,研究人员已经陆续开发了500多对微卫星标记[9-11]。最初微卫星标记主要运用在五大湖区三角帆蚌野生资源的调查工作,逐步明晰了我国三角帆蚌资源的分布与亲缘关系等[4]。但是,相比野生群体,养殖群体总存在哈迪—温伯格平衡偏离和变异性下降的问题。三角帆蚌人工繁殖实验成功以后,许多苗种供应商只依靠少量野生亲本进行多代的连续繁殖,并且不控制亲本数量,这就导致了亲本变异性的下降和近交退化。
现阶段改变三角帆蚌养殖场选育策略应该注意群体选育后代的遗传变化情况,适时地引进优异亲本;同时开展有效的家系选育项目,在操作过程中要记录好家系信息,以防止遗传侵蚀和基因流失。利用高效稳定、经济节约的多重PCR技术,监测选育项目进展已成为必要环节。微卫星多重PCR技术已广泛应用于水产动物的遗传分析和亲子鉴定。如斑节对虾(Penaeus monodon)[12]、三疣梭子蟹(Portunus trituberculatus)[13]、中国明对虾(Fenneropenaeus chinensis)[14]、长牡蛎(Crassostrea gigas)[15]、皱纹盘鲍(Haliotis discus hannai)[16]。目前还未见有关三角帆蚌微卫星多重PCR体系建立和应用的报道。本研究拟通过建立一套微卫星多重PCR体系,结合基因扫描技术达到位点的自动化分析和检测,给三角帆蚌群体选育和家系管理提供了一种稳定而廉价的标记工具。
1 材料与方法
1.1 材料
实验蚌采集自3个养殖群体,首先是开发适用于三角帆蚌的微卫星多重PCR的实验样品来自实验室2014年4月份采集的浙江武义伟民水产养殖公司的三角帆蚌15只;将构建好的微卫星多重PCR体系运用于太湖群体紫色选育系F1代(50雄性,36雌性)遗传多样性研究;最后,利用本实验室经过多年选育得到的三角帆蚌F5代家系进行亲子鉴定,随机挑选其中3个家系(编号P1、D1、K2),每个家系取样 36 个个体,以及各家系对应的亲本。所有样品剪取斧足,将其固定在无水乙醇中备用。
1.2 方法
1.2.1 基因组DNA的提取和检测 用苯酚/氯仿法抽提三角帆蚌基因组DNA,100 μL双蒸水溶解。DNA浓度和纯度通过NanoDrop 2000C分光光度计测量,完整性用1% 琼脂糖凝胶检测,随后对应稀释成40-50 ng/μL的实验浓度,-20℃保存备用。
1.2.2 微卫星多重PCR的设计和优化 首先根据实验室绘制遗传图谱提供的微卫星位点信息,主要包括所在连锁群的位置、位点多态性和片段大小等特性,选取40个微卫星候选位点,所选微卫星位点的最适退火温度均为55℃。之后,对所有40个位点进行单一引物扩增,以扩增效率,条带清晰程度以及亮度为条件,挑选出最好的24个微卫星标记用于多重PCR的开发。用荧光基团FAM 或 HEX 修饰其上游引物 5' 端,荧光基团的修饰引物由上海迈浦生物科技有限公司合成。初始20 μL的PCR反应体系:10 μL 2 x Taq PCR Mastermix(0.1 U Taq Polymerase / μL、500 μmol/L dNTP、20 mmol/L Tris-HCl pH 8.3、100 mmol/L KCl、3 mmol/L MgCl2),10 mmol / L 引物对各 0.25 μL,基因组DNA 2 μL,无菌水补充到20 μL,其中2×Taq PCR Mastermix 购自天根生化科技(北京)有限公司。PCR 扩增程序:94℃ 预变性 3 min;94℃ 变性1 min,退火 1 min,72℃ 延伸1 min,扩增 35 个循环;再 72℃ 延伸 10 min,最后10℃ 保存。PCR扩增反应在Eppendorf Mastercycler pro 384梯度PCR仪(Eppendorf,德国)上进行。荧光标记的 PCR 产物由上海迈浦生物科技有限公司进行STR 测序分析(毛细管电泳检测ABI 3730XL全自动DNA测序仪)。
微卫星多重PCR反应体系的组合。根据单个微卫星位点的 PCR 扩增结果,挑选没有非特异性条带的24对引物。将最适的退火温度、目的片段大小范围以及引物序列等信息输入到Multiplex Manager1.0软件[17],设置相应的参数,所用的荧光标记设置为2种、单个循环体系中能够容纳的最大引物数为4、引物序列互补的临界值以及相同染料颜色等位基因最小差距等。将相应的参数设置完成后,输出软件计算结果,对于相应结果挑选目的片段长度差异大于50 bp的微卫星组合进行实验。首先进行3个位点的组合实验,获得清晰的目的条带后再添加一条引物进行四重PCR扩增,反复实验直到能够扩增得到条带均一且清晰组合为止。
微卫星多重PCR反应体系的调整优化。对于实验结果要严格区别多重PCR反应产物中4组峰值是否为对应引物产物,避免出现高度相似的引物间错配;根据分型后峰值的相对高低调整引物的相对浓度,每次调整单个引物浓度的增减量为0.05 μL;为保证添加值极小的引物能够精确点样,可以增加总点样管数,先将多对引物组充分混匀,再平均点样,有效保证极低浓度引物的按量添加,最终使扩增效率达到基本一致。
1.3 数据统计分析
所有的STR 基因型分析,使用GeneMapper v4.0软件读取微卫星等位基因数据,利用 Cervus v3.0 软件分析12个微卫星位点的等位基因数(A)、观测杂合度(Ho)、期望杂合度(He)和多态信息含量(PIC),而有效等位基因数(An)、香农多样性指数(I)通过Popgene v1.32计算。亲子鉴定根据两种方法计算得来:(1)采用x2检验检测在子代中等位基因的分离比是否符合孟德尔遗传规律,显著性水平设为P>0.01;(2)利用Cervus v3.0 的Paternity 功能进行模拟分析完成。
2 结果
2.1 微卫星多重PCR的建立
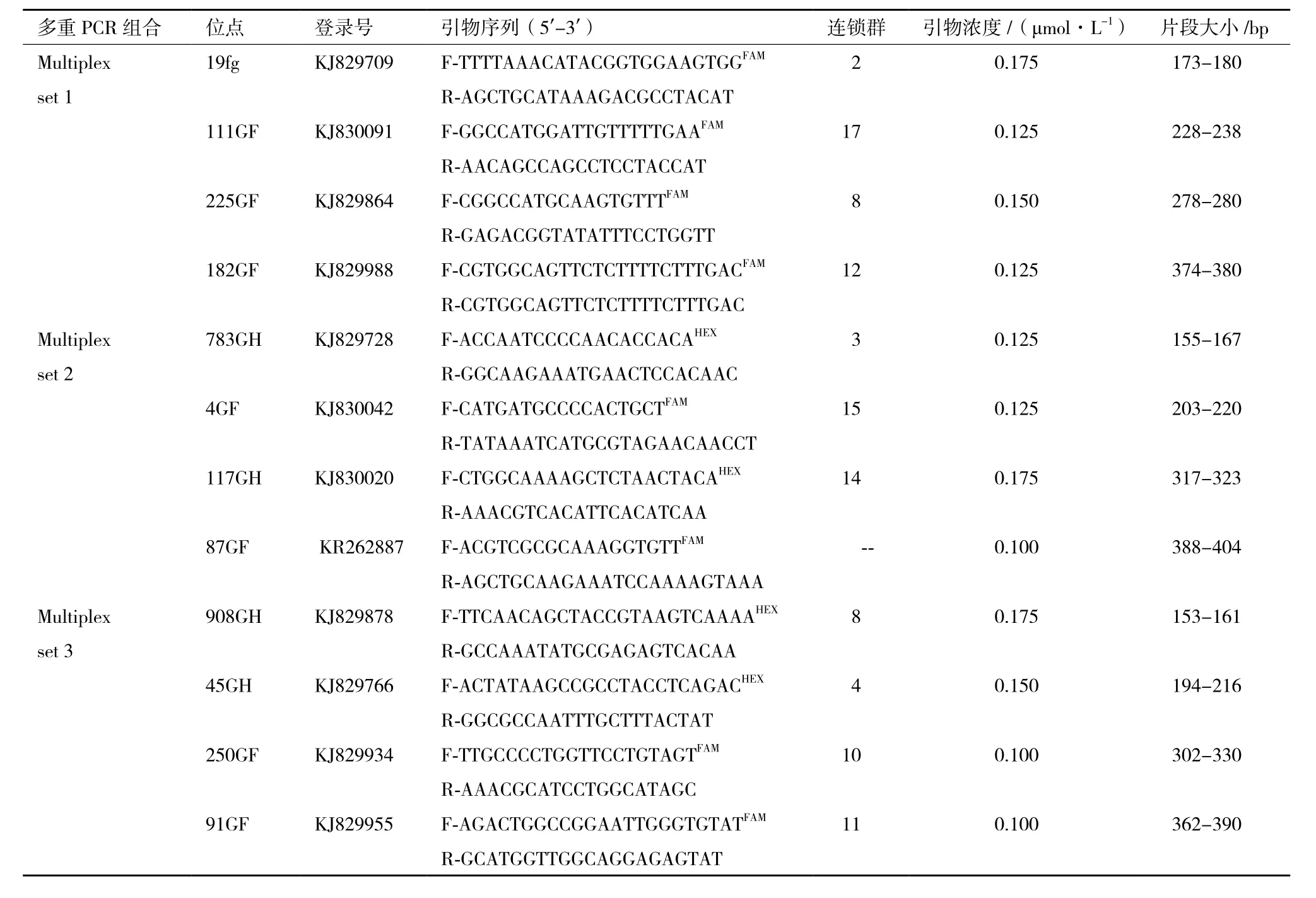
表 1 三角帆蚌 3 组微卫星多重 PCR 的特性
利用Multiplex Manager1.0软件将24对高度多态性的微卫星位点成功的组合出6组的微卫星多重PCR体系。将得到引物组合依次实验,除去没有扩增出来的引物组合、能够得到4条的组合调整引物浓度使得每对引物的扩增效率相似,最后成功组合出3组4重微卫星多重PCR体系(表1)。
2.2 应用微卫星多重PCR分析三角帆蚌紫色选育系遗传多样性
微卫星多重PCR工具对太湖群体紫色选育系F1代56个个体进行分析,12个位点在群体中表现出较高的多态性,平均等位基因数到达了18.75,每个位点的等位基因数(A)12-25;有效等位基因数(An)3.959-15.525,平均有效等位基因数9.010;观测杂合度(Ho)0.518-1.000,平均观测杂合度0.809;期望杂合度(He)0.754-0.944,平均期望杂合度0.876;多态信息含量(PIC)0.728-0.932,平均多态性信息含0.857;香农多样性指数(I)1.907-2.935,平均香农多样性指数2.422(表2)。从STR分型部分峰值图(图1)可以看到,使用的微卫星多重PCR具有稳定和均一的扩增效果,能够适用于三角帆蚌的遗传多样性研究。总体而言,紫壳色三角帆蚌养殖群体的遗传多样性较高,具有较高的改良潜力。
2.3 微卫星多重PCR在家系鉴定中的应用
在3个家系中12个微卫星位点的等位基因数(A)从7到15,平均等位基因数为10.50。多态信息含量(PIC)为0.682-0.912,平均多态信息含量为0.8176,反映了这个多重PCR工具在家系鉴定中具有较高的排除能力。其观测杂合度(Ho)范围为0.265-0.980,平均观测杂合度为0.724,期望杂合度(He)范围为0.720-0.921,平均期望杂合度为0.839。
利用Cervus v3.0 软件进行家系鉴定显示(表3),所有微卫星位点均能匹配,12个微卫星位点亲本基因型未知的累积排除率为99.92%,已知一个亲本基因型时的累积排除率为99.99%;并且子代个体的分离比符合孟德尔遗传定律,在子代中等位基因分别来自父母本。分析3个家系的分离模式发现,如果考虑无效等位基因的存在,所有子代的基因型都符合孟德尔遗传定律。

图1 部分三角帆蚌个体三组多重PCR基因扫描图
3 讨论
微卫星多重PCR要求在一个反应体系中能够稳定、均一地扩增出多条引物,要求所有引物的退火温度相近、片段大小不能重叠、不能有非特异性扩增,因而构建稳定多重PCR体系在技术难度较大,需要全面的分析和反复优化[18]。微卫星多重PCR体系的构建能够极大地减少基因分型成本和时间,尤其是在分析大量实验样本时,多重PCR技术的优势更为明显。结合荧光微卫星的基因扫描技术能够提高分析的效率。多重PCR扫描得到的信息并不是简单地把几对微卫星信息累加,其扫描得到的信息比单纯单对微卫星要得到更多的遗传信息[13]。同时,与传统的非变性聚丙烯酰胺凝胶电泳相比,毛细管电泳检测能够得到更丰富等位基因以及更高的判读准确性。郝晨阳等[19]对SSR荧光标记和银染技术的比较发现,荧光技术较银染方法在每个位点上多检测到3个等位变异,检测效率显著高于银染法7.8倍。利用相同的RSO0683位点,刘萍等[20]通过传统的非变性聚丙烯酰胺凝胶电泳在20个明对虾个体中检测到12个等位基因,而孔杰等[14]采用毛细管电泳检测的等位基因数是上述结果的2倍。
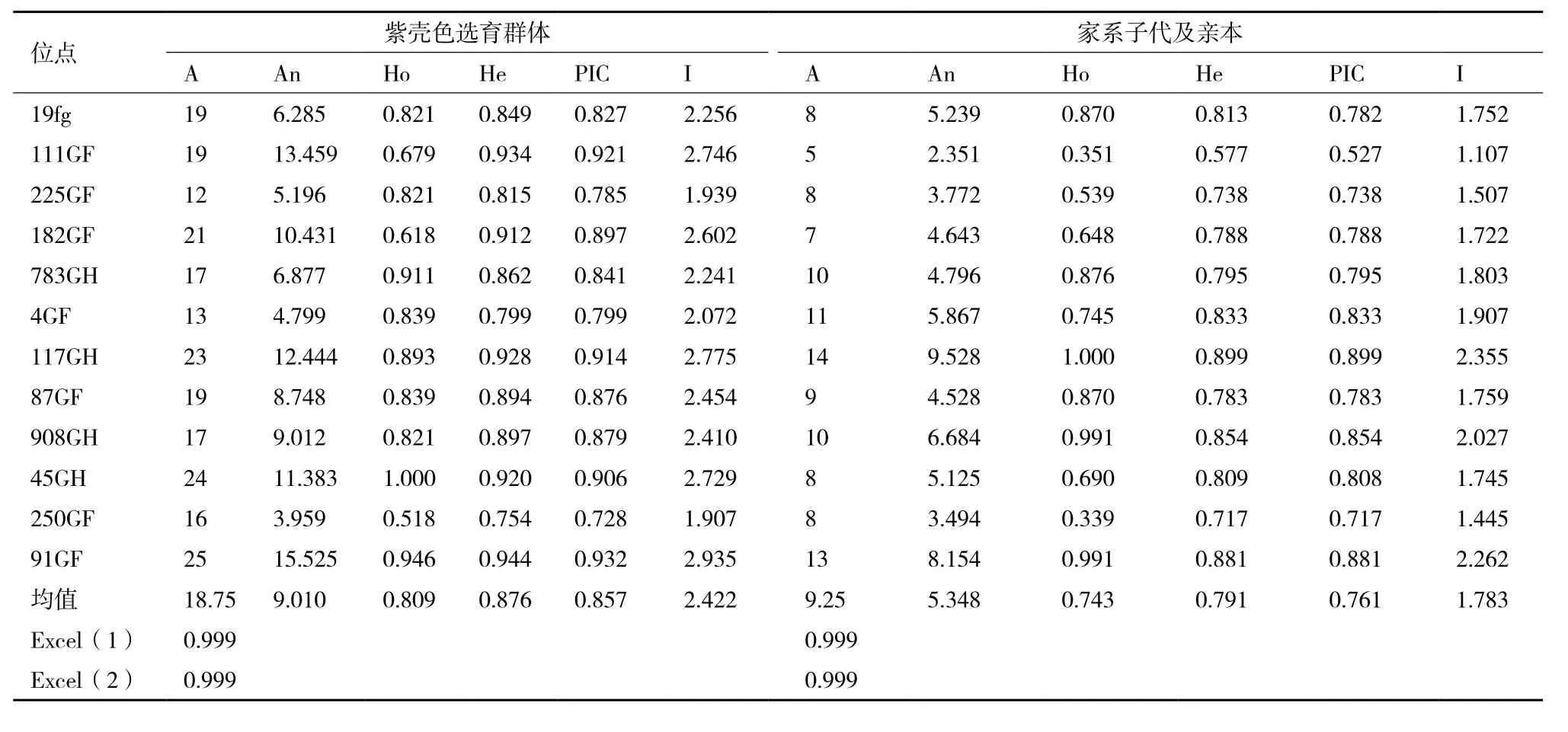
表 2 三角帆蚌紫壳色选育群体和3个家系的遗传参数
微卫星用于三角帆蚌群体的遗传结构的研究较多,最初主要运用于我国五大湖区野生三角帆蚌的亲缘关系分析[4],在野生资源与诸暨和金华的养殖群体比较中发现养殖群体出现了轻微的遗传多样性下降[21]。同样,在很多水产动物的养殖群体和野生群体的比较研究中都发现了遗传多样性下降的现象[22,23]。本研究的结果显示,三角帆蚌群体观察杂合度平均值和期望杂合度平均值分别是0.809和0.876,平均多态性信息含量为0.857,最小值为0.728,具有较高的遗传多样性(PIC>0.5)。这主要是用于繁殖的亲本来自太湖水域的野生三角帆蚌并且大规模养殖在外荡中,在人工选择时没有经过高强度淘汰,这可能是导致该群体遗传多样性较高的主要原因。利用微卫星进行遗传多样性监测有利于在早期淘汰遗传多样性低的群体,减少养殖和管理的成本。
在水产动物的家系选育中通常要了解混养条件下子代的亲子关系,才能在最大程度地避免近交,微卫星标记可以有效地区分混养群体的家系信息。在家系鉴定中所用标记的识别能力和位点的稳定性是家系鉴定成功的主要原因,理论上所选微卫星标记的遗传多样性越丰富,其非亲排除率就越高,越容易通过特异性条带确定亲缘关系。本研究的亲子代具有高的遗传多样性0.818,利用Cervus 3.0软件分析双亲未知时的排除率为99.92%,已知一个亲本时的排除率为99.99%,表明本研究建立的由12个微卫星位点组成的3个多重PCR体系可以实现比普通PCR方法具有更高效的亲子鉴定率。在长牡蛎的亲子鉴定研究中利用具有较高的多态信息含量multiplex set 4 和其余任意一组就可达到100% 的实际鉴定结果[15]。傅建军等[24]使用12个多态微卫星位点对草鱼进行亲权分析发现使用其中8个位点即能达到 100%的亲权鉴定成功率。在3个家系统计分析了亲本和子代的基因型分离情况,12个微卫星标记在考虑无效等位基因的前提下,x2检验没有发现偏离孟德尔遗传比例的情况。但在双壳贝类中也存在偏离孟德尔遗传的现象[15,16],其主要原因普遍认为是存在大量无效等位基因,可能会对家系鉴定结果有一定影响[25,26]。但也和取样家系的数量、DNA的突变、致死基因的存在等因素有关。本研究也检测到两个位点存在无效等位基因,并将其看做隐性基因处理则子代的分离比符合孟德尔遗传规律。

表 3 多重 PCR 在3个三角帆蚌家系中的微卫星基因型分离比
4 结论
本研究构建的3组微卫星多重PCR体系,具有相比普通PCR方法高3-4倍的分析效率;这套微卫星多重PCR体系不仅能够应用于三角帆蚌群体多样性研究和资源管理,而且在家系重建和亲子鉴定上也有较高识别力。
[1]Li JL, Li YS. Aquaculture in China—Freshwater pearl culture[J]. World Aquaculture, 2009, 40(1):60-62.
[2]钱荣华, 李家乐, 董志国. 中国五大湖三角帆蚌形态差异分析[J]. 海洋与湖沼, 2003, 34(4):436-443.
[3]Li JL, Wang GL, Bai ZY. Genetic diversity of freshwater pearl mussel(Hyriosis cumigii)in populations from five largest lakesin China revealed by inter-simple sequences repeat(ISSR)[J]. Aquaculture International, 2009, 17:323-330.
[4]汪桂玲, 袁一鸣, 李家乐. 中国五大湖三角帆蚌群体遗传多样性及亲缘关系的SSR分析[J]. 水产学报, 2007, 31(2):152-158.
[5]李家乐, 白志毅, 钱荣华. 中国五大湖三角帆蚌群体与诸暨养殖群体生长性能的比较研究[J]. 水产科技情报, 2006, 33(6):243-246.
[6] 王照旗, 韩学凯, 等. 三角帆蚌紫色选育系1龄阶段内壳色及生长性状的遗传参数估计[J]. 水产学报, 2014, 38(5):644-650.
[7]Jin W, Bai ZY, Fu LL, et al. Genetic analysis of early growth traits of the triangle shell mussel, Hyriopsis cumingii, as an insight for potential genetic improvement to pearl quality and yield[J]. Aquaculture International, 2012, 20:927-933.
[8]金武, 李家乐, 付龙龙, 等. 三角帆蚌早期阶段生长性状遗传参数估计[J]. 水产学报, 2012, 36(8):1209-1214.
[9]Bai ZY, Han XK, Luo M et al. Constructing a microsatellite-based linkage map and identifying QTL for pearl quality traits in triangle pearl mussel(Hyriopsis cumingii)[J]. Aquaculture, 2015, 437, 1:102-110.
[10]Wang GL, Wang JJ, Li JL. Preliminary study on applicability of microsatellite primers developed from the Crassostrea gigas for genomic analysis of Hyriopsis cumingii[J]. Journal of Fisheries of China, 2006, 30(1):15-20.
[11]Li JL, Wang GL, Bai ZY, et al. Ten polymorphic microsatellites from freshwater pearl mussel, Hyriopsis cumingii[J]. Molecular Ecology Notes, 2007, 7(6):1114-1116.
[12]Li YT, Wongprasert K, Shekhar M, et al. Development of two microsatellite multiplex systems for black tiger shrimp Penaeus monodon and its application in genetic diversity study for two populations[J]. Aquaculture, 2007, 266(1-4):279-288.
[13]任宪云, 刘萍, 高保全, 等. 微卫星多重PCR基因扫描在三疣梭子蟹个体识别中的应用[J]. 水生生物学报, 2012, 36(4):770-778.
[14]孔杰, 高焕, 等. 微卫星三重PCR基因扫描技术在中国明对虾家系标识中的应用[J]. 中国水产科学, 2007, 14(1):59-66.
[15]Li RH, Li Q, Cornette F, et al. Development of four EST-SSR multiplex PCRs in the Pacific oyster(Crassostrea gigas)and their validation in parentage assignment[J]. Aquaculture, 2010, 310(1-2):234-239.
[16]聂鸿涛, 李琪, 等. 皱纹盘鲍微卫星多重 PCR 体系构建及其在家系鉴定中的应用[J]. 水产学报, 2013, 37(2)207-215.
[17]Holleley CE, Geerts PG. Multiplex Manager 1. 0:a cross-platform computer program that plans and optimizes multiplex PCR[J]. Biotechniques, 2009, 46:511-517.
[18]陈明洁, 方倜, 柯涛, 等. 多重PCR—一种高效快速的分子生物学技术[J]. 武汉理工大学学报, 2005, 27(10):33-36.
[19]郝晨阳, 王兰芬, 贾继增, 等. SSR荧光标记和银染技术的比较分析[J]. 作物学报, 2005, 31(2):144-149.
[20]刘萍, 孟宪红, 何玉英, 等. 中国对虾(Fenneropenaeus chinensis)黄、渤海3个野生地理群遗传多样性的微卫星DNA分析[J]. 海洋与湖沼, 2004, 35(3):252-256.
[21]Li JL, Wang GL, Bai ZY. Genetic variability in four wild and two farmed stock s of the Chinese freshwater pearl mussel(Hyriopsis cumingii)estimated by microsatellite DNA markers[J]. Aquaculture, 2008, 287:286-291.
[22]王庆志, 李琪, 孔令锋. 长牡蛎3代人工选育群体的微卫星分析[J]. 水产学报, 2012, 36(10):1529-1536.
[23]Lind CE, Evans BS, Knauer J, et al. Decreased genetic diversity and a reduced effective population size in cultured silver-lipped pearl oysters(Pinctada maxima)[J]. Aquaculture, 2009, 286(1-2):12-19.
[24]Fu JJ, Shen YB, et al. Multiplex microsatellite PCR sets for parentage assignment of grass carp(Ctenopharyngodon idella)[J]. Aquaculture International, 2013, 21(6):1195-1207.
[25]McGoldrick D, Hedgecock D, English LJ, et al. The transmission of microsatellite alleles in Australian and North American stocks of the Pacific oyster(Crassostrea gigas):selection and null alleles[J]. Journal of Shellfish Research, 2000, 19(2):779-788.
[26]Marshall TC, Slate J, Kruuk LEB, et al. Statistical confidence for likelihood-based paternity inference in natural populations[J]. Molecular Ecology, 1998, 7(5):639-655.
(责任编辑李楠)
The Development and Application of Multiplex PCR Panels of Microsatellites in Hyriopsis cumingii
YIN Hao1,2BAI Zhi-yi1,2HAN Xue-kai1,2LI Jia-le1,2
(1. Key Laboratory of Freshwater Fishery Germplasm Resources of Ministry of Agriculture Shanghai Ocean University,Shanghai 201306;2. Shanghai Engineering Research Center of Aquaculture,Shanghai 201306)
In order to increase the analysis efficiency of microsatellite in Hyriopsis cumingii, 3 groups of 4 multiplex PCRs panels were established from the developed microsatellite labels. The stable multiplex PCR panels were used to analyze the genetic diversities of 56 purple breeding lines of H. cumingii. The results showed that the average number of alleles(A)was 18.75, the effective number of alleles(An)was 9.010,the average of polymorphism information content(PIC)was 0.857, the average observed heterozygosity(Ho)and expected heterozygosity(He)were 0.809 and 0.876, respectively, and Shannon information index(I)was 2.422. The Cervus v3.0 software was used to have parentage assignment for 114 individuals in 3 full-sib families, and the results revealed that accuracy of parentage assignment by this 3 groups of multiplex PCR panels was 100%. Therefore, this multiplex panels can be applied in genetic diversity studies of H. cumingii population, parentage assignment and family management with increased detection efficiency and reduced test cost.
Hyriopsis cumingii;microsatellites;multiplex PCR;genetic diversity;parentage assignment
10.13560/j.cnki.biotech.bull.1985.2016.01.026
2015-05-22
国家自然科学基金项目(31272657),国家科技支撑计划课题(2012BAD26B04),水产动物遗传育种中心上海市协同创新中心(ZF1206)
殷浩,男,硕士,研究方向:种质资源与遗传育种;E-mail:yinhao0222@163.com
白志毅,男,博士,副教授,研究方向:种质资源与遗传育种;E-mail:zybai@shou.edu.cn
