单核苷酸多态性(SNP)在近交系小鼠遗传检测中的应用
2016-10-09赵丽亚邢正弘陈国强上海西普尔必凯实验动物有限公司上海201203上海实验动物研究中心上海201203
赵丽亚, 张 蓉, 赵 莹, 邢正弘, 陈国强(1. 上海西普尔-必凯实验动物有限公司, 上海 201203; 2. 上海实验动物研究中心, 上海 201203)
单核苷酸多态性(SNP)在近交系小鼠遗传检测中的应用
赵丽亚1,2, 张 蓉1,2, 赵 莹1,2, 邢正弘1,2, 陈国强1,2
(1. 上海西普尔-必凯实验动物有限公司, 上海 201203; 2. 上海实验动物研究中心, 上海 201203)
目的 利用单核苷酸多态性(SNP)位点构建多重聚合酶链反应和链接酶反应(PCR-LDR)方案,为近交系小鼠的遗传检测提供一种快速、简便的方法。方法 在SNP公共数据库中挑选51 个SNP位点,该51个位点分布于每条常染色体和性染色体,共分为5组,进行多重PCR-LDR方案构建,并将该方案作为遗传质量检测方法对采集的7个近交系样本进行检测。结果 在送检的7个近交系小鼠样本中,纯合度都为100%,相同来源相同品系小鼠的遗传背景一致,但在不同单位的近交系小鼠中,某些SNP位点有差异,CBA/Ca/BKl和CBA/JSlac在a9、a10、b5、b9、c1、c12、e1、e2位点的遗传检测结果不同,C57BL/6/BKl和C57BL/6JSlac在c7、c10位点的遗传检测结果不同。另外,两家公司各有5个位点在所有品系中基因型相同,因此有效位点数各为46个。结论 构建的多重PCR-LDR方案能有效对两家动物生产单位的近交系小鼠进行检测,可用于常见近交系小鼠快速、高通量的基因分型,有利于大规模遗传质量检测和品系鉴定。
遗传质量检测; 近交系小鼠; 单核苷酸多态性(SNP); 多重聚合酶链反应和链接酶反应(PCR-LDR); 分型方案
随着C57BL/6及其它近交系[1,2]序列测序的完成,发现了大量单核苷酸多态性(single nucleotide polymorphisms, SNP),SNP多态性及低检测费用为遗传鉴定提供了有利条件。2001年Grupe等[1]利用SNP检测了15个品系, 2002年Wade等[2]利用SNP 从15个品系中检测出4个品系是多态性, 2012年Cui等[3]利用SNP检测了实验小鼠的遗传状态, 2004年Petkov等[4]从235个SNPs中挑选28个SNPs用于鉴定48个小鼠品系。SNP数目多、分布广, 遍布整个基因组, 具有遗传稳定性,SNP单核苷酸突变率为10-9。另外,SNP基因型分析特别适合模型动物的遗传纯度分析,等位基因位标包含了近交系小鼠中的所有信息,某些位于基因内部的SNP变异有可能直接影响蛋白质结构或表达水平,因此SNP可用于描述近交系小鼠的遗传状态。
1 材料与方法
1.1 实验动物
选择7个品系SPF级成年近交系小鼠,分别是上海西普尔-必凯实验动物有限公司(以下简称BK公司)[SCXK(沪)2008-0016]提供的C57BL/6/BKl、C3H/He/BKl、FVB/NJ/BKl、BALB/c/BKl、DBA/2/ BKl、CBA/Ca/BKl、129/SvEvBrd/BKl和上海斯莱克实验动物有限公司(以下简称BK公司)[SCXK(沪)2012-0002]提供的C57BL/6JSlac、C3H/HeJSlac、FVB/NJSlac、BALB/cAnSlac、DBA/2JSlac、CBA/JSlac,129S1/SVImJSlac, 每个品系分别从繁殖群和种子群中取雌雄各3~4只,两家公司的繁殖群样本都为56个,种子群样本分别为55个和54个,共221个样本。上海西普尔-必凯实验动物有限公司提供的动物来源于英国BK公司,在国内繁殖了约14代。上海斯莱克实验动物有限公司提供的动物来源于Jackson实验室,在国内繁殖了约24代。
1.2 方法
1.2.1 DNA提取 使用动物基因组DNA抽提试剂盒(天根生化科技有限公司), 从成年小鼠尾尖剪取0.5 cm组织用于抽取全基因组DNA,剪取的组织包括骨、血管、皮肤等各种组织,抽提产物用0.8%的琼脂糖凝胶电泳后在Tanon-3500全自动凝胶成像分析系统上扫描,有结果时,可以作为模板用于后续的多重PCR扩增,-20℃保存备用。
1.2.2 SNP位点引物与探针设计 从NCBI(National Center for Biotechnology Information)进入小鼠SNP公共数据库,根据提供的小鼠品系,在每条染色体上选取1~3个SNP(short tandem repeat)位点,共挑选SNP位点51个[5]。
根据挑选的SNP序列,分别设计引物和探针,用primer3在线软件(http://frodo.wi.mit.edu/primer3/)设计引物,选择性扩增包含SNP位点的不同染色体区段。多重引物设计主要借助Oligo6.0程序(Molecular Biology Insights Inc., USA)完成,同时将PCR-LDR产物的范围设计在78~131 bp,每组内各位点差异2 bp, 共设计引物和探针51对,将51对引物和探针分为5组,第1组为10个位点,第2组、第3组、第4组为12个位点,第5组为5个位点,并由上海生工生物工程技术服务有限公司合成(表1、表2, 表中列出第一组的引物和探针序列,其余4组未列出引物和探针,只在结果中附图)。

表 1 第一组PCR扩增引物序列Table 1 Genome primer sequences of the first group
1.2.3 PCR及LDR程序 PCR反应体系(10 μL体系): 10×PCR缓冲溶液1 μL, GC 1μL, 25 mmol/L,Mg2+1.2 μL, 200 μmol/L的dNTP混合液(各2.5 mmol/L)0.8 μL, 各0.5 μmol/L的引物1 μL, 5个单位/μL热启动DNA聚合酶(AmpliTaq Gold®360 DNA Polymerase)0.05 μL, 3~5 ng/μL的模板DNA 1 μL,加水补足10 μL。
反应程序: 95 ℃ 15 min; 94 ℃ 30 s→50 ℃~65 ℃90 s→ 72℃ 1 min, 共35个循环; 72℃ 10 min; 10 ℃持续。其中第1组、第2组、第5组引物的退火温度为56 ℃, 第3组引物的退火温度为58℃, 第4组引物的退火温度为60 ℃。

表 2 第1组LDR探针序列Table 2 Genome probe sequences of the first group
LDR反应体系(10 μL体系): 10× 缓冲溶液1 μL,各0.5 μmol/L的位点特异探针1 μL,模板(PCR产物)4 μL,40 U/μL Taq DNA链接酶0.1 μL,加水补足10 μL。
反应程序:95 ℃ 2 min;94 ℃ 30 s→50 ℃2 min,共30个循环;10℃ 持续。
1.2.4 多重PCR-LDR方案构建 取C57BL/6小鼠构建多重PCR-LDR方案,本实验的多重PCR-LDR方案构建依据:①各LDR产物长度不同,相隔2 bp。②不同PCR引物或探针之间不发生相互作用以致影响某些PCR引物或探针的正常反应。因此将51个 SNP位点合理的分为5组,而后对每一组分别优化PCR反应温度、各引物间浓度比、各探针间浓度比等条件,使每组中所有位点都能被检测出来,最后用5组PCR-LDR方案对待检样本进行检测。该方法可将原来的11 271个反应过程缩减为1 105个PCR-LDR反应过程。
1.2.5 PCR产物的检测 质量分数2%琼脂糖电泳,电压20 V/cm, 泳动方向:负极→正极, 泳动时间:40 min。溴化乙锭(15 μg/mL)染色,紫外灯下观察, 拍照。
利用ABI3730自动测序仪(Applied Biosystems Inc., 美国)对PCR-LDR连接产物进行毛细管电泳。具体过程是将1.0 μL LDR连接产物、1.0 μL ROX分子量标准和8 μL HIDI混匀后,95 ℃,5 min变性,立刻置冰水浴5 min,然后通过ABI3730自动测序仪进行检测,每轮分析48个样本,每轮运行时间为900 s。通过GeneMapperID v3.2软件分析可将各种激光标记的DNA电泳带转变成相应的扫描吸收峰。再通过与已知分子量标准的内参比较,确定LDR连接产物大小。
2 结果
2.1 小鼠基因组DNA抽取
根据动物基因组DNA抽提试剂盒提取DNA,挑选部分品系的抽提产物用质量分数0.8%的琼脂糖凝胶电泳后在Tanon-3500全自动凝胶成像分析系统上扫描(图1),有结果时,可以作为模板用于后续的多重PCR扩增。

M: Marker; 第1道至第5道分别为C57BL/6/BKl、C3H/ He/BKl、FVB/NJ/BKl、BALB/c/BKl、DBA/2/BKl图 1 基因组DNA 检测结果Figure 1 The results of genomic DNA
2.2 多重PCR-LDR方案构建
取本实验室中的C57BL/6/BKl为模板,构建多重PCR-LDR方案,最终将51个SNP位点组合为5组进行多重PCR-LDR扩增,第1组为10个位点(图2A),第2组、第3组、第4组为12个位点(图2B, C, D), 第5组为5个位点(图2E)。PCR-LDR结果表明,C57BL/6/BKl小鼠DNA在目标SNP位置均为单峰,表明其为纯合子。
2.3 近交系小鼠遗传检测
在送检的7个近交系中,采用51个SNP位点检测,其纯合度都为100%,相同来源相同品系的SNP状态完全一致,但在不同单位的近交系小鼠中,某些位点有差异,主要有差异的品系及位点是:CBA/Ca/BKl和CBA/JSlac在a9、a10、b5、b9、c1、c12、e1、e2位点的遗传检测结果不同,C57BL/6/BKl和C57BL/6JSlac在c7、c10位点的遗传检测结果不同(表3)。
2.4 SNP位点在品系间的差异分析
品系间两两比较见表4、表5,BK公司各品系间最大差异位点数为30个,最小差异位点数为7个, 平均差异位点数为23个。SLAC公司各品系最大差异位点数为31个, 最小差异位点数为1个,平均差异位点数为23个。另外, 两家公司各有5个位点在所有品系中基因型相同,分别为a4、b11、c7、c8、d9和a4、b11、c8、d9、e2,因此有效位点数各为46个。
3 讨论
在医学生物研究中,实验动物遗传质量对实验结果的可比性、可重复性和准确性有着重要影响。在长期保种、育种过程及繁殖生产中,近交系动物可能发生遗传污染或遗传突变、漂变,从而影响相关实验结果的科学性、可靠性。所以加强对近交系小鼠进行长期、有效、严格、定时的遗传学检测非常重要。近交系小鼠传代中产生变异的主要原因有基因突变以及遗传污染。本研究提出利用多重PCR-LDR基因分型方案,多个位点可以在同一体系内进行多重PCR扩增、LDR连接与分型,从而降低了费用。另外,通过对7个品系的近交系小鼠进行遗传检测,构建一种可检测近交系小鼠的方案,可一次性检测所有待检样本,并可进行品系间鉴定,有利于动物生产单位的近交系小鼠常规遗传检测。
本实验应用51个SNP位点较全面的反应了小鼠的遗传背景。将这51个SNP位点在7个品系小鼠中的连接产物进行比对,结果各品系基因纯合度都为100%,说明其在传代中没有出现变异现象。但两家公司的CBA/Ca/BKl和CBA/JSlac在a9、a10、b5、b9、c1、c12、e1、e2位点的遗传检测结果不同,C57BL/6/BKl和C57BL/6JSlac在c7、c10位点的遗传检测结果不同。原因可能是两家公司动物来源不同,亚系不同。据报道, 2009和2015年Mekada等[6,7]利用SNP检测C57BL/6的不同亚系,因来源不同,其基因型有差别, 2011年Zurita等[8]利用1 449个SNP检测了C57BL/6的不同亚系,本文的结果与文献报道一致。
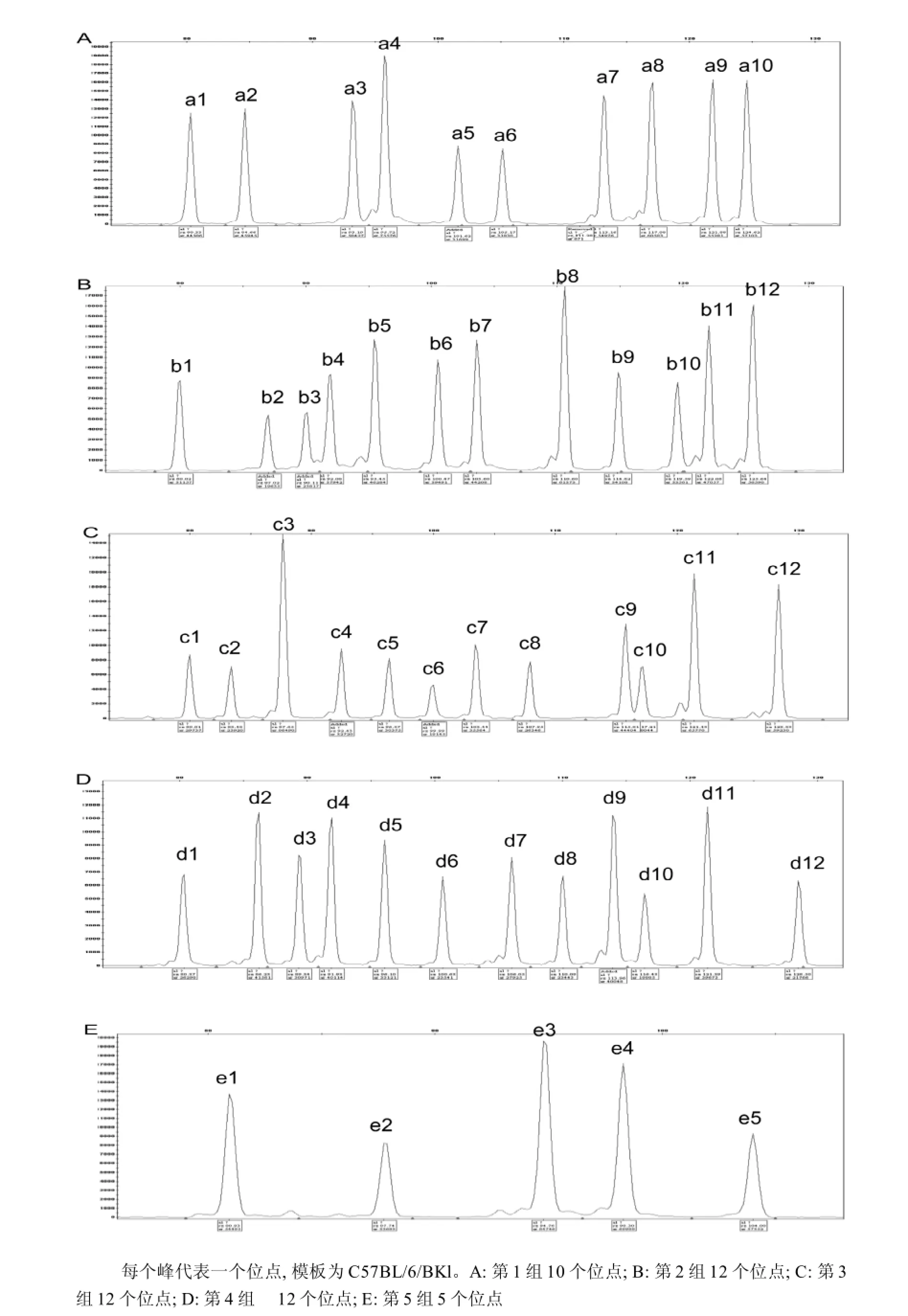
图 2 PCR-LDR分型方案的毛细管电泳图Figure 2 The capillary electrophoresis of products by PCR-LDR

表 3 近交系小鼠遗传检测结果Table 3 The result of genetic monitoring of inbred mice

表 4 BK公司小鼠SNP位点品系间差异Table 4 The difference of SNPs in mouse from BK Co. strains

表 5 SLAC公司小鼠SNP位点品系间差异Table 5 The difference of SNPs in mouse from SLAC Co. strains
同时,实验数据还表明各品系小鼠在某些位点表现一致,如a4、b11、c7、c8、d9或a4、b11、c8、d9、e2在所有品系中的基因型都相同,反应出小鼠品系间有相近的亲缘关系,而在某些位点的不同结果也体现出了不同品系的特异性。另外,两个公司品系间两两比较后,平均差异位点数都为23个。因此,通过对7个品系的近交系小鼠进行遗传检测,得到一种可检测近交系小鼠的方案,可一次性检测所有待检样本,并可进行品系间鉴定。该方案不仅节约了检测时间及成本,更有利于对大规模样本的检测,将会拥有更加广阔的发展前景。
[1] Grupe A, Germer S, Usuka J, et al. In silico mapping of complex disease-related traits in mice[J]. Science, 2001, 292(5523): 1915-1918.
[2] Wade CM, Kulbokas EJ 3rd, Kirby AW, et al. The mosaic structure of variation in the laboratory mouse genome[J]. Nature, 2002, 420(6915):574-578.
[3] Cui SF, Zhou Q, Qu XH. SNP genotyping for the genetic monitoring of laboratory mice by using a microarray-based method with dualcolour fluorescence hybridisation[J]. HYPERLINK “http://www.ncbi.nlm.nih.gov/pubmed/ 22943516”o “Alternatives to laboratory animals: ATLA”. 2012, 40(3):155-163.
[4] Petkov PM, Cassell MA, Sargent EE, et al. Development of a SNP genotyping panel for genetic monitoring of the laboratory mouse[J]. Genomics, 2004, 23(1):4-20.
[5] The Jackson Laboratory. Mouse Genome Database (MGD)at the Mouse Genome Informatics website[DB/OL]. http:// www.informatics.jax.org.
[6] Mekada K, Hirose M, Murakami A, et al. Development of SNP markers for C57BL/6N-derived mouse inbred strains[J]. Exp Anim, 2015, 64(1):91-100.
[7] Mekada K, Abe K, Murakami A, et al. Genetic differences among C57BL/6 substrains[J]. Exp Anim, 2009, 58(2):141-149.
[8] Zurita E, Chagoyen M, Cantero M. Genetic polymorphisms among C57BL/6 mouse inbred strains[J]. Transgenic Res,2011, 20(3):481-489.
Application of Single Nucleotide Polymorphism Genotyping Panel in Genetic Monitoring of Laboratory Mice
ZHAO Li-ya1, ZHANG Rong1, ZHAO Ying1, XING Zheng-hong1, CHEN Guo-qiang1
(1. Sino-British SIPPR/BK Laboratory Animal Co., Ltd., Shanghai 201203, China;
(2. Shanghai Laboratory Animal Research Center, Shanghai 201203, China)
Objective To develop multiple PCR-LDR genotyping protocols for monitoring genetic contamination in inbreed mice in a fast and reliable way. Method In total 51 single nucleotide polymorphisms (SNPs) from public SNP database were chosen and divided into five groups. These 51 SNPs are wildly distributed and there are least one SNP on each autosomal chromosome and the sex chromosome. A multiple PCR-LDR genotyping protocol was established as a genetic quality control approach to monitor the genotypes of 7 inbred mice. Results The homozygosity is 100% for the 7 tested mice. The genetic background of the inbred mice from the same source is identical. For the same inbred mice from different companies, several SNPs are different. The SNPs are different at a9、a10、b5、b9、c1、c12、e1、e2 for CBA/Ca/Bkl and CBA/JSlac, while the SNPs are different at c7, c10 for C57BL/ 6/BKl and C57BL/6JSlac. However, there are five loci which are the same in the 7 inbred mice of different companies. So there are 46 effective loci which are different in the 7 inbred mice of different companies. Conclusion The multiple PCR-LDR genotyping protocol is used to monitor the genetic differences of inbred mice from two companies. The results of this study provide a rapid and highthroughput genotyping approach, which is sufficient for genetic contamination monitoring and strain identification.
Genetic quality monitoring; Inbred mice; Single Nucleotide Polymorphisms (SNP);multiple PCR-LDR; Genotyping protocol
Q95-33
A
1674-5817(2016)01-0024-08
10.3969/j.issn.1674-5817.2016.01.005
2015-06-29
上海市科学技术委员会科研计划项目(12140900400)
赵丽亚(1981-), 女, 硕士。研究方向: 分子遗传学。E-mail: zhaoliya3002@163.com
赵 莹(1982-), 女, 硕士, 助理研究员。研究方向:群体遗传学及药物毒性安全评价。E-mail: 12414863@qq.com
