孕妇血浆游离核酸高通量测序检测胎儿遗传异常
2016-09-22殷旭阳陈芳王威
殷旭阳 陈芳,2 王威
(1.深圳华大基因研究院,广东 深圳 518083;2.哥本哈根大学,丹麦 哥本哈根 2200;3:深圳华大临床检验中心,广东 深圳518083)
孕妇血浆游离核酸高通量测序检测胎儿遗传异常
殷旭阳1陈芳1,2王威3*
(1.深圳华大基因研究院,广东 深圳 518083;2.哥本哈根大学,丹麦 哥本哈根 2200;3:深圳华大临床检验中心,广东 深圳518083)
孕妇血浆中胎儿源性的游离DNA的发现,为无创产前检测(noninvasive prenatal testing,NIPT)奠定了科学基础。通过对孕妇外周血中的游离DNA进行检测,能够以无创的方式分析胎儿的遗传特征,为产前筛查(prenatal screening)和产前诊断(prenatal diagnosis)提供了有效的手段。新一代高通量测序技术(nextgeneration sequencing,NGS)的发展及其在临床的广泛应用,使其在孕妇血浆胎儿游离DNA检测中的应用成为必然趋势,并推动无创产前检测技术的迅猛发展。通过孕妇血浆游离DNA的高通量测序,可实现胎儿的染色体非整倍体、单基因遗传疾病、微缺失微重复的检测,也可以对胎儿基因组、DNA甲基化组以及转录组等信息进行分析。
孕妇血浆; 胎儿游离DNA; 无创产前检测; 产前筛查; 产前诊断; 高通量测序
孕妇血浆中胎儿源性游离DNA的发现,为无创产前检测(noninvasive prenatal testing,NIPT)奠定了科学基础。通过对孕妇外周血中的游离DNA进行检测,能够以无创的方式分析胎儿的遗传特征,为产前筛查(prenatal screening)和产前诊断(prenatal diagnosis)提供了有效的手段。新一代高通量测序技术(nextgeneration sequencing,NGS)的发展及其在临床的广泛应用,使其在孕妇血浆胎儿游离DNA检测中的应用成为必然趋势,并推动无创产前检测技术的迅猛发展。通过孕妇血浆游离DNA的高通量测序,可实现胎儿的染色体非整倍体、单基因遗传疾病、微缺失微重复的检测,也可以对胎儿基因组、DNA甲基化组以及转录组等信息进行分析。
1 孕妇血浆游离DNA测序检测胎儿非整倍体
1997年,研究人员在一位怀男性胎儿的孕妇外周血的血浆中发现了胎儿Y染色体游离DNA[1],从而确定母体血浆中存在胎儿游离DNA这一事实,并检测出胎儿DNA约占母体血浆总游离DNA的3%~6%,其半衰期仅为16分钟,说明此次妊娠的游离DNA不会受到之前妊娠胎儿的影响。之后的研究发现怀有唐氏综合征胎儿的孕妇,其血浆中的总体胎儿游离DNA总量要高于怀有正常胎儿的孕妇[2]。随着孕妇外周血游离DNA的研究,特别是检测技术的不断发展,孕妇外周血的血浆逐渐成为无创检测胎儿遗传特征的理想材料。
妊娠中较常见的胎儿非整倍体,除了唐氏综合征,即21-三体外,还有18-三体、13-三体,以及性染色体异常如X单体(Turner综合征)等。以21-三体为例,原理上唐氏综合征的胎儿应释放相对其他染色体更多的21号染色体DNA序列进入母体血浆。因此通过母体血浆的全基因组高通量测序,并分析比对到每条染色体的数据量,将待测孕妇的数据与一组正常妊娠的孕妇的数据进行统计学比较,即可判定待测孕妇中的胎儿染色体数目异常。这种方法已被国内外多家机构应用并证实其高准确性[3],包括检测21-三体[4-9]、18-三体[4,6-9]、13-三体[6-9],以及性染色体异常[3,8]和其他非整倍体[10,11]。
除全基因组测序,也可采用目标区域测序分析孕妇血浆游离DNA以检测胎儿非整倍体。通过杂交捕获基因组上特定区域的DNA序列继而进行测序[12,13],或者以多重PCR的方法[14-16]将多个SNP位点捕获和扩增,对扩增子进行测序,通过分析杂合SNP位点的等位基因数据量差异,从而检测胎儿目标染色体的数目异常。
2004年,Chan等[17]发现孕妇血浆中胎儿来源的游离DNA长度要短于母体来源的DNA。通过对DNA片段采用双侧末端测序的策略(paired-end),可确定血浆中游离DNA片段的长度。研究发现,孕妇血浆中母体来源的DNA其长度峰值在166bp,而胎儿游离DNA的长度峰值为143bp[18]。在怀有唐氏综合征胎儿的孕妇血浆中,胎儿会释放额外的21号染色体DNA序列,引起21号染色体总游离DNA的长度分布偏短。这种现象在18-三体和13-三体胎儿的孕妇血浆中同样存在。因此可通过孕妇血浆游离DNA长度分布的分析来检测胎儿染色体数目异常。这种片段长度的分析方法也可用于孕妇血浆中胎儿浓度的检测[19]。
DNA甲基化分析方法也可检测孕妇血浆中的胎儿染色体数目异常,根据胎儿和母体在特定基因区的DNA甲基化差异,胎儿DNA多为高甲基化而母体DNA多为低甲基化。通过重亚硫酸盐测序,对每条染色体特定基因区的甲基化程度进行统计学分析来检测胎儿染色体数目异常[20]。DNA甲基化的分析方法也可用于孕妇血浆中的胎儿浓度定量[21]。孕妇血浆中游离RNA的分析也可检测胎儿染色体数目异常,通过对目标染色体上胎盘特异表达RNA在孕妇血浆总RNA中的定量分析,可确定胎儿染色体非整倍体[22]。
2 孕妇血浆游离DNA测序检测胎儿单基因遗传病
单基因遗传病的无创产前检测也有其意义,可以为存在羊膜腔穿刺禁忌证的孕妇提供一个备选方案。单基因病由单一基因突变引起,对检测技术的精度要求高;此外,孕妇血浆中存在大量母源DNA干扰,致使检测难度大大增加。目前,无创产前检测单基因病的方法分为直接法和间接法。直接法是直接对突变位点序列进行检测,通过突变序列和野生型序列的比例推断胎儿基因型;其中胎儿的父源突变很容易被区分,而母源突变则需要区分不同比例基因型的微量差异,胎儿DNA浓度越低,等位基因剂量差异越小,检测越困难。间接法则是根据血浆DNA单体型剂量不平衡原理,通过SNP单体型连锁分析确定胎儿的单体型,从而判定胎儿是否携带父源或母源致病基因;孕妇血浆游离DNA的测序结合夫妇双方以及先证者的基因组DNA测序,计算出夫妇双方的单体型以及致病基因的遗传规律,继而结合孕妇血浆的数据构建出胎儿的单体型和基因型,最终确定胎儿是否患病或是否为携带者。这种基于单体型的无创产前检测策略已在多种单基因疾病中得以实现,包括beta-地中海贫血[23]、α-地中海贫血[24]、先天性肾上腺皮质增生症[25,26]、杜氏肌营养不良[27]、枫糖尿病[28]、先天性耳聋[29]等。
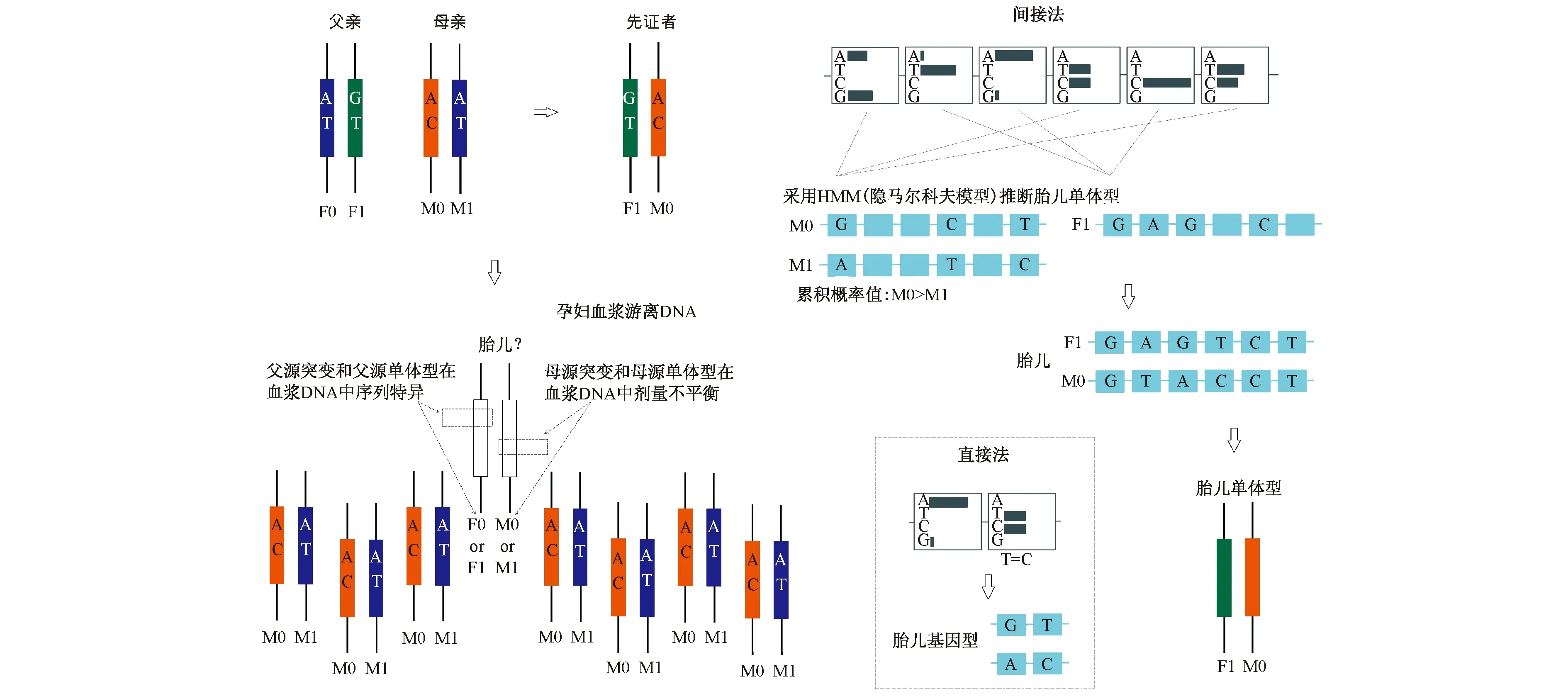
图1 孕妇血浆游离DNA测序检测胎儿单基因遗传病的原理
F0与F1代表父亲基因组的两种单体型组成;M0与M1代表母亲基因组的两种单体型组成;胎儿的单体型分别来自父亲和母亲
地中海贫血是较为常见的单基因病之一,包括β-地贫和α-地贫,分别多由HBB基因点突变和HBA基因的片段缺失引起。Lam等人[23]于2012年采用包含HBB基因的目标区域测序,结合夫妇双方和先证者DNA测序进行单倍型分析,从而实现了1例β-地中海贫血的无创产前检测。Ge等人于2013年实现了5例α-地中海贫血的无创产前检测[24],其设计了包含HBA和HBB基因以及4525个常染色体SNP位点的捕获探针,对5个家系的夫妇双方、孕妇血浆以及羊水或脐血进行目标区域测序。通过孕妇血浆的相对拷贝率(relative copy ratio,RCR)分析以及与正常对照组的R值和L值分析,成功鉴定了1例HBA基因纯合缺失,2例HBA杂合缺失以及另2例正常的胎儿,并与羊水或脐血结果一致,证明了此方法对致病性片段缺失遗传疾病进行无创产前检测的可行性。
杜氏肌营养不良(Duchenne muscular dystrophy,DMD)是一种X性连锁的隐性单基因遗传病,DMD基因包含79个外显子。Xu等[27]设计了涵盖X和22号染色体的定制化芯片,进行8个家系夫妇双方和先证者以及孕妇血浆的目标区域测序,通过夫妇双方和胎儿的单倍型分析结合胎儿性别判定进行检测。原理上,对于男胎,其致病基因只能来自母方,因此只要其DMD基因单倍型与母方突变基因一致,即可判定此男胎患病;而对于女胎,则需结合父方的单倍型进行综合分析。最终在8个家系中,3例女胎中确定1例为携带者,2例为完全正常;另5例男胎,均确定遗传了母方的突变基因,为患者。其8例无创产前检测结果均与羊水结果一致。
CYP21A2,GJB2,BCKDHA基因突变分别可引起先天性肾上腺皮质增生症(congenital adrenal hyperplasia,CAH)、先天性耳聋(congenital deafness)和枫糖尿病(maple syrup urine disease,MSUD),且均为常染色体隐性遗传病。Ma等人[25]和New等人[26]均于2014年建立了涵盖CYP21A2基因的目标区域测序,通过单倍型分析进行孕妇血浆CAH无创产前检测的方法。Meng等[29]也采用半定制化芯片目标区域测序和单倍型方法,在1个家系中实现先天性耳聋的无创产前检测,确定其胎儿为GJB2基因突变的携带者。You等[28]在一个家系中通过目标测序发现了BCKDHA基因上可引起枫糖尿病的新型突变,并以此通过家系单体型方法实现胎儿的无创产前检测,确定胎儿为枫糖尿病患者。
3 孕妇血浆游离DNA测序检测胎儿微缺失微重复
孕妇血浆游离DNA测序不仅能检测胎儿染色体的数目异常,且能检测胎儿染色体片段缺失和重复,包括一些亚显微水平的改变。最早由Peters等人[30]于2011年报道通过孕妇血浆全基因组测序,检出胎儿12p11.22和12p12.1染色体区段间约4.2M的缺失。其后多个机构亦报道孕妇血浆全基因组测序可检测胎儿染色体微缺失与微重复[31-36]。其方法是将人类参考基因组分成许多窗口,将孕妇血浆游离DNA的测序数据与参考基因组比对,计算分布在每个窗口的测序数据量,再经过GC偏差矫正,及与正常对照组的统计学分析,确定哪些窗口存在缺失重复,继而判定胎儿的染色体微缺失微重复的位置和大小。每例血浆的测序数据量、胎儿浓度、缺失重复大小、窗口设置以及正常对照组的选择等因素均会影响胎儿微缺失微重复检测的效能。Chen等人[31]采用低深度基因组测序无创检测10M以上缺失重复,1311例孕妇检出4例阳性,有1例假阳性。Yin等人[35]对1476例B超异常孕妇进行无创缺失重复检测,测序深度10M read检测灵敏度为94.5%(69/73),出现55例假阳性,其中35例为母体缺失重复。Helgeson等[36]以全基因组测序对175393例孕妇进行无创检测5p缺失、22q11缺失、15q缺失、1p36缺失、4p缺失、11q缺失和8q缺失;对于22q11缺失,检出32例阳性(0.02%),其中20例母体染色体缺失,12例胎儿染色体缺失;对于1p36、15q和5p缺失,检出20例阳性(0.01%),其中16例真阳性。
基于SNP的目标测序方法也可用于孕妇血浆中胎儿微缺失微重复的检测。针对经常发生缺失或重复而致病的染色体区域,选取足够多的SNP位点设计引物,多重PCR扩增并测序,根据杂合SNP位点的等位基因剂量差异,可判定此位点是否发生缺失或重复,结合众多SNP位点结果可确定此染色体区域的缺失重复及其大小。Wapner等人[37]已将这种方法应用于22q11.2缺失,Prader-Willi/Angelman综合征,1p36缺失综合征和猫叫综合征的无创检测。Gross等人[38]在20776例孕妇中评价SNP方法无创检测22q11.2缺失,检出97例阳性(0.5%),包括11例真阳性、50例假阳性、34例无临床结局和2例母体缺失。

表1 孕妇血浆游离DNA测序可以检测的胎儿疾病
4 孕妇血浆游离DNA测序分析胎儿基因组,DNA甲基化组与转录组
通过孕妇血浆游离DNA和夫妇双方DNA的全基因组深度测序,可获得胎儿的基因组序列,此结论已在多个机构的研究中得以实现[18,39-41],其胎儿的基因型和单倍型分析方法也各异。Lo等[18]最早尝试无创产前分析胎儿基因组,但对夫妇双方均为杂合的SNP位点无法分析,影响了方法的准确性。Fan等[39]和Kitzman等[40]开发了准确的胎儿单倍型和基因型分析方法,分别通过单细胞实验和fosmid实验方法直接获得母方单倍型,但限于胎儿浓度>6%才可准确分析。Chen等[41]开发了结合家系中其他成员来构建夫妇双方单倍型,继而同时分析胎儿的基因型和单倍型的策略,此方法不依赖于血浆胎儿浓度,相对来说更具实用性[42]。
从孕妇血浆游离DNA中也可分析胎儿的DNA甲基化组。Lun等[43]对孕妇血浆游离DNA进行重亚硫酸盐处理,之后进行高通量测序,可以获得包括胎儿DNA和大量母体背景的基因组甲基化谱。根据夫妇双方SNP信息,可以计算出孕妇血浆中胎儿特异的SNP基因型,并据此将胎儿特异的DNA甲基化谱数据从母体背景中分离出来,构建出胎儿的DNA甲基化组。孕妇血浆中胎儿的DNA甲基化组与胎盘很相似,甲基化程度都比较低,从中Lun等人还发现了印记基因以及胎儿与母体的差异甲基化区域。
2000年在孕妇血浆中发现了胎儿游离RNA[44]。将孕妇血浆游离RNA反转录成cDNA进行转录组测序,再根据胎儿特异SNP基因型,可从血浆数据中选出胎儿来源的转录本,从而分析胎儿的转录组[45,46]。通过产前和产后母体血浆转录组的变化分析,可确定PAPPA等一百余个妊娠相关基因的表达情况[45]。通过与已有组织特异表达基因数据库比较,还可以根据孕妇血浆转录组来分析胎儿的组织特异表达基因在不同孕周的变化情况[46]。
5 孕妇血浆游离DNA测序的临床实践
孕妇血浆游离DNA测序的临床应用主要以胎儿常见非整倍体的无创检测为主,包括21-三体,18-三体和13-三体,以及性染色体异常。无创产前检测的临床应用需与传统B超检查、血清学筛查等结合起来,其阳性结果需经介入性产前检测验证。孕妇血浆DNA无创产前检测已在高危孕妇人群中进行临床验证研究[6]并证实其可靠性,而随着这项新技术临床实践不断深入,其在低危人群中的表现也受到关注,并不断扩展至更广泛的人群[7,9]。孕妇血浆无创产前检测的灵敏度为99.17%(21-三体)、98.24%(18-三体)、100%(13-三体),特异性为99.95%(21-三体)、99.95%(18-三体)、99.96%(13-三体)[9],检测性染色体异常的灵敏度约为88.6%(Turner综合征)、93.8%(其他性染色体异常)[47]。其假阳性率远低于传统血清学联合筛查,因此可大大降低不必要的介入性产前检测,也避免由于介入性手术引发的流产风险。孕妇血浆无创产检也可应用于双胎妊娠,目前已经有一些前瞻性研究报道[48-50],双胎妊娠中的21-三体检出率可达94.4%[47]。
一些生物学原因会导致孕妇血浆无创产检结果与胎儿核型出现不一致的情况。由于孕妇血浆中包含胎儿DNA和大量的母体DNA背景,因此当母体DNA存在染色体异常时,会干扰对胎儿染色体片段的分析,使无创产前检测结果出现假阳性,其假阳性率、或不一致率在临床研究中约为1%~2%水平。除了母体染色体异常[51]外,目前报道可以导致无创产检结果与胎儿核型结果不一致的情况还包括胎盘特异性嵌合[52,53],母体嵌合[54]等。亦有报道母体癌症[11,55,56]也可在无创产检中检出,Bianchi等[55]在3757例NIPT阳性案例中发现10例母体癌症。这些孕妇自身发生肿瘤的病例,孕妇本人在进行无创产前检测时普遍存在非特异性的多条染色体数目异常的结果,从而造成NIPT的假阳性结果。Amant等[56]也发现了类似现象。由于上述生物学因素在临床实践中难以避免,因此业内多将孕妇血浆无创产前检测作为一种产前筛查,而非诊断性检测,其阳性结果必须经介入性产前检测的验证。针对这项新型检测技术,包括我国、美国与欧洲等多个专业协会专家均发表了临床指导与共识意见[57-59],指导其临床应用。
对于无创产前检测胎儿其他染色体非整倍体、微缺失与微重复以及单基因遗传疾病的临床应用,业内多持谨慎态度。染色体微缺失微重复的片段较小,其对应的表型往往难以预测,当产前检出胎儿阳性时,其表型可能是完全正常的[60],因此给临床决策带来困难。且因微缺失微重复发病率低,临床检测中的阳性预测值很低,因此很多专业团体并不建议将无创检测微缺失微重复作为常规的产前检测[59],而是针对特定人群开展[61]。目前对于无创微缺失微重复检测的临床实践仍存争议[62]。而关于其他非整倍体,Snyder等[11]统计了113 415例孕妇,检测其他非整倍体阳性138例(0.12%),在67例进行了核型分析的孕妇中,47例(70%)胎儿染色体正常而为假阳性。国际产前诊断协会(International Society for Prenatal Diagnosis,ISPD)2015年声明[57]指出,无创微缺失微重复检测及其他非整倍体检测应限于临床意义明确或已知严重表型的疾病,每种疾病应有明确的检出率、假阳性率等指标,并配套相应的专业咨询。应由孕妇自己决定是否检测和获知结果,并在临床咨询中详细告知疾病信息和检测的局限性。单基因遗传病患病人群小,市场需求少,因其无创产前检测同时需要家系的遗传信息,分析周期相对较长,同时费用也更加昂贵,所以目前总体上仍处于实验室研究阶段,以个案的形式开展,并未进入常规临床实践中。无论是无创产前检测微缺失微重复、其他非整倍体还是单基因病,均以孕中晚期的介入性产前检测结果作为金标准。关于无创单基因病检测的临床推广仍需继续探讨。而胎儿的无创基因组、无创DNA甲基化和转录组分析亦处于技术研发阶段,有待继续发展。
6 不同基因组分析平台的应用
在无创产前检测中应用的NGS平台有多种,目前主流使用的是Illumina公司的Hiseq2000平台[4,7,12,15,18],此外还有小型化的测序平台MiSeq[63],Life Technologies公司的SOLiD[64]以及半导体测序系统Ion torrent/Ion Proton[8,35,65]。随着测序技术的发展,单分子测序平台Helicos[66]以及新型纳米孔测序平台nanopore[67]也开始应用于无创产前检测的技术研究。2014年,我国自主开发的BGISEQ1000测序平台[68]首次获得国家食品药品监督管理总局(CFDA)认证,对应的胎儿染色体非整倍体(T21、T18、T13)检测试剂盒(联合探针锚定连接测序法)亦获得CFDA审批,继而小型化的BGISEQ500测序平台亦于2015年推出,目前已进入无创产前检测的临床测试阶段。除NGS,亦有将目标染色体片段制作成微阵列芯片进行检测[69]。NGS等基因组分析多种平台的应用及其技术不断创新,为无创产前检测的发展提供强大动力。
7 结论
孕妇血浆游离DNA高通量测序技术的快速发展,使无创产前检测胎儿的非整倍体、单基因遗传病、微缺失微重复以及胎儿基因组、DNA甲基化组和转录组成为现实。胎儿非整倍体无创产前检测的临床实践不断开展,也推动着孕妇血浆游离DNA测序的临床应用不断扩展。同时,我们也应看到,在技术发展同时,其应用规范化也至关重要,要求我们在实践中不断进行评估和总结,使孕妇血浆无创产前检测这项新型技术能够为医学实践带来更多益处。
[1]Lo YM,Corbetta N,Chamberlain PF,et al.Presence of fetal DNA in maternal plasma and serum[J].Lancet,1997,350:485-487.
[2]Lo YM,Lau TK,Zhang J,et al.Increased fetal DNA concentrations in the plasma of pregnant women carrying fetuses with trisomy 21[J].Clin Chem,1999,45:1747-1751.
[3]Jiang F,Ren J,Chen F,et al.Noninvasive Fetal Trisomy (NIFTY) test: an advanced noninvasive prenatal diagnosis methodology for fetal autosomal and sex chromosomal aneuploidies[J].BMC Med Genomics,2012,5:57.
[4]Dan S,Wang W,Ren J,et al.Clinical application of massively parallel sequencing-based prenatal noninvasive fetal trisomy test for trisomies 21 and 18 in 11,105 pregnancies with mixed risk factors[J].Prenat Diagn,2012,32:1225-1232.
[5]Chiu RW,Akolekar R,Zheng YW,et al.Non-invasive prenatal assessment of trisomy 21 by multiplexed maternal plasma DNA sequencing: large scale validity study[J].BMJ,2011,342:c7401.
[6]Bianchi DW,Platt LD,Goldberg JD,et al.Genome-wide fetal aneuploidy detection by maternal plasma DNA sequencing[J].Obstet Gynecol,2012,119:890-901.
[7]Bianchi DW,Parker RL,Wentworth J,et al.DNA sequencing versus standard prenatal aneuploidy screening[J].N Engl J Med,2014,370:799-808.
[8]Liao C,Yin AH,Peng CF,et al.Noninvasive prenatal diagnosis of common aneuploidies by semiconductor sequencing[J].Proc Natl Acad Sci U S A,2014,111:7415-7420.
[9]Zhang H,Gao Y,Jiang F,et al.Non-invasive prenatal testing for trisomies 21,18 and 13: clinical experience from 146,958 pregnancies[J].Ultrasound Obstet Gynecol,2015,45:530-538.
[10]Bayindir B,Dehaspe L,Brison N,et al.Noninvasive prenatal testing using a novel analysis pipeline to screen for all autosomal fetal aneuploidies improves pregnancy management[J].Eur J Hum Genet,2015,23:1286-1293.
[11]Snyder HL,Curnow KJ,Bhatt S,et al.Follow-up of multiple aneuploidies and single monosomies detected by noninvasive prenatal testing: implications for management and counseling[J].Prenat Diagn,2016,36:203-239.
[12]Sparks AB,Struble CA,Wang ET,et al.Noninvasive prenatal detection and selective analysis of cell-free DNA obtained from maternal blood: evaluation for trisomy 21 and trisomy 18[J].Am J Obstet Gynecol,2012,206:319 e1-9.
[13]Norton ME,Brar H,Weiss J,et al.Non-Invasive Chromosomal Evaluation (NICE) Study: results of a multicenter prospective cohort study for detection of fetal trisomy 21 and trisomy 18[J].Am J Obstet Gynecol,2012,207:137 e1-8.
[14]Zimmermann B,Hill M,Gemelos G,et al.Noninvasive prenatal aneuploidy testing of chromosomes 13,18,21,X,and Y,using targeted sequencing of polymorphic loci[J].Prenat Diagn,2012,32:1233-1241.
[15]Hall MP,Hill M,Zimmermann B,et al.Non-invasive prenatal detection of trisomy 13 using a single nucleotide polymorphism-and informatics-based approach[J].PLoS One,2014,9:e96677.
[16]Nicolaides KH,Syngelaki A,Gil M,et al.Validation of targeted sequencing of single-nucleotide polymorphisms for non-invasive prenatal detection of aneuploidy of chromosomes 13,18,21,X,and Y[J].Prenat Diagn,2013,33:575-579.
[17]Chan KC,Zhang J,Hui AB,et al.Size distributions of maternal and fetal DNA in maternal plasma[J].Clin Chem,2004,50:88-92.
[18]Lo YM,Chan KC,Sun H,et al.Maternal plasma DNA sequencing reveals the genome-wide genetic and mutational profile of the fetus[J].Sci Transl Med,2010,2:841-849.
[19]Kim SK,Hannum G,Geis J,et al.Determination of fetal DNA fraction from the plasma of pregnant women using sequence read counts[J].Prenat Diagn,2015,35:810-815.
[20]Tong YK,Jin S,Chiu RW,et al.Noninvasive prenatal detection of trisomy 21 by an epigenetic-genetic chromosome-dosage approach[J].Clin Chem,2010,56:90-98.
[21]Nygren AO,Dean J,Jensen TJ,et al.Quantification of fetal DNA by use of methylation-based DNA discrimination[J].Clin Chem,2010,56:1627-1635.
[22]Lo YM,Tsui NB,Chiu RW,et al.Plasma placental RNA allelic ratio permits noninvasive prenatal chromosomal aneuploidy detection[J].Nat Med,2007,13:218-223.
[23]Lam KW,Jiang P,Liao GJ,et al.Noninvasive prenatal diagnosis of monogenic diseases by targeted massively parallel sequencing of maternal plasma: application to beta-thalassemia[J].Clin Chem,2012,58:1467-1475.
[24]Ge H,Huang X,Li X,et al.Noninvasive prenatal detection for pathogenic CNVs: the application in alpha-thalassemia[J].PLoS One,2013,8:e67464.
[25]Ma D,Ge H,Li X,et al.Haplotype-based approach for noninvasive prenatal diagnosis of congenital adrenal hyperplasia by maternal plasma DNA sequencing[J].Gene,2014,544:252-258.
[26]New MI,Tong YK,Yuen T,et al.Noninvasive prenatal diagnosis of congenital adrenal hyperplasia using cell-free fetal DNA in maternal plasma[J].J Clin Endocrinol Metab,2014,99:E1022-1030.
[27]Xu Y,Li X,Ge HJ,et al.Haplotype-based approach for noninvasive prenatal tests of Duchenne muscular dystrophy using cell-free fetal DNA in maternal plasma[J].Genet Med,2015,17:889-896.
[28]You Y,Sun Y,Li X,et al.Integration of targeted sequencing and NIPT into clinical practice in a Chinese family with maple syrup urine disease[J].Genet Med,2014,16:594-600.
[29]Meng M,Li X,Ge H,et al.Noninvasive prenatal testing for autosomal recessive conditions by maternal plasma sequencing in a case of congenital deafness[J].Genet Med,2014,16:972-976.
[30]Peters D,Chu T,Yatsenko SA,et al.Noninvasive prenatal diagnosis of a fetal microdeletion syndrome[J].N Engl J Med,2011,365:1847-1848.
[31]Chen S,Lau TK,Zhang C,et al.A method for noninvasive detection of fetal large deletions/duplications by low coverage massively parallel sequencing[J].Prenat Diagn,2013,33:584-590.
[32]Yu SC,Jiang P,Choy KW,et al.Noninvasive prenatal molecular karyotyping from maternal plasma[J].PLoS One,2013,8:e60968.
[33]Srinivasan A,Bianchi DW,Huang H,et al.Noninvasive detection of fetal subchromosome abnormalities via deep sequencing of maternal plasma[J].Am J Hum Genet,2013,92:167-176.
[34]Zhao C,Tynan J,Ehrich M,et al.Detection of fetal subchromosomal abnormalities by sequencing circulating cell-free DNA from maternal plasma[J].Clin Chem,2015,61:608-616.
[35]Yin AH,Peng CF,Zhao X,et al.Noninvasive detection of fetal subchromosomal abnormalities by semiconductor sequencing of maternal plasma DNA[J].Proc Natl Acad Sci U S A,2015,112:14670-14675.
[36]Helgeson J,Wardrop J,Boomer T,et al.Clinical outcome of subchromosomal events detected by whole-genome noninvasive prenatal testing[J].Prenat Diagn,2015,35:999-1004.
[37]Wapner RJ,Babiarz JE,Levy B,et al.Expanding the scope of noninvasive prenatal testing: detection of fetal microdeletion syndromes[J].Am J Obstet Gynecol,2015,212:332 e1-9.
[38]Gross SJ,Stosic M,McDonald-McGinn DM,et al.Clinical experience with single-nucleotide polymorphism-based non-invasive prenatal screening for 22q11.2 deletion syndrome[J].Ultrasound Obstet Gynecol,2016,47:177-183.
[39]Fan HC,Gu W,Wang J,et al.Non-invasive prenatal measurement of the fetal genome[J].Nature,2012,487:320-324.
[40]Kitzman JO,Snyder MW,Ventura M,et al.Noninvasive whole-genome sequencing of a human fetus[J].Sci Transl Med,2012,4:137ra76.
[41]Chen S,Ge H,Wang X,et al.Haplotype-assisted accurate non-invasive fetal whole genome recovery through maternal plasma sequencing[J].Genome Med,2013,5:18.
[42]Papageorgiou EA,Patsalis PC.Maternal plasma sequencing: a powerful tool towards fetal whole genome recovery[J].BMC Med,2013,11:56.
[43]Lun FM,Chiu RW,Sun K,et al.Noninvasive prenatal methylomic analysis by genomewide bisulfite sequencing of maternal plasma DNA[J].Clin Chem,2013,59:1583-1594.
[44]Poon LL,Leung TN,Lau TK,et al.Presence of fetal RNA in maternal plasma[J].Clin Chem,2000,46:1832-1834.
[45]Tsui NB,Jiang P,Wong YF,et al.Maternal plasma RNA sequencing for genome-wide transcriptomic profiling and identification of pregnancy-associated transcripts[J].Clin Chem,2014,60:954-962.
[46]Koh W,Pan W,Gawad C,et al.Noninvasive in vivo monitoring of tissue-specific global gene expression in humans[J].Proc Natl Acad Sci U S A,2014,111:7361-7366.
[47]Gil MM,Akolekar R,Quezada MS,et al.Analysis of cell-free DNA in maternal blood in screening for aneuploidies: meta-analysis[J].Fetal Diagn Ther,2014,35:156-173.
[48]Lau TK,Jiang F,Chan MK,et al.Non-invasive prenatal screening of fetal Down syndrome by maternal plasma DNA sequencing in twin pregnancies[J].J Matern Fetal Neonatal Med,2013,26:434-437.
[49]Huang X,Zheng J,Chen M,et al.Noninvasive prenatal testing of trisomies 21 and 18 by massively parallel sequencing of maternal plasma DNA in twin pregnancies[J].Prenat Diagn,2014,34:335-340.
[50]Canick JA,Kloza EM,Lambert-Messerlian GM,et al.DNA sequencing of maternal plasma to identify Down syndrome and other trisomies in multiple gestations[J].Prenat Diagn,2012,32:730-734.
[51]Sehnert AJ,Rhees B,Comstock D,et al.Optimal detection of fetal chromosomal abnormalities by massively parallel DNA sequencing of cell-free fetal DNA from maternal blood[J].Clin Chem,2011,57:1042-1049.
[52]Pan M,Li FT,Li Y,et al.Discordant results between fetal karyotyping and non-invasive prenatal testing by maternal plasma sequencing in a case of uniparental disomy 21 due to trisomic rescue[J].Prenat Diagn,2013,33:598-601.
[53]Gao Y,Stejskal D,Jiang F,et al.False-negative trisomy 18 non-invasive prenatal test result due to 48,XXX,+18 placental mosaicism[J].Ultrasound Obstet Gynecol,2014,43:477-478.
[54]Lau TK,Jiang FM,Stevenson RJ,et al.Secondary findings from non-invasive prenatal testing for common fetal aneuploidies by whole genome sequencing as a clinical service[J].Prenat Diagn,2013,33:602-608.
[55]Bianchi DW,Chudova D,Sehnert AJ,et al.Noninvasive Prenatal Testing and Incidental Detection of Occult Maternal Malignancies[J].JAMA,2015,314:162-169.
[56]Amant F,Verheecke M,Wlodarska I,et al.Presymptomatic identification of cancers in pregnant women during noninvasive prenatal testing[J].JAMA Oncol,2015,1:814-819.
[57]Benn P,Borrell A,Chiu RW,et al.Position statement from the chromosome abnormality screening committee on behalf of the board of the international society for prenatal diagnosis[J].Prenat Diagn,2015,35:725-734.
[58]Dondorp W,de Wert G,Bombard Y,et al.Non-invasive prenatal testing for aneuploidy and beyond: challenges of responsible innovation in prenatal screening.Summary and recommendations[J].Eur J Hum Genet,doi: 10.1038/ejhg.2015.56.[Epub ahead of print].
[59]Committee opinion No.640: Cell-free DNA screening for fetal aneuploidy[J].Obstet Gynecol,2015,126:e31-37.
[60]Jia Y,Zhao H,Shi D,et al.Genetic effects of a 13q31.1 microdeletion detected by noninvasive prenatal testing (NIPT)[J].Int J Clin Exp Pathol,2014,7:7003-7011.
[61]Vora NL,O'Brien BM.Noninvasive prenatal testing for microdeletion syndromes and expanded trisomies: proceed with caution[J].Obstet Gynecol,2014,123:1097-109.
[62]Rose NC,Benn P,Milunsky A.Current controversies in prenatal diagnosis 1: should NIPT routinely include microdeletions/microduplications?[J].Prenat Diagn,2016,36:10-14.
[63]Xie W,Tan Y,Li X,et al.Rapid detection of aneuploidies on a benchtop sequencing platform[J].Prenat Diagn,2013,33:232-237.
[64]Faas BH,de Ligt J,Janssen I,et al.Non-invasive prenatal diagnosis of fetal aneuploidies using massively parallel sequencing-by-ligation and evidence that cell-free fetal DNA in the maternal plasma originates from cytotrophoblastic cells[J].Expert Opin Biol Ther,2012,12 Suppl 1:S19-26.
[65]Yuan Y,Jiang F,Hua S,et al.Feasibility study of semiconductor sequencing for noninvasive prenatal detection of fetal aneuploidy[J].Clin Chem,2013,59:846-849.
[66]van den Oever JM,Balkassmi S,Verweij EJ,et al.Single molecule sequencing of free DNA from maternal plasma for noninvasive trisomy 21 detection[J].Clin Chem,2012,58:699-706.
[67]Cheng SH,Jiang P,Sun K,et al.Noninvasive prenatal testing by nanopore sequencing of maternal plasma DNA: feasibility assessment[J].Clin Chem,2015,61:1305-1306.
[68]Drmanac R,Peters BA,Church GM,et al.Accurate whole genome sequencing as the ultimate genetic test[J].Clin Chem,2015,61:305-306.
[69]Juneau K,Bogard PE,Huang S,et al.Microarray-based cell-free DNA analysis improves noninvasive prenatal testing[J].Fetal Diagn Ther,2014,36:282-286.
编辑:熊诗诣
10.13470/j.cnki.cjpd.2016.02.012
出生缺陷筛查工程实验室项目[JZF No.(2011) 861]
王威,E-mail:wangw@genomics.cn
R714.53
A
2016-04-30)
