苦豆子赖氨酸脱羧酶基因克隆与表达分析
2016-09-14杨毅陆姗姗刘萍田蕾
杨毅,陆姗姗,刘萍,田蕾
(宁夏大学农学院,宁夏 银川 750021)
苦豆子赖氨酸脱羧酶基因克隆与表达分析
杨毅,陆姗姗,刘萍*,田蕾
(宁夏大学农学院,宁夏 银川 750021)
赖氨酸脱羧酶(lysine decarboxylase,LDC)基因是苦豆子中氧化苦参碱(oxymatrine,OMA)生物合成的第一个关键酶基因。根据近缘物种苦参的赖氨酸脱羧酶基因设计特异引物,同源克隆法克隆了苦豆子赖氨酸脱羧酶基因的蛋白质编码区序列,全长1368 bp,命名为Sa-LDC,GenBank登录号为KM249871。生物信息学分析表明Sa-LDC编码区序列无内含子,与苦参和狗苦参的LDC序列一致性均达到97%;属于Ⅲ型5-磷酸吡哆醛依赖酶[type Ⅲ pyridoxal 5-phosphate (PLP)-dependent enzymes,PLPDE-Ⅲ]超基因家族,功能活跃。Sa-LDC编码455个氨基酸残基,其编码的肽链相对分子质量49.14 kD,理论等电点5.63,无信号肽和跨膜结构;在其氨基酸序列中具有产喹诺里西啶生物碱的特征性保守位点Phe340;系统进化树将苦豆子与其他产喹诺里西啶类生物碱的植物聚为一类。qPCR和HPLC检测显示,苦豆子赖氨酸脱羧酶基因的表达和氧化苦参碱的积累均受干旱胁迫的影响,且基因的表达量与氧化苦参碱的积累呈正相关关系。
苦豆子;赖氨酸脱羧酶基因;基因克隆;基因表达;氧化苦参碱
苦豆子(Sophoraalopecuriodes)是豆科槐属多年生草本植物[1],广泛分布于宁夏、新疆、内蒙古、甘肃、西藏等的荒漠和半荒漠地区,由于其根茎繁殖力极强,根系发达,枝叶繁茂,在地表干沙层能抗风蚀,是西北地区生态环境保护中优良的固沙植物[2]。氧化苦参碱(oxymatrine,OMA)又称苦参素,是苦豆子中重要的生物碱之一,不仅在抗肝损伤、抗肿瘤、抗心血管疾病等方面有良好的疗效,还具有镇痛、免疫调节和治疗慢性乙型肝炎等多种功效[3]。
OMA属于喹诺里西啶类生物碱(quinolizidine alkaloids,QAs),赖氨酸脱羧酶(lysine decarboxylase,LDC)是QAs生物合成第一酶,它可催化赖氨酸脱羧生成戊二胺(尸胺),3个戊二胺分子和4个丙酮酸盐在17-氧基-鹰爪豆碱合酶(17-oxosparteine synthase)的作用下生成17-氧基-鹰爪豆碱(也称金雀花碱,17-oxosparteine)和4个丙氨酸,17-氧基-鹰爪豆碱经脱氢金雀花碱生成鹰爪豆碱,再经一系列生化代谢最终转化为OMA[4-7]。LDC作为喹诺里西啶生物合成途径的第一个酶基因,在OMA生物合成过程中有着至关重要的作用。2011年Bunsupa等[8]对产QAs与不产QAs的两种羽扇豆通过差异基因表达谱(differential gene expression profile)分析证明LDC的表达与植物合成QAs直接相关;2012年Bunsupa等[7]分别从苦参(Sophoraflavescens)、狗苦参(Echinosophorakoreensis)、羽扇豆(Lupinusangustifolius)、蓝花赝靛(Baptisiaaustralis)及小叶野决明(Thermopsischinensis)中分离出LDC序列,并将其转入烟草悬浮细胞和毛状根中进行功能验证,同时明确产QAs的植物其LDC中均具有Phe340。杜次等[9]在蛇足石杉(Huperziaserrata)中克隆到2个序列一致性高达95.3%的LDC序列,通过原核表达均得到可催化赖氨酸脱羧形成尸胺的产物。由于苦豆子重要的生态和药用价值,和其他产QAs的植物相比,对苦豆子LDC的研究目前还很少,而关于OMA生物合成与代谢途径中关键基因及其调控机理更是知之甚少。鉴于此,本研究采用同源克隆法克隆苦豆子活性成分OMA生物合成关键酶基因LDC,并进行生物信息学分析,探讨PEG胁迫下苦豆子LDC表达与OMA含量变化的相互关系,为今后详细研究苦豆子LDC的生物学功能和作用机理奠定基础,也为研究氧化苦参碱生物合成和代谢调控机制提供依据。
1 材料与方法
1.1试验材料
苦豆子植株由宁夏大学农学院李晓伟博士鉴定为Sophoraalopecuroides,荚果于2013年采于宁夏永宁县杨和乡(106.14° E,38.14° N),标本号为103,于室温自然风干后脱粒并精选,-20 ℃低温保存。
1.2苦豆子LDC基因gDNA和cDNA全长序列克隆
采用改良的SDS(十二烷基磺酸钠)法[10]提取gDNA。总RNA提取及cDNA第一链合成参照试剂盒说明书进行。
参照NCBI已公布的苦豆子近缘物种苦参LDCcDNA序列设计特异性引物Sa-LDC-Pri(表1),分别以苦豆子gDNA和cDNA为模板进行PCR扩增,扩增产物连接至PMD18-T载体,转化感受态大肠杆菌菌株TOP10,氨苄青霉素抗性平板筛选阳性克隆,阳性单克隆送生工生物工程(上海)股份有限公司测序。
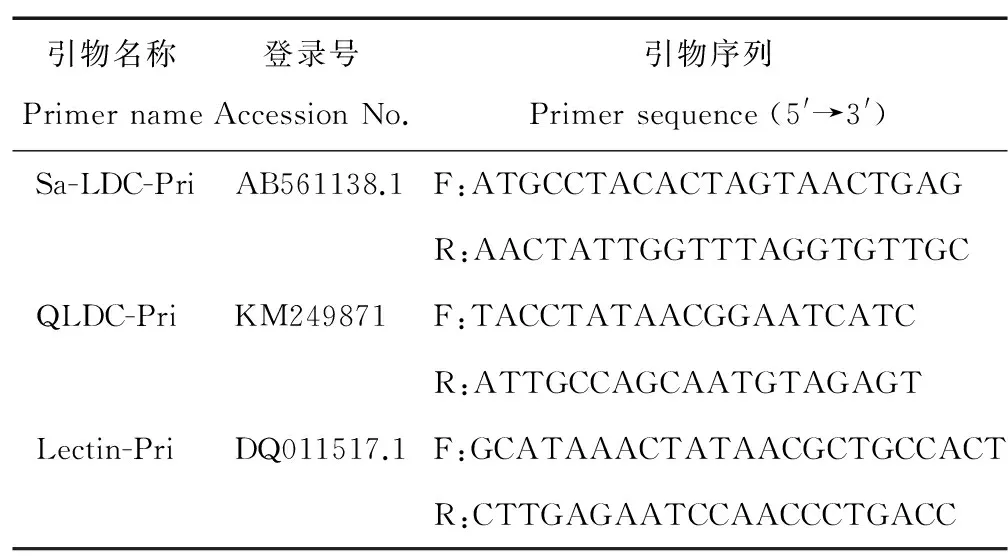
表1 用于基因克隆及荧光定量PCR的引物序列
1.3苦豆子LDC生物信息学分析
将测序获得的赖氨酸脱羧酶基因序列提交至NCBI进行BLAST比对。采用MEGA 5.0 Neighbor-Joining法构建系统进化树[11], ExPASy-ProtParam tool进行蛋白质理化性质分析[12],TMHMM分析跨膜结构[13],SOPMA在线软件预测蛋白的二级结构[14],PSORT在线软件进行亚细胞定位预测[15],www.predictprotein.org在线网站预测蛋白质修饰位点[16],SignalP 4.1预测信号肽[17],Swiss model构建蛋白质三维模型[18]。
1.4苦豆子植株的PEG胁迫、qPCR和OMA含量测定
选取大小均匀、饱满、无虫蛀的苦豆子种子,浓硫酸处理20 min,清水冲洗干净后,置25 ℃恒温黑暗培养箱中萌动种子,待露白后播种于直径40 cm的花盆中,自然条件下生长至幼苗约15 cm高,连根挖出彻底洗净根部泥土,用吸水纸吸干表面水分后,将根系全部浸泡于质量分数为10%,20%和30%的PEG 6000溶液中以胁迫苦豆子植株,以去离子水为对照,每一处理3组重复。分别于0,1,4,8和24 h时采集叶片,一部分液氮速冻后-80 ℃保存用于qPCR,另一部分120 ℃杀青后烘干,用于测定OMA含量。
qPCR反应在Stepone plus型荧光定量PCR仪(ABI)上进行,以苦豆子Lectin序列为模板设计内参引物Lectin-pri,以测序获得LDC序列设计荧光定量引物QLDC-pri(表1),采用20 μL体系:SybrGreen qPCR Master Mix 10 μL,上下游引物各1 μL,cDNA 1 μL,ddH2O 7 μL。扩增程序:95 ℃ 3 min,95 ℃ 15 s,60 ℃ 40 s,40个循环。按照2-ΔΔCT法计算出基因的相对表达量,即ΔCt 目标基因=Ct(LDC)-Ct(Lectin);ΔΔCt(LDC)=处理组(ΔCtLDC)-对照组(ΔCtLDC),相对表达量=2-ΔΔCT(LDC)[19]。
OMA提取和测定参照杨毅等[20]的方法。
1.5统计分析
利用DPS 6.0进行方差分析,Excel作图。
2 结果与分析

图1 Sa-LDC基因gDNA和cDNA扩增结果Fig.1 The electrophoresis results of Sa-LDC genes 1,2分别为以苦豆子cDNA和gDNA为模板扩增的赖氨酸脱羧酶基因; Marker:从上到下依次为2000,1000,750,500,250,100 bp。1: LDC fragment obtained from the template of cDNA; 2: LDC fragment obtained from the template of gDNA; M: 2000,1000,750,500,250,100 bp from top to bottom.
2.1苦豆子LDCgDNA和cDNA的克隆与生物信息学分析
2.1.1苦豆子LDC基因克隆及序列分析分别以苦豆子gDNA和cDNA为模板,利用特异性引物SaLDC-Pri进行PCR扩增,都获得约1400 bp大小的条带(图1)。经测序比对后发现,该基因gDNA和cDNA序列完全一致,编码区全长1368 bp(图2),说明该基因无内含子。将该编码区序列提交至NCBI进行BLAST比对后发现,序列与狗苦参和苦参LDC序列一致性均达到97%,与小叶野决明和蓝花赝靛LDC序列一致性达到86%,与羽扇豆LDC序列一致性达到82%,表明扩增所得序列确实为苦豆子LDC编码区序列。将序列提交至GenBank,命名为Sa-LDC,登录号KM249871。
2.1.2Sa-LDC蛋白结构分析通过对Sa-LDC开放阅读框分析发现,该基因共编码455个氨基酸残基,Sa-LDC蛋白含PLPDE_Ⅲ_ODC、Orn_Arg_deC_N、LysA、PRK08961和PLN02537五个结构功能域,属于Ⅲ型5-磷酸吡哆醛依赖酶[type Ⅲ pyridoxal 5-phosphate (PLP)-dependent enzymes,PLPDE-Ⅲ]超基因家族;在该蛋白第75~405个氨基酸范围内,存在多个活性位点,推测该氨基酸序列范围可能是Sa-LDC的功能区域。理化性质和二级结构预测显示Sa-LDC属于水溶性蛋白,分子式为C2192H3416N574O658S25,分子量为49.14 kD,无信号肽和跨膜结构,理论等电点5.63,带负电残基(Asp+Glu)数49,带正电残基(Arg+Lys)数42,氨基酸整体带负电,为酸性蛋白。不稳定系数为41.92,是一个不稳定蛋白。疏水系数-0.02,脂肪系数为81.08。其二级结构主要由α-螺旋(38.2%)和无规则卷曲(37.8%)构成(图3)。亚细胞定位预测显示Sa-LDC蛋白最可能位于叶绿体中。由于该蛋白N端无信号肽剪切位点,推测其不属于分泌蛋白,在细胞质中合成后不能进行转运。此外,在Sa-LDC蛋白序列中找到产QAs植物的特征性保守位点Phe340。

图2 Sa-LDC基因序列Fig.2 The sequence of Sa-LDC genes

图3 赖氨酸脱羧酶蛋白结构三维模型Fig.3 Three-dimensional model of Sa-LDC protein structure
2.1.3Sa-LDC氨基酸序列比对及系统进化树构建
将推导的氨基酸序列与NCBI上已公布的L/ODC蛋白进行BLASTp同源性比对发现,Sa-LDC氨基酸序列与狗苦参和苦参的LDC氨基酸序列一致性高达97%;但与大豆(Glycinemax)LDC氨基酸序列一致性仅为77%。不同植物L/ODC氨基酸序列在功能区域相对保守,差异主要存在于N-端和C-端。应用Neighbor-Joining法构建的系统进化树结果显示,所有豆科植物被聚为一个大类,苦豆子与苦参遗传距离最近,与百脉根(Lotusjaponicus)遗传距离最远;所有产喹诺里西啶生物碱的植物聚为一类,其氨基酸序列中均有特征性保守位点Phe340;大豆和蒺藜苜蓿(Medicagotruncatula)聚在一起,百脉根则与它们的遗传距离较远,单独分为一类(图4)。总体上,系统进化树与传统分类结果一致。
2.1.4Sa-LDC蛋白修饰位点分析蛋白质翻译后修饰会直接影响其多种属性[21]。对Sa-LDC蛋白修饰位点分析发现,该蛋白共含8种类型修饰位点,分别是N-糖基化位点、cAMP和cGMP依赖型蛋白激酶磷酸化位点、蛋白激酶C磷酸化位点、酪蛋白II磷酸化位点、酪氨酸激酶磷酸化位点、N-豆蔻酰化位点、酰胺化位点以及磷酸吡哆醛结合位点。

图4 苦豆子Sa-LDC和L/ODC同源序列的系统进化树Fig.4 Phylogenetic tree estimated by the amino acid sequences of S. alopecuroides and other plant L/ODCs 粗线所示为产QAs植物;黑框所示为产QAs植物氨基酸序列特征性保守位点Phe340。Bold line indicates the QAs-producing plants; Black box indicates the conserved amino acid residue Phe340.
2.2PEG胁迫下苦豆子叶片中LDC表达量和OMA含量的动态变化
用不同质量分数PEG溶液胁迫苦豆子植株根系,qPCR结果表明苦豆子叶片中LDC表达量在胁迫前处于较低的水平,对照组叶片LDC表达量在去离子水中随时间延长呈上升趋势;在质量分数为10%的PEG胁迫下,叶片中LDC表达量在8 h时骤然升高,与0~4 h时的表达量差异显著,是同时期对照组的1.6倍,胁迫前的110倍,随后表达量开始下降,在胁迫结束时其基因表达量与胁迫前相当;在20%的PEG胁迫下,苦豆子叶片LDC表达量在1 h时达到最高,是同时期对照组的27.5倍,胁迫前的343倍。随胁迫时间延长,表达量下降,但在胁迫结束时其表达量仍然是胁迫前的21倍;在30%的PEG胁迫下,基因表达量一直处于较低水平(图5A)。PEG胁迫也影响了苦豆子叶片中OMA的积累,使其含量发生变化。对照组中苦豆子叶片OMA含量在1 h时最低,随后逐渐升高,8 h后与0 h叶片中OMA含量无显著差异;在10%的PEG胁迫下,苦豆子叶片OMA含量在胁迫8 h时骤然上升,与1~4 h时的OMA含量差异显著且高于同时期对照组的OMA含量;苦豆子叶片OMA积累对20%的PEG胁迫响应迅速,胁迫1 h时OMA含量升至最高,随后开始下降,在胁迫24 h时其含量与胁迫前无差异;30%的PEG胁迫显著抑制叶片中OMA积累,从胁迫开始直到结束,叶片中OMA含量均显著低于胁迫前(图5B)。

图5 PEG胁迫下苦豆子叶片中LDC表达量(A)和OMA含量(B)变化Fig.5 The LDC expression level (A) and OMA content (B) under PEG stress in S. alopecuroides leaves 不同小写字母表示差异显著;图5A中纵坐标为基因表达量的对数值。Different small letters indicate significant differences at 0.05 level; The y-coordinate is logarithm of gene expression level in Fig.5A.
3 讨论
LDC作为QAs生物合成的第一个关键酶基因,在苦豆子OMA生物合成和代谢过程中有至关重要的作用。本研究首次获得了苦豆子LDC编码区序列,命名为Sa-LDC,GenBank登录号KM249871。对推测的Sa-LDC蛋白序列分析表明该蛋白属于Ⅲ型5-磷酸吡哆醛依赖酶超基因家族。亚细胞定位预测显示该蛋白存在于叶绿体中,与Wink等[22]通过同位素示踪法研究的结果一致。除L/ODC外,同属该超基因家族的还有二氨基庚二酸脱羧酶(diaminopimelate decarboxylase,DapDC)、精氨酸脱羧酶(arginine decarboxylase,ADC)以及羧基降亚精胺脱羧酶(carboxynorspermidine decarboxylase,CANSDC)。该家族的酶主要在辅酶5-磷酸吡哆醛的参与下,催化聚胺或赖氨酸脱羧的生化反应[23]。在Sa-LDC蛋白修饰位点中存在多个磷酸化位点和酰胺化位点,说明此蛋白主要由蛋白激酶和蛋白因子所激活以行使生化功能,极易被外界因素所调控。
BLASTp同源性比对表明Sa-LDC氨基酸序列与同是豆科槐属的苦参和狗苦参亲缘关系最近,说明在亲缘关系相近的植物中LDC的保守性强,这与陈春艳等[24]在对甘肃红豆草(Onobrychisviciaefoliacv. “Gansu”)无色花青素还原酶 LAR基因的同源性分析结论相一致。LDC的系统进化树分析表明苦豆子与苦参、狗苦参、蓝花赝靛、小叶野决明和羽扇豆这几种产QAs的植物聚为一类,且其氨基酸序列中都具有共同的特征性保守位点Phe340,说明苦豆子的赖氨酸脱羧酶与这些植物的赖氨酸脱羧酶具有共同的起源。与Bunsupa等[25]研究结果所不同的是,本研究找到的Phe在氨基酸序列的第340位,而Bunsupa等[25]认为在氨基酸序列的第344位。
生物碱在保护植物中有着特殊的作用,有研究表明在遇到重度胁迫和生物侵扰时植物会瞬间大量合成生物碱[26-27]。Christiansen等[28]对3种窄叶羽扇豆3个不同生长阶段的干旱胁迫研究认为,干旱造成窄叶羽扇豆种子中生物总碱含量发生变化,其中营养生长阶段的干旱胁迫促使其生物总碱含量上升;杨毅等[20]用不同质量分数的PEG胁迫刚萌动的苦豆子种子,发现轻度胁迫下子叶中OMA含量降低,但在重度胁迫下OMA含量又有所回升,且轻度和重度胁迫下OMA含量与LDC表达量的变化基本一致。本研究中苦豆子植株在10% PEG中胁迫8 h,叶片中LDC表达量突然升高,与之对应的OMA含量也同时明显上升;同样,20%的PEG胁迫1 h时叶片中LDC表达量的升高也促使OMA含量增加,说明苦豆子LDC的表达受干旱胁迫的调控,轻度的干旱(PEG≤10%)胁迫需要较长时间才能诱导LDC表达,并促使OMA合成;但在中度干旱(10% 本研究首次从苦豆子中克隆获得LDC编码区全长序列,共1368 bp,编码区无内含子,共编码455个氨基酸残基,生物信息学分析表明赖氨酸脱羧酶功能较为活跃,其氨基酸序列中具有产QAs的特征性保守位点Phe340,LDC的表达受干旱胁迫的影响,在一定程度上也影响OMA的积累。 References: [1]Editorial Committee of Flora of China. Flora Reipublicae Popularis Sinicae[M]. Beijing: Science Press, 1998: 80-81. [2]Li A H, Sun Z J. The developmental situation and application potential ofSopharaalopecuroidesL. Journal of Ningxia University: Natural Science Edition, 2000, 21(4): 354-356. [3]Zhou F S, Mu Q. Chemical constituent and pharmacological activities ofSophoraalopecuroidesL.Chinese Wild Plant Resources, 2006, 25(4): 1-3. [4]Waller G R, Nowacki E K. Alkaloid Biology and Metabolism in Plants[M]. Beijing: Science Press, 1984. [5]Hartmann T, Schoofs G, Wink M. A chloroplast-localized lysine decarboxylase ofLupinuspolyphyllus. FEBS Letters, 1980, 115: 35-38. [6]Wink M, Hartmann T, Witte L. Enzymatic synthesis of quinolizidine alkaloids inLupinchloroplasts. Zeitschrift Fur Naturforschung, 1980, 35: 93-97.[7]Bunsupa S, Katayama K, Ikeura E,etal. Lysine decarboxylase catalyzes the first step of quinolizidine alkaloid biosynthesis and coevolved with alkaloid production in leguminosae. The Plant Cell Online, 2012, 24(3): 1202-1216. [8]Bunsupa S, Okada T, Saito K,etal. An acyltransferase-like gene obtained by differential gene expression profiles of quinolizidine alkaloid-producing and nonproducing cultivars ofLupinusangustifolius. Plant Biotechnology, 2011, 28(1): 89-94. [9]Du C, Li J, Tang Y T,etal. Cloning, prokaryotic expression and characterization of lysine decarboxylase gene fromHuperziaserrata. Chinese Journal of Biotechnology, 2014, 30(8): 1299-1307. [10]Huang L X, Luo J, Li W C,etal. Comparison study on effects of different methods extracted fromSophoraalopecuroidesDNA. Ningxia Journal of Agriculture and Forestry Science and Technology, 2009, (3): 8-9. [11]Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution, 1987, 4(4): 406-425. [12]Gasteiger E, Hoogland C, Gattiker A,etal. Protein identification and analysis tools on the ExPASy Server[M]// The Proteomics Protocols Handbook. New York: Humana Press, 2005: 571-607. [13]Möller S, Croning M D R, Apweiler R. Evaluation of methods for the prediction of membrane spanning regions. Bioinformatics, 2001, 17(7): 646-653. [14]Sapay N, Guermeur Y, Deléage G. Prediction of amphipathic in-plane membrane anchors in monotopic proteins using a SVM classifier. BMC Bioinformatics, 2006, 7(1): 255. [15]Nakai K, Horton P. PSORT: a program for detecting sorting signals in proteins and predicting their subcellular localization. Trends in Biochemical Sciences, 1999, 24(1): 34-35. [16]Yachdav G, Kloppmann E, Kajan L,etal. Predict protein—an open resource for online prediction of protein structural and functional features. Nucleic Acids Research, 2014, doi: 10.1093/nar/gku366. [17]Petersen T N, Brunak S, von Heijne G,etal. SignalP 4.0: discriminating signal peptides from transmembrane regions. Nature Methods, 2011, 8(10): 785-786. [18]Biasini M, Bienert S, Waterhouse A,etal. SWISS-MODEL: modelling protein tertiary and quaternary structure using evolutionary information. Nucleic Acids Research, 2014, doi: 10.1093/nar/gku340. [19]Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod. Methods, 2001, 25(4): 402-408. [20]Yang Y, Tian L, Liu P,etal. The relationship between gene expression of lysine decarboxylase and matrine, oxymatrine accumulation inSophoraalopecuroides. Chinese Pharmaceutical Journal, 2015, 50(10): 846-849. [21]Ruan B J, Dai P, Wang W,etal. Progress on post-translational modification of proteins. Chinese Journal of Cell Biology, 2014, 36(7): 1027-1037. [22]Wink M, Hartmann T. Localization of the enzymes of quinolizidine alkaloid biosynthesis in leaf chloroplasts ofLupinuspolyphyllus. Plant Physiology, 1982, 70(1): 74-77. [23]Schneider G, Käck H, Lindqvist Y. The manifold of vitamin B6dependent enzymes. Structure, 2000, 8(1): R1-R6. [24]Chen C Y, Ma H L, Dong W K. Cloning and expression analysis of a leucoanthocyanidin reductase (LAR) gene fromOnobrychisviciifoliacv. Gansu. Acta Prataculturae Sinica, 2015, 24(6): 177-187. [25]Bunsupa S, Yamazaki M, Saito K. Quinolizidine alkaloid biosynthesis: recent advances and future prospects. Frontiers in Plant Science, 2012, 3(4): 279-286. [26]Walter L.Plant Eco Physiology[M]. Beijing: China Agriculture University Press, 1997: 246. [27]Tang Z H, Yu J H, Yang F,etal. Metabolic biology of plant alkaloids. Chinese Bulletin of Botany, 2003, 20(6): 696-702. [28]Christiansen J L, Jornsgard B, Buskov S,etal. Effect of drought stress on content and composition of seed alkaloids in narrow-leafed lupin,LupinusangustijdiusL. European Journal of Agronomy, 1997, 7(4): 307-314. [29]Canel C, Lopes-Cardoso M I, Whitmer S,etal. Effects of over-expression of strictosidine synthase and tryptophan decarboxylase on alkaloid production by cell cultures ofCatharanthusroseus. Planta, 1998, 205(3): 414-419. [1]中国科学院中国植物志编辑委员会. 中国植物志(第四十卷)[M]. 北京: 科学出版社, 1998: 80-81. [2]李爱华, 孙兆军. 苦豆子资源开发现状及前景初探. 宁夏大学学报: 自然科学版, 2000, 21(4): 354-356. [3]周福生, 穆青. 野生植物苦豆子的化学成分和主要药理作用. 中国野生植物资源, 2006, 25(4): 1-3. [4]沃勒 G R, 诺瓦茨基 E E. 生物碱的生物学及其在植物中的代谢作用[M]. 朱太平, 佟绍华, 马忠武, 译. 北京: 科学出版社, 1984. [9]杜次, 李菁, 唐云涛, 等. 蛇足石杉LDC的克隆、原核表达及其功能分析. 生物工程学报, 2014, 30(8): 1299-1307. [10]黄丽霞, 罗静, 李文超, 等. 不同方法从苦豆子中提取DNA的效果比较研究. 宁夏农林科技, 2009, (3): 8-9. [20]杨毅, 田蕾, 刘萍, 等. 苦豆子LDC表达与苦参碱和氧化苦参碱含量的关系. 中国药学杂志, 2015, 50(10): 846-849. [21]阮班军, 代鹏, 王伟, 等. 蛋白质翻译后修饰研究进展. 中国细胞生物学学报, 2014, 36(7): 1027-1037. [24]陈春艳, 马晖玲, 董文科.甘肃红豆草无色花青素还原酶LAR基因的克隆和表达分析. 草业学报, 2015, 24(6): 177-187. [26]Walter L.植物生态生理学[M]. 北京: 中国农业大学出版社, 1997: 246. [27]唐中华, 于景华, 杨逢, 等. 植物生物碱代谢生物学研究进展. 植物学通报, 2003, 20(6): 696-702. Cloning and expression analysis of a lysine decarboxylase gene inSophoraalopecuroides YANG Yi, LU Shan-Shan, LIU Ping*, TIAN Lei CollegeofAgronomy,NingxiaUniversity,Yinchuan750021,China In the biochemical metabolic processes ofSophoraalopecuroides, a lysine decarboxylase (LDC) gene is one of the key enzyme genes involved in the process of Oxymatrine biosynthesis. In the present study, the full length of the LDC coding sequence inS.alopecuroideswas cloned using a pair of specific primers designed based on theLDCsequence ofSophoraflavescensand was named Sa-LDC(gene bank accession number: KM249871). Sa-LDCbelongs to the Type Ⅲ Pyridoxal 5-phosphate (PLP)-Dependent enzyme supergene family, is comprised of a 1368 bps open reading frame (ORF) without intron, and has 97% identity with theLDCofEchinosophorakoreensisandS.flavescensin GeneBank. Its nucleotide sequence encodes 455 amino acid residues whose putative protein had a relative molecular mass of 49.14 kD and the theoretical isoelectric point of 5.63 without signal peptide and transmembrane structure. Interestingly, the deduced amino acid sequence of Sa-LDChad the conserved amino acid residue (Phe340) in quinolizidine alkaloid producing plants. Therefore, theS.alopecuroidesand other quinolizidine alkaloid producing plants were placed into a single group in the phylogenetic tree. In addition, the real time fluorescence quantitative PCR (qPCR) and high performance liquid chromatography (HPLC) results showed that both theLDCexpression level and oxymatrine content were influenced by polyethylene glycol (PEG) stress, and thatLDCexpression and oxymatrine accumulation inS.alopecuroideswere correlated. Sophoraalopecuroides; lysine decarboxylase gene; gene clone; gene expression; oxymatrine 10.11686/cyxb2015477http://cyxb.lzu.edu.cn 杨毅, 陆姗姗, 刘萍, 田蕾. 苦豆子赖氨酸脱羧酶基因克隆与表达分析. 草业学报, 2016, 25(8): 128-135. YANG Yi, LU Shan-Shan, LIU Ping, TIAN Lei. Cloning and expression analysis of a lysine decarboxylase gene inSophoraalopecuroides. Acta Prataculturae Sinica, 2016, 25(8): 128-135. 2015-10-12;改回日期:2016-01-04 宁夏回族自治区自然科学基金(NZ14033)资助。 杨毅(1989-),男,四川内江人,硕士。E-mail:kismet.young@163.com Corresponding author. E-mail: liupnxdx@126.com4 结论
