PMA-PCR方法快速检测VBNC状态青枯菌
2016-09-14于小龙许景升胡晓梅王学利
于小龙, 徐 进, 张 昊, 许景升,黄 雯, 胡晓梅, 冯 洁*, 王学利
(1. 天津农学院园艺园林学院, 天津 300384; 2. 中国农业科学院植物保护研究所,植物病虫害生物学国家重点实验室, 北京 100193)

PMA-PCR方法快速检测VBNC状态青枯菌
于小龙1,2,徐进2,张昊2,许景升2,黄雯2,胡晓梅2,冯洁2*,王学利1*
(1. 天津农学院园艺园林学院, 天津300384; 2. 中国农业科学院植物保护研究所,植物病虫害生物学国家重点实验室, 北京100193)
青枯菌为应对逆境胁迫,可进入活的但非可培养状态(viablebutnon-culturable,VBNC)。本文利用叠氮溴化丙锭(PMA)与PCR技术相结合,建立了一种快速有效区分青枯菌死活细胞的分子检测方法。基于hrcS基因序列,设计了一对青枯菌种特异性检测引物hrcSf/hrcSr;利用PMA对青枯菌Po82菌株的细胞悬浮液样品进行预处理,随后进行常规PCR扩增。结果表明,当样品中PMA质量浓度为3μg/mL、曝光时间大于5min时,PMA可有效抑制死亡菌体细胞中的DNA扩增;且对可培养和VBNC状态细胞中的DNA扩增没有影响;本试验建立的PMA-PCR方法能有效对包括VBNC状态在内的青枯菌活菌进行检测,避免了假阳性与假阴性结果的产生。
青枯菌;叠氮溴化丙锭;活的非可培养状态
茄科雷尔氏菌[Ralstonia solanacearum (Smith, 1896)Yabuuchietal. 1996]简称青枯菌,引起植物细菌性青枯病。青枯菌可以侵染54个科,超过450种寄主植物,包括马铃薯、香蕉、番茄、烟草、豇豆、花生、腰果、木瓜、油橄榄和生姜等诸多重要粮食及经济作物[1]。在我国,青枯病自20世纪30年代首次于长江中下游地区被报道以来,目前已向南扩散至20°N的海南省、向北扩散至42°N河北坝上地区[2],成为我国农业生产上的重要限制因子。
作为研究寄主植物与病原细菌互作的模式系统之一,青枯菌毒力调控网络及致病分子机理等方面的研究已取得了长足的进展。但作为土壤习居菌,青枯菌于土壤及水体环境中生态流行学研究领域的工作则相对滞后。一种观点认为青枯菌利用植物残体降解后产生的芳香族化合物作为营养代谢底物,从而得以在土壤中长期存活[3];另一种观点则认为青枯菌,特别是3号小种,在土壤环境中可进入活的非可培养状态(viablebutnon-culturableVBNC),并于该状态下长期存活[4]。VBNC状态是细菌为应对极端温度、渗透压、寡营养和重金属等逆境胁迫而普遍采用的一种生存策略。1982年,VBNC状态细菌首次被报道。目前已知85种细菌存在VBNC状态,其中仅有5种植物相关细菌,分别为荧光假单胞菌(Pseudomonas fluorenscence)、地毯草黄单胞菌柑橘致病变种(Xanthomonas axonopodispv. citri)、大豆根瘤菌(Rhizobium leguminosarum)[5]、根癌农杆菌(Agrobacterium tumefaciens)和茄科雷尔氏菌(R.solanacearum)[6-8]。
由于进入VBNC状态的细菌无法采用常规平板画线技术培养,故而造成VBNC状态细菌的假阴性检测结果,致使以该状态存在的细菌成为隐性初侵染源,进而对公共卫生安全、粮食安全以及食品安全构成潜在的威胁。目前常用于VBNC状态细菌检测的方法分别基于显微光学、免疫学以及分子检测技术[8]。PMA-PCR和EMA-PCR是近年来基于常规PCR技术发展起来用于VBNC状态细菌的检测方法。这两种方法克服了常规PCR无法区分活体细菌与死亡菌体释放出的游离DNA的缺陷,可有效避免假阳性检测结果。
叠氮溴化丙锭(propidiummonoazide,PMA)与溴化乙锭单叠氮溴(ethidiummonoazidebromide,EMA)是光敏DNA染料,无法透过拥有完整结构的细胞膜,只能修饰细胞死亡后暴露出来的DNA,并与之形成稳定的共价氮碳键,从而阻断DNA分子的聚合酶链式反应,在强光照射下剩余的PMA和EMA与水分子反应生成没有活性的羟胺[9]。PMA与EMA较其他方法更快捷,并且能与PCR结合,达到快速检测的目的,但由于溴化乙锭单叠氮溴(EMA)具有细胞毒性[10]并且其检测上限为108cfu/mL[11],故本试验选用PMA为光敏DNA染料。
本研究以用常规方法难以检测的VBNC状态青枯菌为切入点,旨在建立VBNC状态青枯菌的快速分子检测技术体系,藉此为植物繁殖材料的带菌检测与防控策略的效果评价提供技术支持,并为进一步深入解析青枯菌生态流行学规律奠定基础。
1 材料与方法
1.1材料
供试菌株及培养方法:本研究使用的青枯菌菌株与环境菌株材料均来自本实验室,详见表1。

表1 特异性验证所用青枯菌菌株Table 1 Ralstonia solanacearum strains used in this study
参试菌株均采用NA平板于28℃条件下培养,培养基配方如下:葡萄糖10g、多聚蛋白胨5g、牛肉膏3g、酵母提取物0.5g、琼脂18g,调pH至7.0。定容至1L,121℃灭菌20min。
试剂和耗材:叠氮溴化丙锭(propidiummonoazide)(BiotiumCat.No.40013)、 细菌基因组DNA提取试剂盒(BioTekeCat.No.DP2002)、MarkerI(BingDa)、PCR仪(AppliedBiosystems)、Advantage2PolymeraseMix(Cat.No.639201);其余试剂为国产分析纯。
1.2方法
1.2.1青枯菌死菌悬浮液的制备
分别挑取室温条件下保存于灭菌去离子水中的各供试菌株,于红四氮唑(triphenyltetrazoliumchloride,TZC)培养平板上画线,30℃条件下培养48h后,挑取典型的青枯菌野生型菌落,转至普通NA培养平板,30℃条件下培养48h。采用比浊法,挑取各纯培养物,分别于灭菌去离子水中配制成浓度为3×108cfu/mL的细菌悬浮液, 100℃水浴处理10min,获得青枯菌死菌细胞悬浮液。并于NA平板上画线,验证致死效果后备用。
1.2.2PCR引物设计
根据GenBank中登录的青枯菌Po82菌株的hrcS基因核苷酸序列(NC_017575.1 1330556-1330816),利用NCBI在线引物设计软件PrimerBLAST进行青枯菌种特异性引物设计,交由上海生工生物工程有限公司合成。
1.2.3PCR扩增条件
根据预试验,确立反应体系。25μL体系中包括PCRMix10μL,hrcSf(0.099nmol/μL)1μL,hrcSr(0.106nmol/μL) 1μL,样品1μL,ddH2O12μL。
PCR反应程序为:94℃预变性5min;94℃变性30s,59℃退火30s,72℃延伸30s,共30个循环;最后72℃延伸10min。
1.2.4PCR特异性试验
分别以52个青枯菌菌株和3个参照环境菌株DNA为扩增模板,用特异性引物hrcSf/hrcSr进行PCR扩增,扩增体系及程序同1.2.3,以灭菌去离子水为阴性对照,扩增产物用2.0%琼脂凝胶电泳检测,观察结果。
1.2.5引物扩增灵敏度
用核酸浓度测定仪(Gene公司,NanoVueplus)测定所提取的Po82基因组DNA浓度,并10倍梯度依次稀释为25ng/μL、2.5ng/μL、250pg/μL、25pg/μL、2.5pg/μL;按1.2.1方法制备浓度为3×108cfu/mL的Po82菌悬液,10倍梯度稀释至3×101cfu/mL。利用特异性引物hrcSf/hrcSr,按1.2.3的反应体系和程序分别对梯度稀释的Po82基因组DNA进行PCR扩增,2.0%琼脂糖凝胶电泳检测,观察结果。
1.2.6样品的PMA处理方法
PMA溶液于-20℃避光保存。取500μL制备好的菌悬液置于1.5mL离心管中,加入2μL浓度为 1mg/mL的PMA溶液,使PMA的终浓度为4μg/mL;PMA与菌悬液混合均匀后在室温条件下避光放置5min,利用500W的卤素灯曝光5min,光照交联时样品置于冰上(避免过热),且在距光源50cm处;交联后的悬浮液1 000g离心5min,所得沉淀溶于500μL无菌水中。
1.2.7青枯菌VBNC状态的诱导
高温处理:将1mL3×108cfu/mL的菌悬液分别以50、52、54、56、58、60、62、64和66℃处理30min后,分别取1μL涂布于NA平板,48h后查看菌落数。
确定青枯菌进入VBNC状态后,采用PMA-PCR进行检测,对照组为同浓度的青枯菌死菌和可培养状态的活菌。
2 结果与分析
2.1特异性引物设计
基于我们早期发表的Po82全基因组序列中hrcS基因,设计了12对引物,从中筛选获得了一对特异性引物(hrcSf/hrcSr)(表2),可扩增获得大小为246bp的目的片段。

表2 青枯菌hrcS基因PCR扩增引物序列Table 2 Primer sequences of Ralstonia solanacearum hrcS gene
2.2PCR条件的确立
在反应体系中分别加入7.5、10、12.5μLPCRMix,扩增条带无明显差异;不同退火温度:56、57、58、59、60、61℃所产生的扩增条带无明显差异;引物含量分别为0.6、0.8、1.0、1.2μL所得到的扩增条带也无明显差异(图1);由此拟定25μL反应体系组成为:PCRMix10μL、上下游引物各1μL、样品1μL、ddH2O12μL、退火温度59℃。

图1 引物hrcSf/hrcSr PCR条件验证结果Fig.1 PCR conditions validation with primers hrcSf/hrcSr
2.3PCR特异性与灵敏度验证
2.3.1特异性验证
以55个参试菌株的DNA为模板(表1),采用特异性引物hrcSf/hrcSr进行PCR扩增。结果显示,所有青枯菌DNA均能扩增出246bp左右的单一特异性条带,与预期结果一致。而甘露醇雷尔氏菌、肠杆菌和金黄杆菌等对照细菌则未产生扩增条带(图2)。
2.3.2灵敏度验证
以特异性引物hrcSf/hrcSr分别对不同浓度梯度的Po82基因组DNA与菌悬液进行扩增,设置无菌水为模板的阴性对照,结果显示:最低DNA和菌体悬浮液的检测浓度分别为25pg/μL和3×103cfu/mL(图3)。
2.4高温胁迫诱导青枯菌进入VBNC状态
如表3所示,菌悬液经52℃以上的高温处理30min后,按常规方法涂板培养,其菌落计数为0。表明处理后的青枯病菌进入了VBNC状态,将样品室温静置35d后,所有样品恢复可培养状态。

图2 引物hrcSf/hrcSr特异性验证结果Fig.2 Specificity of the primer set hrcSf/hrcSr for detection of Ralstonia solanacearum


图3 引物hrcSf/hrcSr灵敏度验证结果Fig.3 Sensitivity of the primer set hrcSf/hrcSr for detection of cell suspension and total DNA of Ralstonia solanacearum
2.5处于VBNC状态青枯菌的PMA-PCR检测
对进入VBNC状态的菌悬液进行PMA-PCR检测,结果显示(图4),经过PMA预处理的样品1~9产生特异性扩增条带, 而灭活组11则无特异性条带,表明PMA-PCR能检测处于VBNC状态的青枯病菌,并有效避免了常规平板计数法检测出现的假阴性结果。
2.6PMA-PCR 检测方法的假阳性验证
为验证PMA-PCR是否产生假阳性检测结果,分别采用常规平板计数、PCR与PMA-PCR对3组死菌样品进行检测。结果(表4)表明,常规PCR能以死菌DNA作为模板正常扩增,检测结果均为阳性;而PMA-PCR仅以活菌为检测靶标,未出现假阳性检测结果(图5),平板计数进一步验证了PMA-PCR的可靠性。

表3 不同温度处理后青枯菌Po82涂板计数结果Table 3 Colony number of bacterial suspensionsof Ralstonia solanacearum strain Po82 treated bydifferent temperatures

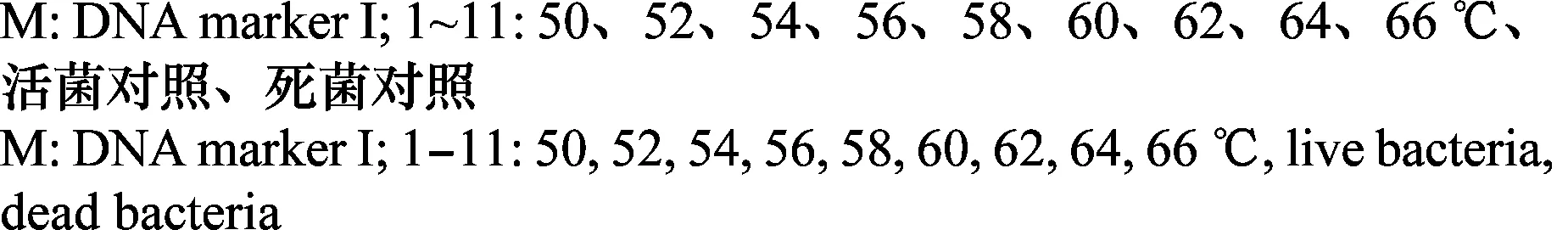
图4 不同温度处理样品PMA-PCR扩增结果Fig.4 PMA-PCR products of bacterial suspensions of Ralstonia solanacearum strain Po82 treated by different temperatures


图5死菌样品PCR与PMA-PCR扩增结果
Fig.5Comparison of PCR and PMA-PCR for detection of dilution series of dead Ralstonia solanacearum cells

表4 常规平板培养计数法、常规PCR法、PMA-PCR测定灭活青枯菌的比较Table 4 Comparison of plate counting, PCR andPMA-PCR for detection of dead Ralstonia solanacearum cells
3 讨论
VBNC状态是细菌特有的逆境生理生态响应机制,该状态下的细菌具有完整细胞结构、维持低水平呼吸与代谢速率、保持高ATP等级,且基因转录表达以及蛋白翻译合成均发生了相应改变,并可于适当条件下恢复可培养状态。VBNC状态的细菌与环境监测,食品加工和检测技术以及疾病控制等诸多领域均密切相关,且目前对细菌可培养与VBNC两者状态之间相互转换的分子调控机理尚不清楚,因此研究VBNC状态的细菌对于了解细菌的致病机理及生态适应性至关重要[12-14]。
Grey等2001年首次报道了采用铜制剂诱导青枯菌进入VBNC状态[8]。本研究尝试了采用高温胁迫诱导青枯菌2号小种Po82菌株快速进入VBNC状态,并使用常规平板培养与PMA-PCR结合验证其是否被成功诱导进入VBNC状态。初步试验结果显示52℃及以上温度可成功诱导Po82进入VBNC状态,68℃为青枯菌的致死温度。去除胁迫后,进入VBNC状态的Po82菌株可复苏至可培养状态。但随着胁迫温度的升高,复苏时间相应延长:52~56℃处理,室温下静置数小时后即可复苏;58~62℃处理,2d后可复苏;64~66℃,复苏时间则延长至35d。该结果与OliverandBockian等描述的通过简单温度变化可引起VBNC状态细菌复苏基本吻合[15-16],本研究结果同时提示我们应重新审视澳大利亚政府生物安全官方文件报道的青枯菌致死温度为52℃的结论,以及闷棚、生物熏蒸和50℃温汤浸种等高温物理防治措施能否达到预期的青枯病防治效果[17-19]。
本研究锚定与致病密切相关的hrp基因簇中保守基因hrcS作为青枯菌种水平检测靶标。hrp基因簇广泛存在于植物与动物病原细菌中,其编码产物参与组装形成Ⅲ型分泌系统,并通过该系统直接将Ⅲ型效应子注入寄主细胞,以抑制植(动)物的防御反应。与野油菜黄单胞菌(Xanthomonas campestris)等植物病原细菌不同,青枯菌的hrp基因簇是与祖先基因共同进化的核心基因组成员,而非通过基因水平转移获得的致病岛[20]。细菌自然进化过程中核心基因经受了更大的选择压力,因此在种水平上具有高度保守性,以此类基因作为检测靶标,比利用基因间隔区序列和未知功能片段更为可靠。
综上所述,本研究建立的PMA-PCR检测方法能从混合状态下的纯培养菌中特异性检测出1×103cfu/mL的青枯菌活菌,并能够有效区分死细胞和VBNC状态的菌体,为处于VBNC状态的青枯菌检测提供了更为准确、科学的检测方法,具有一定的实用价值和应用前景。
[1]SchellMA.ControlofvirulenceandpathogenicitygenesofRalstonia solanacearumbyanelaboratesensorynetwork[J].AnnualReviewofPhytopathology,2000,38: 263-292.
[2]徐进, 冯洁. 植物青枯菌遗传多样性及致病基因组学研究进展[J]. 中国农业科学, 2013, 46(14): 2902-2909.
[3]GeninS,BoucherC.LessonslearnedfromthegenomeanalysisofRalstonia solanacearum [J].AnnualReviewofPhytopathology, 2004, 42:107-134.
[4]MillingA,MengFanhong,DennyTP,etal.Interactionswithhostsatcooltemperatures,notcoldtolerance,explaintheuniqueepidemiologyofRalstonia solanacearumrace3biovar2 [J].TheAmericanPhytopathologicalSociety, 2009, 99(10):1127-1134.
[5]蒋娜, 李健强, 罗来鑫. 植物病原细菌的VBNC状态研究进展[J].植物病理学报, 2013, 43(3): 249-257.
[6]delCampoR,RussiP,MaraP,etal. Xanthomonas axonopodispv. citrienterstheVBNCstateaftercoppertreatmentandretainsitsvirulence[J].FEMSMicrobiologyLetters, 2009, 298(2): 143-148.
[7]AlexanderE,PhamD,SteckTR.TheviablebutnonculturableconditionisinducedbycopperinAgrobacterium tumefaciensandRhizobium leguminosarum [J].AppliedandEnvironmentalMicrobiology, 1999, 65(8): 3754-3756.
[8]GreyBE,SteckTR.TheviablebutnonculturablestateofRalstonia solanacearummaybeinvolvedinlong-termsurvivalandplantinfection[J].AppliedandEnvironmentalMicrobiology, 2001, 67(9): 3866-3872.
[9]潘乐, 孟庆峰, 钱爱东, 等.VBNC状态细菌检测方法的研究进展[J]. 中国农学通报, 2012, 28(5): 32-35.
[10]NockerA,CheungCY,CamperAK.ComparisonofpropidiummonoazidewithethidiummonoazidefordifferentiationoflivevsdeadbacteriabyselectiveremovalofDNAfromdeadcells[J].JournalofMicrobiologicalMethods, 2006, 67(2): 310-320.
[11]WangLuxin,MustaphaA.EMA-real-timePCRasareliablemethodfordetectionofviablesalmonellainchickenandeggs[J].JournalofFoodScience, 2010, 75(3):M134-M139.
[12]熊书, 殷幼平, 王芳, 等. 基于EMA-qPCR的茄科青枯菌活体检测技术的建立[J]. 微生物学通报, 2013, 40(9): 1723-1732.
[13]RamamurthyT,GhoshA,PazhaniGP,etal.Currentperspectivesonviablebutnonculturable(VBNC)pathogenicbacteria[J].FrontiersinPublicHealth, 2014, 2:103.
[14]TrevorsJT.Viablebutnonculturable(VBNC)bacteria:Geneexpressioninplanktonicandbiofilmcells[J].JournalofMicrobiologicalMethods, 2011, 86(2): 266-273.
[15]OliverJD.Theviablebutnonculturablestateinbacteria[J].TheJournalofMicrobiology, 2005, 43:93-100.
[16]OliverJD,BockianR.In vivoresuscitation,andvirulencetowardsmice,ofviablebutnonculturablecellsofVibrio vulnificus[J].AppliedandEnvironmentMicrobiology,1995,61(7):2620-2623.
[17]BiosecurityAustralia.Reviseddraftimportriskanalysisreportfortheimportationofcavendishbananasfromthephilippines,partC[R].Canberra:BiosecurityAustralia, 2007.
[18]ZanónMJ,FontMI,JordáC.UseoftomatocropresiduesintosoilforcontrolofbacterialwiltcausedbyRalstonia solanacearum [J].CropProtection, 2011, 30(9):1138-1143.
[19]ParetML.Managementofbacterialwiltofginger(Zingiber officinaleR.)causedbyRalstonia solanacearumwithplantessentialoils[D].Hawaii:UniversityofHawaii, 2009.
[20]AlfanoJR,CollmerA.ThetypeⅢ (Hrp)secretionpathwayofplantpathogenicbacteria:traffickingharpins,Avrproteins,anddeath[J].JournalofBacteriology,1997,179(18):5655-5662.
(责任编辑:杨明丽)
Rapiddetectionofviablebutnon-culturable(VBNC) Ralstonia solanacearumbyPMA-PCRmethod
YuXiaolong1,2,XuJin2,ZhangHao2,XuJingsheng2,HuangWen2,HuXiaomei2,FengJie2,WangXueli1
(1.CollegeofHorticultureandLandscape,TianjinAgriculturalUniversity,Tianjin300384,China;2.InstituteofPlantProtection,ChineseAcademyofAgriculturalSciences,StateKeyLaboratoryforBiologyofPlantDiseasesandInsectPests,Beijing100193,China)
Ralstonia solanacearumcanenterintotheVBNCstate(viablebutnon-culturable)inresponsetoadverseenvironmentalconditions.Anovelmethodtodifferentiateviable/deadcellsofR.solanacearumwasestablishedbyusingaDNAdyeofpropidiummonoazide(PMA)incombinationwiththepolymerasechainreaction(PMA-PCR).OnthebasisofhrcSgenesequence,aspecies-specificprimersethrcSf/hrcSrwasdesignedfordetectionofR.solanacearum.Sampleswerepre-treatedwithPMAwhichboundDNAfromdeadcellssothatonlyviablecellscanbeamplifiedbyPCR.Afinalconcentrationof3μg/mLPMAand5-minexposuretimewasfoundtocompletelyinhibitPCRamplificationfromDNAofdeadcells,andhadnoinhibitioneffectonviableandviablebutnon-culturable(VBNC)cells.ThePMA-PCRmethodestablishedinthisworkcandetectviableandviablebutnon-culturable(VBNC)cellsandavoidfalsepositiveandfalsenegationresultofR.solanacerum.
Ralstonia solanacerum;propidummonoazide;viablebutnon-culturable
2014-12-31
2015-02-09
公益性行业(农业)科研专项(201303015); 国家自然科学基金(31272008,31371908); 天津市农业科技成果转化与推广项目(201002250)
E-mail:wxlzxp@sohu.com;jfeng@ippcaas.cn
S436.411
ADOI:10.3969/j.issn.0529-1542.2016.01.026
