定量PCR检测HLA-C等位基因表达方法的建立和初步应用①
2016-08-29王文君吴士超缪凤琴张建琼
王文君 陆 森 吴士超 缪凤琴 潘 宁 张建琼
(东南大学医学院,南京210009)
·免疫学技术与方法·
定量PCR检测HLA-C等位基因表达方法的建立和初步应用①
王文君陆森②吴士超缪凤琴潘宁张建琼
(东南大学医学院,南京210009)
①本文受国家自然基金青年科学基金项目(81201601)资助。
②南京医科大学第一附属医院普外科肝脏外科,南京210029。
目的:建立实时定量PCR方法检测同一个体两条不同的HLA-C等位基因的表达水平。方法:构建HLAⅠ类等位基因外显子2和3数据库,设计等位基因特异性系列引物,建立等位基因定量PCR方法;在数据库和835例汉族人外周血标本中对方法进行理论和实验验证;检测并比较20对肝癌和癌旁组织标本中HLA-C等位基因的表达水平。结果:本实验设计的20对引物能检测23种杂合基因型中不同的HLA-C等位基因,覆盖汉族人群中出现频率大于0.96%的个体;55%(11/20)的肝癌组织中HLA-C位点两条等位基因表达水平变化不一致。结论:成功建立了针对汉族人群的HLA-C等位基因表达水平的检测方法;肝癌组织中普遍存在着两条HLA-C等位基因表达水平变化不一致的现象。
定量PCR;HLA-C;等位基因特异性;肝癌
经典HLA(Human leucocyte antigen,HLA)Ⅰ类分子在免疫学上的主要功能是通过与相应受体结合调节CD8+T细胞、NK细胞等效应细胞的活化,大量研究表明经典HLAⅠ类分子表达水平异常是肿瘤细胞、病毒感染细胞的常见免疫逃逸方式,显著影响着肿瘤免疫、感染免疫以及自身免疫等疾病进程[1,2]。
经典HLAⅠ类基因包括HLA-A、-B、-C三个基因座位,在人群中具有高度多态性,绝大多数个体的这三个位点为杂合子。经典HLAⅠ类基因呈共显性表达,即来自父母的两种等位基因产物均表达于有核细胞表面。因此,除了少数纯合个体外,大部分个体的HLA-A、-B、-C位点在细胞表面表达两种不同的等位基因。然而,以往研究在检测经典HLAⅠ类分子表达水平时,通常对一个位点的两种不同等位基因产物并不加以区分,即检测到的是同一基因座位两种等位基因叠加的表达水平。
现有研究已明确,不同等位基因编码的HLA Ⅰ类分子在调节免疫效应细胞功能中常常发挥不同作用。比如,某些等位基因的产物和NK细胞上的受体分子结合,参与调节NK细胞的活化[3];特定的等位基因产物结合特定抗原肽,与TCR结合,在特异性T细胞的活化中发挥关键作用[4]。而且,针对白血病细胞的研究发现,不同等位基因编码的HLAⅠ类分子表达水平变化确实不同[5]。因此,区分检测同一基因座位不同等位基因的表达水平,对于理解机体在生理和病理条件下的免疫调节机制非常必要。
由于HLAⅠ类等位基因间具有高度多态性和相似性,目前尚无足够的抗体可以区分各等位基因所编码的蛋白,因此蛋白水平的检测无法覆盖绝大多数个体。实时定量PCR方法敏感性高,且依赖引物序列特异性检测目标基因。现有的实时定量PCR方法仅能区分HLA-A、-B、-C不同基因座位的mRNA表达水平,不能区分同一基因座位两条等位基因的mRNA表达水平[6]。因此,需要建立新的实时定量PCR方法,用于检测绝大多数个体的HLA等位基因mRNA表达水平。
经典HLAⅠ类等位基因数高达9 000多个,主要集中在外显子2和3区域,具有高度多态性和相似性,为保证仅扩增两条等位基因中的一条,需要设计高度特异性的系列引物对大多数个体进行等位基因mRNA表达水平的定量分析。本实验室已经成功建立了HLA-A、-B等位基因mRNA表达水平的定量PCR方法,本实验旨在建立检测HLA-C等位基因mRNA表达水平的定量PCR方法,为后续经典HLAⅠ类等位基因mRNA表达水平的研究提供实验基础。
1 材料与方法
1.1材料
1.1.1血标本DNA实验室现存835例中国汉族人群外周血标本DNA,所有标本HLA-C位点经过PCR-SBT(聚合酶链反应-测序分型方法)高分辨率分型[7-10]。
1.1.2肝癌标本cDNA来自实验室收取新鲜肝癌及癌旁组织,采用PCR-SBT方法进行HLA-C位点高分辨率分型[7],随机选取20例HLA-C位点杂合的组织。Trizol法提取标本总RNA,DNA酶处理去除基因组DNA,加入Oligo(dT),M-MLV,dNTP,RNAase抑制剂,DEPC水,总共20 μl逆转录反应体系,获得CDNA保存于-20℃。
1.1.3主要仪器试剂和引物设计软件Real-time qPCR仪 (ABI Stepone Plus,Applied Biosystems,美国); Beacon Designer 8.02定量PCR引物设计软件;SYBR Green PCR Master Mix (ABI, 美国;Vazyme,中国);HiScriptTMQ RT SuperMix for qPCR(Vazyme,中国)。
1.2方法
1.2.1构建经典HLAⅠ类等位基因位点特异性碱基数据库IMGT/HLA数据库收录了经WHO命名委员会确认的全部HLA 基因数据,以IMGT/HLA(Release3.19.0)数据库为基础,构建包含HLA-A,-B,-C位点所有等位基因的位点特异性碱基数据库。
1.2.2引物设计根据构建的数据库寻找特异性位点,作为引物3′末端碱基,设计等位基因特异性引物(一般16~24 bp),然后使用Beacon DesignerTM8.02定量PCR引物设计软件,设计出另外一条对应引物,参照软件的评分选择最佳引物。
1.2.3方法建立和验证通过比对NCBI/Primer-Blast数据库和HLA数据库中的核酸序列,在理论上验证所设计引物的特异性。以本实验室HLA-C已分型的汉族人群为目标样本,优化退火温度及引物浓度,确定最适PCR反应条件;选取适宜阴性对照从实验上验证方法特异性。目标标本扩增曲线典型,溶解曲线为单峰,阴性参照未出现明显扩增曲线,则视为该引物验证成功。采用系列稀释法检测扩增效率。
1.2.4实时定量PCR反应体系及条件本实验采用SYBR Green染料法进行Real-time PCR,反应体系20 μl,包括:2×SYBR® Green PCR Master Mix(Vazyme)10 μl,10 μmol/L上下游引物200 nmol/L,cDNA模板1 μl,加双蒸水补足20 μl。PCR反应条件:95℃ 10 min;95℃ 15 s,退火(各引物最适退火温度)15 s,72℃30 s,40个循环。
1.2.5肝癌及癌旁组织定量PCR内参引物选择在检测肝癌及癌旁标本时,选用TBP和HPRT-1作为内参基因,取二者Ct值的几何平均值作为内参基因的Ct值,内参引物序列如表1所示。
1.2.6肝癌及癌旁组织定量PCR结果的数据处理方法检测肝癌及癌旁HLA-C等位基因cDNA水平时,每对等位基因特异性的引物和内参引物定量PCR扩增时均作3个复孔,结果取其平均值。采用相对定量的方法分析数据,即2-△△Ct法。其中:△△Ct=△Ct肝癌-△Ct癌旁,△Ct=Ct目的基因-Ct内参基因。

表1 内参基因引物序列及产物长度Tab.1 Sequences and product length of reference gene primers

图1 经典HLAⅠ等位基因位点特异性碱基数据库Fig.1 Classical HLA class Ⅰ alleles database
2 结果
2.1构建经典HLAⅠ类等位基因位点特异性碱基数据库以IMGT/HLA(Release3.19.0)数据库为基础,将经典HLAⅠ类基因的外显子2和3的基因序列(共546 bp),从1到546进行排列,每一个位点可能出现1~4个不同的碱基;列出每个位点上不同碱基对应的所有等位基因以及对应的所有等位基因亚型及数目(如图1),用于后续引物设计过程中查找和确定特异性的位点。
2.2设计等位基因特异性引物根据构建的经典HLAⅠ类等位基因位点特异性碱基数据库,查找具有多态性的碱基位点,以该位点作为引物3′末端碱基。引物特异性碱基筛选遵循以下标准:①理论上这一位点碱基对应的所有等位基因包括同一血清型的所有亚型,图1中是外显子2的第216位点对应的经典HLAⅠ类等位基因,灰色阴影标注区域,216位碱基是G,以该位点作为特异性碱基位点,可扩增C*01、C*04、C*06和C*07,并不混杂其他HLA-C等位基因及HLA-A,-B等位基因(其他位点208位碱基为A);②上游引物位于外显子2区域,下游引物位于外显子3区域,跨内含子2从而排除基因组DNA的影响,产物长度在80~240 bp之间;③设计的引物GC含量在30%~70%之间,无发夹二级结构,引物长度保持在16~24 bp之间,且上下游引物Tm值相差不超过3℃,以保证引物扩增效率;④设计的引物满足通用SYBR Green染料法的要求。

表2 等位基因特异性验证方法举例Tab.2 An example of how to verify allelic specificity
根据选定的特异性3′末端碱基设计一条引物,使用Beacon DesignerTM8.02定量PCR引物设计软件,设计出另外一条引物。根据以上标准及软件评分选择最佳引物。最终设计完成20对符合以上原则的HLA-C等位基因定量PCR引物。
2.3定量PCR方法的验证
2.3.1数据库验证引物特异性本实验所构建的基于HLA-A、-B、-C等位基因数据库已经避开三个位点之间的非特异扩增,但仍需检验引物与其他基因的互补程度。①通过NCBI/Primer-Blast数据库验证,检验是否能扩增出其他非HLA基因。②非经典HLAⅠ类基因与经典基因相似度亦非常高,通过IMGT/HLA专业数据库,验证所得引物是否能够扩增HLA-E、-F、-G等非经典等位基因。20对引物均在以上两个数据库完成验证,未发现与以上非目的基因匹配。
2.3.2实验验证验证方法的特异性和扩增效率。
2.3.2.1验证方法的等位基因特异性在已知HLA-C等位基因的835例汉族人群标本中按照出现频率从高到低依次选取样本,作为目标样本。根据等位基因序列选择适宜引物,逐一扩增每个目标样本的两条等位基因,同时验证每条等位基因扩增的特异性。表2以其中一份标本为例(C*01:02:01G;C*08:01:01G)说明验证过程:序列分析显示引物对C-07仅能扩增C*01:02:01G,而不能扩增C*08:01:01G,因此选取引物对C-07扩增C*01:02:01G。阴性对照选择一条等位基因为C*08:01:01G,且另一条等位基因亦不能被C-07扩增的标本(C*07:02:01G;C*08:01:01G)。
采用C-07同时扩增目标标本和阴性对照(图2),扩增曲线和产物溶解曲线均显示,目标标本得到扩增,阴性对照未有扩增,证明该反应仅扩增目标标本中的C*01:02:01G,而未扩增其另一条等位基因C*08:01:01G。
采用以上流程,同样方法扩增目标标本另一等位基因C*08:01:01G,并验证其特异性。选取20对引物的不同组合,能够扩增出汉族人群中23种基因型的每条等位基因,且相应阴性参照均无扩增。
2.3.2.2检测扩增效率将目标标本cDNA系列稀释,实时定量PCR扩增目标基因并计算扩增效率。本研究所有体系扩增效率在90%~110%之间。
2.4引物序列及应用范围本实验最终采用20对HLA-C等位基因定量PCR引物(表3、4),能够特异性扩增835例汉族人群中HLA-C基因型频率大于0.96%个体的所有等位基因(共23种基因型)。表3列出了按照基因型频率从高到低排列的汉族人群中23种基因型,以及扩增每条等位基因所选取的引物对及其退火温度。
2.5肝癌及癌旁组织中HLA-C等位基因mRNA水平检测选取20对HLA-C位点为杂合子的肝癌及癌旁组织cDNA,根据其等位基因选取适宜的等位基因特异性定量PCR引物,检测并比较HLA-C等位基因的mRNA表达水平(表5)。
实时定量PCR结果显示,所设计等位基因特异性定量PCR引物能特异性区分不同HLA-C等位基因。在20例肝癌组织中,与癌旁组织相比,肝癌组织中HLA-C等位基因上调表达17.50%,表达一致47.50%,下调表达35.0%;同时发现,在HLA-C位点杂合的个体中,55.0%个体存在着两条等位基因表达水平变化不一致的现象。
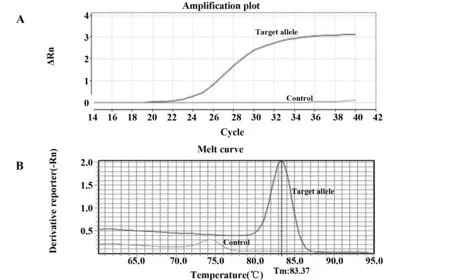
图2 目标标本和阴性对照的扩增结果Fig.2 Amplification of target sample and negative control Note: The amplification plot(A) and melt curve (B) of target sample and negative control.

表3 HLA-C等位基因特异性定量PCR引物可区分汉族人群基因型Tab.3 Allelic primers distinguishing two HLA-C alleles in Han population

表4 HLA-C等位基因特异性定量PCR引物序列Tab.4 Sequences of HLA-C allelic primers

表5 肝癌相比癌旁组织HLA-C等位基因mRNA表达变化Tab.5 mRNA expression level of HLA-C alleles in HCC tissue
3 讨论
本实验设计的HLA-C引物能区分汉族人群常见等位基因,对于低频等位基因或杂合子中两条等位基因不能用以上引物区分,可以通过相似流程对个别等位基因进行单独引物设计(如等位基因C*06:53,C*14:03,C*15:04)。本文建立的方法能区分HLA-C等位基因mRNA水平的表达,但是其局限在于无法反映转录后加工过程。
以往对于白血病细胞的研究发现,同一白血病细胞表面的不同等位基因编码的HLAⅠ类分子,其表达水平变化并不相同:其规律是作为NK细胞抑制性配体的某些HLA等位基因产物在表面表达水平正常,而其他非抑制性HLA等位基因产物则存在明显下调,可能反映了白血病细胞在NK细胞和T细胞双重免疫监视压力下精细的逃逸方式[5]。在肝癌中,本实验室以往研究发现,HLA及其配体基因多态性影响了肝癌的发生[7,10],且其表面HLAⅠ类分子也存在着明显的异常表达,但从未有研究对其等位基因表达水平进行检测。本次对20例新鲜肝癌和癌旁组织的检测结果发现,同一例肝癌组织HLA-C位点的两条等位基因表达水平变化存在明显差异,一方面表明在肝癌中区分检测同一基因座位不同等位基因表达水平非常必要,另一方面也为我们进一步研究肝癌细胞精细的免疫逃逸机制奠定了实验基础。
本实验建立了针对汉族人群的HLA-C等位基因特异性定量PCR检测方法,设计、验证等位基因定量PCR引物并明确引物实际应用范围,为经典HLAⅠ类基因mRNA表达水平的研究建立实验方法和提供了技术基础,在HLA表达相关的病毒免疫、肿瘤免疫、自身免疫等领域研究中具有应用价值。
[1]Angell TE,Lechner MG,Jang JK,etal.MHC class I loss is a frequent mechanism of immune escape in papillary thyroid cancer that is reversed by interferon and selumetinib treatment in vitro [J].Clin Cancer Res,2014,20(23):6034-6044.
[2]Klitz W,Gragert L,Trachtenberg E.Spectrum of HLA associations:the case of medically refractory pediatric acute lymphoblastic leukemia [J].Immunogenetics,2012,64:409-419.
[3]Jodie P.Goodridge,Aura Burian,etal.HLA-F and MHC class I open conformers are ligands for NK cell Ig-like receptors [J].J Immunol,2013,191:3553-3562.
[4]Acevedo-Sáenz L,Carmona-Pérez L,Velilla-Hernández PA,etal.The APPEESFRS peptide,restricted by the HLA-B*35:01 molecule,and the APPEESFRF variant derived from an autologous HIV-1 strain induces polyfunctional responses in CD8+T cells [J].Biores Open Access,2015,4(1):115-120.
[5]Demanet C,Mulder A,Deneys V,etal.Down-regulation of HLA-A and HLA-Bw6,but not HLA-Bw4,allospecificities in leukemic cells:an escape mechanism from CTL and NK attack?[J].Blood,2004,103:3122-3130.
[6]Hong HA,Loubser AS,de Assis Rosa D,etal.Killer-cell immunoglobulin-like receptor genotyping and HLA killer-cell immunoglobulin-like receptor-ligand identification by real-time polymerase chain reaction [J].Tissue Antigens,2011,78(3):185-194.
[7]Ning Pan,Wei Jiang,Hang Sun,etal.KIR and HLA Loci are associated with hepatocellular carcinoma development in patients with hepatitis B virus infection:a case-control study [J].PLoS One,2011,6(10):e25682.
[8]Ning Pan,Jie Qiu,Hang Sun,etal.Combination of human leukocyte antigen and killer cell immunoglobulin-like receptor genetic background influences the onset age of hepatocellular carcinoma in male patients with hepatitis B virus infection [J].Clin Developmental Immunol,2013,4(3):6-354.
[9]Miao F,Sun H,Pan N,etal.Association of human leukocyte antigen class I polymorphism with spontaneous clearance of hepatitis B surface antigen in Qidong Han population [J].Clin Dev Immunol,2013,2013:145725.
[10]Ning Pan,Keping Chen,Jie Qiu,etal.Human leukocyte antigen class I alleles and haplotypes associated with primary hepatocellular carcinoma in persistent HBV-infected patients [J].Human Immunol,2013,74:758-763.
[收稿2015-12-31修回2016-04-01]
(编辑倪鹏)
A novel real-time quantitative PCR method for analyzing HLA-C allele expression level
WANG Wen-Jun,LU Sen,WU Shi-Chao,MIAO Feng-Qin,PAN Ning,ZHANG Jian-Qiong.Southeast University Medical School,Nanjing 210009,China
Objective:To establish real-time qPCR method to analyze each HLA-C allele expression level of individual.Methods: Database including exon 2 & 3 sequences of HLA class Ⅰ alleles was built,HLA-C allelic specific primers were designed and the real-time quantitative PCR method for analyzing HLA-C allele expression level was built.The allelic specificity of these primers were confirmed in database and 835 normal peripheral blood samples of Han population.The mRNA level of each HLA-C allele from 20 pairs of liver tumor tissues and non-tumor tissues was analyzed by the qPCR method we built.Results: 20 pairs of allelic specific primers were designed to distinguish the two HLA-C alleles of each individual with frequency over 0.96% in 835 cases of Han population.Among 55% of the liver cancer tissues,the expression levels of the two HLA-C alleles from the same tumor tissue changes differently compared to that of the relevant non-tumor tissue.Conclusion: This study provides method for HLA-C allele expression level analysis of Han population and each HLA-C allele expression level is inconsistent of liver cancer tissue.
Real time qPCR;HLA-C;Allelic specific;Liver cancer
10.3969/j.issn.1000-484X.2016.08.018
R392.33文献标志码A
1000-484X(2016)08-1165-06
王文君(1989年-),女,硕士,主要从事肿瘤免疫学方面研究,E-mail:wwj2832@163.com。
及指导教师:潘宁(1976年-),女,博士,讲师,主要从事肿瘤免疫学方面研究,E-mail:panningmicro@aliyun.com。
