贵州省两株山羊源羊种布鲁菌MLST分析
2016-08-25李沛丽李世军周敬祝唐光鹏王定明周碧君
李沛丽,李世军,刘 英,王 月,周敬祝,唐光鹏,王定明,周碧君
贵州省两株山羊源羊种布鲁菌MLST分析
李沛丽1,李世军2,刘英2,王月2,周敬祝2,唐光鹏2,王定明2,周碧君1
目的了解贵州省2010年分离自山羊的两株羊种布鲁菌的等位基因序列型(Sequence type, ST)及遗传学特征,为动物间和人间布病疫情的预防和控制提供科学依据。方法应用布鲁菌属特异性PCR(BCSP31-PCR)和种/型特异性PCR(AMOS-PCR)对2010年分离自山羊的两株布鲁菌进行鉴定,采用多位点序列分型(Multiple locus sequence typing, MLST)技术对两株布鲁菌菌株的7个管家基因、1个外膜蛋白基因及1个基因间区的序列进行测定,将各个基因的序列与参考菌株的等位基因型进行比对,确定其等位基因谱及序列型(STs),分析与两株菌ST型与各种型布鲁菌代表菌株的遗传进化关系。结果来自贵州省山羊的两株布鲁菌株经BCSP31-PCR鉴定为布鲁菌属细菌,AMOS-PCR将其鉴定为羊种布鲁菌。MLST分析显示,两株菌的9个等位基因序列型为ST8型,与同属羊种布鲁菌的ST7、ST9~ST12、ST34和ST35遗传关系最近。结论贵州省2010年分离的2株布鲁菌分离株均为ST8型布鲁菌,与同属羊种布鲁菌的ST型遗传关系最近,结果为贵州省人间和动物间布病疫情的预防和控制提供了科学依据。
山羊;布鲁菌; PCR;MLST;遗传特征
Supported by the grant from the Science and Technology Projects for Social Development in Guizhou Province (No. Guizhou science co-word SY[2013]3049), the Project of Special Fund for the Cultivation of Outstanding Youth Talents of Science and Technology in Guizhou Province (No. Guizhou Science Renhezi[2015]09), and the Found for the Research Team of Natural Foci and Vector-borne Infectious Disease Control and Prevention (No. RCJD1401)
布鲁菌病(Brucellosis)(以下简称布病)是由布鲁菌属(Brucella)的细菌侵入机体引起世界性、多宿主感染的人兽共患病。本病历史悠久,分布广泛,于1860年发现于地中海的马耳他岛,故又称为马耳他热、地中海热[1]。早在20世纪50年代,布鲁菌即为美军成功研发的第一个细菌战武器,因此也是生物反恐的一个重要潜在对象[2]。布病危害严重,家畜罹患布病,怀孕母畜常流产,公畜发生睾丸炎和附睾炎引起不育,病畜分泌物(如乳汁等)及脏器带菌,并发生关节炎、淋巴结炎和滑液囊炎等引起痛疼,严重影响家畜的生产力和动物性产品的生物安全,给畜牧业生产带来巨大损失[3]。对人而言,布病并不同于一般的疾病,布鲁菌侵入机体引起变态反应性全身病变,急性期可以治愈,但不易被确诊,常被拖延为慢性期,慢性期布病患者可有肝、脾及睾丸肿大,关节和脊柱强直,肌腱挛缩变硬等,很难治愈,患脑炎和心肌炎可致命,严重影响人类健康[4]。
贵州省于2010年首次从患病动物(山羊)分离出羊种布鲁菌,从病原学角度证实了布病在贵州省动物的存在[5],随后人间布病疫情在贵州省迅速扩散,散发和暴发疫情时有发生,防控形势非常严峻[6-8]。了解动物布病病原特点和流行特征是动物间和人间布病防控的基础,本项研究采用基于布鲁菌属外膜蛋白31基因的PCR(BCSP31-PCR)和基于插入序列IS711的牛、羊、绵羊附睾和猪种布鲁菌特异的PCR(AMOS-PCR)技术对2010年分离自山羊的两株布鲁菌进行属和种(型)鉴定,采用多位点序列分型(Multiple locus sequence typing, MLST)技术对2株布鲁菌菌株的7个管家基因、1个外膜蛋白基因及1个基因间区的序列进行测定[9-11],将各个等位基因的序列与参考菌株的等位基因型进行比对,确定其等位基因谱型及菌株序列型(STs),分析其与布鲁菌不同ST型的遗传进化关系,为贵州省动物间和人间布病的防控提供科学依据。
1 材料与方法
1.1实验试剂布鲁菌培养基采用进口布鲁菌肉汤(BD公司,货号为211088)和布鲁菌琼脂(BD公司,货号为211086)成品培养基按照说明书配制。PCR引物由大连宝生物科技有限公司合成。PCR相关试剂购买于大连宝生物公司。
1.2实验菌株实验菌株为分离自贵州省2010年黔南市龙里县动物(山羊)血液并经传统方法鉴定为羊种生物3型布鲁菌菌株(命名为LL3和LL4)。阳性对照菌株为布鲁菌疫苗株A19(牛种)、M5(羊种)和S2(猪种)(购自兰州生物制品研究所)。
1.3DNA提取挑取布鲁菌可疑菌落接种于布鲁菌琼脂平板,培养48 h后采用水煮法提取细菌核酸。
1.4BCSP31-PCR法鉴定布鲁菌属采用布鲁菌属特异性基因BCSP31作为定属基因,按照参考文献[12-13]提供的B4和B5引物序列和参数进行布鲁菌属的鉴定。
1.5AMOS-PCR法鉴定布鲁菌种/型采用参考文献[12-13]提供的以布鲁菌属IS711插入序列为基础建立的AMOS-PCR引物及参数进行布鲁菌种/型的鉴定。
1.6MLST分析采用参考文献[9-11]提供的布鲁菌7个管家基因(dnaK、gyrB、trpE、aroA、cobQ、gap、glk)、1个外膜蛋白基因(omp25)和1个基因间区int-hyp作为MLST的靶标基因,选取的靶标基因、引物、序列及扩增长度见表1。PCR反应条件为:95 ℃预变性5 min;94 ℃变性30 s,63 ℃退火30 s,72 ℃延伸l min,30个循环;72 ℃延伸10 min。PCR产物采用1%琼脂糖凝胶电泳检测。PCR产物委托北京天一辉生物科技公司进行双向测序。采用DNAStar软件包分析整理和编辑序列,使序列长度范围与文献中给出的序列一致。将测定序列分别与相应基因的等位基因型的序列进行比较,明确序列是否与某个等位基因型的序列一致,如果一致,则定义为某个等位基因型,否则定义为一个新的等位基因型。利用MLST在线工具对序列的等位基因型、进化关系等进行分析,确定分离菌株的ST型及与文献[8-10]报道的36个ST型菌株(见表2)间的进化关系。
2 结 果
2.1BCSP31-PCR鉴定BCSP31-PCR方法对分离株和对照菌株核酸进行扩增,产物采用1.5%琼脂糖电泳进行检测,结果显示两株分离菌株及阳性对照菌株核酸均扩增出布鲁菌属特异的223 bp的条带,而阴性对照无条带出现。
2.2AMSO-PCR检测结果AMOS-PCR方法对分离株和对照菌株核酸进行扩增,产物采用1.5%琼脂糖电泳进行检测,结果显示两株分离菌株及阳性对照菌株核酸均扩增出羊种布鲁菌特异的731 bp的条带,而阴性对照无条带出现。
表1用于扩增和测序分析的9对引物序列Tab.1Primer sequences used for the amplification and sequencing of nine genetic loci
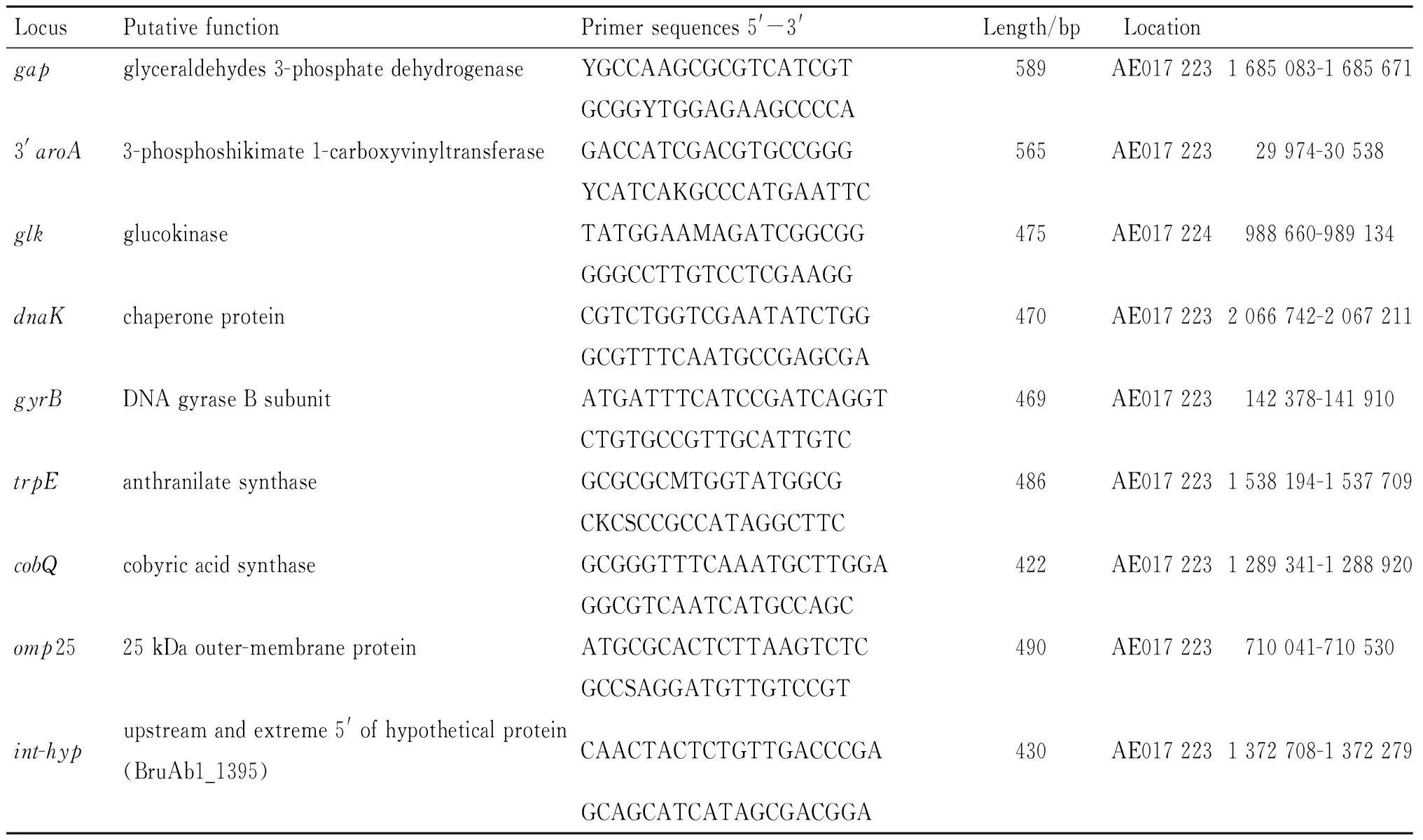
LocusPutativefunctionPrimersequences5'-3'Length/bpLocationgapglyceraldehydes3-phosphatedehydrogenaseYGCCAAGCGCGTCATCGT589AE0172231685083-1685671GCGGYTGGAGAAGCCCCA3'aroA3-phosphoshikimate1-carboxyvinyltransferaseGACCATCGACGTGCCGGG565AE01722329974-30538YCATCAKGCCCATGAATTCglkglucokinaseTATGGAAMAGATCGGCGG475AE017224988660-989134GGGCCTTGTCCTCGAAGGdnaKchaperoneproteinCGTCTGGTCGAATATCTGG470AE0172232066742-2067211GCGTTTCAATGCCGAGCGAgyrBDNAgyraseBsubunitATGATTTCATCCGATCAGGT469AE017223142378-141910CTGTGCCGTTGCATTGTCtrpEanthranilatesynthaseGCGCGCMTGGTATGGCG486AE0172231538194-1537709CKCSCCGCCATAGGCTTCcobQcobyricacidsynthaseGCGGGTTTCAAATGCTTGGA422AE0172231289341-1288920GGCGTCAATCATGCCAGComp2525kDaouter-membraneproteinATGCGCACTCTTAAGTCTC490AE017223710041-710530GCCSAGGATGTTGTCCGTint-hypupstreamandextreme5'ofhypotheticalprotein(BruAb1_1395)CAACTACTCTGTTGACCCGA430AE0172231372708-1372279GCAGCATCATAGCGACGGA
2.3MLST分析结果MLST分析显示,两株布鲁菌dnaK、gyrB、trpE、aroA、cobQ、gap、glk、omp25和int-hyp位点的等位基因序列号分别为3、2、3、2、1、5、3、8和2,聚类分析显示,两株菌株等位基因位点序列型别与ST8型相同(表2和图1)。Split tree分析显示,至今已发现的36个ST型所涵盖的布鲁菌6个种呈现出相同种的ST型菌株聚类相对较近,本研究株的两株分离株与羊种布鲁菌ST7、ST9、ST10、ST11、ST12、ST34和ST35遗传关系最近,见图2。
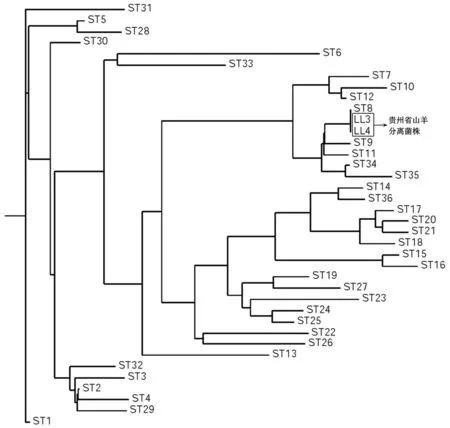
图1 贵州省两株山羊来源羊种布鲁菌与不同种型布鲁菌ST型聚类分析图Fig.1 Clustering tree of ST of two strains of B. melitensis isolated from goats and different species of Brucella
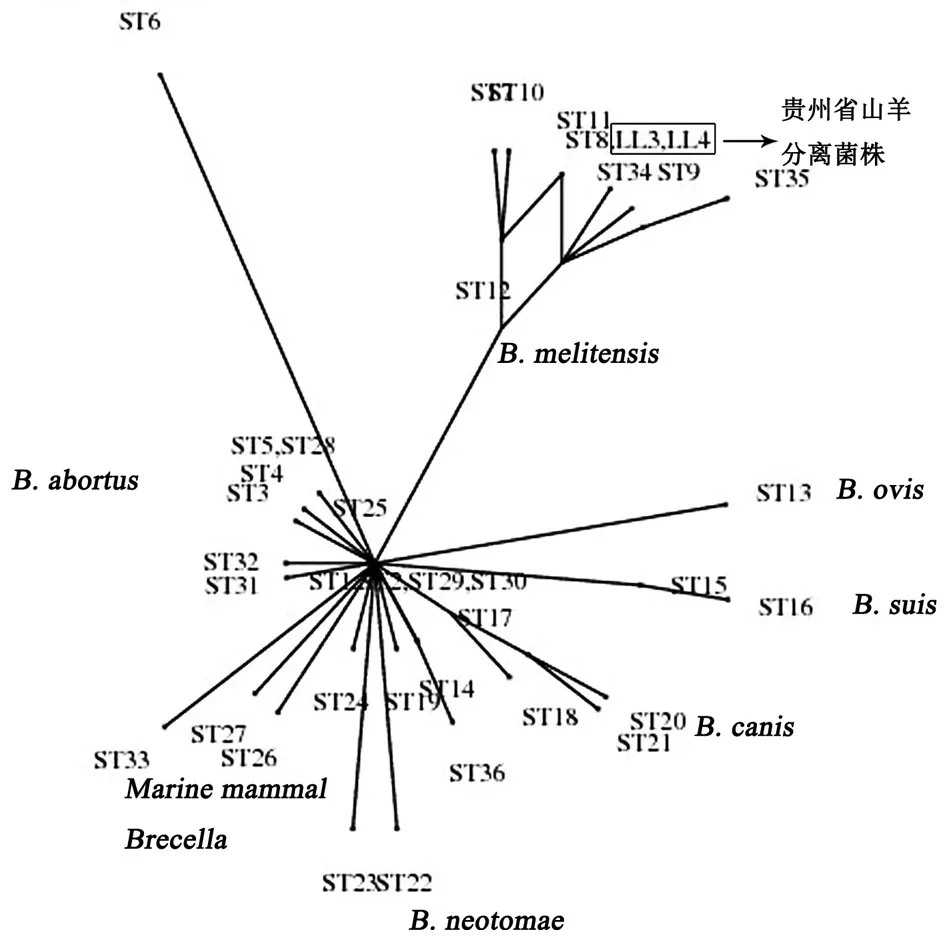
图2 贵州省两株山羊来源羊种布鲁菌与不同种型布鲁菌ST型Spilt TreeFig.2 Split tree of ST of two strains of B. melitensis isolated from goats and different species of Brucella
表2布鲁菌各等位基因谱
Tab.2Allele profiles of Brucella
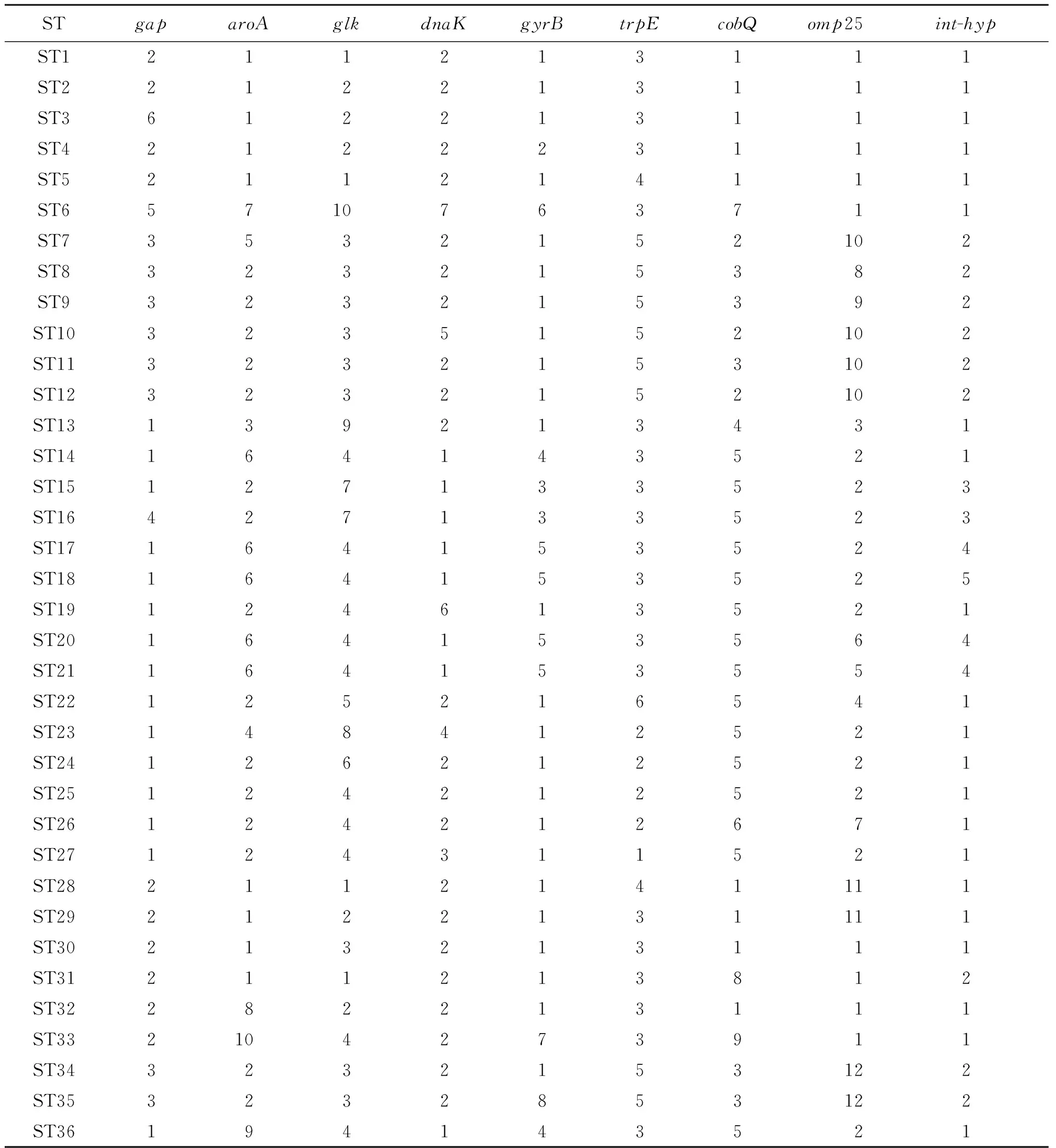
STgaparoAglkdnaKgyrBtrpEcobQomp25int-hypST1211213111ST2212213111ST3612213111ST4212223111ST5211214111ST65710763711ST73532152102ST8323215382ST9323215392ST103235152102ST113232153102ST123232152102ST13139213431ST14164143521ST15127133523ST16427133523ST17164153524ST18164153525ST19124613521ST20164153564ST21164153554ST22125216541ST23148412521ST24126212521ST25124212521ST26124212671ST27124311521ST282112141111ST292122131111ST30213213111ST31211213812ST32282213111ST332104273911ST343232153122ST353232853122ST36194143521
3 讨 论
传统的分类方法将布鲁菌分为羊、牛、猪、犬、绵羊附睾和沙林鼠6个生物种19个生物型,即牛种布鲁菌B.abortus8个型;猪种布鲁菌B.suis5个型;羊种布鲁菌B.melitensis3个型;犬种布鲁菌B.canis、绵羊附睾种布鲁菌B.ovis和沙林鼠种布鲁菌B.neotomae各1型。因其生物种、型和菌株的不同,致病力各异,对人致病力最强的为羊种菌,其次为猪种菌,牛种菌对人致病力弱。动物布病主要由羊种、牛种和猪种菌感染羊、牛和猪等动物呈急性或慢性经过[4]。因此,了解布病病原学特征对布病的有效预防和控制至关重要。贵州省分别于2010年首次从患病动物(羊)和病人分离出羊种布鲁菌[5-6],此后,布病疫情在贵州省迅速扩散,散发和暴发疫情时有发生[7-8]。本研究将来源于贵州省山羊的两株菌株采用属特异性BCSP31-PCR和AMOS-PCR进行鉴定,结果显示本次分离的可疑细菌为羊种布鲁菌。
传统血清学方法鉴定只能将布鲁菌鉴定到种(型),无法对菌株进行溯源分析,一些低分辨率的分子分型方法如PCR产物限制性片段长度低多态性(PCR-RFIP)、随机扩增多态性DNA(RAPD-PCR)和扩增片断长度多态性分析(AFLP)等在分子流行病学中应用价值十分局限。由于布鲁菌属种间DNA同源性高达90%,需要高分辨力的分型方法用来提供疾病传播模式和分子流行病学信息,随着分子生物学的发展,分子分型技术被广泛应用于传染病病原菌的溯源。布鲁菌分子分型方法如脉冲场凝胶电泳(pulsed-field gel electrophoresis, PFGE)[13]、多位点可变数目串联重复序列分析(multilocus variable-Humber tandem repeat analysis, MLVA-16)技术[14]应运而生和MLST技术[10]在细菌性传染病的病原鉴定、传染源追踪与病原体的溯源等方面发挥了重要作用。然而,PFGE实验过程中活菌操作繁多,实验室感染风险较大,与MLVA-16技术相比,MLST方法更能反映菌株的遗传特征,在实验过程的可操作性及实验结果的可靠性之间取得了平衡,在不同的实验室之间具有良好的可比性[10]。Adrian等[9]2006年采用MLST技术对来自不同国家的160株不同种型布鲁菌分为27个ST型,Chen YF等[11]对来自中国的60株布鲁菌进行了MLST分析,结果在Adrian等发现的27个ST型的基础上增加了9个新的ST型,分别命名为ST28~ST36。因此,本研究中我们采用布鲁菌MLST分子分型方法对2010年分离自贵州省山羊的菌株进行分子分型分析并与已发现的ST型进行比对,结果两株菌株均为ST8型,聚类分析结果显示(图1),本次分离菌株与同属羊种布鲁菌几个ST型聚类处于同一分支,其中与ST8型聚类最近,从遗传学水平证实了两株布鲁菌为羊种布鲁菌,这与全国布鲁菌病原体种型分布一致[15-16],同时与Chen YF以及周晓艳[10-11]等发现的中国菌株以ST8为优势序列型的结果一致,提示ST8(表2)序列型可能为贵州省动物间和人间布病病原体的优势序列型。流行病学调查资料显示,本研究中的患者均为养羊户,所饲养的羊多数为我省因畜牧业发展需求而从外地引进羊或是来历不明的山羊,本研究分析证实两株布鲁菌序列型与我国其他地区的具有流行优势的ST8型一致,提示我省动物间布鲁菌感染为输入性传播。
为了进一步了解分离株的遗传学特征,我们利用Splits tree工具,对ST型的数据进行分裂分解分析(Split decomposition analysis),从图2中可以看出,ST8与其它几个同属于羊种布鲁菌的ST型(ST7~ST11、ST34和ST35)相距较近,它们均由ST12分离而来。与其它种的布鲁菌相比,羊种菌的这几个ST型是聚集较近,说明羊种布鲁菌在基因水平上是高度保守的。
贵州省近年正值畜牧业大力发展时期,羊、牛等种畜大多从国内布病高发的区域引进,在引种的同时有可能导致布病传染源输入贵州省引发畜间或人间布病流行。2010年首次从山羊分离出羊种布鲁菌,从病原学角度证实了布病在畜间存在[2],随后的2011年相继在患者血液分离出羊种布鲁菌[6],人间布病疫情迅速扩散,疫情中多数患者均为养羊户或与羊具有接触史,且患者分离流行菌株均为羊种布鲁菌,本研究结果显示2株山羊来源菌株为羊种布鲁菌的ST8型,提示ST8型菌株很有可能是引起人间布病疫情的病原菌,然而,人间布病流行菌株序列型是否同为ST8型尚需进一步研究。此外,本研究分析的动物来源菌株数量受限,结果是否具有代表性尚需进一步研究。本研究对贵州省2010年分离自山羊的的两株布鲁菌采用PCR方法和MLST技术进行分析,揭示了菌株序列型为ST8,结果为贵州省布病的防控提供了科学依据,在保护人群健康和保障畜牧业发展方面具有重要意义。
[1] Shang DQ. Brucellosis raging again and the reasons[J]. Chin J Ctrl Endem Dis, 2001, 16(1): 29-34. DOI:10.3969/j.issn.1001-1889.2001.02.016.(in Chinese)
尚德秋. 布鲁氏菌病再度肆虐及其原因[J]. 中国地方病防治杂志,2001, 16(1): 29-34.
[2] Pappas G, Panagopoulou P, Christou L, et al.Brucellaas a biological weapon [J]. Cell Mol Life Sci, 2006, 63(19/20): 2229-2236.DOI:10.1007/s00018-006-6311-4.
[3] Seleem MN, Boyle SM, Sriranganathan N. Brucellosis: a re-emerging zoonosis[J]. Vet Microbiol, 2010, 140(3/4): 392-398.DOI:10.1016/j.vetmic.2009.06.021.
[4] Franco MP, Mulder M, Gilman RH, et al. Human brucellosis[J]. Lancet Infect Dis, 2007, 7(12): 775-786. DOI: 10.1016/S1473-3099(07)70286-4.
[5] Li SJ, Wang Y, Chen H, et al. Isolation and identification ofBrucellamelitensisfirstly isolated from goat in Guizhou province[J]. Chin J Zoonoses, 2011, 27(6): 515-518. (in Chinese)
李世军,王月,陈红,等. 贵州省首次从山羊分离到布鲁氏菌及其种型鉴定[J]. 中国人兽共患病学报,2011, 27(6): 515-518.
[6] Li SJ, Wang Y, WANG DM, et al. Etiologic diagnosis and analysis of the first case of human brucellosis in Guizhou Province[J]. Chin J Endem Dis, 2012, 31 (5): 69-71. DOI:10.3760/cma.j.issn.1000-4955.2013.06.013.(in Chinese)
李世军,王月,王定明,等.贵州省首例人间布鲁氏菌病病例的病原学诊断与分析[J]. 中国地方病学杂志,2012, 31 (5) : 69-71.
[7] Liu Y, Wang Y, Ma Q, et al. Etiologic identification and analysis of the human brucellosis in Guizhou Province, China, 2013[J]. Chin J Zoonoses, 2015, 31(1): 45-48. DOI:10.3969/j.issn.1002-2694.2015.01010.(in Chinese)
刘英,王月,马青,等.贵州省2013年人间布鲁氏菌病病原体种例鉴定与分析[J].中国人兽共患病学报,2015,31(1):45-48.
[8] Li SJ, Wang Y, Wang DM, et al. Identification and genetic characteristics on the bacteria isolate from a case of human brucellosis in Guizhou province[J]. Chin J Epidemiol, 2013, 34(7): 717-720. DOI:10.3760/cma.j.issn.0254-6450.2013.07.013.(in Chinese)
李世军,王月,王定明,等. 贵州省一起人间布鲁氏菌病疫情的菌株鉴定及遗传特征研究[J].中华流行病学杂志,2013, 34(7): 717-720.
[9] Whatmore AM, Perrett LL, MacMillan AP. Characterisation of the genetic diversity ofBrucellaby multilocus sequencing[J]. BMC Microbiol, 2007, 20(7): 34.DOI: 10.1186/1471-2180-7-34.
[10] Zhou HY, Chen YF, Cui BY, et al. Multiple sequence typing analysis ofBrucellamelitensisserotype 3 strains[J]. Chin J Zoonoses, 2011, 27(5): 371-375. DOI:10.3969/j.issn.1002-2694.2011.05.002.(in Chinese).
周海艳,陈燕芬,崔步云,等. 我国羊种3型布鲁氏菌的多位点序列分型研究[J].中国人兽共患病学报, 2011, 27(5):371-375.
[11] Chen Y, Ke Y, Wang Y, et al. Changes of predominant species/biovars and sequence types ofBrucellaisolates, Inner Mongolia, China[J]. BMC Infect Dis, 2013, 13: 514. DOI: 10.1186/1471-2334-13-514.
[12] Jiang H, Cui BY, Zhao HY, et al. Use of AMOS-PCR assay for the species identification ofBrucella[J]. Chin J Zoonoses, 2009, 25(2): 107-109. DOI:10.3969/j.issn.1002-2694.2009.02.004.(in Chinese)
姜海,崔步云,赵鸿雁,等. AMOS-PCR对布鲁氏菌种型鉴定的应用[J]. 中国人兽共患病学报,2009, 25(2): 107-109.
[13] Cespedes S, Salgado P, Valenzuela P, et al. Characterization of genomic island 3 and genetic variability of Chilean field strains ofBrucellaabortus[J]. J Clin Microbiol, 2011, 49(7): 2461-2469.DOI:10.3969/j.issn.1002-2694.2009.07.006.
[14] Her M, Kang SI, Cho DH, et al. Application and evaluation of the MLVA typing assay for theBrucellaabortusstrains isolated in Korea[J]. BMC Microbiol, 2009, 9: 230.
[15] Deng YQ, Wang JX, Lin DH, et al. Molecular identification of theBrucellastrains isolated in Fujian province[J]. Chin J Zoonoses, 2009, 25(7): 636-639.DOI:10.3969/j.issn.1002-2694.2015.01.11 (in Chinese)
邓艳琴,王加熊,林代华,等. 福建省布鲁氏菌分离株的分子生物学鉴定[J]. 中国人兽共患病学报,2009, 25(7): 636-639.
[16] Cui BY.Brucellaepidemic situation and vaccine research in China[J]. Chin J Epidemiol, 2012, 31(4): 355-356. (in Chinese)
崔步云.关注中国布鲁杆菌病疫情发展和疫苗研究[J].中国地方病学杂志,2012, 31(4): 355-356.
Multiple locus sequence typing of two strains ofBrucellamelitensisisolated from goats in Guizhou Province
LI Pei-li1, LI Shi-jun2, LIU Ying2,WANG Yue2, ZHOU Jing-zhu2,TANG Guang-peng2,WANG Ding-ming2,ZHOU Bi-jun1
(1.CollegeofAnimalScience,GuizhouUniversity,Guiyang550025,China;2.GuizhouProvincialCenterforDiseaseControlandPrevention,Guiyang550004,China)
In order to understand the allele locus gene sequence type (ST) and the genetic characteristic of twoBrucellamelitensisstrains isolated from goats in Guizhou Province in 2010 and provide scientific basis for the control and prevention of animal and human brucellosis, the two isolates were identified by usingBrucellagenus specific BCSP31-PCR and species-specific AMOS-PCR, and the seven house-keeping genes and one out membrane protein gene and one gene -inter- region sequence of the strains were further analyzed using multiple locus sequence typing (MLST). Each gene sequence was compared with the reference strains to determine the allelic profile and sequence type, and the ST data-based genetic relationship of the two isolates andBrucellastrains representing different species were analyzed. The results showed that the twoBrucellastrains were identified asB.melitensiswith BCSP31-PCR and AMOS-PCR. MLST analysis revealed that the 9 alleles based sequence type of the two strains was ST8, which was genetically closed to the STs (ST7, ST8-ST12, ST34 and ST35) belonging toB.melitensis. The results indicate that the sequence type of the two strains ofB.melitensisisolated from goats in Guizhou Province in 2010 is ST8, close to the STs belonging toB.melitensis.
goats;Brucellamelitensis; PCR; MLST; genetic characteristic
Zhou Bi-jun, Email: as.bjzhou@gzu.edu.cn;Li Shi-jun,Email:zjumedjum@163.com
周碧君,Email:as.bjzhou@gzu.edu.cn;李世军,Email:zjumedjun@163.com
1.贵州大学动物科学院,贵阳550025;2.贵州省疾病预防控制中心,贵阳550004
R378.5
A
1002-2694(2016)06-0547-06
2015-07-28;
2015-11-19
DOI:10.3969/j.issn.1002-2694.2016.06.008
贵州省科技公关计划社会发展项目(No.黔科合SY[2013]3049)、贵州省优秀青年科技人才培养对象专项资金项目(黔科合人字[2015]09号)和贵州省传染病人才培养基地项目(黔人领发[2013]15)“自然疫源性和虫媒传染病”研究团队(RCJD1401)经费联合资助
