一起诺如病毒GⅡ.17型引起的急性胃肠炎病原学诊断及基因特征分析
2016-08-24陈静芳欧新华
姚 栋,陈静芳,叶 文,欧新华
一起诺如病毒GⅡ.17型引起的急性胃肠炎病原学诊断及基因特征分析
姚栋,陈静芳,叶文,欧新华
目的对长沙市2014年12月某厂区暴发的一起急性胃肠炎疫情进行病原学诊断及对其致病原进一步的基因分型研究。方法共采集 6例病人肛拭子标本和可疑水源标本4份,提取核酸后,诺如病毒GI/GII型real-time RT-PCR试剂盒检测;阳性标本普通RT-PCR扩增VP1基因片段,产物测序后BLAST比对确定其型别,并构建进化树分析其进化关系。结果10份标本real-time RT-PCR扩增结果显示为诺如病毒GII型,VP1区片段RT-PCR扩增后产物测序,BLAST比对发现与2014年香港诺如病毒株GII/Hu/HKG/2014/GII.17/CUHK-NS-463同源性最高,达99%,证实为诺如病毒GII.17型;构建系统进化树分析显示本次疫情分离的毒株与日本、香港、台湾等亚洲地区的毒株亲缘关系更为接近,而与美国、法国等地区的毒株亲缘关系则较远。结论诺如病毒是引起此次急性胃肠炎疫情的病原体,且引起暴发的病毒株属于GII.17型。
诺如病毒;胃肠炎;分子分型
诺如病毒(Norovirus, NoV),又称诺瓦克样病毒,属杯状病毒科诺如病毒属,是非细菌性急性胃肠炎的主要病原之一,被世界卫生组织定为B类病原,主要导致食源性及水源性急性腹泻。目前世界范围内超过50%的急性胃肠炎均是由NoV引起的,各个年龄段人群均普遍易感,在国内多个季节都可出现流行高峰[1-2]。NoV为单正链RNA病毒,基因组全长约7.7kb,包含3个开放阅读框(open reading frame, ORF)。ORF1编码非结构蛋白,包括RNA聚合酶,ORF2和ORF3分别编码主要(VP1)和次要(VP2)衣壳蛋白。根据VP1区基因序列的差异,NoV可分为GI-GVI 6个基因群,感染人类的主要为GI、GII群,其中GI群至少可分为14个基因型,GII群至少可分为21个基因型[3]。
研究显示由NoV引起的感染性腹泻中80%由GII群引起,而GII群中又以GII.4 型为主要基因型。自20世纪90年代诺如病毒首次导致世界性大流行以来,GII.4型一直在流行中占据主导地位[4],其他基因型如GI、GII.3等型别也有报道,但多为小范围流行或散发病例报道[5]。
2014年12月,长沙市暮云区一厂医院报告近2周多名村民陆续出现呕吐、腹胀腹泻等胃肠道症状的病例,调查显示该厂区及周边居民采用自备水源(280 m深地下井)集中供水,根据临床特点怀疑为水源性NoV引起的感染性腹泻,采集6名患者肛拭子标本,4户居民末梢水送实验室进行相关检测。
1 材料与方法
1.1标本采集6名患者肛拭子标本,Hank’s液中保存;250 mL无菌瓶分别在4户居民家中采集末梢水200 mL,运送至实验室进行检测。
1.2主要试剂病毒RNA提取试剂盒QIAamp viral mini kit购自QIAGEN公司;诺如病毒GI/GII型real-time RT-PCR检测试剂盒购自江苏硕世生物有限公司;SuperScript○RⅢ One-Step RT-PCR with Platinum○RTaq试剂盒购自Invitrogen公司。
1.3病毒核酸提取末梢水经抽滤浓缩后用于核酸提取,肛拭子标本振荡混匀后直接用于核酸提取,具体操作方法参照试剂盒说明书进行。
1.4Real-time RT-PCR检测取病毒RNA提取液5 μL用于检测,诺如病毒real-time RT-PCR试剂盒定性检测病毒GI/GII型,所用仪器为LC 480Ⅱ实时荧光PCR仪,检测条件及GI、GII型结果的判断参照试剂盒说明书进行。
1.5基因型别确定及序列分析参照相关文献方法[6]扩增病毒VP1基因部分序列,所用引物序列如下,mon381: 5′-CCAGAATGTACAATGGTTATGC-3′,mon383: 5′-CAAGAG ACTGTGAAGACATCATC-3′,RT-PCR反应条件为:55 ℃ 30 min,94 ℃ 5 min,1个循环;94 ℃ 30 s,55 ℃ 30 s,68 ℃ 30 s,35个循环,68 ℃ 10 min。扩增片段大小为323 bp,
PCR产物送上海英俊公司进行序列测定,测序结果采用BLAST程序比对鉴定其型别,并上传GenBank数据库,序列登录号为KR269694~ KR269703。选取GenBank中GII型诺如病毒VP1区序列,采用DNA Star软件进行序列同源性分析,MEGA软件通过邻位法(Neighbor-Joining method)进行系统发生学分析,建树的可靠性通过1000 bootstrap评估。
2 结 果
2.1Real-time RT-PCR核酸检测10份标本核酸经real-time RT-PCR检测,所有标本均出现典型“S”型扩增曲线,根据检测试剂盒说明书判断为诺如病毒GII型阳性。
2.2病毒型别的确定为确定病毒基因型别,采用特异性引物扩增病毒ORF2的VP1基因部分区域(图1),PCR产物测序后,与GenBank数据库BLAST 比对分析,发现与2014年香港NoV GII.17型毒株 GII/Hu/HKG/2014/GII.17/CUHK-NS-463同源性最高,达99%,证实导致本次疫情暴发的诺如病毒基因型别为GII.17型。10份标本核苷酸序列同源性在96%~100%间,其中7份标本碱基序列完全一致;氨基酸序列与上述香港地区GII.17型毒株VP1蛋白序列相比(序列号AKB94550),肛拭子标本Hu/14Y03/CS/CHN/2014第196、197位氨基酸发生变异,末梢水标本water/14Y03/CS/CHN、water/14Y04/CS/CHN第197位氨基酸发生变异(图2)。

图1 PCR扩增区域ORF结构示意图Fig.1 ORF structures of VP1 gene PCR amplification fragments used in this study

图2 NoV GII.17型VP1蛋白氨基酸序列比对图Fig.2 Amino acid sequences alignment of NoV GII.17 VP1 protein
2.3遗传进化树分析从GenBank中选取NoV GII.17型、GI群及GII群其他型别病毒VP1基因序列,构建系统进化树如图3。本次疫情检测到的10株GII.17型NoV在系统进化树中形成一独立的小分支,与2013年台湾、2014年香港及2013-2015年日本等地区分离到的毒株亲缘关系较近,而与美国、法国等国家则距离较远,可能由于空间的距离,以及分离时间间隔较远而导致系统进化上的差异。
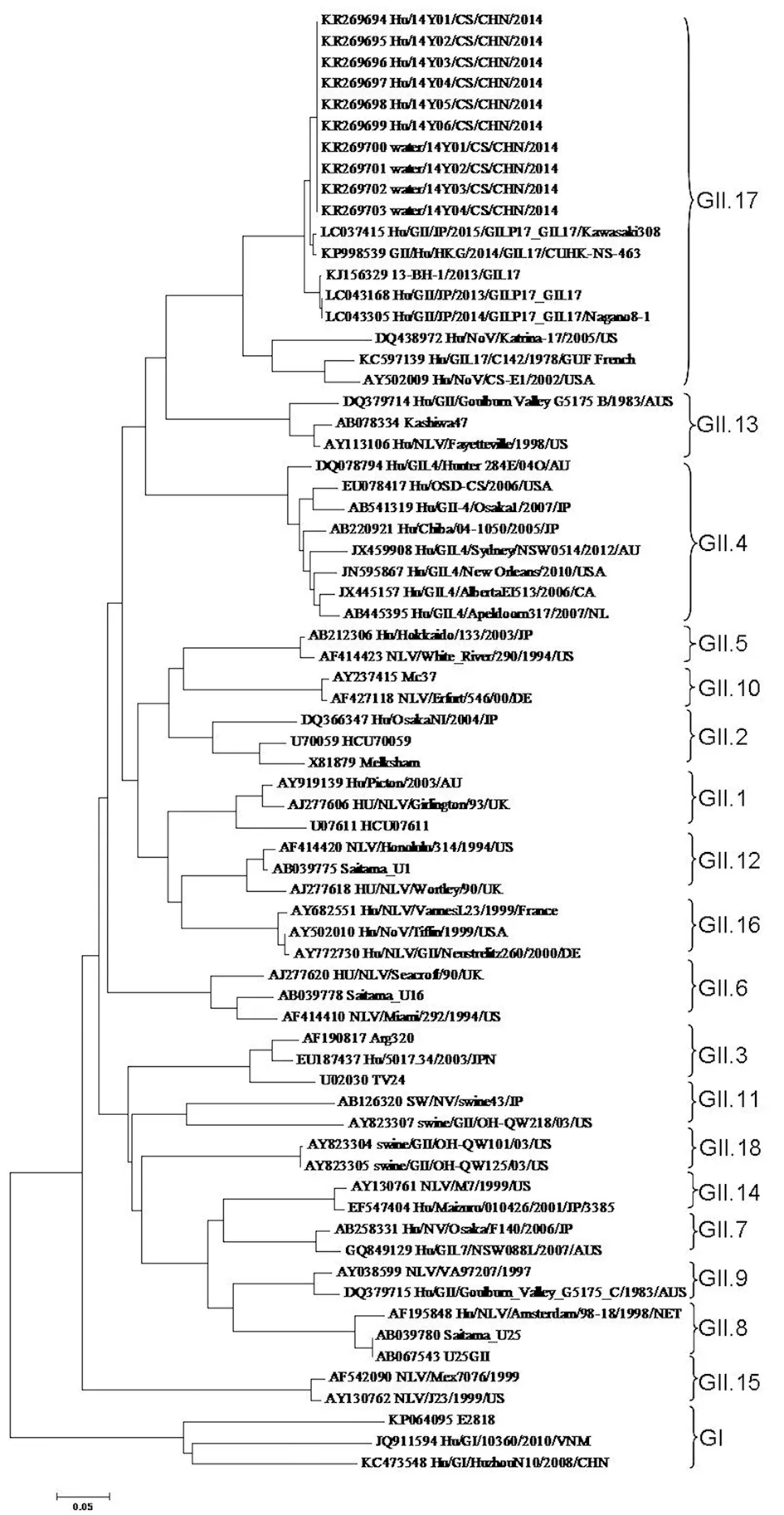
图3 NoV GII.17型VP1基因进化树Fig.3 Phylogenetic analysis based on partial VP1 gene sequences of GII.17 NoV
3 讨 论
NoV是一种变异频繁的RNA病毒,其中GII.4型的变异频率比其它基因型更高[7],导致其在全世
界范围内的广泛流行,甚至暴发流行,自1995年以来共监测到7种引起大范围暴发流行的GII.4型变异株;中国许多地区如北京、上海等地的报道也显示GII.4型在NoV引起的急性胃肠炎中占大部分比例[8-9],其他型别如GI型、GII.3、GII.14型等则流行程度较低,散发病例报道更常见[10],而GII.17型流行的报道则较少见。本次由NoV引起的急性胃肠炎暴发疫情经序列分析证实为GII.17型,搜索GenBank中提交的NoV GII.17型序列,我们发现从2012年开始,大多提交的GII.17型序列来自韩国、日本、香港、台湾、中国广东等亚洲地区国家,说明从2012年开始,NoV GII.17型在亚洲地区流行开始增强,应引起重视。根据部分VP1基因序列构建的遗传进化树(图3)显示本次疫情NoV GII.17型与香港、日本、台湾地区分离株亲缘关系接近,可能为同一遗传进化来源。
VP1蛋白是NoV的主要衣壳蛋白,主要与病毒结构的组装、与宿主间的相互作用以及免疫应答的产生等生物学功能有关[11];同时VP1蛋白也是病毒最易发生变异的区域,通过多序列比对我们发现污染的水样标本中病毒VP1蛋白本身存在个别氨基酸的差异,而从一患者体内检测到的病毒VP1蛋白则出现新的氨基酸突变位点(图2,第196位氨基酸),说明病毒在感染患者后,在与宿主相互作用过程中有新的突变产生。
NoV引起的肠胃炎可以通过人与人直接接触、通过接触污染的环境如食品、水等途径传播,NoV不同基因型对外界环境的抵抗力、与受体结合能力等生物学特性存在明显区别[12],导致不同基因型的流行病学特征不同,如GI型食源性传播更常见,而GII.4型人与人之间直接传播较为常见[13]。调查显示本次疫情是由于NoV GII.17型污染末梢水管网引起,通过对末梢水消毒后发病例数明显减少;有研究报道非洲肯尼亚河流中NoV分型显示GII.17型占检出阳性率的76%[14],说明NoV GII.17型可能通过水源性的传播风险较高,但具体流行病学特征还需大量的监测数据反映。
NoV在遗传上的多样性,主要是因为VP1蛋白的频繁变异以及不同基因型间ORF1和ORF2的重组现象,本实验中我们仅对NoV GII.17型VP1区部分序列进行了分析,ORF1区基因分型及全基因组序列还有待于进一步的研究。
[1] Patel MM, Widdowson MA, Glass RI, et al. Systematic literature review of role of noroviruses in sporadic gastroenteritis[J]. Emerg Infect Dis, 2008, 14(8): 1224-1231.
[2] Jin M, Sun JL, Chang ZR, et al. Outbreaks of noroviral gastroenteritis and their molecular characteristics in China, 2006-2007[J]. Chin J Epidemiol, 2010, 31(5): 549-553. (in Chinese)
靳淼, 孙军玲, 常昭瑞, 等. 中国2006-2007年诺如病毒胃肠炎暴发及其病原学特征分析[J]. 中华流行病学杂志, 2010, 31(5): 549-553.
[3] Kroneman A, Vennema H, Deforche K, et al. An automated genotyping tool for enteroviruses and noroviruses[J]. J Clin Virol, 2011, 51(2): 121-125.
[4] Siebenga JJ, Vennema H, Zheng DP, et al. Norovirus illness is a global problem: emergence and spread of norovirus GII.4 variants, 2001-2007[J]. J Infect Dis, 2009, 200(5): 802-812.
[5] Chen YZ, Chen XY. Epidemiological situation of Norovirus outbreaks[J]. Chin J Zoonoses, 2012, 28(4): 398-402. (in Chinese)
陈志永, 陈小岳. 诺如病毒暴发的流行病学研究进展[J]. 中国人兽共患病学报, 2012, 28(4): 398-402.
[6] Noel JS, Ando T, Leite JP, et al. Correlation of patient immune responses with genetically characterized small round-structured viruses involved in outbreaks of nonbacterial acute gastroenteritis in the United States, 1990 to 1995[J]. J Med Virol, 1997, 53(4): 372-383.
[7] Bull RA, Eden JS, Rawlinson WD, et al. Rapid evolution of pandemic noroviruses of the GII.4 lineage[J]. PLoS Pathog, 2010, 6(3): e1000831.
[8] Jia LP, Qian Y, Zhang Y, et al. Prevalence and genetic diversity of noroviruses in outpatient pediatric clinics in Beijing, China 2010-2012[J]. Infect Genet Evol, 2014, 28: 71-77.
[9] Shen Z, Wang G, Zai SB, et al. The emergence of novel GII.4 Norovirus variant, Sydney-2012, in Shanghai, China[J]. Chin J Virol, 2013, 29(6): 608-614. (in Chinese)
沈震, 王刚, 宰淑蓓, 等. 诺如病毒新型GII.4流行株Sydney-2012在上海地区的检出和鉴定[J]. 病毒学报, 2013, 29(6): 608-614.
[10] Liu X, Liu P, Wang J, et al. Seroepidemiology of norovirus GII.3 and GII.4 infections in children with diarrhea in Xi’an, China[J]. Foodborne Pathog Dis, 2015, 12(6): 500-505.
[11] Chen R, Neill JD, Estes MK, et al. X-ray structure of a native calicivirus: structural insights into antigenic diversity and host specificity[J]. Proc Natl Acad Sci U S A, 2006, 103(21): 8048-8053.
[12] Gentry J, Vinje J, Guadagnoli D, et al. Norovirus distribution within an estuarine environment[J]. Appl Environ Microbiol, 2009, 75(17): 5474-5480.
[13] Verhoef L, Vennema H, van Pelt W, et al. Use of norovirus genotype profiles to differentiate origins of foodborne outbreaks[J]. Emerg Infect Dis, 2010, 16(4): 617-624.
[14] Kiulia NM, Mans J, Mwenda JM, et al. Norovirus GII.17 predominates in selected surface water sources in Kenya[J]. Food Environ Virol, 2014, 6(4): 221-231.
Etiology and genotype features analysis of an acute gastroenteritis outbreak associated with Norovirus GII.17
YAO Dong, CHEN Jing-fang, YE wen, OU Xin-hua
(ChangshaCenterforDiseaseControlandPrevention,Changsha410001,China)
To identify the pathogens of an acute gastroenteritis outbreak in a factory of Changsha in December, 2014 and further analyze the genotypes of the pathogens. GI/GII norovirus of six anal swabs and four water samples were detected by real-time RT-PCR. The VP1 gene fragments of norovirus positive samples were then amplified and sequenced by RT-PCR with specific primers. BLAST alignment and phylogenetic analysis were performed based on the VP1 sequences. The ten specimens were tested norovirus GII positive and the VP1 sequences showed 99% homology with strain GII/Hu/HKG/2014/ GII.17/CUHK-NS-463 isolated in Hong Kong in 2014. It’s confirmed the norovirus genotype in this outbreak was GII.17. Phylogenetic analysis of viral VP1 sequences showed that the GII.17 norovirus in this study were closely related to strains circulating in Asian areas like Japan, Hong Kong and Taiwan, but a little far from those isolated in U.S.A and French which formed another cluster. The pathogen of this acute gastroenteritis outbreak was norovirus and the genotype was GII.17.
norovirus; gastroenteritis; genotype
Ou Xin-hua, Email: xhouteam@163.com
欧新华,Email: xhouteam@163.com
长沙市疾病预防控制中心,长沙410001
R373.3
A
1002-2694(2016)07-0641-03
2015-10-19;
2016-05-09
DOI:10.3969/j.issn.1002-2694.2016.07.010
