利用微滴数字PCR方法快速分析转基因玉米中外源基因的拷贝数
2016-08-15姜志军徐摇光张立全张晓东
姜志军, 江 颖, 徐摇光, 张立全, 张晓东*
1.北京市农林科学院北京农业生物技术研究中心, 北京 100097;2.首都师范大学生命科学学院, 北京 100037
利用微滴数字PCR方法快速分析转基因玉米中外源基因的拷贝数
姜志军1,2,江颖1,徐摇光1,张立全1,张晓东1,2*
1.北京市农林科学院北京农业生物技术研究中心, 北京 100097;2.首都师范大学生命科学学院, 北京 100037
以玉米自交系501幼胚为受体材料,首先将来自球形节杆菌的EPSPS基因(G23V)按玉米密码子偏爱性进行优化与人工合成,并且将其克隆到表达载体pBAC9200中;然后利用农杆菌介导法将质粒载体转入玉米自交系501的幼胚中。经过愈伤诱导、草甘膦抗性筛选和分化培养最终获得14株转化再生植株。经PCR、RT-PCR检测表明,其中5株目的基因G23V-EPSPS稳定整合且在转录水平获得表达。随后,利用微滴数字PCR技术对外源基因拷贝数进行了检测分析,分析结果表明在5株阳性转基因植株中,外源基因G23V-EPSPS拷贝数分别为0.12、1.0、0.9、1.89和0.66,介于0~2之间。成功建立了草甘膦抗性基因G23V-EPSPS在玉米中的遗传转化体系,为以新型高抗草甘膦G23V-EPSPS基因作为转基因玉米筛选标记基因奠定了基础;而且以微滴数字PCR技术代替传统的Southern Blot简便快速的完成外源基因拷贝数的分析,为微滴数字PCR技术在转基因外源基因拷贝数检测上的广泛应用做了初步的探索。
转基因玉米;G23V-EPSPS基因;抗草甘膦;ddPCR
玉米(ZeamaysL.)是世界三大粮食作物之一,在农业、畜牧业和工业中占据重要地位,全球对玉米的需求量正在不断攀升。近年来,玉米作为转基因技术主要的研究对象受到世界各国科学家的广泛关注。应用转基因技术已获得多个可用的农艺性状,如在玉米螟和食根害虫的防治、除草剂的抗性以及玉米营养品质的改善等方面都取得了一定的成果[1,2]。近些年,尽管转基因技术在玉米中的应用已较为成熟,但在转基因阳性植株的筛选过程中仍然存在着筛选效率低、假阳性高的问题,所以探索一种高效筛选转基因阳性植株的方法意义重大。当前所报道的转基因玉米成功案例中大部分都是以抗除草剂基因作为筛选标记基因如bar、CP4-EPSPS等,其中CP4-EPSPS已商业化应用多年[3]。虽然以bar和CP4-EPSPS基因作为转基因玉米阳性植株的筛选标记基因已比较成熟且筛选效率有所改善,但在实际转基因研究中假阳率性仍然很高,因此探索一个能在玉米中高效表达且高抗草甘膦的基因对于进一步提高转基因阳性植株的筛选效率至关重要。
数字PCR(digital PCR)是近年来迅速发展起来的一种突破性的核酸定量分析技术。 其技术原理是先将核酸模板进行大量稀释,使其分配到多个独立的反应单元中,每个反应单元中只有单个核酸模板分子,然后以每个独立的反应单元进行PCR扩增反应,扩增结束后对每个反应室的荧光信号进行统计学分析,最终实现对核酸的绝对定量。微滴数字PCR(droplet digital PCR, ddPCR)是数字PCR技术的一种,它主要是以形成大量油包水微滴的形式对核酸进行成千上万倍稀释,然后以每个小微滴为独立的反应单元进行PCR反应,最后利用泊松分布原理对核酸分子进行绝对定量分析[4~8]。微滴数字PCR技术在转基因外源基因拷贝数分析方面具有诸多优点,与Southern Blot相比,它工作量小、周期短、操作要求低、准确性高;与实时荧光定量PCR相比,它灵敏度高、检测限度低至1/104、无需标准品不依赖于标准曲线、采用终点PCR信号计数检测不依赖于Ct值、能有效克服PCR抑制剂的影响,是一种更加理想的进行外源基因拷贝数分析的新方法[9,10]。本研究探索将一种新的具有高抗草甘膦功能和超强热稳定性的G23V-EPSPS(G23V 5-enolpyruvylshikimate-3-phosphate synthase)基因[11,12]利用农杆菌介导法转入玉米中以验证该基因在玉米中能否顺利整合并表达;另外借助微滴数字PCR技术对外源基因拷贝数进行快速鉴定,为利用微滴数字PCR技术分析转基因植物外源基因拷贝数做了初步的探索。
1 材料与方法
1.1仪器与试剂
1.1.1实验仪器Nanodrop ND-2000核酸蛋白定量仪(Thermo Scientific);QX200 Droplet Digital PCR系统(Bio-Rad 包括微滴生成仪、盖膜仪、PCR仪和微滴读取仪 )。
1.1.2微滴数字PCR实验中所需的试剂2×QX200 ddPCR EvaGreen Supermix(Bio-Rad);微滴生成油 droplet generation oil/(Bio-Rad)。微滴数字PCR反应中内参基因为HMGA(GenBank: AJ131373.1)。
1.2实验材料
1.2.1转基因受体材料转基因受体材料为玉米(ZeamaysL.)自交系501幼胚,玉米幼穗取于自交授粉后11~12 d,幼胚大小为0.8~1.5 mm。
1.2.2菌株和植物表达载体构建[13,14]农杆菌(Agrobacteriumtumefaciens)菌株为EHA105;目的基因依据GenBank:GM718572.1序列,按照玉米密码子进行优化与人工合成(由上海捷瑞生物工程公司合成),优化后的G23V-EPSPS基因为1 242 bp,翻译蛋白氨基酸413个,用花椰菜花叶病毒CaMV35S启动子与玉米Adh1基因的第1个内含子驱动G23V-EPSPS基因,获得植物表达载体pBAC 9200(图1)。

图1 质粒pBAC 9200的T-DNA区Fig.1 The map of plasmid pBAC 9200 T-DNA.P35S:花椰菜花叶病毒35S RNA的启动子; adh1 intron:玉米Adh1基因的第一个内含子; G23V-EPSPS:G23V型5-烯醇式丙酮酸莽草酸-3-磷酸合酶基因; Tnos:农杆菌胭脂碱合成酶基因终止子
1.3实验方法
1.3.1农杆菌转化[15~19]利用携带表达载体pBAC 9200的EHA105农杆菌菌株介导玉米自交系501幼胚的遗传转化。具体实验步骤如下:①农杆菌侵染液的制备:将-80℃保存的包含表达载体的EHA105农杆菌在含有抗生素的YEP固体培养基上28℃划线培养3 d;挑取单菌落涂于含有抗生素的YEP固体培养基中20℃培养3 d;将培养好的农杆菌用接种环直接刮取到配好的侵染液中,并用侵染液调节OD600=0.35,放于摇床中,22℃,180 r/min,振荡4 h;4 h后侵染液直接用于幼胚的侵染。②遗传转化及植株再生过程:将玉米幼穗于75%乙醇中5 min,无菌水洗3次, 然后于25%NaClO中消毒3 min,无菌水洗3次;用无菌刀片将玉米籽粒幼胚逐个剥出放到已配制好的农杆菌侵染液(培养基配方见表1)中30 min;用无菌滤纸吸干幼胚表面浮液,将幼胚转入共培养培养基中22℃进行暗培养3 d。
1.3.2除草剂抗性愈伤筛选与植株再生将幼胚转入愈伤诱导培养基(培养基配方见表1)中25℃暗培养15 d进行愈伤诱导;将愈伤转入筛选培养基中25℃暗培养筛选2次,每次15 d;然后将筛选后生长状态良好的愈伤转入分化培养基中,25℃光照培养进行分化成苗;将分化的幼苗转入壮苗培养基中25℃光照培养进行壮苗;最后将获得的转化苗室温条件下进行炼苗,7 d后移入大田中。

表1 玉米遗传转化再生培养基
1.3.3T0代转化玉米植株分子生物学检测经筛选分化的转化再生植株,分别进行PCR、G23V-EPSPS插入拷贝数与RT-PCR等分子生物学检测分析。
①G23V-EPSPS基因的PCR检测。采用天根生化科技有限公司的DNA Secure Plant Kit提取玉米叶片基因组DNA,运用Primer 5.0设计G23V-EPSPS基因的特异引物(表2)。PCR反应体系:DNA模板3 μL,2×TaqMaster Mix 12.5 μL,正、反向引物(10 μmol/L)各1 μL,RNase-Free-water(购自TaKaRa公司)7.5 μL,共25 μL。PCR程序如下:95℃ 5 min;94℃ 30 s,62℃ 30 s,72℃ 30 s, 28个循环;72℃ 5 min,16℃保存。
②G23V-EPSPS基因的插入拷贝数分析。微滴数字PCR完整的实验流程包括:PCR反应体系的配制、生成微滴、PCR扩增和信号读取4个步骤[20]。微滴数字PCR体系:DNA模板30 ng/20 μL体系,2×QX200 ddPCR EvaGreen Supermix 10 μL,正、反向引物(10 μmol/L)各0.2 μL,加RNase-Free-water直至总体系20 μL。生成微滴需要使用仪器专配的微滴生成卡和微滴生成仪,将20 μL PCR反应体系与70 μL微滴生成油一同加入微滴生成卡中,盖上专用胶垫后放入微滴生成仪中生成微滴。微滴数字PCR反应程序:95℃ 5 min;95℃ 30 s,60℃ 30 s,72℃ 30 s, 40个循环;4℃ 5 min,90℃ 5 min,12℃保存。每个样品进行3个平行重复实验,PCR扩增完毕后,将PCR产物放入微滴读取仪中依据荧光信号的有无对阳性微滴和阴性微滴进行判定读值。最后,在电脑上利用软件QuantaSoft V1.7.4对数据进行分析,最终得到核酸绝对定量结果。
③G23V-EPSPS基因的RT-PCR检测。用Trizol Reagent(购自Ambion公司)提取玉米叶片总RNA。采用逆转录试剂盒(Prime ScriptTMRT reagent Kit with gDNA Eraser,购自TaKaRa公司)将RNA逆转录成cDNA。以cDNA为模板进行PCR扩增,采用Primer 5.0设计G23V-EPSPS基因和内参Actin基因的半定量特异引物(表2)。 RT-PCR反应体系:稀释5倍的cDNA 模板1 μL,2×TaqMaster Mix 12.5 μL,正、反向引物(10 μmol/L)各1 μL,RNase-Free-water 9.5 μL,总共25 μL。Actin基因RT-PCR程序如下:95℃ 5 min;95℃ 20 s,54℃ 20 s,72℃ 20 s,30个循环;72℃ 5 min,16℃保存。G23V-EPSPS基因RT-PCR程序如下:95℃ 5 min;95℃ 30 s,58℃ 30 s,72℃ 30 s,40个循环;72℃ 5 min,16℃保存。

表2 PCR检测中的引物序列
2 结果与分析
2.1G23V-EPSPS基因的PCR检测结果
对经草甘膦抗性筛选分化再生获得的14株转化玉米苗进行初步的PCR检测,以检测外源G23V-EPSPS基因是否转入并整合到玉米基因组中(图2)。14株转化苗中共有5株即2、9、10、11、12号植株表现出PCR阳性,初步确认这5颗植株发生了G23V-EPSPS基因的整合,阳性率为35.7%。
2.2G23V-EPSPS基因插入拷贝数检测结果
利用微滴数字PCR技术对PCR表现阳性的5株转基因植株进一步进行拷贝数检测。本研究中计算外源基因拷贝数的方法为:
外源基因拷贝数=外源基因浓度/内参基因浓度(本研究中所选的内参基因HMGA在玉米中的拷贝数为1)。首先,对微滴数字PCR实验中玉米内参基因HMGA与G23V-EPSPS基因相应引物的特异性进行检测(图3),二者的阳性微滴(蓝色带)与阴性微滴(黑色带)都能够显著的区分,证明内参基因HMGA与G23V-EPSPS基因引物的特异性没有问题,且说明系统可以准确判读出阳性微滴与阴性微滴的数目;其次,对微滴数字PCR的重复性进行分析(表3),所有样品内参HMGA基因和G23V-EPSPS基因的实验微滴总数均大于12 000,满足微滴数字PCR微滴的分析要求,同时,实验中生成微滴的RSD(相对标准偏差)介于2%~14%之间,小于25%,符合欧盟核酸定量检测的要求,说明实验中建立的微滴数字PCR体系微滴生成稳定,重复性良好,数据可靠性高;最后,完成对5株转基因植株中G23V-EPSPS基因拷贝数的分析(表4),G23V-EPSPS基因的插入拷贝数在0~2之间。

图2 T0代转基因玉米G23V-EPSPS基因的PCR检测Fig.2 Analysis of G23V-EPSP gene’s integration in T0 transgenic maize genome with PCR.M:DNA marker;+:阳性质粒;H2O:空白对照;WT:阴性对照;1~14:获得的转化再生植株
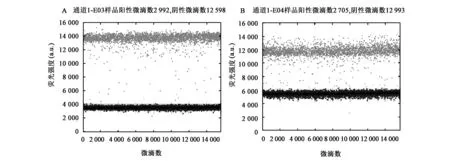
图3 微滴数字PCR引物特异性检测Fig.3 Droplet digital PCR specificity detection of primers.A.内参基因;B.G23V-EPSPS基因。注:图中灰色信号点代表发生PCR扩增的微滴,系统判读为阳性信号;黑色的信号点代表未发生PCR扩增的微滴,系统判读为阴性信号。

样品基因微滴数总数123平均值标准偏差SD相对标准偏差RSDH2OHMGA1403416997-1551520950.14G23V1576516383-160744360.03WTHMGA1454317120168101615714060.09G23V155401708115839161538170.051HMGA156081713817338166949460.06G23V1521617323177761677113360.082HMGA1494717866169501658714920.09G23V1325716708154431513617450.123HMGA1557915590181981645515080.09G23V156071569816134158132810.024HMGA1472317401151161574614460.09G23V152101622715002154796550.045HMGA167631598417292166796570.04G23V164241697615965164555060.03
2.3G23V-EPSPS基因的RT-PCR检测结果
对PCR表现阳性的5株转基因植株进行RT-PCR检测,以检测目的G23V-EPSPS基因在玉米机体中是否顺利转录(图4)。首先,对RNA完整性进行验证(图4A),28S、18S和5.8S RNA完整性良好;其次,对逆转录后获得的cDNA质量进行验证(图4B),所有样品均得到了特异的目的DNA片段,cDNA没有问题;最后,完成对G23V-EPSPS基因的RT-PCR检测(图4C),所获得的5株转化再生植株,G23V-EPSPS基因都在转录水平获得了表达。
3 讨论
G23V-EPSPS基因是一种来源于原核细菌中的基因,由于原核生物与真核生物在一些氨基酸上具有不同的密码子偏爱性,所以为了提高G23V-EPSPS基因在玉米中的表达效率,本研究依据玉米中密码子的偏爱性对其进行了密码子优化处理[21]。有文献报道在常用的植物转基因方法中,基因枪转化法与农杆菌介导转化法相比,农杆菌介导的外源基因转化系统所产生的外源基因低拷贝事件高于基因枪转化法[22],并且外源基因的表达水平也要高于基因枪转化法,造成外源基因沉默的几率较基因枪法低[23]。所以本研究采用农杆菌EHA105介导的遗传转化法将外源G23V-EPSPS基因转入玉米自交系501幼胚中,经过愈伤诱导草甘膦抗性筛选与分化等过程,最终获得5个在转录水平表达的株系。运用ddPCR技术对外源基因拷贝数进行快速分析,结果表明5个转基因株系都是外源基因低拷贝事件,插入拷贝数在0~2之间并成功获得了转G23V-EPSPS基因的T0代转基因植株。最后,尽管ddPCR的灵敏度和精确性相对较高,但是如需准确的确定外源基因的插入拷贝数,仍需要进一步通过Southern Blot进行验证。

表4 ddPCR G23V-EPSPS基因拷贝数分析

图4 5株转基因阳性植株RT-PCR扩增结果Fig.4 RT-PCR results of PCR positive maize lines.A.RNA 1%琼脂糖凝胶电泳图;B.内参基因Actin的RT-PCR扩增结果;C.G23V-EPSPS基因的RT-PCR扩增结果。M:DNA marker;+:阳性质粒对照;H2O:空白对照;WT:非转基因阴性对照;1~5:获得的5株PCR阳性植株
[1]李余良,胡建广. 转基因玉米研究进展[J]. 中国农学通报,2006, 22(2): 71-75.
[2]尹祥佳,翁建峰,谢传晓,等. 玉米转基因技术研究及其应用[J]. 作物杂志, 2010,(6): 1-9.
[3]Glyphosate: A unique global herbicide[J]. Am. Chem. Soc. Monog., 1997, 189: 521-615.
[4]孙 越,刘秀霞,李丽莉,等. 抗亚洲玉米螟、抗草甘膦转基因玉米的培育[J]. 农业生物技术学报,2015,(1): 52-60.
[5]葛 敏,张体付,王 华,等. 农杆菌介导Cry1Ab/Ac抗虫转基因玉米植株的获得[J]. 江苏农业学报, 2012,(2): 254-258.
[6]徐丹丹,王丕武,赵邯郸,等. 农杆菌介导phyA基因转化玉米愈伤组织培养体系的优化[J]. 湖北农业科学,2015,(2): 465-467.
[7]王宏伟,梁业红,史振声,等. 农杆菌介导玉米幼胚遗传转化体系的研究[J]. 玉米科学, 2011, 19(3): 73-75.
[8]王洪红. 农杆菌介导的玉米幼胚遗传转化体系的优化[J]. 生物技术世界,2012,(6): 59-61.
[9]孙传波,郭 嘉,袁 英. 农杆菌介导玉米幼胚遗传转化体系的建立[J]. 湖北农业科学, 2014,(12):2743-2746.
[10]Yu G, Liu Y, Du W,etal.. Optimization ofAgrobacteriumtumefaciens-mediated immature embryo transformation system and transformation of glyphosate-resistant gene 2mG2-EPSPS in maize (ZeamaysL.)[J]. J. Integr. Agric., 2013, 12(12): 2134-2142.
[11]Mu D, Yan L, Tang H,etal.. A sensitive and accurate quantification method for the detection of hepatitis B virus covalently closed circular DNA by the application of a droplet digital polymerase chain reaction amplification system[J]. Biotechnol. Lett., 2015, 37(10): 2063-2073.
[12]Brunetto G S, Massoud R, Leibovitch E C,etal.. Digital droplet PCR (ddPCR) for the precise quantification of human T-lymphotropic virus 1 proviral loads in peripheral blood and cerebrospinal fluid of HAM/TSP patients and identification of viral mutations[J]. J. Neuro. Virol., 2014, 20(4): 341-351.
[14]李春勇. 数字PCR技术原理及应用[J]. 生物技术世界, 2014,(11): 10-13.
[15]林彩琴,姚 波. 数字PCR技术进展[J]. 化学进展, 2012,(12): 2415-2423.
[16]White T B, Mccoy A M, Streva V A,etal.. A droplet digital PCR detection method for rare L1 insertions in tumors[J]. Mobile DNA, 2014, doi:10.1186/s13100-014-0030-4.
[17]Corbisier P, Pinheiro L, Mazoua S,etal.. DNA copy number concentration measured by digital and droplet digital quantitative PCR using certified reference materials[J]. Anal. Bioanaly. Chem., 2015, 407(7): 1831-1840.
[18]Pipke R, Amrhein N. Isolation and characterization of a mutant ofArthrobactersp. strain GLP-1 which utilizes the herbicide glyphosate as its sole source of phosphorus and nitrogen[J]. Appl. Environ. Microbiol., 1988, 54(11): 2868-2870.
[19]Pipke R, Amrhein N. Degradation of the phosphonate herbicide glyphosate byArthrobacteratrocyaneusATCC 13752[J]. Appl. Environ. Microbiol., 1988, 54(5): 1293-1296.
[20]姜 羽,胡佳莹,杨立桃. 利用微滴数字PCR分析转基因生物外源基因拷贝数[J]. 农业生物技术学报, 2014, 22(10): 1298-1305.
[21]Liu D. Design of gene constructs for transgenic maize: methods in molecular biology[J]. Methods Mol. Biol., 2009,526:3-20.
[22]Shou H, Frame B, Whitham S,etal.. Assessment of transgenic maize events produced by particle bombardment orAgrobacterium-mediated transformation[J]. Mol. Breed., 2004, 13: 201-208.
[23]Vaucheret H, Beclin C, Elmayan T,etal.. Transgene induced gene silencing in plants[J]. Plant J., 1998,16: 651-659.
Rapid Analysis of Exogenous Gene’s Copy Number in Transgenic Maize with Droplet Digital PCR
JIANG Zhi-jun1,2, JIANG Ying1, XU Yao-guang1, ZHANG Li-quan1, ZHANG Xiao-dong1,2*
1.BeijingAgriculturalBiotechnologyResearchCenter,BeijingAcademyofAgricultureandForestryScience,Beijing100097,China;2.CollegeofLifeScience,CapitalNormalUniversity,Beijing100037,China
In this study,EPSPSgene deriving fromArthrobacterglobiformiswas optimized with maize biased codons and synthesized. The synthesized gene named asG23Vwas cloned into plant expression vector pBAC9200. The vector was transformed into immature embryos of maize (Zeamays) inbred lines 501 withAgrobacterium-mediated transformation method. Total 14 regenerated plants were obtained following steps of callus induction, glyphosate resistance screening and differentiation culture. PCR, RT-PCR detection showed that five transformed plants possessedG23V-EPSPSgene’s integration and expression on the transcriptional level. Finally,G23V-EPSPSgene’s insertion copy number was detected using droplet digital PCR technology. The results showed that, in the five confirmed genetically modified plants, the copy number ofG23V-EPSPSgene was 0.12, 1.0, 0.9, 1.89, 0.66, respectively, ranging between 0 to 2. The study established genetic transformation system of glyphosate resistance gene in maize, which laid the foundation forEPSPSgene as selection marker in transgenosis. Besides, droplet digital PCR technology is a simple and rapid method to analyze foreign gene’s insertion copy number, and could replace traditional Southern Blot method in the near future.
transgenic maize;G23V-EPSPSgene; glyphosate resistance; ddPCR
10.3969/j.issn.2095-2341.2016.04.12
2016-04-21; 接受日期:2016-05-28
国家转基因生物新品种培育重大专项(2013ZX08003-001);北京市农林科学院创新团队(JNKST201617)资助。
姜志军,硕士研究生,研究方向为植物遗传学。E-mail:jiangzhijun12345@126.com。*通信作者:张晓东,研究员,博士,主要从事作物遗传育种研究。E-mail:zhangxiaodong@baafs.net.cn
