水稻转录因子基因OsPHR3在磷素利用过程中的作用
2016-08-09张亮孙文献孙雅菲裴文霞罗闻真孙瑞张占田徐国华孙淑斌
张亮 孙文献 孙雅菲 裴文霞 罗闻真 孙瑞 张占田 徐国华 孙淑斌
(南京农业大学 资源与环境科学学院, 南京210095;*通讯联系人, E-mail: sunshubin@njau.edu.cn)
水稻转录因子基因OsPHR3在磷素利用过程中的作用
张亮孙文献孙雅菲裴文霞罗闻真孙瑞张占田徐国华孙淑斌*
(南京农业大学 资源与环境科学学院, 南京210095;*通讯联系人,E-mail:sunshubin@njau.edu.cn)
ZHANGLiang,SUNWenxian,SUNYafei,etal.RolesoftranscriptionfactorgeneOsPHR3ontheutilizationofphosphateinrice.ChinJRiceSci, 2016, 30(4): 397-405.
张亮, 孙文献, 孙雅菲, 等. 水稻转录因子基因OsPHR3在磷素利用过程中的作用. 中国水稻科学, 2016, 30(4): 397-405.
摘要:磷是植物体生长发育所必需的大量营养元素之一,广泛参与植物体多种生命活动。土壤中磷的有效性很低,是农业生产中限制作物产量的重要因素。OsPHR3(LOC_Os02g04640)属于MYB-CC家族,与水稻中磷信号途径中心调控因子OsPHR2是同源基因,且具有部分功能重叠。本研究利用转基因技术获得OsPHR3基因的突变体和超表达材料,通过水培实验、32Pi同位素实验以及桶培实验来研究该基因在吸收利用磷素过程中的作用。水培实验表明,与野生型相比,突变体磷含量无明显差异,基因超表达能够提高水稻体内磷含量。32Pi同位素实验显示,与野生型相比,缺磷时突变体吸收速率降低,而该基因超表达能够促进磷素的吸收与转运。桶培实验表明,该基因超表达能够增加水稻有效分蘖数,提高种子中磷含量,该基因缺失使得穗长变短。OsPHR3基因可能调控促进磷的吸收与向地上部转运。该研究将为以后分子育种提供依据。
关键词:水稻; 磷酸盐; OsPHR3
水稻是全球最重要的粮食作物之一,占世界谷类作物种植面积的1/3,约有一半的人口以稻米为主食。中国作为世界上水稻种植面积最大的国家,目前种植面积达3100多万hm2,占世界总量的20% , 水稻产量占全国粮食总产的50%,是保证我国粮食安全的最重要作物[1,2]。世界上约有43%的耕地缺磷,我国的缺磷耕地占到2/3左右。因此,研究水稻对磷素的吸收利用机制,提高磷素利用率,对于提高作物产量具有重要的现实意义。
磷是植物生长发育所必需的大量营养元素之一,占植物体干质量的0.05%~0.5%[3]。是许多重要有机化合物(如核酸、蛋白、磷脂、ATP等)的组分,并且广泛参与了植物体内的信号转导、光合作用、呼吸作用、糖酵解、三羧酸循环等代谢过程[4]。土壤中可以直接被植物吸收利用的磷的浓度一般不超过10μmol/L,有的甚至低于1μmol/L,远低于植物正常生长发育所需的磷浓度[5]。植物为了适应土壤中的缺磷环境,经过长期进化形成了一系列的适应性反应,包括改变根系形态结构、增加有机酸的分泌、与菌根真菌形成菌根共生体以及增强或诱导基因的表达等途径来活化和提高土壤中磷的生物有效性[6-9]。
2001 年,Rubio等报道了拟南芥缺磷信号转导途径的中心调控因子 AtPHR1[10]。此后不同物种中 AtPHR1 的功能性同源基因相继被发现,拟南芥中MYB-CC转录因子基因家族共有 15 个成员,其中与 AtPHR1 亲缘关系最近的为At5g29000,命名为AtPHR1 。EMSA等实验表明AtPHR1 具有类似 AtPHR1 的体外结合P1BS的能力,说明MYB-CC家族转录因子间存在部分功能冗余[11]。2008年,Zhou等[13]用拟南芥AtPHR1(At4g28610)蛋白序列进行BLAST分析,在水稻基因组中发现了两个AtPHR1 的同源基因AK063486 和AK100065,分别命名OsPHR1 和 OsPHR2, 在转录水平基本不响应缺磷诱导。正常供磷条件下,OsPHR2超表达会导致地上部磷的过量积累,表现出磷中毒症状,且根毛显著多于野生型[12,13],同时OsPHR2是水稻磷信号转导途径的中心调控因子[13]。OsPHR3(LOC_Os02g04640)是水稻中OsPHR2的同源基因,氨基酸序列同源性为45.21%,具有部分相似功能,与OsPHR1 和 OsPHR2共同参与下游磷相关基因的调控[13,14]。本研究通过转基因技术获得该基因的超表达和敲除材料,通过水培实验、32Pi同位素吸收与转运实验以及桶培实验研究了该基因在磷素利用过程中的作用,以期为后续的分子育种提供依据。
1材料与方法
1.1试验材料
供试材料:日本晴野生型(WildType;WT),纯合突变体osphr3和 OsPHR3超表达材料。
1.2水稻和拟南芥中MYB-CC家族成员进化树分析
水稻OsPHR3与OsPHR1、OsPHR2一样,同属于MYB-CC家族成员。为了研究该基因在MYB-CC家族的进化关系,根据水稻和拟南芥中MYB-CC家族相关基因登录号,在NCBI(http://www.ncbi.nlm.nih.gov/)和TAIR(http://www.arabidopsis.org/)网站上获得相应的氨基酸序列,利用ClustalX和MEGA5.0软件做进化树,引导程序设为 1000。
1.3 缺磷条件下OsPHR3的表达模式
以粳稻品种日本晴野生型为试验材料,1/2MS培养基发苗,7d后选择生长一致的幼苗去种子移入7L培养箱中,每箱20棵苗,先清水缓苗然后全营养液培养7d进行处理。设置为正常供磷营养液(0.30mmol/LKH2PO4,+P)和缺磷营养液(0.00mmol/LKH2PO4,-P)处理,缺磷处理的营养液中用0.30mmol/L的KCl补足K+的含量,其他营养成分参照IRRI水稻完全营养液配方。每天调pH至5.5,每3d换一次营养液,每个处理4个重复,处理14d后分为地上部和根系采样,贮存于-70℃冰箱。采用Trizol法提取总RNA,cDNA合成用TaKaRa公司的定量反转录试剂盒(RR047A),使用SYBY定量RT-PCR试剂(DRR041A)进行实时PCR检测。
1.4转基因材料水培实验
根据OsPHR3基因序列信息,从韩国突变体网站SIGnALSalk(http://signal.salk.edu/)查询突变体信息,鉴定后获得3个株系的Tos17纯合突变体,沉默效果很好,分别命名为osphr3-1、osphr3-2、osphr3-3。同时,利用转基因技术得到该基因的超表达材料,经潮霉素筛选和GUS染色鉴定,得到日本晴阳性转基因材料29个株系。然后定量RT-PCR鉴定超表达效果,选择效果较好的3个株系Ox1、Ox2、Ox3进行后续的实验。
转基因水稻株系和野生型材料,用1/2MS培养基发苗,7d后选择生长一致的幼苗去种子移入7L培养箱中,每箱16棵苗,先清水缓苗然后全营养液培养7d进行处理。设置为正常供磷营养液(0.30mmol/LKH2PO4,+P)和缺磷营养液(0.00mmol/LKH2PO4,-P)处理,其中缺磷处理的营养液用0.30mmol/L的KCl补足K+的含量,每个处理4个重复,其他营养成分参照IRRI水稻完全营养液配方。每天调pH至5.5,每3d换一次营养液,处理21d后观察表型并统计相关生理数据。
1.5水培条件下转基因材料磷含量的测定
转基因材料经水培缺磷处理21d后收获,分为地上部和根系两部分,鲜样用液氮磨成粉末后称取0.5g左右,用高氯酸提取法和硫酸-钼酸铵比色法测定可提取磷含量;另外一批样品105°C恒温箱杀青30min,然后于70°C恒温箱烘至恒重磨碎,称取磨碎的植株样品0.05g左右,采用硫酸-双氧水消煮法和钼锑抗比色法测定总磷含量。具体参照土壤农化分析第三版进行测定。
1.632Pi同位素的吸收和转运实验
正常供磷和缺磷条件下处理14d后的转基因苗转移到300mL的一次性杯子中进行同位素示踪实验,每杯3穴,每穴1株,每杯装有160mL含100mg/LKH232PO4的IRRI营养液,每杯营养液中实际加入总量2.96×105Bq32Pi放射性原液。植株消解样品鲜质量控制在0.3g以下,消解过程使用2mL高氯酸和1mL双氧水,室温过夜消解样品至清亮。取300μL消解样进行放射性测定,300μL消解样加入5mL离心管中(管壁完全光滑无刻度无磨砂面),再加入3.5mL闪烁液(型号OPTIPHASE“HISAFE”-3),加入闪烁液后强烈摇晃离心管,放置后观察混合液是否澄清均一,如无分层现象混匀后放置4h以上,在南京农业大学同位素实验室进行测定。
1.7转基因材料在正常供磷条件下的全生育期实验
为了观察OsPHR3基因对整个生育期的影响,在南京农业大学牌楼基地进行桶培实验,所用土壤取自江苏省南京市牌楼地区酸性黄棕壤,每桶15kg土壤,每1kg土壤施肥量为尿素220mg/kg,KH2PO4120mg/kg,不施钾肥。每个株系6个重复,统计相关生理数据,成熟后收获材料,把野生型和转基因材料分为茎秆、叶鞘、穗柄3个部位,样品在105 ℃下杀青30min,70 ℃下烘至恒重(3d左右)。总磷测定采用硫酸-双氧水消煮和钼锑抗比色法,具体参照土壤农化分析第三版进行测定。
1.8成熟期转基因材料相关农艺性状统计
为了进一步研究OsPHR3基因对水稻生殖器官的影响,成熟后收获材料测定种子中总磷含量,并对水稻的穗长、一次枝梗数、单株产量等农艺性状进行统计。
1.9数据统计分析
用软件Excel和SigmaPlot10.0进行数据统计分析和绘图。
2结果与分析
2.1水稻和拟南芥中MYB-CC家族成员进化树分析
OsPHR3位于水稻第2条染色体上,cDNA全长1404bp,编码467个氨基酸,有7个外显子和6个内含子。根据已报道的拟南芥中MYB-CC家族的15个成员和 OsPHR1、OsPHR2的氨基酸序列进行进化树分析。从图1可以看出,水稻中OsPHR1和OsPHR2亲缘关系较近,OsPHR3与拟南芥中At3g13040亲缘关系较近。相对于OsPHR1来说,OsPHR3与OsPHR2的亲缘关系更近一些。同时,可以看出OsPHR3与OsPHR1和OsPHR2不在一个分支上,即OsPHR3与OsPHR1和OsPHR2的功能可能存在一定的差异。
2.2缺磷条件下OsPHR3的表达模式
如图2所示,在正常供磷和缺磷处理14d后,分别为地上部和根系采样提取RNA,反转录成cDNA后定量RT-PCR检测OsPHR3基因的表达情况。从表达部位上来说,该基因在地上部的表达量高于根系。与正常供磷相比,缺磷条件下OsPHR3基因地上部和根系的相对表达量分别增加了40.6%和52.9%。说明该基因不论是地上部还是根系均受缺磷诱导。
2.3OsPHR3基因的缺失和超表达对水稻苗期生长的影响
为了研究OsPHR3基因对水稻苗期生长的影响,选取苗龄7d的水稻幼苗正常供磷和缺磷处理,14d后统计并观察表型。如图3所示,与野生型相比,不论是在正常供磷还是缺磷条件下,突变体株高和根长均无明显差异。在正常供磷条件下与野生型相比,超表达植株变得矮小,3个株系Ox1、Ox2和Ox3的株高分别比野生型降低了26.3%、17.3%和19.2%;根长也微弱降低(图3-A、B、C);而在缺磷条件下,与野生型相比,超表达材料株高和根长无明显变化(图3-D、E、F)。由此可见,与野生型相比,正常供磷条件下超表达植株生长受到抑制,而缺磷条件下并无明显差异。这可能是由于正常供磷下,OsPHR3基因超表达导致水稻体内磷过度积累,进而导致超表达株系的生长受到抑制。
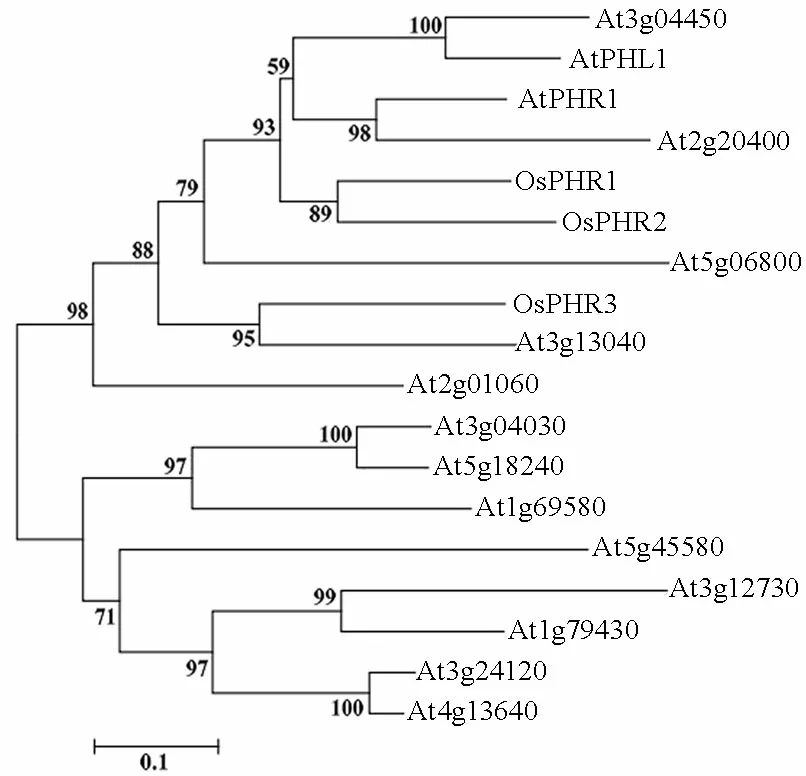
图1水稻和拟南芥中MYB-CC家族成员进化树分析
Fig. 1.PhylogeneticanalysisofMYB-CCfamilymembersinriceandArabidopsis.
2.4水培条件下OsPHR3基因对水稻苗期磷含量的影响
为了解该基因的缺失和超表达后是否影响水稻苗期地上部和根系的磷含量,我们测定了野生型和转基因株系在正常供磷和缺磷条件下地上部和根系的磷含量。我们发现(图4),不论是正常供磷还是缺磷条件下,突变体地上部和根系中磷含量均无明显变化。正常供磷条件下,与野生型相比,超表达株系Ox1、Ox2地上部可提取磷含量分别增加了42.4%、35.2%(图4-A);而总磷含量的变化趋势与可提取磷含量的变化趋势相似,超表达Ox1、Ox2地上部的总磷含量分别增加了29.4%、28.8%,根系分别增加了48.8%、41.1%(图4-C)。缺磷条件下,与与野生型相比较,超表达地上部和根系可提取磷含量无明显变化(图4-B);而超表达Ox1、Ox2根系中总磷含量分别增加了58.3%、45.2%(图4-D)。可以看出,不论是正常供磷还是缺磷条件下,OsPHR3的超表达均能促进水稻磷素的吸收,提高水稻体内的磷含量;但缺磷条件下,超表达株系只有根部总磷增加,而地上部变化不明显。可能是该基因的超表达虽然促进水稻对磷的吸收,但是由于磷的转运相对滞后,且磷素的利用先发生于根部,因此,该基因的超表达未能影响其在缺磷条件下的转运。为此我们设计了32Pi同位素吸收和转运实验来进一步验证这个结果。
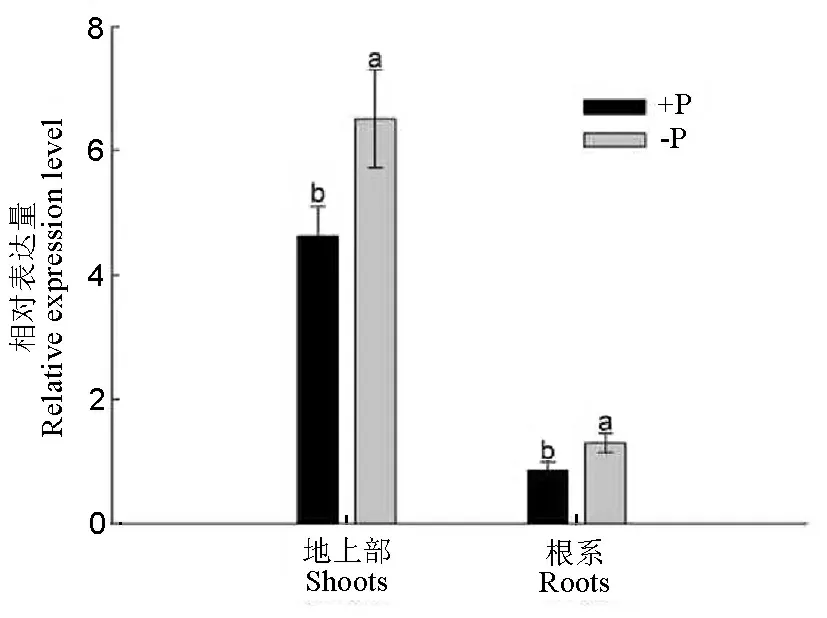
图2OsPHR3基因在正常供磷和缺磷条件下的相对表达量
Fig. 2.RelativeexpressionofOsPHR3underPi-sufficientandPi-deficientconditions.
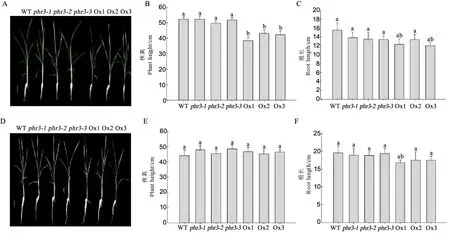
A-正常供磷条件下野生型和转基因材料的表型,A的标尺为5cm;B,C-正常供磷条件下野生型和转基因材料的株高和根长;D-缺磷条件下野生型和转基因材料的表型,D的标尺为5cm;E,F-缺磷条件下野生型和转基因材料的株高和根长。
A,PhenotypeofWTandtransgenicplantsunderPi-sufficientcondition,bar=5cm;B,C,PlantheightandrootlengthofWTandtransgenicplantsunderPi-sufficientcondition;D,PhenotypeofWTandtransgenicplantsunderPi-deficientcondition,bar=5cm;E,F,PlantheightandrootlengthofWTandtransgenicplantsunderPi-deficientcondition.
图3不同供磷条件下野生型和转基因材料表型分析
Fig. 3.ThephenotypicanalysisofWTandtransgenicplantsunderPi-sufficientandPi-deficientconditions.
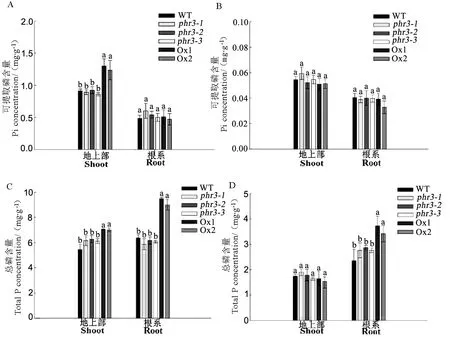
A、C-正常供磷;B、D-缺磷。
A,C,UnderPi-sufficientcondition;B,D,UnderPi-deficientcondition.
图4不同供磷条件下野生型和转基因材料磷含量
Fig. 4.TotalPandPiconcentrationofWTandtransgenicplantsunderPi-sufficientandPi-deficientconditions.
A、C-正常供磷;B、D-缺磷。
A,C,UnderPi-sufficientcondition;B,D,UnderPi-deficientcondition.
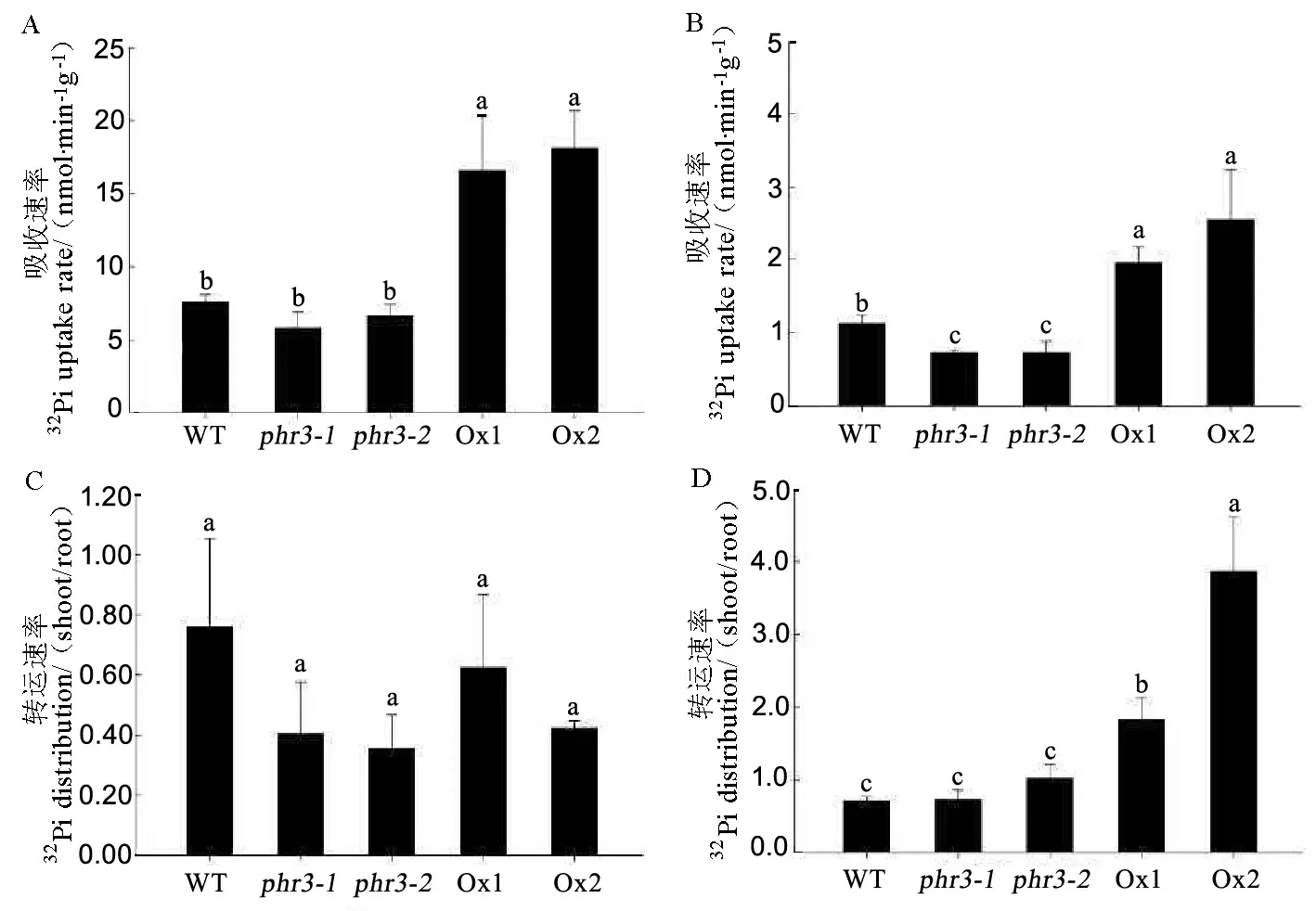
图532Pi同位素吸收和转运速率
Fig. 5.RateofPiuptakebyrootsandtransportationfromroottoshootsusing32Piisotope.
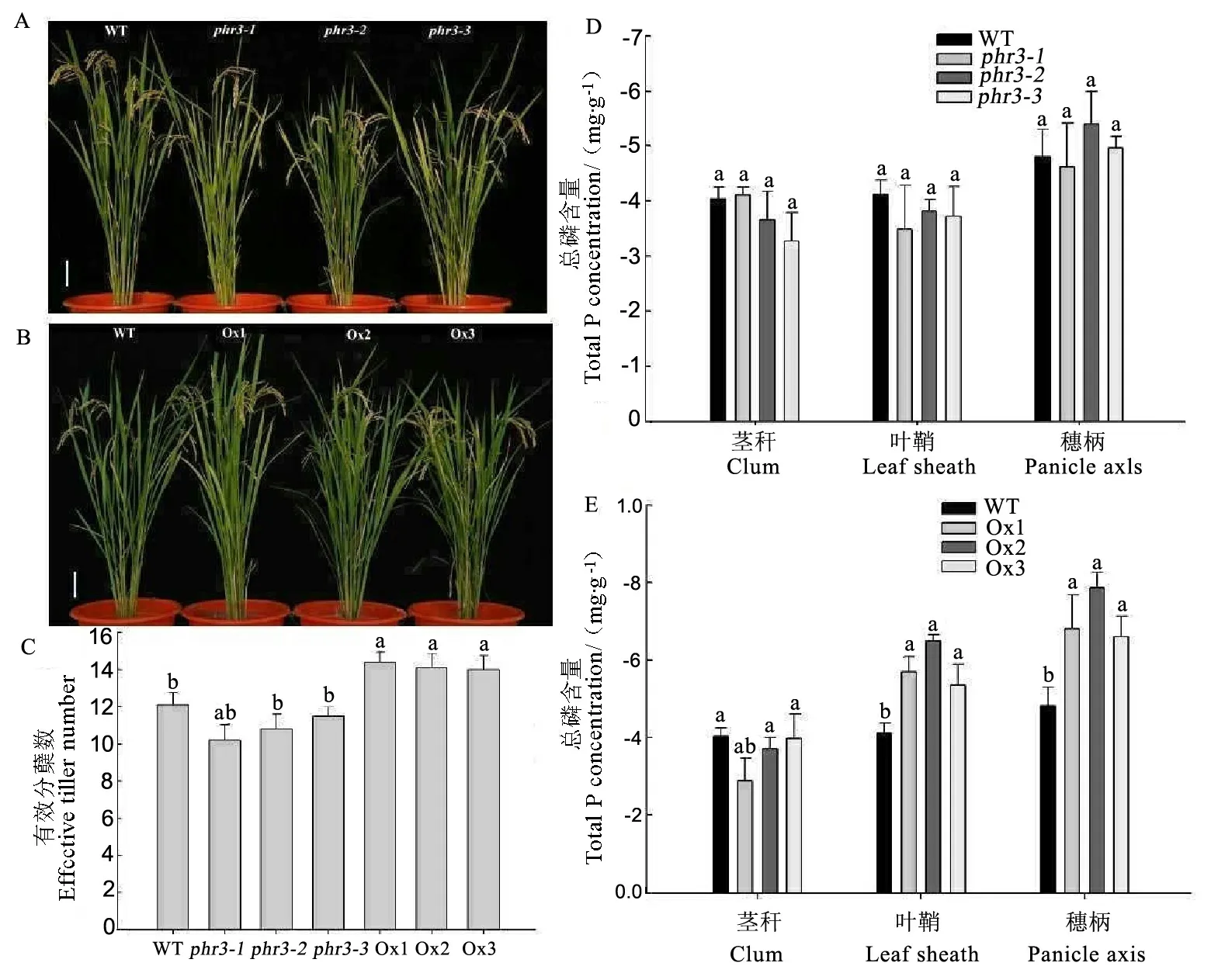
A-野生型和突变体桶培表型,标尺为5cm;B-野生型和超表达桶培表型,标尺为5cm。
A,ThephenotypeofWTandmutantmaterials,Bar= 5cm;B,ThephenotypeofWTandover-expressionmaterials,Bar= 5cm.
图6正常供磷条件下桶培实验中野生型和转基因材料表型及各部位总磷含量
Fig. 6.PhenotypeandtotalPconcentrationindifferenttissuesofWTandtransgenicplantsunderPi-sufficientpotexperiments.
2.5OsPHR3基因对水稻磷素吸收和转运能力的影响
为了验证水培实验结果,确定OsPHR3基因缺失和超表达对水稻吸收及转运磷素能力的影响,我们做了32Pi同位素的吸收和转运实验。转基因材料在正常供磷和缺磷处理14d后,在加入100mg/LKH232PO4的IRRI营养液处理3h,测定吸收和转运速率。结果如图5所示,在缺磷条件下,与野生型相比,突变体材料吸收速率降低,其他无明显变化。正常供磷条件下,与野生型相比,超表达材料的吸收速率明显升高,但是转运速率无明显变化(图5-A、C);缺磷条件下,超表达吸收和转运速率明显升高(图5-B、D)。可见,不论是在正常供磷还是缺磷条件下,OsPHR3超表达都能促进磷的吸收,缺磷时转运速率明显提高。
2.6OsPHR3基因缺失和超表达对水稻成熟期表型及各部位磷含量的影响
为了进一步研究该基因在水稻整个生育期中的作用,我们对桶培至成熟期的野生型、突变体和超表达材料农艺性状进行统计与观测,并测定了野生型和转基因材料茎秆、叶鞘和穗柄3个部分的总磷含量。如图6所示,与野生型相比,突变体株高有变矮趋势,但是有效分蘖数和各部位总磷含量无明显变化。与野生型相比,超表达株高升高,三个株系Ox1、Ox2、Ox3的有效分蘖数分别增加了18.8%、16.3%、8.9%; 叶鞘总磷含量分别增加了38.3%、57.8%、30.3%,穗柄总磷含量分别增加了41.8%、63.4%、37.4%。可见,OsPHR3基因超表达能够提高水稻有效分蘖数,可能通过提高磷的吸收和向地上部转运,从而提高地上部磷含量。
2.7OsPHR3基因缺失和超表达对水稻繁殖器官的影响
为了研究该基因对水稻繁殖器官的影响,我们在水稻成熟期统计并观察野生型、突变体及超表达材料繁殖器官的变化,并测定种子中的磷含量。如图7所示,与野生型相比,突变体三个株系phr3-1、phr3-2、phr3-3穗长分别变短了17.6%、17.5%、13.3%, 一次枝梗数微弱降低,种子总磷含量无明显变化。与野生型相比,超表达材料3个株系Ox1、Ox2、Ox3种子中总磷含量分别增加了22.2%、18.3%、21.6%,穗长和一次枝梗数无明显变化。此外,与野生型相比,超表达材料3个株系Ox1、Ox2、Ox3的单株产量分别增加了15.8%、12.1%、7.7% 。可见,成熟期突变体有株高、穗长、一次枝梗数降低的趋势,而超表达种子中磷含量和单株产量增加。推测OsPHR3超表达材料可能通过增加种子中磷含量,影响水稻单株产量。
3讨论
拟南芥中AtPHR1的发现是植物磷信号调控研究的重要节点,PHR1-miR399-PHO2调控模块逐渐被验证[15,16],并且以AtPHR1为中心的磷信号调控网络越来越清晰[10,11,17,18]。在水稻基因组中发现了AtPHR1的同源基因OsPHR1和OsPHR2,它们共同参与水稻中磷信号的调控,其中OsPHR2是水稻中磷信号转导途径的中心调控因子[13]。虽然水稻和拟南芥中PHR家族成员都存在着功能冗余,调节机制相似[19,20,21],但是家族成员的功能上也存在着差异[14,22,23]。 例如,拟南芥中高亲和磷转运蛋白AtPht1;1、AtPht1;4以及AtPHF1直接被AtPHR1调控[24,25],但水稻中重要的磷转运蛋白调控基因OsPHF1,却不是直接被OsPHR2调控[14,26]。水稻是单子叶植物,磷信号的调控过程十分复杂,许多基因是在转录或转录后水平发生作用,进一步通过生理和形态学上的改变来适应环境的变化[14,27-29]。
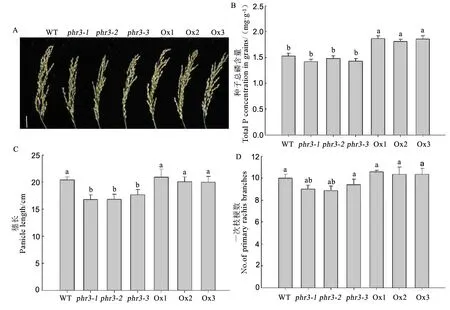
A-标尺为2.5cm。
A,Bar= 2.5cm.
图7野生型和转基因材料穗型及种子总磷含量
Fig. 7.PaniclephenotypeandtotalPconcentrationingrainsofWTandtransgenicplants.
OsPHR3是OsPHR2的同源基因,氨基酸序列同源性为45.21%。进化树分析表明, 相对于OsPHR1来说,OsPHR3与OsPHR2的亲缘关系更近一些(图1),推测两者在调控水稻磷素利用过程中的功能相近。OsPHR3与OsPHR1和OsPHR2在表达模式上不同,后两者在转录水平上不受缺磷诱导,而OsPHR3地上部和根系均受缺磷诱导(图2),这3个基因共同参与水稻下游磷相关基因的调控[13,14]。AtPHR1和OsPHR2超表达材料能够导致地上部磷积累,从而使叶片坏死和生长受到抑制,出现磷中毒现象[10,12,13,30]。正常供磷条件下,OsPHR3超表达地上部可提取磷含量明显高于野生型,同时地上部和根系总磷含量升高,但并未表现出磷中毒现象(图4)。32Pi同位素吸收和转运实验表明超表达在不同供磷条件下都能促进磷的吸收,缺磷条件下能够提高磷的转运速率(图5)。一般来说,拟南芥和水稻在缺磷条件下会出现根毛变长的现象[31-33]。最近,Guo等研究表明,与野生型相比,水稻OsPHR1、OsPHR2和OsPHR3基因的突变体根毛变短,而超表达根毛变长[14,31]。因此,水培条件下超表达株系的根毛变长,可以促进对磷的吸收与转运,从而提高植株体内磷含量。OsPHR2超表达材料桶培高磷条件下表现出地上部磷中毒,结实率和产量下降的现象[13],但Guo等磷梯度田间实验显示OsPHR3基因超表达能够提高磷的利用效率,增强其耐低磷的能力,提高水稻产量[14]。本研究正常供磷桶培至成熟期,与野生型相比突变体材料无明显变化;而超表达材料叶鞘、穗柄和种子等地上部总磷含量显著提高,有效分蘖数和单株产量也明显升高(图6,图7)。因此,OsPHR3有可能通过该基因超表达提高磷素的吸收和利用效率,从而影响水稻单株产量。
OsPHR3基因缺失后对水稻整个生长发育过程没有显著的影响,只有在成熟期水稻的株高、穗长等发生变化。可能是水稻中存在OsPHR1、OsPHR2和OsPHR3等磷调控基因且部分功能冗余[12-14,34],OsPHR3基因缺失后代替该基因发挥功能,从而弥补该基因缺失带来的影响。而OsPHR3超表达对水稻整个生长发育过程产生显著影响,苗期地上部磷积累,成熟期种子中磷含量升高。可见该基因超表达对于磷素的利用具有重要的作用。
综上所述,水稻OsPHR3基因超表达能够促进磷素的吸收与转运,提高水稻有效分蘖数以及地上部和种子中的磷含量,提高水稻磷素利用效率。对于分子育种具有重要的借鉴意义,在减少肥料的施用和保护环境等方面具有重大的经济和社会意义。
参考文献:
[1]胡培松, 翟虎渠. 中国水稻生产新特点与稻米品质改良. 中国农业科技导报, 2002, 4(4):33-39.
HuPS,ZhaiHQ.NewcharacteristicsofriceproductionandqualityimprovementinChina.Rev China Agric sci Techhol,2002,4(4):33-39.(inChinesewithEnglishabstract).
[2]彭少兵,黄见良,钟旭华,等.提高中国稻田氮肥利用率的研究策略.中国农业科学, 2002, 35(9):1095-1103.
PengSB,HuangJL,ZhangXH,etal.Researchstrategyinimprovingfertilizer-nitrogenuseefficiencyofirrigatedriceinChina,Sci Agric Sin, 2002,35(9):1095-1103.(inChinesewithEnglishabstract).
[3]VanceCP,Uhde-StoneC,AllanDL.Phosphorusacquisitionanduse:Criticaladaptationsbyplantsforsecuringanonrenewableresource. New Phytol, 2003, 157: 423-447.
[4]AbelS,TicconiCA,DelatorreCA.Phosphatesensinginhigherplants. Physiol Plant, 2002, 115(1): 1-8.
[5]BieleskiRL.Phosphatepools,phosphatetransport,andphosphateavailability. Ann Rev Plant Physiol, 1973, 24(1): 225-252.
[6]PéretB,ClémentM,NussaumeL,etal.Rootdevelopmentaladaptationtophosphatestarvation:Bettersafethansorry. Trends Plant Sci, 2011, 16(8): 442-450.
[7]BonfanteP,GenreA.Mechanismsunderlyingbeneficialplant-fungusinteractionsinmycorrhizalsymbiosis. Nat Commun, 2010, 1: 48.
[8]HinsingerP.BioavailabilityofsoilinorganicPintherhizosphereasaffectedbyroot-inducedchemicalchanges:Areview. Plant Soil, 2001, 237(2): 173-195.
[9]López-ArredondoDL,Herrera-EstrellaL.Engineeringphosphorusmetabolisminplantstoproduceadualfertilizationandweedcontrolsystem. Nat Biotechnol, 2012, 30(9): 889-893.
[10]RubioV,LinharesF,SolanoR,etal.AconservedMYBtranscriptionfactorinvolvedinphosphatestarvationsignalingbothinvascularplantsandinunicellularalgae. Genes & Devel, 2001, 15: 2122-2133.
[11]BustosR,CastrilloG,LinharesF,etal.AcentralregulatorysystemlargelycontrolstranscriptionalactivationandrepressionresponsestophosphatestarvationinArabidopsis. PLoS Genet, 2010, 6(9):e1001102.
[12]WuP,WangXM.RoleofOsPHR2onphosphorushomeostasisandroothairsdevelopmentinrice.Plant Signal & Behav, 2008, 3(9): 674-675.
[13]ZhouJ,JiaoFC,WuZC,etal. OsPHR2isinvolvedinphosphate-starvationsignalingandexcessivephosphateaccumulationinshootsofplants. Plant Physiol, 2008, 146(4)1673-1686.
[14]GuoMN,RuanWY,LiCY,etal.IntegrativecomparisonoftheroleofthePHR1subfamilyinphosphatesignalingandhomeostasisinrice. Plant Physiol, 2015, 168(4):1762-76.
[15]AungK,LinSI,WuCC,etal.Pho2,aphosphateoveraccumulator,iscausedbyanonsensemutationinamicroRNA399targetgene. Plant Physiol, 2006, 141: 1000-1011.
[16]BariR,PantBD,StittM,etal.PHO2,microRNA399,andPHR1defineaphosphate-signalingpathwayinPlants. Plant Physiol, 2006, 141: 988-999.
[17]GonzálezE,SolanoR,RubioV,etal.Phosphatetransportertrafficfacilitator1isaplant-specificSEC12-relatedproteinthatenablestheendoplasmicreticulumexitofahigh-affinityphosphatetransporterinArabidopsis. Plant Cell, 2005, 17: 3500-3512.
[18]SchachtmanDP,ShinR.Nutrientsensingandsignaling:NPKS. Annu Rev Plant Biol, 2007, 58: 47-69.
[19]NilssonL,MullerR,NielsenTH.IncreasedexpressionoftheMYB-relatedtranscriptionfactor,PHR1,leadstoenhancedphosphateuptakeinArabidopsis thaliana. Plant Cell Environ, 2007, 30: 1499-1512.
[20]ChiouTJ,LinSI.Signalingnetworkinsensingphosphateavailabilityinplants. Anu Rev Plant Biol, 2011, 62:185-206.
[21]WuP,ShouHX,XuGH,etal.Improvementofphosphorusaffinityinriceonthebasisofunderstandingphosphatesignalingandhomeostasis. Curr Opin Plant Biol, 2013, 16:1-8.
[22]LiuF,WangZY,RenHY,etal.OsSPX1suppressesthefunctionofOsPHR2intheregulationofexpressionofOsPT2andphosphatehomeostasisinshootsofrice. Plant J, 2010, 62: 508-517.
[23]BayleV,ArrighiJF,CreffA,etal. Arabidopsis thalianahigh-affinityphosphatetransportersexhibitmultiplelevelsofposttranslationalregulation. Plant Cell, 2011, 23:1523.
[24]ShinH,ShinHS,DewbreGR,HarrisonMJ.PhosphatetransportinArabidopsis:Pht1;1andPht1;4playamajorroleinphosphateacquisitionfrombothlow-andhigh-phosphateenvironments. Plant J, 2004, 39: 629-642.
[25]KhanGA,BouraineS,WegeS,etal.CoordinationbetweenzincandphosphatehomeostasisinvolvesthetranscriptionfactorPHR1,thephosphateexporterPHO1,anditshomologuePHO1;H3inArabidopsis. J Exp Bot, 2014, 65: 871-884.
[26]ChenJY,LiuY,NiJ,etal.OsPHF1regulatestheplasmamembranelocalizationoflow-andhigh-affinityinorganicphosphatetransportersanddeterminesinorganicphosphateuptakeandtranslocationinRice. Plant Physiol, 2011, 157: 269-278.
[27]BangXJ,Finnegan.Regulationofphosphatestarvationresponsesinhigherplants. Ann Bot (Lond) PM, 2010, 105: 513-526.
[28]Franco-ZorrillaJM,GonzálezE,BustosR,etal.Thetranscriptionalcontrolofplantresponsestophosphatelimitation. J Exp Bot, 2004, 55: 285-293.
[29]YangXJ.FinneganRegulationofphosphatestarvationresponsesinhigherplants. Ann Bot (Lond) PM, 2010, 105: 513-526.
[30]RenF,GuoQQ,ChangLL,etal.BrassicanapusPHR1geneencodingaMYB-likeproteinfunctionsinresponsetophosphatestarvation. PLoS ONE, 2012, 7:e44005.
[31]WuP,XuJI.DoesOsPHR2,centralPi-signalingregulator,regulatesomeunknownfactorscrucialforplantgrowth? Plant Signal & Beh, 2010, 5:6, 712-714.
[32]WissuwaM.Howdoplantsachievetolerancetophosphorusdeficiency?Smallcauseswithbigeffects. Plant Physiol, 2003, 133:1947-1958.
[33]YiKK,WuZC,ZhouJ,etal.OsPTF1,anoveltranscriptionfactorinvolvedintolerancetophosphatestarvationinrice. Plant Physiol, 2005, 138:2087-2096.
[34]WangZY,RuanWY,ShiJ,etal.RiceSPX1andSPX2inhibitphosphatestarvationresponsesthroughinteractingwithPHR2inaphosphate-dependentmanner. Proc Natl Acad Sci, 2014, 11: 14953-14958.
收稿日期:2016-01-22; 修改稿收到日期: 2016-02-04。
基金项目:国家自然科学基金资助项目(31172014);江苏省自然科学基金资助项目(BK20141367);转基因生物新品种培育科技重大专项(2014ZX0800931B; 2016ZX08009-003-005)。
中图分类号:Q755; Q945.1
文献标识码:A
文章编号:1001-7216(2016)04-0397-09
RolesofTranscriptionFactorGeneOsPHR3ontheUtilizationofPhosphateinRice
ZHANGLiang,SUNWen-xian,SUNYa-fei,PEIWen-xia,LUOWen-zhen,SUNRui,ZHANGZhan-tian,XUGuo-hua,SUNShu-bin*
(College of Resources and Environmental Science, Nanjing Agricultural University, Nanjing 210095, China;*Correspondingauthor,E-mail:sunshubin@njau.edu.cn)
Abstract:Phosphorus, an essential macronutrient element for plant growth and development, is broadly participating in various plant life activities. However, low availability of soil phosphate is a major constraint for crop production in many agricultural systems. OsPHR3(LOC_Os02g04640)is a homolog of OsPHR2, the central regulator of Pi-signaling in rice. Both OsPHR2and OsPHR3 belong to the MYB-CC family members and have functional redundancy in part. In this study, OsPHR3 knock-out and over-expression transgenic plants were obtained. The hydroponic culture,32Pi isotopic and pot experiments were used to demonstrate the role of OsPHR3 in rice phosphate untilization. First, hydroponic experiments showed that over-expression of OsPHR3increased the phosphorus content of the rice, although the knock-out lines had no significant difference with wild type plants. Second,32Pi isotopic experiments revealed that over-expression of the gene promoted the absorption and distribution of phosphate, while the absorption rate in knock-out lines decreased in comparison with wild type plants under Pi-deficient condition. Third, pot experiments demonstrated that over-expression of OsPHR3 enhanced the effective tillers and phosphorus content in seed. However, the panicle length in knock-out lines is shorter than in wild type plants. In conclusion, OsPHR3 is a potential transcription factor for improving P efficiency in rice through molecular breeding.
Key words:rice; phosphate; OsPHR3
