牦牛轮状病毒VP6基因序列分析及RT-PCR检测方法的建立与应用
2016-08-09周芳,岳华,张斌,李凡,陈曦,汤承
周 芳,岳 华,张 斌,李 凡,陈 曦,汤 承
(西南民族大学生命科学与技术学院/四川省教育厅重大动物疫病防控技术创新团队,成都 610041)
牦牛轮状病毒VP6基因序列分析及RT-PCR检测方法的建立与应用
周芳,岳华,张斌,李凡,陈曦,汤承*
(西南民族大学生命科学与技术学院/四川省教育厅重大动物疫病防控技术创新团队,成都 610041)
摘要:轮状病毒(rotavirus,RV)VP6基因是RV的主要分子检测靶点,但该基因的点突变会影响RV的分子检测。本试验的目的是在研究牦牛RV VP6基因遗传变异的基础上,建立检测牦牛RV的RT-PCR方法并应用于临床样本检测。采用Prime 5.0设计引物,从30个牦牛腹泻样本中扩增得到14个位于931—1 338 bp牦牛RV VP6基因片段。序列分析结果发现,与牛轮状病毒(bovine rotavirus,BRV)相比,牦牛RV VP6基因在931—1 110 bp间出现多处点突变,而在1 100—1 338 bp之间序列高度保守。据此设计检测引物,成功建立了检测牦牛RV的RT-PCR方法,具有良好的特异性和稳定性,只扩增出牦牛RV及BRV的特异片段,对其他无关病原无扩增;检测下限为0.45 pg·μL-1,灵敏性较好。所建立方法对牦牛RV的检出率明显优于现有检测BRV的RT-PCR方法,为牦牛RV病的诊断和流行病学调查提供了可靠的方法;对BRV的检出也与其他方法具有很好的符合率,也可以用于BRV的检测。对234份犊牦牛腹泻粪便样本的RV检出率:西藏为60.00%(36/60)、青海为95.00%(57/60);四川为85.19%(46/54);云南为90.00%(54/60),证明牦牛RV感染是当前导致犊牦牛腹泻的重要原因。
关键词:牦牛;轮状病毒;VP6基因;点突变;RT-PCR
国内外研究表明,牛轮状病毒(bovine rotavirus,BRV)是引起犊牛腹泻的主要病因,世界范围内流行,给全球养牛业带来巨大的经济损失[1-2]。BRV为呼肠孤病毒科轮状病毒属,无囊膜双链RNA病毒,分11个基因节段,由三层衣壳组成,分别编码6种结构蛋白(VP1~VP4、VP6、VP7)和6种非结构蛋白(NSP1~NSP6),其中VP1~VP3为病毒核心蛋白;VP6为内衣壳蛋白;VP4和VP7蛋白决定病毒的毒力和P型与G型[3]。VP6作为病毒分组的特异性抗原,具有较强的免疫原性,是RV感染血清抗体检测的重要候选抗原;因其基因序列在各毒株间高度保守,目前VP6基因已成为RV的主要分子检测靶点[4-6]。
PCR方法是病原检测和流行病学调查的主要方法。目前国内外已建立了多种检测BRV的RT-PCR方法,靶基因的选择多是VP6[7-9]。研究表明:尽管VP6基因高度保守,但可以快速发生点突变而引起抗原漂移[10]。故应加强对VP6基因实时监测,了解其基因序列的变异状况,以便及时改进PCR方法,提高检测效率[11]。最近,作者实验室利用二代测序技术从牦牛腹泻粪便样本鉴定病毒种类时,证实样本中存在RV[12],但采用国内外文献报道的三种检测牛RV的RT-PCR方法对该份牦牛RV阳性样本的检测结果却为阴性,推测其原因可能是牦牛RVVP6基因序列发生了变异。本研究旨在分析牦牛RVVP6基因分子特征并基于此建立RT-PCR检测方法,并应用该方法对青藏高原腹泻牦牛粪便样本进行流行病学调查。
1材料与方法
1.1病毒(菌)株、临床样本
牦牛RV分离株,牛轮状病毒标准毒株(NCDV1,由本实验室保存);用于特异性验证的病毒(菌)株及核酸样本:牦牛源星状病毒(bovine astrovirus,BAstV)Swun0201、牦牛源牛病毒性腹泻-黏膜病病毒(bovine viral diarrhea virus,BVDV)swun0603、牦牛源肠炎病毒(bovine enterovirus,BEV)swun0510;牦牛源K99大肠杆菌swun4025、牦牛源都柏林沙门菌swun3736、牦牛源产气荚膜梭菌swun2930、牦牛源空肠弯曲杆菌swun1639;牦牛源冠状病毒(bovine coronavirus,BCV)、牦牛源细小病毒、牦牛源kobu病毒核酸样本以及牦牛源艾美尔球虫、牦牛源瑞氏隐孢子虫的DNA样本由本实验室保存。
检测样本:50份牦犊牛腹泻粪便样本(2014年6月采自于四川省阿坝州红原县)、38份肉牛腹泻粪便样本(2015年9月采集自重庆纂江县)用于不同检测方法的比较;234份犊牦牛(≤3月龄)腹泻粪便样本分别于2015年8月采集于四川省阿坝州(n=54)、云南省香格里拉市(n=60)、青海省玉树市(n=60)、西藏藏族自治区亚东县(n=60),所有样本于-80 ℃保存备用。
1.2主要试剂及仪器
TrizolTMReagent、PrimescriptTM、pMD19-T克隆载体等购于TaKaRa公司;AXY prepTMDNA胶回收试剂盒购于Axygen公司。DNA Marker、DH5ɑ感受态细胞、质粒提取试剂盒购于宝生物工程(大连)股份有限公司;凝胶成像系统Doc2000(Bio-Rad公司,美国);紫外分光光度计Cary50Probe(Vatian公司,美国);高速冷冻离心机(Eppendorf公司,德国)。
1.3引物的设计与合成
根据GenBank收录的牛A群轮状病毒的VP6基因序列(登录号为JF693031)及本实验室宏病毒基因组测序结果(938 bp-ATTTATATTTCATGCTACAGTTGGACTCACACTACGAATTGAATC-TGCAGTTTGTGAATCTGTGCTTGCGGACGC-TTCCGAAACTTTATT-1 029 bp),利用Primer5.0引物设计软件,设计扩增引物,从30份牦牛腹泻样本中扩增长度为407 bp位于931—1 338 bp位点的RVVP6基因片段,用于遗传演化分析并筛选分子检测靶点,引物序列如下,F:5′-GGTAGCGGCGTTATTTCC-3′、R:5′-CGCCATCTGAGTG-ATTACTC-3′。
特异性检测引物的设计:基于上述筛选出的分子检测靶点,设计特异性检测引物,扩增长度为231 bp位于牦牛RVVP6基因1 100—1 338 bp位点的目的片段,引物信息见表1。以上引物均由大连宝生物工程有限公司合成。
1.4RNA的提取与cDNA合成
将临床粪便样本与PBS(1∶5)充分重悬混匀,-80 ℃冰箱中反复冻融3次,3 000 r·min-1离心10 min,弃沉淀,再以12 000 r·min-1离心30 min,取上清,然后按照Trizol Reagent说明书提取总RNA,并按照反转录试剂盒说明书合成cDNA,-20 ℃保存备用。细菌DNA采用酚-氯仿法提取,-20 ℃保存备用。
1.5牦牛RV阳性标准品的制备
以牦牛RV cDNA为模板,加入检测引物扩增大小为231 bp的牦牛RVVP6基因片段,PCR产物经2.0%琼脂糖凝胶电泳鉴定,用胶回收试剂盒回收目的片段,并将其克隆至pMD19-T载体,并转化大肠杆菌DH5α感受态细胞,筛选出阳性克隆接入含氨苄青霉素的LB液体培养基中,37 ℃培养8 h,用质粒提取试剂盒提取重组质粒,送擎科生物有限公司测序。测序正确的阳性质粒作为RV阳性标准品,核酸蛋白质检测仪测定其浓度。
1.6反应体系及条件的优化
采用25 μL体系(预混酶12.5 μL,牦牛RV阳性标准品1.5 μL,上、下游引物各1 μL,ddH2O补足至25 μL),对退火温度从50~60 ℃进行优化,再以优化的退火温度对浓度为10 μmol·μL-1的引物用量(0.5~1.5 μL)进行优化。
1.7敏感性测定
将牦牛RV阳性标准品10倍递增稀释后作为模板(1×100、1×10-1~1×10-6),用建立的RT-PCR进行检测,确定其检测限。
1.8特异性评价
用建立的RT-PCR对“1.1”中待检病原的核酸样本进行检测,并设立标准阳性样本和阴性对照,以评价该方法的特异性。
1.9稳定性评价
用建立的RT-PCR对3个阳性模板及3份阴性样本进行3次重复检测,以评价方法的稳定性。
1.10四种RT-PCR方法的比较
采用所建立的RT-PCR方法和文献报道的3种检测BRV的RT-PCR方法(表1),同时对50份牦牛腹泻粪便样本进行检测,比较这4种方法的检出率。引用的3种方法完全按照文献报道的最适反应条件进行。在此基础上又选取上述三种方法中检出率最高的方法和本实验室建立RT-PCR方法同时对38份肉牛腹泻样本进行检测,比较对BRV的检出率。每个方法中检出的阳性样本,对其PCR产物进行测序。
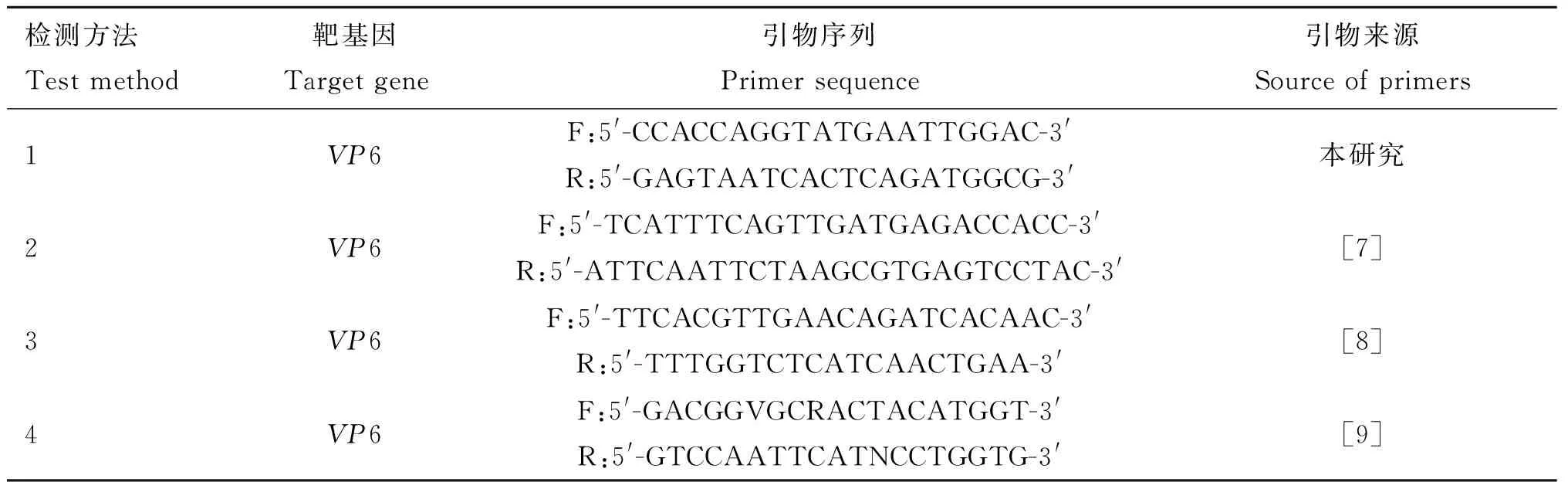
表1 四种RT-PCR方法的引物信息
1.11流行病学调查
流行病学调查样本为2015年8月四川省阿坝州地区4个牧场、云南香格里拉地区6个牧场、青海玉树地区6个牧场、西藏远东地区8个牧场,共计234份犊牦牛(≤3月龄)腹泻粪便样本,见表2。采用本研究建立好的RT-PCR方法对234份牦牛腹泻样本进行检测,分析牦牛RV在青藏高原的流行情况,并且每个地区随机选取4份检出的阳性样本的PCR产物进行测序,获得的VP6基因片段用于遗传演化分析。
2结果
2.1牦牛RVVP6基因片段序列的克隆及系统发育分析
从30份腹泻粪便样本中扩增出14个407 bp的牦牛RVVP6基因片段,相似性比对发现:14个VP6基因片段间的相似性为94.1%~100%,与GenBank中的BRV相似性为93.4%~97.8%,与其相似性最高的是南非BRV HM988974毒株;与国内仅有的一株BRV GU384194相似性为93.4%~96.3%。
系统发育分析表明:这14株牦牛RV单独的聚为3支(图1),第Ⅰ小支包含5株,与中国牛源RV(GU384194)遗传演化关系最近,共同聚为一大支;第Ⅱ小支包含3株,与美国恒河猴源RV (FJ422136)遗传演化关系最近,共同聚为一大支;第Ⅲ小支包含6株与比利时人源RV(EF554119)遗传演化关系最近,共同聚为一大支。
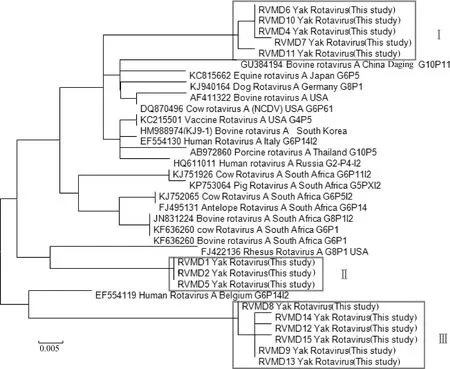
图1 牦牛RV 大小为407 bp的VP6基因片段的邻近法演化树Fig.1 Neighbor-joining consensus tree for size of 407 bp partial VP6 gene fragment of yak RV
14株牦牛RVVP6基因片段与标准毒株(NCDV株,DQ870496)序列比对发现:在931—1 100 bp之间发生的点突变位点有7~13处,在1 100—1 338 bp之间有4~6处;与国内BRV GU384194毒株的比对结果是:在931—1 100 bp之间发生的点突变位点有11~19处,在1 100—1 338 bp之间有4~8处;14个牦牛RVVP6基因序列之间比较,Ⅰ组共同的变异位点有11处,其中6处位于931—1 100 bp,5处位于1 100—1 338 bp。Ⅱ组共同的变异位点有7处,其中6处位于931—1 100 bp,1处位于1 100—1 338 bp;Ⅲ组共同的变异位点有11处,其中9处位于931—1 100 bp,2处位于1 100—1 338 bp。
2.2牦牛RV检测引物的特异性
采用自行设计的检测引物对牦牛RV核酸阳性样本进行PCR扩增,获得的目的片段大小与预期结果一致(图2 A)。测序结果证明目的片段长度为231 bp,为牦牛RV的特异性序列,与牦牛RVVP6基因序列的相似性为100%。
2.3优化的RT-PCR反应体系及条件
优化的反应体系:预混酶12.5 μL,上、下游引物(10 μmol·L-1)各1 μL,cDNA 1.5 μL,ddH2O补足25 μL。反应条件:94 ℃预变性5 min;94 ℃变性30 s;52 ℃退火30 s;72 ℃延伸30 s,共35个循环;72 ℃延伸10 min;16 ℃反应结束。
2.4敏感性
牦牛RV阳性标准品的浓度为45.2 ng·μL-1,敏感性检测结果显示,1×10-5稀释度阳性标准品仍可见目的条带,对RV的核酸最低检测限为0.45 pg·μL-1(图3)。

M.DNA相对分子质量标准MarkerⅠ;+.阳性样品;-.阴性对照;1~16.阳性模板、阴性对照、牦牛轮状病毒、牛轮状病毒BRV、牛星状病毒AstV、牛病毒性腹泻病毒BVDV、牛冠状病毒BCV、牛肠炎病毒BEV、细小病毒parvovirus、Kobu病毒、K99大肠杆菌、都柏林沙门氏菌、产气荚膜梭菌、空肠弯曲杆菌、艾美尔球虫、瑞氏隐孢子虫M.DNA marker Ⅰ;+:DNA sample of Yak RV;-.Negative control;1-16.DNA templete of Yak RV,Negative control,Yak Rotavirus,BRV,AstV,BVDV,BCV,BEV,PV,Kobu virus,E.coli K99,S.Dublin,C.perfringens,CJE,Eimeria,Swiss cryptosporidium图2 引物和方法的特异性检测结果Fig.2 The specificity of the primers and method
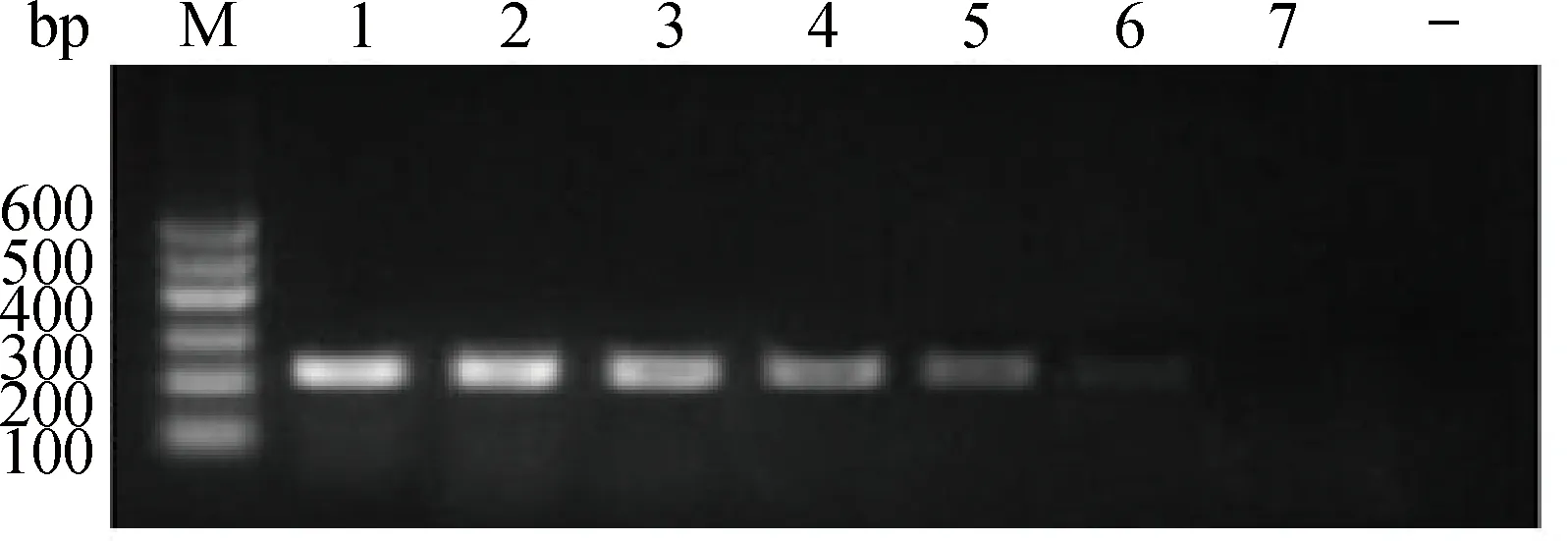
M.DNA相对分子质量标准MarkerⅠ;1~7.阳性模板10倍连续稀释样本(1×100、1×10-1~1×10-6);—.阴性对照M.DNA marker Ⅰ;1-7.10-fold serial dilution of standard temper(1×100,1×10-1-1×10-6);—.Negative control图3 敏感性检测结果Fig.3 Sensitivity test of the RT-PCR
2.5特异性
该方法能从牦牛RV分离株及BRV标准毒株中检出目的基因片段,对AstV、BVDV、BCV、BEV、细小病毒、Kobu病毒、K99大肠杆菌、都柏林沙门菌、产气荚膜梭菌、空肠弯曲杆菌以及艾美尔球虫、瑞氏隐孢子虫的核酸样本无扩增(图2B)。可见本研究建立的RT-PCR方法不仅能适合牦牛RV的检测,也适用于BRV的检测。
2.6稳定性
对同一模板的3次重复检测结果一致,证明该方法具有良好的稳定性和重复性。
2.7四种检测BRV方法的比较
检测方法1~4对50份牦牛腹泻粪便样本中牦牛RV阳性的检出率分别为90.0%、0、0、12.0%,且方法1检出的阳性样本包含了方法4检测的所有阳性样本。可见建立的RT-PCR方法对牦牛RV的检测效率明显优于引用的3种检测BRV的方法。方法1和方法4均检出的阳性样本以及另外随机挑选的10个方法1中检出的阳性样本的PCR产物进行测序,结果显示扩增出的基因片段为轮状病毒VP6基因片段,证实该RT-PCR方法检测结果的准确性。
同时使用检测方法1和方法4对38份肉牛腹泻粪便样本检测,结果显示两种方法检出BRV的阳性符合率为100%,证实了建立的RT-PCR方法不仅适用于牦牛RV的检测,也可以适用于BRV的检测。
2.8牦牛轮状病毒感染的流行病学调查
利用本研究建立的牦牛轮状病毒RT-PCR检测方法对青藏高原牦牛腹泻粪便样本进行轮状病毒的检测,结果(表2)显示:青海地区样本的检出率最高,为95.00%(57/60);四川地区样本的检出率为85.19%(46/54);云南地区样本的检出率为90.00%(54/60);西藏地区样本的检出率最低,为60.00%(36/60),这一结果表明,青藏高原牦牛RV感染率很高。对随机挑选的16份阳性样本的PCR产物测序结果进行分析,证实均为牦牛RV特异片段,同时也进一步验证本研究建立的RT-PCR方法检测结果的准确性。
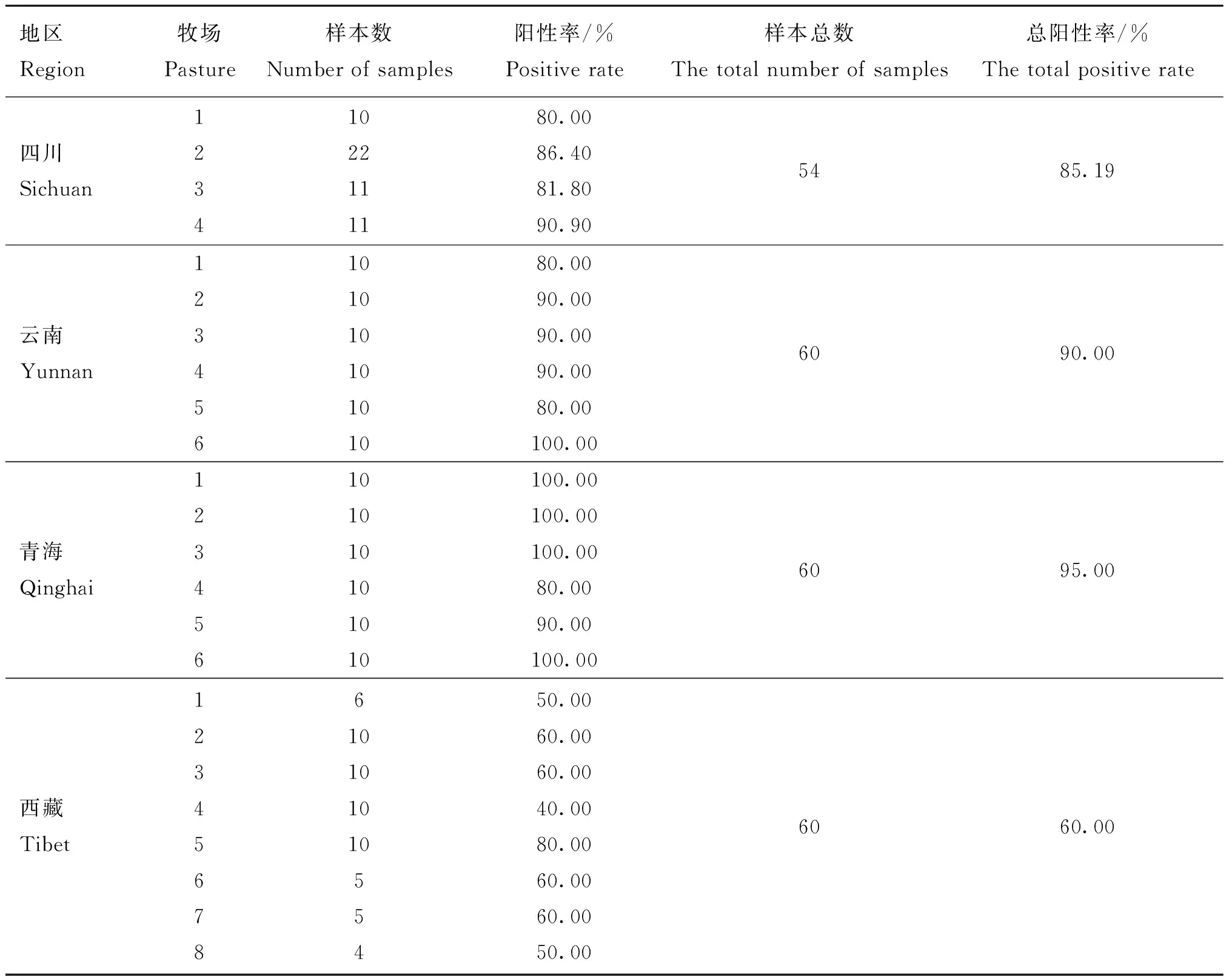
表2 牦牛轮状病毒感染的流行病学调查结果
2.9青藏高原四个地区的牦牛轮状病毒VP6基因片段的系统发育分析
系统发育分析(图4)表明:四个地区牦牛轮状病毒单独的聚在一起,与牛轮状病毒有明显的遗传距离,表明VP6的序列的变异与宿主有关。从四个地区的毒株分布来看,青海、西藏、云南的毒株分别单独聚在一起,四川的毒株分布在西藏和云南的毒株形成的分支中,似乎有一定的地域分布的趋势,但由于样本数量有限,还需要更多不同地域的毒株来验证。
3讨论
由于VP6基因序列的保守性,目前已成为国内外检测RV最重要的分子检测靶点。但2007年T.K.Kerin等在研究美国人源RV时发现:VP6基因序列由于发生点突变、碱基插入等变异能使其发生快速的变异;新分离到的人源RVVP6基因序列与数十年前分离到的参考毒株发生了很大的遗传变异,22株RV中只有3株与标准参考毒株遗传演化关系较近,共同聚为一支,其余占近86.4%的毒株与参考毒株发生了遗传偏倚,单独聚成支;这种基因序列的变异导致2%~3%的氨基酸残基被替换,同时也影响了PCR方法的检测结果[11]。目前国内对轮状病毒VP6遗传变异的研究资料很少。从研究扩增出的14个VP6基因片段序列比对分析结果来看,牦牛RVVP6基因序列在遗传演化的过程中也发生了较多点突变,主要发生区域为931—1 100 bp。这些点突变频率高的位置是其他毒株的高保守区域,也是国内外建立检测RV RT-PCR方法的常用引物设计位点[13-15]。因此,这种点突变对检测方法造成的影响值得我们思考。鉴于VP6基因既是主要的分子检测靶点,又是制备亚单位疫苗的重要候选抗原[16-17],进一步研究牦牛RVVP6基因的遗传演化的特点和对抗原特性影响,这对牦牛轮状病毒病的分子诊断和亚单位疫苗的研制有重要意义。
本研究基于对牦牛RVVP6基因序列的分析,选择在最保守区域设计检测引物,成功建立检测牦牛RV的RT-PCR 方法。该方法特异性好,只能扩增牦牛RV和BRVVP6基因片段,对一些常见的引起犊牛腹泻的其他病毒、细菌和寄生虫等病原无扩增,且具有良好的重复性;从灵敏度上来看,该方法的检测限为0.45 pg·μL-1,与方法4中检测最低检测限处于同一个数量级[9],具有较好的灵敏性,完全能够满足临床样本的检测需求;同时,本方法对BRV的检出也与国外报道的方法有很好的符合率。因此,本研究建立的RT-PCR方法不仅为牦牛RV的检测和流行病学调查提供有力的工具,同时也可以检测BRV,具有良好的通用性。
比较试验表明,其他三种检测BRV的RT-PCR方法对临床样本牦牛RV检出率显著低于本研究建立的RT-PCR方法,分析其原因可能与牦牛RVVP6基因核苷酸序列变异有关。通过把这3种方法中引物序列与作者扩增出来的牦牛RVVP6基因序列比对发现:方法2中[7]下游引物结合位点发生了3处点突变:997 bp处A→G;1 000 bp处C→T;1 003 bp处C→T。方法3中[8]下游引物结合位点发生了7处点突变:916 bp处T→G;918 bp处A→T;920 bp处A→T;921 bp处A→C;922 bp处T→A;926 bp处T→A;928 bp处A→T。方法4虽然为解决点突变问题设计了简并引物[9],但这对简并引物在下游引物结合位点也有一处发生了变异,可能是导致对牦牛RV的阳性检出率低的一个原因。这一结果也表明了VP6基因序列尽管保守,但也有突变的发生,我们应对VP6进行实时监测,不断地改进检测方法。
犊牦牛腹泻一直是危害青藏高原地区牦牛养殖业的主要疾病[12]。国内外流行病学调查表明其病因是多方面的,归纳为两大类:一类为非感染性因素,一类为感染性因素[18-19]。本实验室从牦犊牛腹泻粪便中检测到的病毒多达9种[12]。本研究对青藏高原牦牛主产区的犊牛腹泻样本进行流行病学调查结果表明,牦牛RV感染在青藏高原普遍存在,是当前导致犊牛腹泻的重要病原,为牦牛腹泻病的防治提供了科学依据。有资料显示从人腹泻粪便中分离到BRV,BRV可以直接感染人引发疾病[20-21]。本研究建立的遗传演化树中第Ⅲ支与人源RV有较近的遗传演化关系(图1),故牦牛RV的公共卫生学意义值得关注。
4结论
成功建立检测牦牛轮状病毒的RT-PCR检测方法,具有良好的特异性和稳定性,灵敏性也较好。建立的方法对牦牛RV的检出率明显优于现有检测BRV的RT-PCR方法,为牦牛RV病的诊断和流行病学调查提供了可靠的方法;对BRV的检出也与其他方法具有很好的符合率,也可以用于BRV的检测。对青藏高原牦牛轮状病感染的病原进行流行病学调查表明牦牛RV感染是当前导致犊牦牛腹泻的重要原因。
参考文献(References):
[1]BADARACCO A,GARAICOECHEA L,RODRGUEZ D,et al.Bovine rotavirus strains circulating in beef and dairy herds in Argentina from 2004 to 2010[J].VetMicrobiol,2012,158(3-4):394-399.
[2]MINAMI-FUKUDA F,NAGAI M,TAKAI H,et al.Detection of bovine group A rotavirus using rapid antigen detection kits,RT-PCR and next-generation DNA sequencing[J].JVetMedSci,2013,75(12):1651-1655.
[3]ALFIERI A F,ALFIERI A A,BARREIROS M A,et al.G and P genotypes of group A rotavirus strains circulating in calves in Brazil 1996-1999[J].VetMicrobiol,2004,99(3-4):167-173.
[4]GONZALEZ D D,MOZGOVOJ M V,BELLIDO D,et al.Evaluation of a bovine rotavirus VP6 vaccine efficacy in the calf model of infection and disease[J].VetImmunolImmunopathol,2010,137(1-2):155-160.
[5]WISE A G,SMEDLEY R C,KIUPEL M,et al.Detection of group C rotavirus in juvenile ferrets (Mustela putorius furo) with diarrhea by reverse transcription polymerase chain reaction:sequencing and analysis of the complete coding region of the VP6 gene[J].VetPathol,2009,46(5):985-991.
[6]STIPP D T,ALFIERI A F,LORENZETTI E,et al.VP6 gene diversity in 11 Brazilian strains of porcine group C rotavirus[J].VirusGenes,2015,50(1):142-146.
[7]范晴,谢芝勋,刘加波,等.牛轮状病毒TaqMan实时荧光RT-PCR快速检测方法的建立[J].中国畜牧兽医,2011,38(4):105-108.
FAN Q,XIE Z X,LIU J B,et al.Detection of bovine rotavirus by TaqMan based real-time reverse transcription polymerase chain reaction assay[J].ChinaAnimalHusbandry&VeterinaryMedicine,2011,38(4):105-108.(in Chinese)
[8]王洁清.牛轮状病毒RT-PCR检测和基于VP6重组抗原检测血清抗体ELISA方法初步建立[D].哈尔滨:东北农业大学,2009.
WANG J Q.Detection of bovine rotavirus by RT-PCR and establishment of indirect ELISA to detect bovine rotavirus antibody with VP6 protein[D].Harbin:Northeast Agricultural University,2009.(in Chinese)
[9]BASERA S S,SINGH R,VAID N,et al.Detection of rotavirus infection in bovine calves by RNA-PAGE and RT-PCR[J].IndianJVirol,2010,21(2):144-147.
[10]NYAGA M M,ESONA M D,JERE K C,et al.Genetic diversity of rotavirus genome segment 6(encoding VP6) in Pretoria,South Africa[J].Springerplus,2014,3:179.
[11]KERIN T K,KANE E M,GLASS R I,et al.Characterization of VP6 genes from rotavirus strains collected in the United States from 1996-2002[J].VirusGenes,2007,35(3):489-495.
[12]CHEN X,ZHANG B,YUE H,et al.A novel astrovirus species in the gut of yaks with diarrhea in the Qinghai Tibetan plateau,2013[J].JGenVirol,2015.doi:10.1099/gv.0.000303.
[14]GAUTAM R,ESONA M D,MIJATOVIC-RUSTEMPASIC S,et al.Real-time RT-PCR assays to differentiate wild-type group A rotavirus strains from Rotarix®and Rota Teq®vaccine strains in stool samples[J].HumVaccinImmunother,2014,10(3):767-777.
[15]LIN Y P,KAO C L,CHANG S Y,et al.Determination of human rotavirus VP6 genogroups Ⅰ and Ⅱ by reverse transcription-PCR[J].JClinMicrobiol,2008,46(10):3330-3337.
[16]DENNEHY P H.Rotavirus vaccines:an overview[J].ClinMicrobiolRev,2008,21(1):198-208.
[17]LAPPALAINEN S,PASTOR A R,MALM M,et al.Protection against live rotavirus chanllenge in mice induced by parenteral and mucosal delivery of VP6 subunit rotavirus vaccine[J].ArchVirol,2015,160(8):2075-2078.
[18]MATTHIJNSSENS J,TARAPOREWALA Z F,YANG H,et al.Simian rotaviruses possess divergent gene constellations that originated from interspecies transmission and reassortment[J].JVirol,2010,84(4):2013-2026.
[19]MATTHIJNSSENS J,CIARLET M,HEIMAN E,et al.Full genome-based classification of rotaviruses reveals a common origin between human Wa-Like and procine rotavirus strains and human DS-1-like and bovine rotavirus strains[J].JVirol,2008,82(7):3204-3219.
[20]DURMAZ R,KALAYCIOGLU A T,ACAR S,et al.Prevalence of rotavirus genotypes in children younger than 5 years of age before the introduction of a universal rotavirus vaccination program:report of rotavirus surveillance in Turkey[J].PLoSOne,2014,9(12):e113674.
[21]DOAN Y H,NAKAGOMI T,ABOUDY Y,et al.Identification by full-genome analysis of a bovine rotavirus transmitted directly to and causing diarrhea in a human child[J].JClinMicrobiol,2013,51(1):182-189.
(编辑白永平)
doi:10.11843/j.issn.0366-6964.2016.07.021
收稿日期:2016-01-21
基金项目:“十二五”科技支撑计划项目(2012BAD13B06);四川省科技计划项目青年基金(2014JQ0044)
作者简介:周芳(1990-),女,黑龙江德都人,硕士生,主要从事动物病原分子生物学研究,E-mail:1103306587@qq.com *通信作者:汤承,博士,教授,Tel/Fax:+86-28-85528276,E-mail:tangcheng101@163.com
中图分类号:S852.653;S852.659.3
文献标志码:A
文章编号:0366-6964(2016)07-1465-09
Establishment and Application of an RT-PCR Assay for Yak Rotavirus Based on the Sequence Analysis of Yak RotavirusVP6 gene
ZHOU Fang,YUE Hua,ZHANG Bin,LI Fan,CHEN Xi,TANG Cheng*
(CollegeofLifeScienceandTechnology,SouthwestUniversityforNationalities/InnovativeResearchTeamonAnimalEpidemicPreventionandControlofEducationDepartmentofSichuan,Chengdu610041,China)
Abstract:Rotavirus (RV) VP6 gene is the main target gene of molecular detection,but the gene point mutations affects the molecular detection of RV.The purpose of this experiment was to establish a detection method of yak RV by RT-PCR method which applied in clinical samples based on the study of genetic variation yak RV VP6.The fragments of yak RV VP6 ranged in 931-1 338 bp were obtained with the primers designed by Prime 5.0 software from 30 samples of yak diarrhea.Sequence analysis showed that,yak RV VP6 genes represented multiple point mutations in the section of 931-1110 bp compared with bovine rotavirus (BRV),while the section between 1 100-1 338bp was highly conserved.According to this result,the assay has successfully established a method with good specificity and stability.The primer could amplify the specific fragment of yak RV and BRV,with no amplification of other unrelated pathogens.The detection limit of viral nucleic acid of the assays was 0.45 pg·μL-1.With a remarkable detection rate in yak RV,the RT-PCR method provided a useful tool for the diagnosis and epidemiological investigation of yak RV disease.Additionally,this method can also be used in BRV detection.The RV detection rate in 234 samples of yak diarrhea from different provinces were 60.00% in Tibet (36/60),95.00% in Qinghai (57/60),85.19% in Sichuan(46/54) and 90.00% in Yunnan (54/60),respectively.The result shows that RV infection is an important cause of yak diarrhea.
Key words:yak;rotavirus;VP6 gene;point mutation;RT-PCR
