如何工程化生物学
2016-07-08肖文海周嗣杰王颖元英进
肖文海,周嗣杰,王颖,元英进
(1天津大学系统生物工程教育部重点实验室,天津 300072;2天津化学化工协同创新中心合成生物学平台,天津 300072)
如何工程化生物学
肖文海1,2,周嗣杰1,2,王颖1,2,元英进1,2
(1天津大学系统生物工程教育部重点实验室,天津 300072;2天津化学化工协同创新中心合成生物学平台,天津 300072)
摘要:随着化学学科的发展和应用需求,化学工程应运而生;随着生物学的发展和工程化需求,代表着“生物学第三次革命”的合成生物学也随之诞生。合成生物学,即生物学的工程化,它从工程学角度设计创建元件、器件或模块,以及通过这些元器件改造和优化现有自然生物体系,但是如何对复杂生命进行工程化一直是合成生物学工作者不断探索的重大科学问题。本文系统地阐释了迄今为止工程化生物学的4个特点:①模块化和标准化;②正交性;③鲁棒性;④适配性及其对应研究进展。最后从“设计-构建-测试”循环的研究模式入手提出了今后如何进一步有效地工程化生物学。
关键词:合成生物学;模块化;正交性;鲁棒性;适配性;“设计-构建-检测”循环
第一作者:肖文海(1982—),男,研究员,研究方向为合成生物学。
联系人:元英进,教授,博士生导师。E-mail yjyuan@tju.edu.cn。
生物学研究多年来一直沿着从整体到局部,从个体到器官、组织直到各种细胞和分子的模式,各种计算生物学和实验系统生物学的方法得到了很大发展,促进了人们对微观生物分子(DNA、蛋白质、RNA、代谢物)之间的相互作用和复杂表现的更好理解。然而,即使人们对所有这些细节都研究清楚了,仍然不理解生物作为一个整体是如何协调发挥作用的,生物从最开始是如何从简单到复杂进化的。类比化学学科,随着化学学科的发展和应用需求,化学工程应运而生;随着生物学的发展和对其应用需求的增加,生物学的工程化成为必然。合成生物学,即生物学的工程化,从工程学角度设计创建元件、器件或模块,以及通过这些元器件改造和优化现有自然生物体系(图1),或者设计合成具有预定功能的全新人工生物体系,实现合成生物体系在化学品、医药、重大疾病的诊断与治疗、农业、能源、环境等领域的规模化应用,同时加深人类对生命本质的理解[1-6]。合成生物学从电气工程、化学工程、土木工程和机械工程等工程科学进行了借鉴(图1),提供一条从“局部到整体”的研究思路和方法,即“自下而上”的途径,通过构筑人工生物系统以更好地理解天然生物系统的工作原理,从单一离散的基因元器件到模块,再到整个生物的网络,对原有生物体系自然功能进行改造或使生物系统实现全新的功能,直至“人造生命”。从这一角度来说,合成生物学理论和方法的引入,将会极大地促进人们对生物学重大基本问题的研究和解析,拓展生命科学的视野。合成生物学促进了对生命密码从“读”到“写”的质变,打开了从非生命化学物质向生命物质转化的大门,将生命科学引入“多学科会聚”研究的第三范式,催生了继DNA双螺旋结构发现和基因组测序之后的“第三次生物科学革命”。世界主要发达国家纷纷制定新的生物技术战略,合成生物学已经成为各国明确争抢的科技高地。2012年美国国防部率先启动了一个1.4亿美元的“生命铸造厂”计划(Living Foundries),将细胞作为工厂制造新分子、新材料,并强调更加广泛地利用合成生物学,集中创建一个以生物学为基础的生产平台,以期创造其再工业化、领先国际的新实力[7]。2014年6月,世界经济合作与发展组织简称经合组织(OECD)发表题为“Emerging Policy Issues in Synthetic Biology”的报告,建议各国政府以创新方式促进合成生物学发展[8]。2013年美国能源部对美国国会发表合成生物学国会报告[9];2014年5月美国国防部系统介绍了其2013 —2017年科技发展计划,提出了未来重点关注的六大颠覆性基础研究领域,合成生物学是其中之一[10];2015年美国发布了《生物技术工业化:化学品先进制造路线图》,将合成生物学列为核心发展技术之一[11]。麦肯锡全球研究所发布的研究报告将合成生物学评价为未来的十二大颠覆性技术之一,预测2025年合成生物学和工业生物技术产值将达到1000亿美元左右[12]。英国商业创新技能部将合成生物学列为支撑该国未来经济成长的八大技术之一,预测2020年合成生物学产业规模将达620亿英镑[13]。
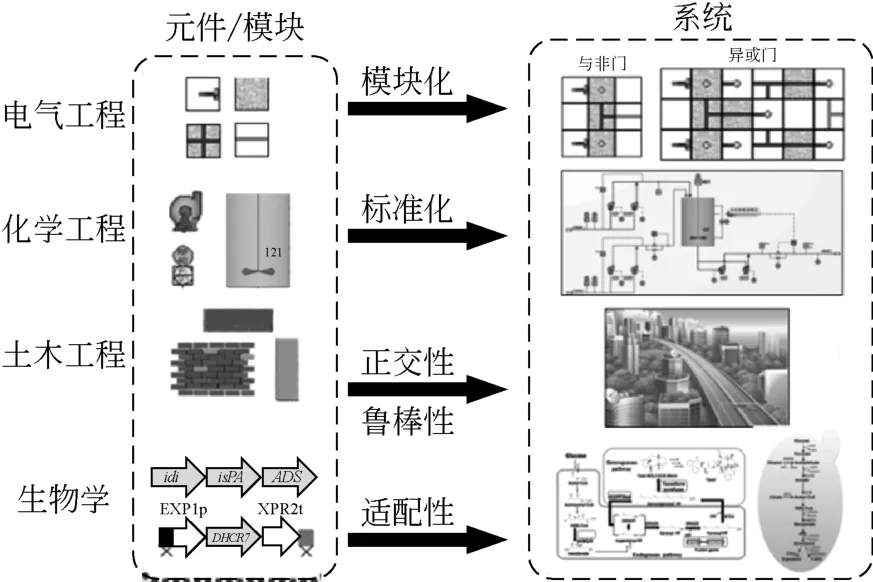
图1 生物学工程化——合成生物学
作为工程化的科学,合成生物学的发展符合我国经济转型升级的战略需求。随着我国经济的高速发展,高能耗、高污染、大规模的传统工业发展已经不适应社会发展的需求。而合成生物学正在对现代工业产生革命性的影响。人类赖以生存的化学品、燃料等制造,正面临着原料路线从化石资源向可再生生物资源转移、加工路线从化学制造向生物制造转移两个不可避免的变革。我国资源对外依存度不断加深、节能减排压力巨大。利用合成生物学设计有机化学品的高效合成路线和人工生物体系,不仅有可能高效利用原来不能利用的生物质资源,也有可能高效合成原来不能生物合成或者原来生物合成效率很低的化工产品。这将为突破自然生物体合成功能与范围的局限,打通传统化学品的生物合成通道,为发展先进生物制造技术、促进可持续经济体系形成与发展,提供重大机遇。2015年8月3日美国市场研究咨询机构透明市场研究(Transparency Market Research)公司最新研究报告显示:从2013年到2019年合成生物学技术市场将保持32.6%的年增长速率(是世界经济增长率的10倍),最终达到134亿美元的规模[14]。合成生物学将以重新“设计合成”的视角创新化工产业、医药产业、能源资源产业、环境产业等的发展,推动关键技术瓶颈的突破和新型技术的引入,重点推进我国在生物制造、生物医学、农业领域战略性新兴产业及技术转让、产业化过程的发展。
1 生物学工程化特性及研究进展
作为工程化的生物学,与其他工程科学类似,合成生物学具备4个方面的工程化特点(图1):①功能元件的模块化和标准化;②人工元器件与底盘的正交性;③人工合成系统的鲁棒性;④人工合成系统的物质代谢与能量代谢适配性。下文将对这4个方面的工程化特性及其研究进展进行阐释和说明。
1.1 功能元件的标准化
合成生物学的本质是生物学的工程化,其核心思想就是设计和构建元件模块或者改造现有的模块,然后再与底盘集成从而获得工程化人工生物体系,因此元件、模块和底盘是合成生物学的物质基础。模块的设计和构建包括元器件优化设计和模块设计组装。功能元件设计的最终目标是实现如电子工程和机械工程一样的“即插即用”。要实现“即插即用”,功能元件必须具备如下4个特点,即标准化(如具有类似的序列结构或接口)、信息化(生物学数据要详尽)、分工化(执行特定的生物学功能)、可替换(如某个元件可被强度不同的同类元件替代,实现微调),以满足实现生物功能从头设计与构建的需要。在构建合成模块时,需将不同的启动子、核糖体结合位点、终止子、功能基因、选择标签等具有特定动能的序列进行标准化、元件化[15]。需要设计和构建如下元件并建库:①基本基因元件;②基因表达的调控元件;③降解控制元件;④信号转导元件;⑤物质转运元件等。同时根据合成生物系统构建的需求来设计功能模块,除了一般的逻辑模块、代谢模块和调控模块以外,还将关注以下模块并建库:①糖转运和代谢模块;②能量(辅因子)代谢模块;③耐受性和抗毒性模块;④响应供氧模块;⑤活性结构单元合成模块;⑥产物及特殊中间代谢物转运模块;⑦纤维素降解模块;⑧光合作用固碳模块;⑨人工固氮模块;⑩疾病的特异性识别模块;疾病治疗模块;疾病治疗的调控模块;表观遗传修饰模块等。然后按照既定设计,选取特定的元器件进行拼接组装[16],进而构建合成模块。
建立开放的、通用的、可以和其他数据库兼容的元件模块库是是促进元件标准化蓬勃发展的重要途径和基石[图2(a)]。2003年成立于MIT的BioBricks元件库(2006年已经更名为BioBricks Foundation,现在是基金会形式运作,主页为http://partsregistry.org/)[17-18]是合成生物学标准化元件库历史上里程碑意义的事件[图2(a)]。随着国际基因组设计大赛(iGEM,International Genetically Engineered Machine Competition)的影响日益广泛,BioBricks元件库已经拥有超过2万个元件和模块。在美国自然基金会资助下,由加州理工大学伯克利分校和斯坦福大学于2009年合作建立的另外一个大型元件库(Biofab,International Open Facility Advancing Biotechnology,主页为http://biofab. synberc.org/),在元件收集的基础上,同时侧重于标准化元件设计构建和表征等方面的研究,并且和美国劳伦斯伯克利国家实验室、BioBricks Foundation等建立了很好的合作关系。我国在元件库建设方面,计算设计与合成表征了满足不同化学品生物合成的功能元件,形成了包含数千个功能特性清晰的功能元件和模块的实体库[图2(a)],尤其天津大学在国家重大项目支持下建立了目前世界上最大的生物元件和模块的实体库,现阶段已经包含超过一万个生物元件和模块,涵盖基础代谢、异源表达和分解代谢3个主要方面,并且已经和国内多家单位建立了良好的共享合作机制(www.synbioml.org) 。该元件模块库有着如下4个方面的优点:①标准化,具有统一的内切酶位点的元件模块化设计;②信息化,开放式元件模块库的信息管理;③分工化,以功能为单位的元件模块库分工构建管理;④可替换,高度可操纵性的模块无缝集成组装。
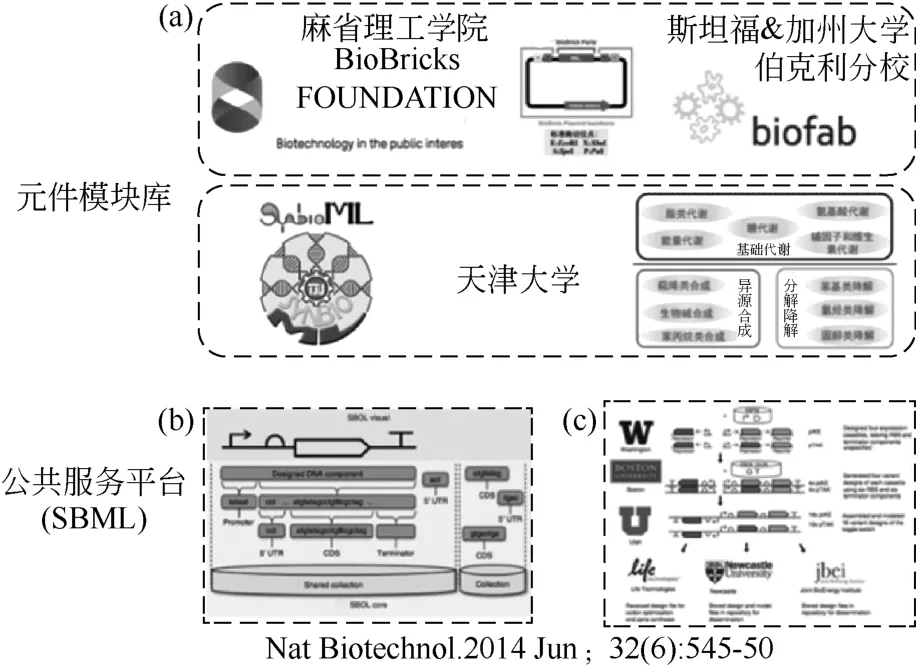
图2 元件模块标准化进展
基于合成生物学是生物学,工程学、计算机、物理等多学科交叉的特点,成立包含学术界、政府和商业化组织在内的公共交流和服务平台也是促进元件标准化和资源共享的有效途径。2008年成立的SBOL(The Synthetic Biology Open Language)给合成生物学领域的研究者提供了一个大型的国际化、标准化交流平台[19][图2(b)和(c)]。成立之初有37个组织参与,其中学术组织23个,商业组织11个,政府组织2个以及1个独立组织。该平台成立的初衷在于建立一个国际化的、跨组织的标准化、基因设计交流和分享平台,同时也能结合多学科交叉优势进行系统的基因设计。其核心数据模型如图2(b)所示。该平台集合平台中6家不同背景的单位快速的、全链条和系统的从设计、优化、合成、元件模块储藏等方面合作重构了James COLLIN 2000年发表在《Nature》杂志上的稳态开关。这也从侧面说明了建立这种标准化的公共服务和交流平台对合成生物学的巨大促进作用。该平台也从当初的1.0版本迅速的升级到了2.0版本,应用和受众更广泛[20]。
1.2 人工元器件与底盘的正交性
导入的人工元器件与底盘网络保持一定的正交性,不干扰底盘已有调控网络,可以减少人工设计的复杂性。目前可用于合成的正交生物元器件与生物模块不论在质量上还是在数量上还远远不能满足合成生物学的要求,需进一步研究不同层次(基因、RNA、蛋白等)生物元器件的正交设计、定量表征与实验验证,例如蛋白调控转录的正交元件、RNA聚合酶的正交元件、核糖体结合功能位点的正交元件等。下文将从DNA、RNA和蛋白正交元件3个方面的研究进展(图3)来阐述人工生物体系正交性。
在DNA正交元件研究中,RAO[21]从正交转录因子[22]、转录调节的正交核糖体[23]、翻译调控的正交核糖体[24]和正交的两组分信号转导体系[25]等方面系统综述了在细菌中如何同时实现对多个基因的正交调控;位点特异性重组酶是实现在真核体系中基因编辑的有力工具。如重组酶Cre将反转或删除由两个loxP位点侧接靶向DNA区域,当重组酶Cre执行基因反转时将实现基因重排。LIN等[26]设计了两套位点特异性重组酶系统最终实现对酿酒酵母基因组的正交重组。这两个重组酶系统分别是Cre-loxP和Vika-VOX。这两个系统属于同一个酪氨酸重组酶家族,虽然它们在DNA识别位点的序列和二级结构上有较高相似性,但是验证它们的识别过程是正交的。该工作为在真核体系实现对基因组的正交编辑提供了很好的研究方向。

图3 人工元器件与底盘的正交性研究进展
在RNA正交元件研究方面,各种正交核糖体开关在原核和真核体系中得到了广泛的应用。CHIN 等[27]在大肠杆菌中重构了rRNA的操纵子,使得用于正交核糖体合成的16S rRNA的合成过程与使用其他rRNA的操纵子的过程独立,进而获得了最小的调控模块可以用于正交核糖体的表达调控。这个最小的正交核糖体元件可以用于理性的改善正交转录-翻译前馈控制中的延迟问题。研究发现虽然核糖体及延伸因子(elongation factors,EF)具有比较宽广的识别tRNA能力,但是识别效率会下降。例如采用琥珀密码子编码非天然氨基酸时,由于受到释放因子的干扰,识别效率会下降至正常的20%~30%。所以需要筛选的核糖体不仅是正交的而且还能够高效识别这些扩展的密码子。NEUMANN等[28]用定向进化方法获得了一个正交核糖体(ribo-Q1),能有效解码一系列四联密码子和琥珀密码子,通过正交的aaRS/tRNA组合并结合正交核糖体,直接在对应mRNA上两个新空白密码子的位置掺入了不同的UAA。
在蛋白正交元件研究方面,各种正交蛋白体系被广泛应用于细胞基因编辑和表达调控中。正交的Cas9蛋白就是其中典型的例子。酿脓链球菌来源的Cas9蛋白可以在在基因组的多点实现一种功能,如编辑、激活或者抑制,但是不能同时实现上述所有的功能。CHURCH等[29]在大肠杆菌和人体细胞中构建和筛选出了正交的Cas9核酸酶体系,可以实现单个细胞基因组的多点同时进行编辑、RNA介导的激活和抑制等功能。同时细胞内信号转导依赖于调控蛋白之间的相互作用,更加精确地控制这些相互反应是工程化细胞功能的关键。人工设计正交的蛋白质接口,能够有效地避免来自于宿主细胞本身信号转导系统的干扰,进而实现蛋白质信号网络的工程简化和提升。KAPP等[30]通过计算机辅助设计了正交的重要信号蛋白GTPase (orthoCdc42)和它的特异激活因子。经过在哺乳动物细胞实验验证表明,这个正交的蛋白体系在实现其信号转导功能的同时而不受细胞本身信号系统的影响。
1.3 人工合成系统的鲁棒性
人工合成系统的鲁棒性是指生物在受到外部或内部扰动时,生物系统保持其结构和功能稳定的特性。与稳定性强调功能的维持相比,鲁棒性更强调功能的迅速恢复。产生鲁棒性的作用机制是生物系统的协调、冗余、网络结构和功能稳定。一方面,获得鲁棒性的合成生物系统,可通过细胞进化手段,结合高通量筛选,获得优势细胞株。尤其是代谢路径的异源表达方面,宿主细胞系统对新代谢路径的识别和配合是实现在环境扰动情况下人工合成细胞体系中目标路径代谢流稳定和最大化的关键。其中通过人工设计元器件来实现对代谢网络的多点调控就是其中有效的增强体系鲁棒性的重要途径之一。LIM等[31]通过构建被Cas9蛋白诱导的同时编码目标位点和调控的RNA支架,在酵母体内实现了对代谢网络中目标代谢多基因激活的同时对其他一些支路基因的抑制。这种RNA支架的应用能够对于复杂代谢网络中代谢的调控和重构起到巨大的促进作用(图4)。通过降低生物系统本身的系统噪声进而减弱其对目标路径的干扰也是提高系统整体鲁棒性的重要手段。微RNA虽然是非编码的小RNA,却能通过增强信使RNA的降解或者翻译抑制来调节很多蛋白的表达水平[32],对生物体内微RNA进行人工调控是能够有效降低系统噪声的重要研究方向之一。美国麻省理工学院DI BERNARDO等[33]首先在中国仓鼠卵巢细胞中建立了微RNA介导的基于蛋白表达水平的双稳态开关。数学建模和实验都发现微RNA的介导使得该双稳态的状态维持以及切换鲁棒性增强。一旦缺失微RNA的介导,在蛋白水平的噪声会大幅度上升,进而导致体系会随机在“开”和“关”的两个状态之间切换,鲁棒性下降(图4)。群体响应因子(QS)的不同密度会对细胞整体的生长和生产特性产生极大的影响,这也为研究人员通过人工设计QS介导的控制系统来提高体系的鲁棒性奠定了理论基础。美国杜克大学的YOU研究团队[34]在大肠杆菌体系通过比较3种调控策略:①细胞处在胁迫条件下,生长缓慢且胞外酶停止表达,整个体系处于“关”的状态;②胞外酶高效表达且有利于缓解外界胁迫,整个体系处于“开”的状态;③群体响应因子LuxR/LuxI体系介导的控制体系。结果证明,第三种策略对于不同的接种量和培养时间的变化都显现出更好的系统鲁棒性(图4)。
图4 人工合成体系鲁棒性研究进展
另一方面,如何设计合理“冗余性”的功能模块,理性构建出人工系统的冗余性,也是需要探索的方向。其中关于最小基因组的研究就是有效的手段之一。无论从物质代谢还是从能量代谢经济性的角度,含有去除了冗余基因的较小基因组的细胞都是更为鲁棒和高效的。OGASAWARA等[35]通过逐步删减的办法将基因组大小从原始大肠杆菌菌株(MGF-01)的3.62Mb删减到3.27Mbp和2.98Mbp,同时在构建过程中插入了两个突变ilvG 和 rph 来加速细胞在延滞期的生长,最终获得了基因组较小,但是在富营养培养基中生长特性更佳的宿主细胞(图4)。
1.4 人工合成系统的物质代谢与能量代谢适配性
人工合成系统的物质代谢与能量代谢适配性即是底盘物质代谢与能量代谢的改变与人工线路的互动、造成的相互影响。不同的模块以及模块之间,在不同的底盘生物中存在着复杂的交互影响,很大部分是不利于系统高效发挥功能的。合成生物系统需具备高效发挥功能的适配性。从代谢网络、调控网络、蛋白作用网络和信号转导网络等多个层次,对模块与底盘的内在联系、调控规律进行发掘,深入解析适配机理,最终实现模块功能的最大化和稳定发挥之间的平衡点。下文将从模块和模块之间、模块和底盘细胞之间以及人工混菌体系中细胞和细胞之间的适配性3个方面来进行阐述。
模块和模块之间的适配性研究方面,主要通过调节模块之间的表达强度以及对关键酶的改造等手段来实现目标代谢路径代谢流的最优化。KOFFAS 等[36](图5)在大肠杆菌体内模块化的构建了3个模块:乙酰辅酶A生成模块、乙酰辅酶A激活模块和脂肪酸合成模块来实现脂肪酸的生物合成。通过对变化不同的启动子和拷贝数来实现对上述3个模块的表达调控和适配,最终使得脂肪酸产量达到8.6g/L,这是当时最高的产量。AJIKUMAR等[37]将紫杉二烯的合成途径通过模块化构建到大肠杆菌体系中,分为上游前体IPP生成模块和下游紫杉二烯合成模块,并通过组合设计的原则将不同强度启动子和不同拷贝数质粒作为变量进行上下游模块表达强度的适配,最终将紫杉二烯产量比文献报道提高了6000倍,这个工作也成为模块之间适配性研究的典范。
在模块和底盘适配中,异源表达的模块和底盘细胞内环境之间的相互作用是研究适配性的理论基础。通常采取底盘细胞和外源模块同时改造,逐步适配的策略,最终使得目标路径代谢实现最优化和对底盘细胞的生长特性的影响最小化的终极目标。ÖZAYDIN等[38]以β-胡萝卜素为报告基因,筛选出酿酒酵母基因单敲库中甲羟戊酸积累最高的底盘菌,再通过调整甲羟戊酸代谢途径的表达与底盘菌适配,最终获得了高产红没药烯的生产菌。该研究通过对底盘细胞和代谢路径的同时微调和适配,使得红没药烯产量提高达20倍之多。YUAN课题组[39]在酿酒酵母体内表达7-脱氢胆固醇时发现,单纯的过表达前体乙酰辅酶A模块和7-脱氢胆固醇合成模块,在增加产物积累的同时会加剧细胞内氧化还原的不平衡,这会反过来抑制产物的生成。通过辅因子再生策略对底盘细胞进行改造以缓解氧化还原不平衡状态,最终使7-脱氢胆固醇的积累提高了75%。
除了上述的单菌(细胞)体系,人工混菌体系也是一类很好的底盘体系。人工混菌体系与单细胞体系相比,尤其是在构建长的代谢通路或者完成更为复杂的功能方面有着单据体系无可比拟的优势[40]。在人工多细胞体系内,除了单细胞体内模块和模块之间以及模块和底盘之间的适配之外,如何提高不同细胞(菌)之间在物质代谢和能量代谢的适配性就显得尤为重要。很多研究者在这方面做了大量的工作,如COLLINS等[41]通过来自于金黄葡萄球菌的基于多肽诱导的agr群体响应体系在作为革兰氏阴性菌的大肠杆菌和作为革兰氏阳性菌的巨大芽孢杆菌之间建立正交的混菌体系,大肠杆菌作为多肽的“供给者”,而巨大芽胞杆菌作为多肽的“接受者”。这个研究的成功为今后不同菌之间在基因表达、代谢路径和生长等方面的协调和适配指出了重要的研究方向。2015年美国麻省理工学院的Gregory STEPHANOPOULOS教授课题组[42]将萜类代谢途径构建到大肠杆菌-酿酒酵母的人工混菌体系中,并且建立互利共生的菌群适配关系:改造酿酒酵母只利用来自于大肠杆菌的乙酸为单一碳源,这样同时有利于缓解乙酸对大肠杆菌生长的抑制,成功实现了抗癌药物紫杉醇重要前体Taxa-4(20),11(12)-dien-5alpha-acetoxy-10beta-ol的大量积累,产量达到33mg/L,为最终实现紫杉醇的异源生物合成奠定了坚实的基础。

图5 人工合成体系适配性研究进展
2 如何进一步促进生物学的工程化
合成生物学作为工程化科学,遵循从设计到构建到测试再到重设计的循环模式。合成生物学的工程化特性,推动整个产业链条从依赖于手工技艺发展为高通量、自动化的系统流水线制造模式(图6),即由计算机辅助设计(computer aided design,CAD)人工合成系统,交由机器人平台上进行组装,最终由高通量分析仪器进行测试与评估。该链条的循环发展,将有效地解决拓宽微生物细胞生产化学品的品种以及提高人工合成体系的生产效率这两个限制细胞工厂产业化的两个关键技术问题,最终有利于细胞工厂产业化的实现。
2.1 设计
设计这一环节是整个人工合成系统产业链条的基础与核心。它包含对人工合成系统的设计以及DNA组装系统的设计。对人工合成系统的设计包含系统的设计和元件的选择两个层次。在第一层次对系统的设计中,不仅涉及异源合成路径的设计,还要包括对宿主底盘的改造。这需要根据特定需求,规划目标产物的合成路径,同时设计相关的基因电路和寻找适当的内源靶标,来满足异源路径与宿主以及宿主与环境的试配。在第一层次设计的基础上,针对设计的具体线路或宿主改造靶点,选择适当的核心基因,以及与之表达相关的顺式元件(启动子核心区、增强子、核糖体结合位点、终止子等)。在设计的过程中,需要根据预期需求,有目的性地在庞大的代谢途径、元件、基因线路等数据库中进行搜索。传统依赖于研究者知识与经验的手工设计,已经不能满足复杂的生物系统的设计工作,尤其在合成非天然分子或构建非天然存在的代谢路径上造成瓶颈。而CAD可大幅提升设计效率,甚至对设计结果进行模拟和优化,是这一环节发展的必然趋势。目前,现已开发的计算工具(例如BNICE、PPS)可根据现有的数据库或以酶促反应机理和热力学参数,计算出理论上可实现预设的产物合成和生物降解途径[43-46]。COBRA等工具可对现有设计进行建模预测,针对现有代谢网络从能量供给、氧化还原平衡以及碳代谢流向等几个方面,提供对宿主的改造方案,促进代谢流最大化地流向产物[47]。一些综述总结了现有的关于元件及基因线路设计的工具[48-50]。此外通过计算机的模拟可以对现有的蛋白进行优化甚至设计全新的蛋白质,以加快反应速率、提高底物特异性或催化非天然反应。

图6 合成生物学展望
合成生物学发展至今,已形成大量高效率、高保真的模块化DNA组装方法[51-52],例如依赖于限制性内切酶和连接酶的BioBricksTM[53]、BglBricks[54]和Golden Gate[55];基于体外序列同源重组的Gibson组装[56];基于宿主体内序列同源重组的DNA Assembler[57];以及基于寡核苷酸连接的连接酶循环反应(ligase cycling reaction,LCR)[58]。此外,还可通过CRISPR-Cas系统[59]、TALEN[60]、MAGE[61]等工具在基因组水平完成对染色体进行编辑或细胞的定向进化[62]。传统序列分析和简单克隆中的人工手段已不能胜任对整个构建蓝图复杂繁琐的设计工作。而零件的标准化、模块化的特性,使得相关CAD工具应运而生。例如,j5可实现元件的选择和组装过程的自动化设计[63]。而RAVEN在此基础上,还可根据后续实验进程和检测结果的反馈,对设计方案进行优化[64]。
2.2 构建
构建针对设计蓝图,对合成路径的构建与底盘细胞的改造。这一环节是整个链条中最为耗时耗力的一步。由于生物系统的复性和理性设计的低成功率,往往需要大量的元件与元件之间,靶点与靶点之间进行排列组合。元件的模块化和组装方法的标准化,高通量、可靠性不断提高的DNA自动化组装将科学工作者从机械化的重复劳动中解放出来,节约了大量的人力成本,避免人为实验错误,符合工业大规模生产的要求。伊利诺伊大学赵惠民课题组创建的伊利诺伊iBioFAB自动化平台,其构建系统以在轨道上可控行使的机械手臂穿梭于不同仪器之间,在96孔板中自动完成DNA的合成与组装(http://chbe.illinois.edu/ research/ faculty/huimin-zhao-research)。将该平台利用到染色体编辑工具TALEN的合成中,可使成本从>$1000降低到<$10[65]。预计未来大多数分子生物学实验操作均可通过自动化实验系统实现。研究者将有更多的时间和精力投入到创新性设计中。
2.3 检测
检测分为基因型检测和表现型检测。新一代测序技术[66]的迅猛发展,在提高测序速度的同时,降低单位测序成本,使DNA测序成为验证构建产物的首选手段。目前常规Illumina HiSeq2500微生物基因组重测序的国内市场报价平均为¥2000/M基因组。而最新的Illumina HiSeqXTen每天可产生约6,000 Gbp数据,人类基因组的测序费用降至$1000以下(http://www.illumina.com/systems/hiseq-x -sequencing-system.html)。在完成组装验证后,需通过检测胞内或胞外的代谢产物对所构建的人工合成系统进行评估。NMR、GC-MS、LC-MS是分析代谢物的常用检测手段。最新的分析系统配备了自动样品制备(需要时包括自动衍生化)和自动进样装置,可实现培养后样品自动化高通量检测。最新的质谱技术可实时检测胞内代谢物的变化[67],甚至可对单细胞进行分析[68-69]。检测所获得的数据可反馈给最先的CAD系统,以优化设计方案,或对现有数据库进行补充。CAD工具设计、机器人构建平台,联合高通量分析仪器测试,共同组成新一代合成生物学人工合成体系开发系统,自动展开设计-构建-测试的自动化循环研究。在上文中提到的伊利诺伊大学iBioFAB系统正式基于这种理念所建设。赵惠民课题组[66]将该平台利用到微生物基因组资源挖掘的工作中,已发现3种未知的具有生物活性的多环稠合大环内酰胺类天然产物。
3 结 语
近年来,合成生物学的快速发展和关键科学问题不断凝练,使人们对工程化生物学的特点有了较为广泛和深入的认识,如标准化元件库建立、正交基因线路、鲁棒的人工多细胞体系等。但是距离实现最终完全按照人为目的设计和构建生物体系的目标还有很长的路要走。遵循从设计到构建到测试再到重设计的循环模式,进一步和其他工程科学如数学、物理学、计算机学等深入交叉融合,同时按照从原料多样化、产品的合成与底盘细胞的选择全链条布局,将对最终阐明人工生物体系设计构建原则,也就是如何更好地工程化生物学奠定基础。
参 考 文 献
[1] KEASLING J D. Synthetic biology for synthetic chemistry[J]. ACS Chemical Biology,2008,3(1):64-76.
[2] KHALIL A S,Collins J J. Synthetic biology:applications come of age[J]. Nature Reviews Genetics,2010,11(5):367-379.
[3] FORSTER A C,LEE S Y. Editorial:NextGen SynBio has arrived[J]. Biotechnology Journal,2012,7(7):827-827.
[4] WANG Y H,WEI K Y,SMOLKE C D. Synthetic biology:advancing the design of diverse genetic systems[J]. Annual Review of Chemical and Biomolecular Engineering,2013,4:69-102.
[5] SINGH V. Recent advancements in synthetic biology:current status and challenges[J]. Gene,2014,535(1):1-11.
[6] AGAPAKIS C M. Designing synthetic biology[J]. ACS Synthetic Biology,2013,3(3):121-128.
[7] US.Living Foundries [EB/OL]. [2012] http://nas-sites.org/ synbioroadmap /files/2014/03/5.Alica-Jackson-Program-Manager-DARPA.pdf.
[8] Organization for Economic Cooperation and Development (OECD). Emerging policy issues in synthetic biology [EB/OL] . [2014]http://www.oecd-ilibrary.org/science-and-technology/emerging-policy-iss ues-in-synthetic-biology_9789264208421-en.
[9] US Department of Energy. Synthetic biology,report to congress [EB/OL]. [2013]. http://www.sumberc.org/sites/default/file/DOE% 20Synthetic%20Biology%20Report%20to%20Congress Fn1.pdf.
[10] U.S. Department of Defense. DOD science & technology priorities [EB/OL]. [2014]. http://community.apan.org/afosr/m/alea_stewart/ 135113/download.aspx.
[11] US National Academy of Sciences. Industrialization of biology:a roadmap to accelerate the advanced manufacturing of chemicals [EB/OL]. [2015]. www.nap.edu/catalogy/19001.
[12] The McKinsey Global Institute. Disruptive technologies:advances that will transform life,business,and the global economy[EB/OL]. [2013]. http://www.mckinsey.com/insights/business_technology/ disruptive_technologies.
[13] UK Department for Business,Innovation & Skills. Eight Great Technologies:infographics[EB/OL]. [2013]. https://www.gov.uk/ government/uploads/system/uploads/attachment_data/file/249268/syn thetic_biology_infographic.pdf.
[14] Transparency Market Research. US synthetic biology market (synthetic DNA,synthetic Genes,synthetic cells,XNA,chassis organisms,DNA synthesis,oligonucleotide synthesis)——Global industry analysis,size,share,growth,trends and forecast 2013-2019 [EB/OL]. [2014-04-08]. http://www.transparencymarketresearch. com/synthetic-biology-market.html.
[15] QI H,LI B Z,ZHANG W Q,et al. Modularization of genetic elements promotes synthetic metabolic engineering[J]. Biotechnology Advances,2015,33(7):1412-1419.
[16] ELLIS T,ADIE T,BALDWIN G S. DNA assembly for synthetic biology:from parts to pathways and beyond[J]. Integrative Biology,2011,3(2):109-118.
[17] KNIGHT T F. Idempotent vector design for standard assembly of BioBricks. MIT Synthetic Biology Working Group Technical Reports[R]. Knight,Thomas,MIT Artificial Intelligence Laboratory:2003. http:// hdl.handle.net/1721.1/21168.
[18] CANTON B,LABNO A,ENDY D. Refinement and standardization of synthetic biological parts and devices[J]. Nature Biotechnology,2008,26(7):787-793.
[19] GALDZICKI M,CLANCY K P,OBERORTNER E,et al. The Synthetic Biology Open Language (SBOL) provides a community standard for communicating designs in synthetic biology[J]. Nature Biotechnology,2014,32(6):545-550.
[20] BARTLEY B,BEAL J,CLANCY K,et al. Synthetic biology open language (SBOL) Version 2.0.0.[J]. J. Int. Bioinformatics,2015,12 (2):272.
[21] RAO C V. Expanding the synthetic biology toolbox:engineering orthogonal regulators of gene expression[J]. Current opinion in biotechnology,2012,23(5):689-694.
[22] DESAI T A,RODIONOV D A,GELFAND M S,et al. Engineering transcription factors with novel DNA-binding specificity using comparative genomics[J]. Nucleic Acids Research,2009,37(8):2493-2503.
[23] LIANG J C,BLOOM R J,SMOLKE C D. Engineering biological systems with synthetic RNA molecules[J]. Molecular Cell,2011,43 (6):915-926.
[24] CHUBIZ L M,RAO C V. Computational design of orthogonal ribosomes[J]. Nucleic Acids Research,2008,36(12):4038-4046.
[25] STOCK A M,ROBINSON V L,Goudreau P N. Two-component signal transduction[J]. Annual Review of Biochemistry,2000,69(1):183-215.
[26] LIN Q H,QI H,WU Y,YUAN Y J. Robust orthogonal recombination system for versatile genomic elements rearrangement in yeast Saccharomyces Cerevisiae[J]. Scientific Reports,2015(5):15249.
[27] AN W,CHIN J W. Synthesis of orthogonal transcription-translation networks[J]. Proceedings of the National Academy of Sciences,2009,106(21):8477-8482.
[28] NEUMANN H,WANG K,DAVIS L,et al. Encoding multiple unnatural amino acids via evolution of a quadruplet-decoding ribosome[J]. Nature,2010,464(7287):441-444.
[29] ESVELT K M,MALI P,BRAFF J L,et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing[J]. Nature Methods,2013,10(11):1116-1121.
[30] KAPP G T,LIU S,STEIN A,et al. Control of protein signaling using a computationally designed GTPase/GEF orthogonal pair[J]. Proceedings of the National Academy of Sciences,2012,109(14):5277-5282.
[31] ZALATAN J G,LEE M E,ALMEIDA R,et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds[J]. Cell,2015 160(1-2):339-350.
[32] INUI M,MARTELLO G,PICCOLO S. MicroRNA control of signal transduction[J]. Nature Reviews Molecular Cell Biology,2010,11(4):252-263.
[33] SICILIANO V,GARZILLI I,FRACASSI C,et al. MiRNAs confer phenotypic robustness to gene networks by suppressing biologyical noise[J]. Nature Communications,2013,4:2364.
[34] PAI A,TANOUCHI Y,YOU L. Optimality and robustness in quorum sensing (QS)-mediated regulation of a costly public good enzyme[J]. Proceedings of the National Academy of Sciences,2012,109(48):19810-19815.
[35] HIROKAWA Y,KAWANO H,TANAKA-MASUDA K,et al. Genetic manipulations restored the growth fitness of reduced-genome Escherichia coli[J]. Journal of Bioscience and Bioengineering,2013,116(1):52-58.
[36] XU P,GU Q,WANG W,et al. Modular optimization of multi-gene pathways for fatty acids production in E. coli[J]. Nature Communications,2013,4:1409.
[37] AJIKUMAR P K,XIAO W H,TYO K E J,et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli[J]. Science,2010,330(6000):70-74.
[38] ÖZAYDIN B,BURD H,LEE T S,et al. Carotenoid-based phenotypic screen of the yeast deletion collection reveals new genes with roles in isoprenoid production[J]. Metabolic Engineering,2013,15:174-183.
[39] SU W,XIAO W H,WANG Y,et al. Alleviating redox imbalance enhances 7-dehydrocholesterol production in engineered Saccharomyces cerevisiae[J]. PloS One,2015,10(6):e0130840.
[40] SHONG J,DIAZ M R J,COLLINS C H. Towards synthetic microbial consortia for bioprocessing[J]. Current Opinion in Biotechnology,2012,23(5):798-802.
[41] MARCHAND N,COLLINS C H. Peptide‐based communication system enables Escherichia coli to Bacillus megaterium interspecies signaling[J]. Biotechnology and Bioengineering,2013,110(11):3003-3012.
[42] ZHOU K,QIAO K,EDGAR S,et al. Distributing a metabolic pathway among a microbial consortium enhances production of natural products[J]. Nature Biotechnology,2015,33(4):377-383.
[43] HOU B K,ELLIS L B M,WACKETT L P. Encoding microbial metabolic logic:predicting biodegradation[J]. Journal of Industrial Microbiology and Biotechnology,2004,31(6):261-272.
[44] PRATHER K L,MARTIN C H. De novo biosynthetic pathways:rational design of microbial chemical factories[J]. Current Opinion in Biotechnology,2008,19(5):468-474.
[45] GONZALEZ-LERGIER J,BROADBELT L J,HATZIMANIKATIS V. Theoretical considerations and computational analysis of the complexity in polyketide synthesis pathways[J]. Journal of the American Chemical Society,2005,127(27):9930-9938.
[46] HATZIMANIKATIS V,LI C,IONITA J A,et al. Exploring the diversity of complex metabolic networks[J]. Bioinformatics,2005,21(8):1603-1609.
[47] LEWIS N E,NAGARAJAN H,PALSSON B O. Constraining the metabolic genotype-phenotype relationship using a phylogeny of in silico methods[J]. Nature Reviews Microbiology,2012,10(4):291-305.
[48] QI H,LI B Z,ZHANG W Q,et al. Modularization of genetic elements promotes synthetic metabolic engineering[J]. Biotechnology Advances,2015,33(7):1412-1419.
[49] BROPHY J A,VOIGT C A. Principles of genetic circuit design[J]. Nature Methods,2014,11(5):508-520.
[50] MACDONALD J T,BARNES C,KITNEY R I,et al. Computational design approaches and tools for synthetic biology[J]. Integrative Biology :Quantitative Biosciences from Nano to Macro,2011,3 (2):97-108.
[51] ELLIS T,ADIE T,BALDWIN G S. DNA assembly for synthetic biology:from parts to pathways and beyond[J]. Integrative Biology :Quantitative Biosciences from Nano to Macro,2011,3(2):109-118.
[52] MA S,TANG N,TIAN J. DNA synthesis,assembly and applications in synthetic biology[J]. Current Opinion in Chemical Biology,2012,16(3-4):260-267.
[53] SMOLKE C D. Building outside of the box:iGEM and the bioBricks foundation[J]. Nature Biotechnology,2009,27(12):1099-1102.
[54] ANDERSON J C,DUEBER J E,LEGUIA M,et al. BglBricks:a flexible standard for biological part assembly[J]. Journal of Biological Engineering,2010,4(1):1.
[55] ENGLER C,KANDZIA R,MARILLONNET S. A one pot,one step,precision cloning method with high throughput capability[J]. PloS One,2008,3(11):e3647.
[56] GIBSON D G,SMITH H O,HUTCHISON C A,et al. Chemical synthesis of the mouse mitochondrial genome[J]. Nature Methods,2010,7(11):901-903.
[57] SHAO Z,ZHAO H,ZHAO H. DNA assembler,an in vivo genetic method for rapid construction of biochemical pathways[J]. Nucleic Acids Research,2009,37(2):e16.
[58] DE KOK S,STANTON L H,SLABY T,et al. Rapid and reliable DNA assembly via ligase cycling reaction[J]. ACS Synthetic Biology,2014,3(2):97-106.
[59] SANDER J D,JOUNG J K. CRISPR-Cas systems for editing,regulating and targeting genomes[J]. Nature Biotechnology,2014,32(4):347-355.
[60] WOOD A J,LO T W,ZEITLER B,et al. Targeted genome editing across species using ZFNs and TALENs[J]. Science,2011,333 (6040):307-307.
[61] WANG H H,ISAACS F J,CARR P A,et al. Programming cells by multiplex genome engineering and accelerated evolution[J]. Nature,2009,460(7257):894-898.
[62] DAVID F,SIEWERS V. Advances in yeast genome engineering[J]. FEMS Yeast Research,2015,15(1):1-14.
[63] HILLSON N J,ROSENGARTEN R D,KEASLING J D. j5 DNA assembly design automation software[J]. ACS Synthetic Biology,2012,1(1):14-21.
[64] APPLETON E,TAO J,HADDOCK T,et al. Interactive assembly algorithms for molecular cloning[J]. Nature Methods,2014,11(6):657-662.
[65] SUN N,ZHAO H. A single-chain TALEN architecture for genome engineering[J]. Molecular BioSystems,2014,10(3):446-453.
[66] METZKER M L. Sequencing technologies ——the next generation[J]. Nature Reviews Genetics,2010,11(1):31-46.
[67] YUKIHIRA D,MIURA D,SAITO K,et al. MALDI-MS-based high-throughput metabolite analysis for intracellular metabolic dynamics[J]. Analytical Chemistry,2010,82(10):4278-4282.
[68] YANES O. Metabolomics:playing pinata with single cells[J]. Nature Chemical Biology,2013,9(8):471-473.
[69] RUBAKHIN S S,ROMANOVA E V,NEMES P,et al. Profiling metabolites and peptides in single cells[J]. Nature Methods,2011,8 (4s):S20-S29.
How to make biology more “engineering”
XIAO Wenhai1,2,ZHOU Sijie1,2,WANG Ying1,2,YUAN Yingjin1,2
(1Key Laboratory of Systems Bioengineering(Ministry of Education),Tianjin University,Tianjin 300072,China;
2SynBio Research Platform,Collaborative Innovation Center of Chemical Science and Engineering(Tianjin),Tianjin 300072,China)
Abstract:With the development and application of chemistry,chemical engineering arises at the historic moment. Similarly,with the development of biology and engineering requirement,synthetic biology,known as“the third revolutions of biology”,was born. Synthetic biology is the engineering of biology. From the point of view of engineering,it designs and creates parts,devices or modules,and employs them to modify and optimization of existing natural biological system,but how to engineer complex life system is the key scientific problem which synthetic biology researchers have long been exploring. In this paper,the main four characteristics of engineering biology are systematically illustrated:①modularization and standardization;②orthogonality;③robustness;④compatibility,as well as the corresponding research progress. Finally,from the point of view of“Design-Build -Test”cycle,proposals for how to further engineer biology in the future are addressed.
Key words:synthetic biology;modularization;orthogonality;robustness;compatibility;“design-bulid-test”cycle
中图分类号:TQ 033
文献标志码:A
文章编号:1000–6613(2016)06–1827–10
DOI:10.16085/j.issn.1000-6613.2016.06.024
收稿日期:2016-01-22;修改稿日期:2016-03-11。
基金项目:国家高技术研究发展计划(国家863计划)重大项目(2012AA02A701)。
