家蚕微粒体GST的鉴定与序列分析
2016-07-05房守敏
房守敏
(西华师范大学生命科学学院,四川 南充 637002)
家蚕微粒体GST的鉴定与序列分析
房守敏*
(西华师范大学生命科学学院,四川 南充637002)
摘要微粒体GSTs(microsomal glutathione S-transferases,mGSTs)是广泛分布于动物、植物、细菌等生物体中的多功能酶类,具有对药物解毒和抗氧化等功能。作为一类膜蛋白,在研究手段上难于胞质型GSTs(cytosolic GSTs),迄今为止在昆虫中较少展开功能研究。本研究在基因组水平对家蚕mGSTs进行了分析,共鉴定出BmmGST1和BmmGST2两个基因,它们位于家蚕15号染色体,呈串联排列,无内含子插入。BmmGST1和BmmGST2编码的蛋白分别含有151和150个氨基酸残基,分子量为16.9 kD和16.7 kD。进化分析表明,家蚕mGSTs可能是由于世系特异扩增而产生的两个拷贝。分析表达序列标签证实BmmGST1在5龄3 d幼虫的翅原基、丝腺和卵巢组织表达。基于组织和发育时期芯片,BmmGST2在各组织和发育期均有表达信号。家蚕与其他昆虫、大鼠的mGSTs序列高度保守,是否也有类似于哺乳动物中的功能值得进一步研究。
关键词家蚕;微粒体GST;表达模式;序列分析
谷胱甘肽-S-转移酶(glutathione S-transferases, GSTs)是一个多功能的超基因家族,广泛分布于各类生物体[1]。根据其细胞定位,GSTs可分为胞质(cytosolic GSTs)、微粒体(microsomal GSTs,mGSTs)和线粒体(mitochondrial GSTs)三种类型,它们均起源于同一祖先序列[2]。胞质型GST的基因数目相对较多,分布于动物、植物、昆虫、细菌及真菌[2]。昆虫胞质型GST研究主要集中于与有机磷(organophosphorus,OPs)、有机氯(organochlorine)和拟除虫菊酯(pyrethroid)等杀虫剂抗性研究[3-6]。线粒体GSTs也叫Kappa GSTs,主要分布于哺乳动物的线粒体和过氧化酶体中,在植物和昆虫中并无此类GSTs的存在。微粒体GSTs是一类膜蛋白,现在已被归类为参与花生四烯酸和GSH代谢的膜相关蛋白(Membrane Associated Proteins in Eicosanoid and Glutathione metabolism,MAPEG)超家族[7]。尽管微粒体GST与胞质和线粒体GST具有共同的祖先,但在序列和结构上已发生了较大的分化,如:微粒体GST的亚基一级结构相对较小,大致由150个氨基酸组成;由同源三聚体构成一个功能单位[8],线粒体和胞质GSTs基本则为同源或异源的二聚体,仅有植物的Lambda GSTs为单聚体[9]。
氧化应激是需氧生物体中普遍存在的自然现象。如杀虫剂等有害物质进入生物体内,能打破体内氧化和抗氧化的动态平衡,大量产生的活性氧(reactive oxygen species, ROS)对膜脂质、DNA、蛋白质及糖类等生物大分子造成氧化损伤,进而产生新的细胞毒素和诱变剂引发氧化应激反应[10-13]。在该链式反应中,降低脂质过氧化物的含量显得尤为重要。氧化应激反应中还会伴随其他有害的醛类物质,如丙二醛(malondialdehyde, MDA)和4-羟基壬烯醛(4-hydroxynonenal, 4-HNE)。尽管低含量的HNE在细胞中具有信号分子的作用,然而高浓度的HNE则对细胞产生毒性[14]。在人类和其他哺乳动物中的研究证实,微粒体mGST具有谷胱甘肽过氧化物酶活性对应激反应中的脂质过氧化氢和醛类等有害物质进行解毒[15],从而降低杀虫剂等对生物体引起的氧化应激损伤。同时,mGST在药物经肝微粒体解毒代谢、抗癌药物耐药性中的作用等亦在哺乳动物中有广泛研究[16]。
微粒体mGST作为一类膜蛋白,相对于可溶性的胞质型而言,在昆虫中的功能研究相对滞后。Shi等[17]对赤拟谷盗(Triboliumcastaneum)和其他几种模式昆虫的GST基因进行了鉴定和比较分析,同时对微粒体mGST进行了预测。本研究基于上述研究基础之上,对家蚕的微粒体mGST进行了全基因组鉴定、基因结构、表达模式和进化分析。旨在为后续研究家蚕mGST的功能等奠定基础。
1材料与方法
1.1软件
NCBI下载的BLAST(Basic local alignment search tool)用于同源比对搜索。ESTs序列的拼接使用DNASTAR中的SeqMan软件。进化树构建采用MEGA6.0[18]。蛋白分子量MW/等电点pI的预测使用在线工具http://www.expasy.ch/tools/pi_tool.html。
1.2mGST的鉴定
以Shi等[17]鉴定的其他昆虫mGST氨基酸序列作为query,与家蚕基因组进行tblastn搜索。截取与已知mGST同源的家蚕scaffold区段,用在线工具FGENESH(http://linux1.softberry.com/)预测家蚕潜在的mGST编码区序列(Coding sequence,CDS)和推导的氨基酸序列。将预测得到的家蚕mGST与NCBI(http://www.ncbi.nlm.nih.gov/)非冗余蛋白数据库(non-redundant protein database,nr)比对,验证是否为真实的mGST。
家蚕mGST编码区序列与表达序列标签(Expressed sequence tags,ESTs)数据库进行Blastn比对,获得其对应的ESTs,并利用DNASTAR中的SeqMan软件对ESTs序列电子延伸,获得家蚕mGST 较完整的cDNA序列。延长后的cDNA序列与家蚕基因组Blastn比对,分析mGST是否存在内含子及插入位置。
2结果与讨论
2.1家蚕mGST的鉴定
以果蝇等昆虫微粒体mGSTs(表1)氨基酸序列与家蚕基因组进行比对,对存在候选mGST的家蚕Scaffold区段进行基因预测。预测得到的mGST与NCBI nr数据库同源搜索,鉴定是否与已知的其他昆虫mGST同源,最终鉴定出BmmGST1和BmmGST2基因。我们将BmmGST1和BmmGST2的CDS序列与家蚕基因组预测的所有蛋白编码基因数据库(http://www.silkdb.org/silkdb/)比对分析,两基因均对应于编号为BGIBMGA007779的预测基因(表1),推测是由于家蚕基因组基因预测错误所致。
BmmGST1和BmmGST2呈串联排列的方式分布于家蚕15号染色体上。家蚕mGST的CDS与EST数据库比对,鉴定出对应的ESTs序列,经拼接获得cDNA长度分别为537bp和571bp。cDNA序列与家蚕基因组序列比对发现,均无内含子插入,基因结构简图见图1。BmmGST1和BmmGST2编码的蛋白分别含有151和150个氨基酸残基,分子量为16.9kD和16.7kD,等电点pI为9.99和9.73。
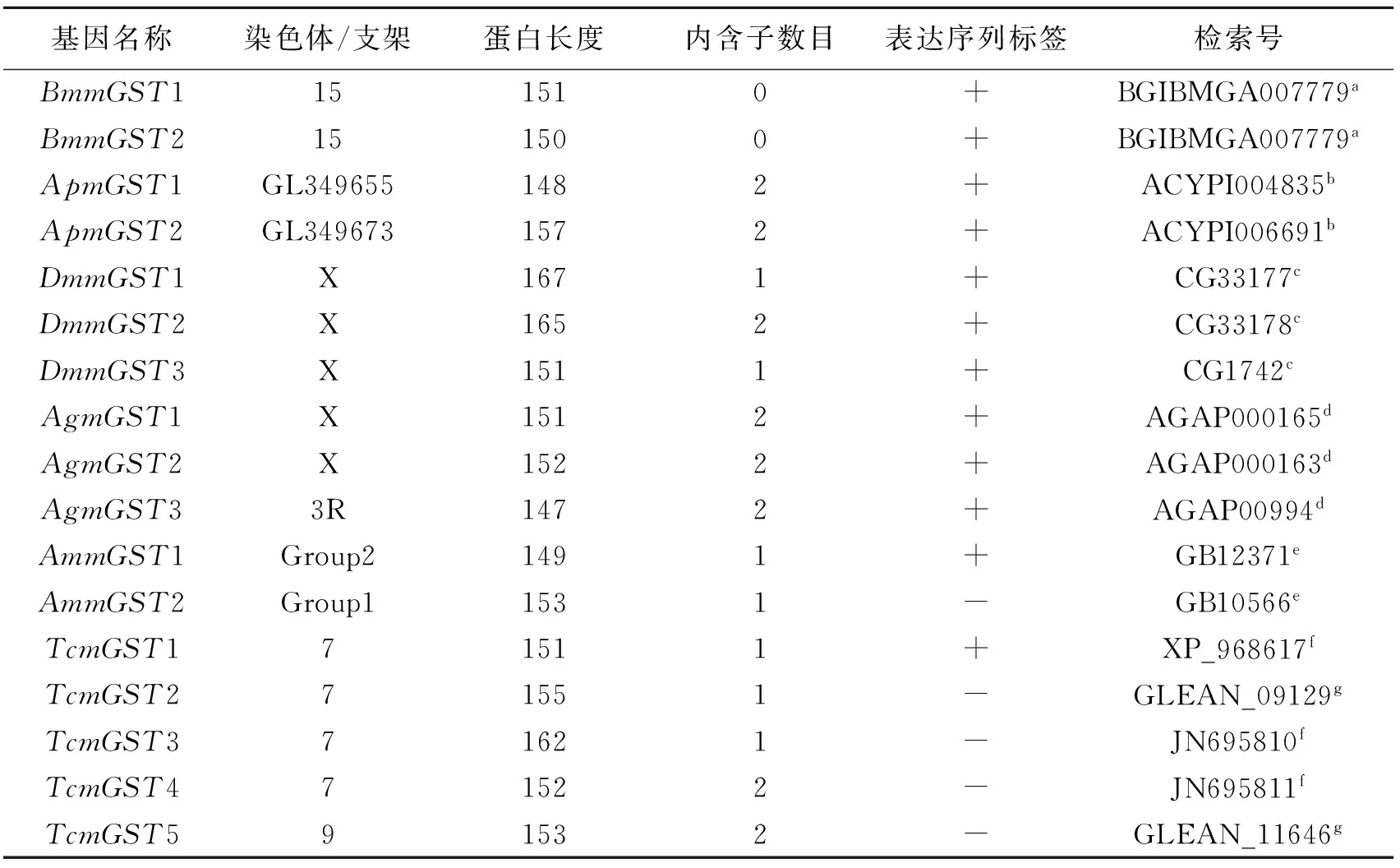
表1 家蚕与其他模式昆虫mGST基因信息
家蚕mGST为本研究鉴定,其他昆虫mGST基因的信息引自Shi等[17]。a为SilkDB(http://www.silkdb.org/silkdb/)检索号;b为Acyrthosiphonpisum基因组数据库 (http://www.aphidbase.com/aphidbase/);c为FlyBase(http://flybase.org/);d为VectorBase (http://www.vectorbase.org/);e为BeeBase(http://hymenopteragenome.org/beebase/);f为NCBI(http://www.ncbi.nlm.nih.gov/);g为T.castaneum基因组数据库(http://beetlebase.org/cgi-bin/gbrowse/BeetleBase3.gff3/)。

灰色框表示非编码区,黑色框表示外显子,箭头表示转录方向和起始密码子位置。
2.2mGST的进化与序列保守性分析
以家蚕微粒体GSTs的蛋白序列与其他模式昆虫mGSTs构建邻近系统发生树(图2),结果表明同一物种的mGST往往聚在一起,暗示在物种分化形成后,mGST发生了少量的世系特异扩增,家蚕由最初的1个基因扩增为2个mGST。有趣地是,赤拟谷盗的mGST基因数目最多,含有5个基因,尽管我们并不清楚mGST为什么会大量扩增,这些扩增是否跟仓储害虫的抵御氧化应激压力相关还值得进一步研究。
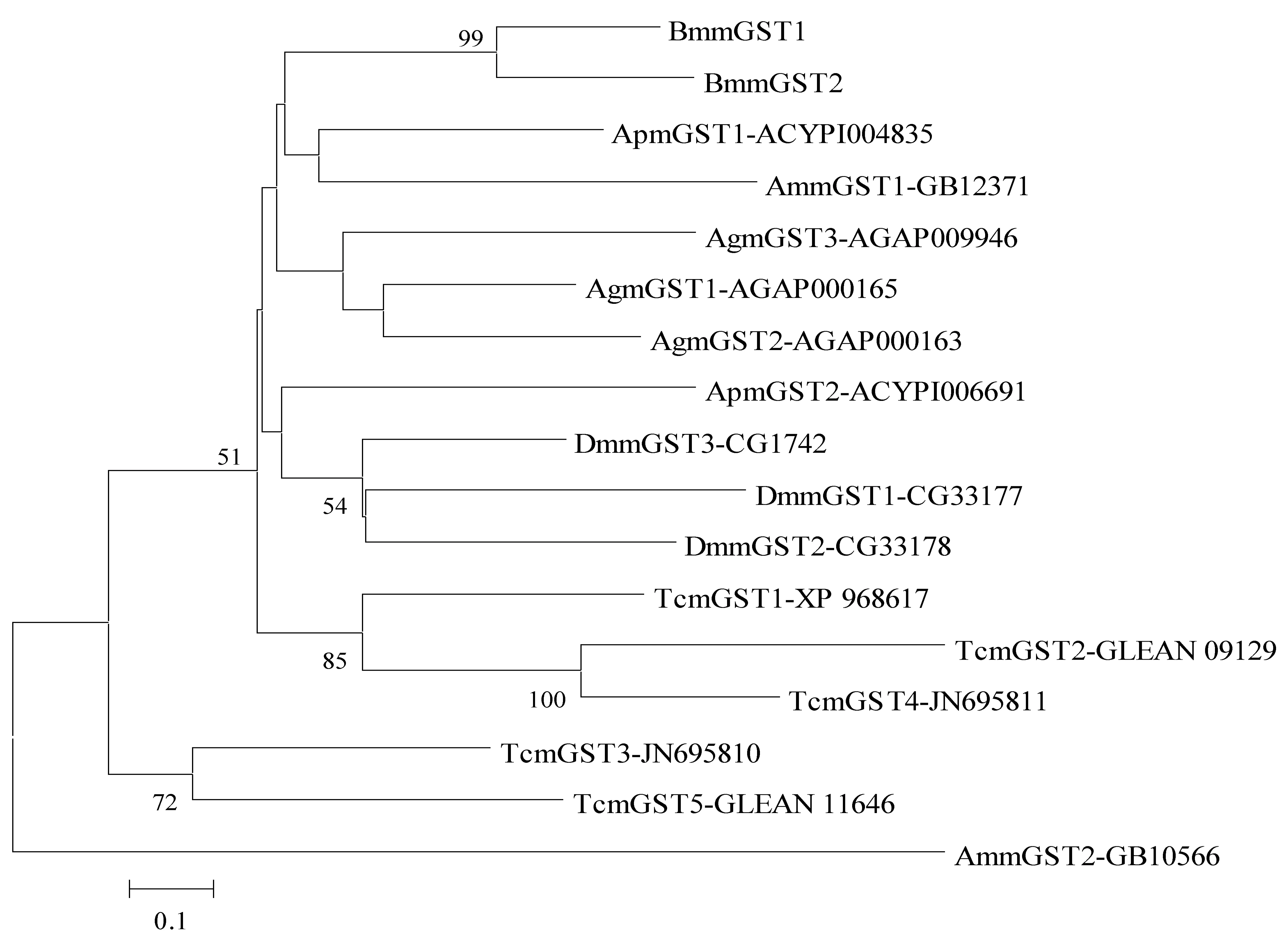
基于MEGA 6.0 构建的邻近(Neighbor-Joining,NJ)树,

方框表示昆虫微粒体mGSTs特异的特征基序,下划横线为跨膜域(transmembrane regions,TM),
昆虫微粒体mGST编码的蛋白长度非常相似,为150个氨基酸左右(表1)。家蚕与其他昆虫微粒体GSTs的多序列比对分析(图3),发现昆虫微粒体mGSTs 含有高度保守的V-E-R-V-R-R-A-H-x-N-D-x-E-N-I结构域,这与以前的研究结果相一致[19]。另外,微粒体mGST作为一种膜蛋白,它们均含有4个跨膜域,贯穿于整条序列中(图3)。家蚕BmmGST1和BmmGST2基因编码蛋白的氨基酸一致性65.3%。BmmGST1和BmmGST2与双翅目昆虫微粒体GSTs的氨基酸序列一致性分别为42.4%-50.0%和44.2%-49.3%,与蜜蜂AmGSTmic-GB12371的一致性分别为43.9%和40.8%。因此,昆虫微粒体mGSTs在结构和一级序列上均高度保守。
2.3家蚕mGST的表达模式
为分析家蚕mGSTs的表达模式,我们将家蚕mGSTs的CDS序列与家蚕芯片探针数据库[20]进行Blastn比对检索,仅BmmGST2有对应的探针sw13395。因此,我们对BmmGST2在组织和发育时期中的表达进行了分析(图4)。结果表明BmmGST2为一个组成型表达的基因,在各组织和发育时期均有表达信号,并在雌雄个体中具有相似的表达模式。但仍有少量的发育时间点雌雄个体间呈现表达差异(t-test,P<0.05),如在雄蛾的表达信号值为6905.0±722.7,雌蛾为1 272.6±703.6;5龄3d幼虫精巢的信号值为2 298.7±988.6,而在卵巢的信号值为5 236.8±870.0。这些表达差异是否与BmmGST2在卵的形成和成熟过程中,防止氧化应激带来损伤有关值得进一步研究。
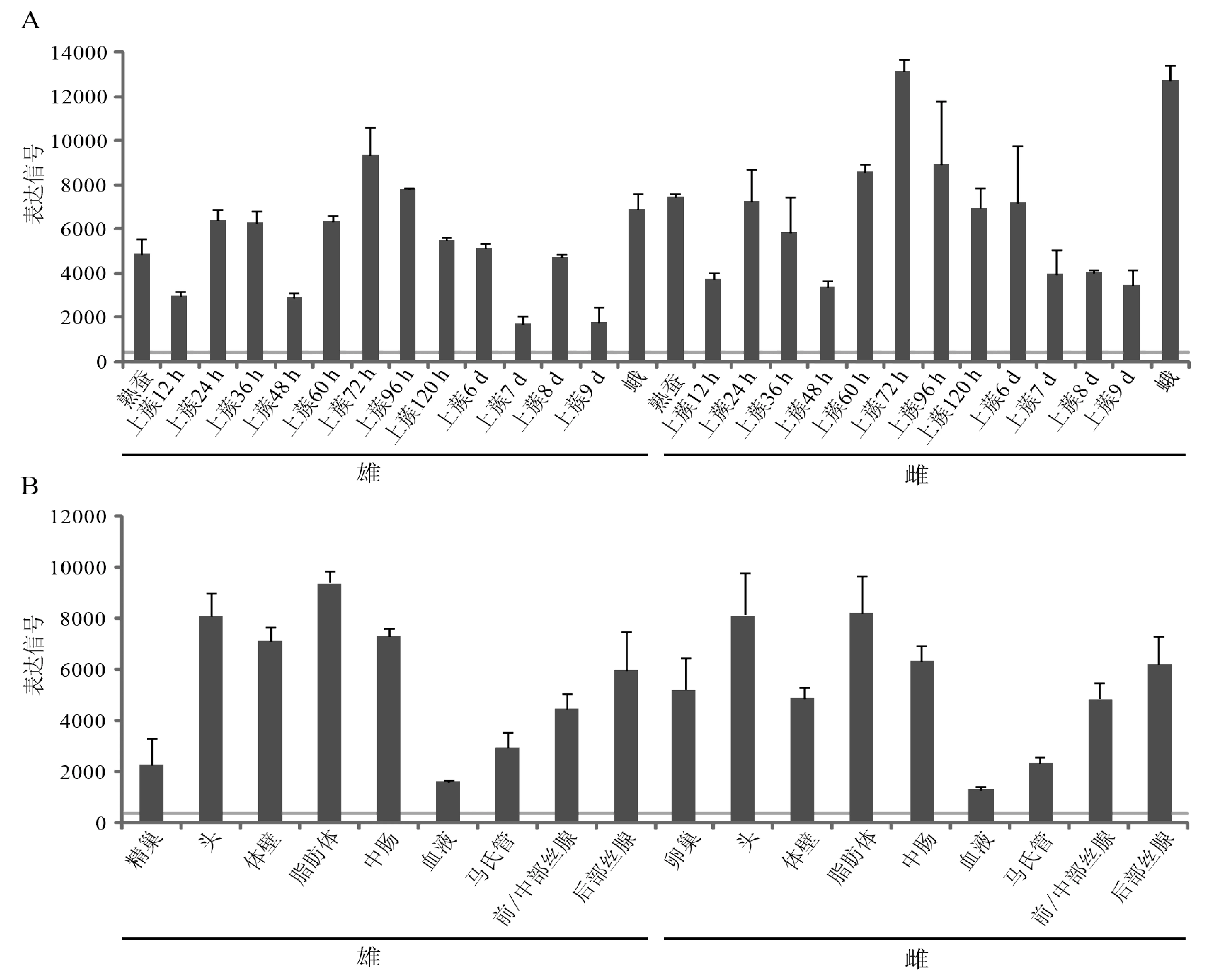
芯片数据下载自SilkDB。A)发育时期芯片数据;B)5龄3d幼虫各组织中的表达。
通过与家蚕ESTs数据比对搜索,BmmGST1鉴定出4条ESTs序列,其GenBank的检索号为AV405705.1、CK485397.1、BB987310.1和BP179853.1。这些EST分别来源于5龄3d幼虫的翅原基、丝腺和卵巢组织[21]。因此,BmmGST1在家蚕中仍具有表达信号,是一个具有功能的基因。
经生物信息学分析,家蚕基因组中存在2个具有功能性的微粒体BmmGST1和BmmGST2基因,其编码的蛋白分别含有151和150氨基酸。它们在基因组中呈串联排列,位于15号染色体上。家蚕微粒体GST与其他昆虫mGSTs保守性高,即使与大鼠的mGST1也具有保守的结构域(图3)。在人类和哺乳动物的研究表明微粒体GST与药物代谢、抗氧化等功能相关[16],这些高度保守的基因是否在昆虫中也具有相似的功能值得进步研究。
参考文献
[1]SALINAS A E, WONG M G. Glutathione S-transferases-a review[J]. Curr Med Chem, 1999,6(4):279-309.
[2]SHEEHAN D, MEADE G, FOLEY V M, et al. Structure, function and evolution of glutathione transferases:implications for classification of non-mammalian members of an ancient enzyme superfamily[J]. Biochem J, 2001,360(Pt 1):1-16.
[3]ENAYATI A A, RANSON H, HEMINGWAY J. Insect glutathione transferases and insecticide resistance[J]. Insect Mol Biol, 2005,14(1):3-8.
[4]吕敏, 刘惠霞, 吴文君. 谷胱甘肽S-转移酶与昆虫抗药性的关系[J]. 昆虫知识, 2003,40(3):204-207.
[5]RANSON H, HEMINGWAY J. Glutathione transferases In: comprehensive molecular insect science-pharmacology[M]. Oxford: Elsevier, 2005: 383-398.
[6]LI X, SCHULER M A, BERENBAUM M R. Molecular mechanisms of metabolic resistance to synthetic and natural xenobiotics[J]. Annu Rev Entomol, 2007,52:231-253.
[7]JAKOBSSON PJ, MORGENSTERN R, MANCINI J, et al. Common structural features of MAPEG-a widespread superfamily of membrane associated proteins with highly divergent functions in eicosanoid and glutathione metabolism[J]. Protein Sci, 1999,8(3):689-692.
[8]SCHMIDT-KREY I, MITSUOKA K, HIRAI T, et al. The three-dimensional map of microsomal glutathione transferase 1 at 6A resolution[J]. EMBO J, 2000,19(23):6311-6316.
[9]FROVA C. Glutathione transferases in the genomics era:new insights and perspectives[J]. Biomol Eng, 2006,23(4):149-169.
[10]GIORDANO G, AFSHARINEJAD Z, GUIZZETTI M, et al. Organophosphorus insecticides chlorpyrifos and diazinon and oxidative stress in neuronal cells in a genetic model of glutathione deficiency[J]. Toxicol Appl Pharmacol, 2007,219(2-3):181-189.
[11]VONTAS J G, SMALL G J, HEMINGWAY J. Glutathione S-transferases as antioxidant defence agents confer pyrethroid resistance inNilaparvatalugens[J]. Biochem J, 2001,357(Pt 1):65-72.
[12]PARKES T L, HILLIKER A J, PHILLIPS J P. Genetic and biochemical analysis of glutathione S-transferases in the oxygen defence system ofDrosophilamelanogaster[J]. Genome, 1993, 36(6):1007-1014.
[13]MARNETT L J, RIGGINS J N, WEST J D. Endogenous generation of reactive oxidants and electrophiles and their reactions with DNA and protein[J]. J Clin Invest, 2003,111(5):583-593.
[14]NEGRE-SALVAYRE A, COATRIEUX C, INGUENEAU C, et al. Advanced lipid peroxidation end products in oxidative damage to proteins. Potential role in diseases and therapeutic prospects for the inhibitors. Br J Pharmacol, 2008,153(1):6-20.
[15]JOHANSSON K, J RVLIDEN J, GOGVADZE V, et al. Multiple roles of microsomal glutathione transferase 1 in cellular protection:a mechanistic study[J]. Free Radic Biol Med, 2010,49(11):1638-1645.
[16]郑英, 楼宜嘉. 微粒体谷胱甘肽S-转移酶与药物代谢[J]. 中国药学杂志, 2003, 38(7):484-487.
[17]SHI H, PEI L, GU S, et al. Glutathione S-transferase (GST) genes in the red flour beetle, Tribolium castaneum, and comparative analysis with five additional insects[J]. Genomics, 2012,100(5):327-335.
[18]TAMURA K, STECHER G, PETERSON D, et al. MEGA6:Molecular Evolutionary Genetics Analysis version 6.0[J]. Mol Biol Evol, 2013,30(12):2725-2729.
[19]BRESELL A, WEINANDER R, LUNDQVIST G, et al. Bioinformatic and enzymatic characterization of the MAPEG superfamily[J]. FEBS J, 2005,272(7):1688-1703.
[20]XIA Q, CHENG D, DUAN J, et al. Microarray-based gene expression profiles in multiple tissues of the domesticated silkworm,Bombyxmori[J]. Genome Biol,2007,8(8):R162.
[21]程道军, 夏庆友, 周泽扬,等. 家蚕cDNA文库构建及大规模EST测序[J]. 蚕业科学, 2003,29(4):335-339.
Microsomal GSTs (mGSTs) are a family of multifunctional enzymes found in animals, plants and bacteria, which contain the activity of detoxification and glutathione peroxidase. As one of the membrane proteins, it is difficult to study compared with cytosolic GSTs in insects. In present study, we analyzed putative mGSTs in the silkworm genome, and identifiedBmmGST1 andBmmGST2. They were tandemly located on chromosome 15 and encoded 151 and 150 amino acids, respectively. Molecular weights of them are 16.9 kD and 16.7 kD. Phylogenetic analysis suggested that twoBmmGSTswere the two copies from linage-specific duplication. The putative coding sequence of silkworm mGSTs was used to do Blastn search in the silkworm expressed sequence tags (ESTs) database.BmmGST1 expressed in the wing disc, silkgland and ovary on day 3 of fifth instar larvae. Based on the microarray datasets,BmmGST2 was found to express in all tissues on day 3 of fifth instar larvae and all the developmental stages. Silkworm mGSTs were highly conserved with the other insect andRattusnorvegicusmicrosomal GSTs. It is worth to study whether the silkworm mGSTs contain the similar functions in mammals.
Key wordsBombyxmori; Microsomal GSTs; Expression pattern; Sequence analysis
Identification and sequence analysis of the microsomal GSTs in the silkworm
FANG Shou-min
(CollegeofLifeScience,ChinaWestNormalUniversity,Nanchong637002)
ABSTRACT
资助项目:四川省教育厅自然科学一般项目(14ZB0139)。
