蒙古沙冬青伴生植物AM真菌多样性
2016-07-02胡从从,郭清华,贺学礼等
蒙古沙冬青伴生植物AM真菌多样性
曹翠兰,女,高级实验师,主要从事植物学研究。E-mail:466213016@qq.com
胡从从1,郭清华1,贺学礼1,赵丽莉1,李英鹏1,曹翠兰2
(1.河北大学 生命科学学院,河北保定071002; 2.西北农林科技大学 生命科学学院,陕西杨凌712100)
摘要为探究蒙古沙冬青(Ammopiptanthus mongolicus)伴生植物丛枝菌根(AM)真菌遗传多样性特征,于2013年7月随机取样采集内蒙古乌海、磴口和乌拉特后旗3个样地霸王(Zygophyllum xanthoxylum)和白刺(Nitraria tangutorum)根围土壤,利用变性梯度凝胶电泳(DGGE)技术对AM真菌群落结构、生态分布及与土壤因子的相关性进行研究。结果表明:(1)共分离鉴定球囊霉属(Glomus)、管柄囊霉属(Funneliformis)和根内球囊霉属(Rhizophagus)3属AM真菌,其中,Glomus为所有样地和土层共有优势属,G.coronatum(AJ276086.2)和G. sp.(KC558550.1)为霸王根围优势菌种,Uncultured Funneliformis(HG004455.1)和Uncultured Rhizophagus(KC797139.1)为白刺根围优势菌种;G.coronatum(FR773145.1)是乌海特有菌种,G.sp.(AJ315521.1),Uncultured Rhizophagus(KF386282.1)和Uncultured Glomus(HG972422.1)为磴口特有菌种。(2)不同宿主、样地、土层AM真菌具有不同的DGGE图谱特征,丰度和多样性指数呈现蒙古沙冬青>霸王>白刺;同一样地,0~30 cm土层高于30~50 cm土层;同一土层,乌海、磴口均高于乌拉特后旗样地,其中乌海20~30 cm土层AM真菌丰度和多样性指数最高,分别为16和2.06。(3)土壤速效磷与AM真菌多样性显著正相关,碱解氮、碱性磷酸酶与AM真菌群落多样性显著负相关。可见,AM真菌能与宿主植物形成良好共生关系,具有丰富的遗传多样性;宿主植物、环境因子对AM真菌群落多样性有重要影响。
关键词蒙古沙冬青;伴生植物;AM真菌;PCR-DGGE;遗传多样性
中国西北荒漠带气候条件恶劣,使得该地区植被稀疏,植物种类组成与群落结构简单。蒙古沙冬青(Ammopiptanthusmongolicus)作为西北荒漠地区独有的旱生常绿豆科阔叶灌木,是砾质及沙砾质荒漠化草原的建群种,也是第三纪孑遗物种,具有古老的生长史和发展史[1]。霸王(Zygophyllumxanthoxylum)和白刺(Nitrariatangutorum)与蒙古沙冬青相伴而生,既有竞争抑制,又有协同进化,其根系发达,具有抗寒、抗旱、耐瘠薄等特点,是防风固沙的理想树种。
从枝菌根(Arbuscular Mycorrhiza,AM)真菌属于真菌界球囊霉门(Glomeromycota),目前包括3纲、5目、14科、26属[2],是一类能与高等植物根系形成共生体的土壤有益微生物。AM真菌作为生态系统的重要组成成分,在生物量生产、营养关系、物质循环、改善土壤理化性质、稳定和修复生态系统、提高植物生态系统生产力等方面起着重要作用,其种群演替对植物种间竞争、群落结构、植物多样性等有重要意义。
AM真菌资源和多样性研究主要通过形态学鉴定和分类,然而这种方法并不能客观反映AM真菌群落结构和生态分布的实际状况。近年来,运用分子生物学技术获得遗传信息,可用于认定新种,分析各种属之间的系统发育关系,确定某种真菌的分类地位,对形态学分类进行补充和完善[3]。变性梯度凝胶电泳(DGGE)技术在分析微生物区系遗传多样性方面发挥着重要作用,能够直接检测AM真菌群落结构变化和多样性的丰度[4]。目前,已有很多关于荒漠地区AM真菌在群落结构[5]、生态功能[6]及其与环境因子关系[7]等方面的研究。然而,利用现代分子手段对荒漠土壤AM真菌资源和多样性研究还很薄弱。
本试验利用PCR-DGGE及DNA克隆等技术,对荒漠植物根围土壤AM真菌分子特征进行研究,探讨AM真菌遗传多样性在宿主植物、样地和土层之间的变化规律,为进一步明确AM真菌群落组成和生态分布,以及与荒漠植物共生关系提供依据。
1材料与方法
1.1研究样地概况
本试验选取内蒙古乌海、磴口、乌拉特后旗3个样地,具体情况如表1所示。

表1 样地概况
1.2样品采集
在乌海、磴口、乌拉特后旗分别选取3个小样地,每个小样地选取长势良好的霸王、白刺各3株,在距植株主干0~30 cm内挖土壤剖面,分别采集0~10、10~20、20~30、30~40、40~50 cm土层土样。将土壤样品装入隔热袋中保存,风干后筛除土中的根与砂石,用于土壤理化性质分析,再将每个样地5个土层土样分别混匀,用于提取土壤总DNA。
1.3土壤DNA提取
提取方法采用已修改的Bead-Beating法[8]。
1.4巢式PCR与琼脂糖凝胶电泳
第1轮扩增采用引物对GeoA2 (5′-CCAGTAGTCATATGCTTGTCTC-3′) 和Geo11 (5′-ACCTTGTTACGACTTTTACTTCC-3′)扩增真菌18S rDNA序列;第2轮扩增以第1次PCR产物为模板进行,AM真菌特异性引物AM1(5′-GTTTCCCGTAAGGCGCCGAA-3′) 和NS31(5′-TTGGAGGGCAAGTCTGGTGCC-3′);第3轮以第2轮扩增产物为模板,真菌 18S rDNA NS31/Glo1 区通用引物对NS31-GC(5′-CGCCCGGGGCGCGCCCCGGGCGGGGCGGGGGC- ACGGGGGTTGGAGGGCAAGTCTGGTGCC-3′) ,Glo1 (5′-GCCTGCTTTAAACACTCTA-3′)。
利用8 g/L琼脂糖凝胶电泳检测扩增结果,取PCR扩增产物5 μL,90 V恒压电泳30 min,EB染色15 min。
1.5DGGE变性梯度凝胶电泳
1.5.1DGGE条件丙烯酰胺凝胶溶液体积分数为8%(丙烯酰胺∶双丙烯酰胺=37.5∶1),变性剂范围为30%~70%(100%变性剂为40%甲酰胺,7 mol/L尿素),电泳缓冲液为1×TAE,130 V、60 ℃电泳,时间8 h,银染法染色,用Bio-1D凝胶定量分析软件进行DGGE分析。
1.5.2DGGE图谱分析利用Quantity One软件对DGGE图谱的优势度、丰度(S)、均匀度(EH)和Shannon-Wiener多样性指数(H)进行统计分析,公式计算:
H=-∑Si=1(Pi)(lnPi)
EH=H/Hmax=H/lnS
式中,S表示每个样品条带数,Pi表示第i条条带优势度。
1.5.3DGGE胶回收与测序分析切割粗亮清晰、具有代表性的条带,加入30 μL ddH2O,4℃过夜,以上清液为模板,NS31/Glo1为引物进行PCR扩增,回收试剂盒回收纯化。将纯化产物连接到pGM-T载体上,转入E.coliDH5α感受态细胞中,挑选阳性克隆,检测后将菌液送去测序,测序结果在数据库中进行同源序列比对,并用MEGA6建立系统发育树。
1.6土壤理化性质测定
土壤理化性质依据鲁如坤[9]方法测定,土壤有机质采用重铬酸钾氧化法测定,速效磷用碳酸氢钠浸提-钼锑抗比色法测定,速效钾用火焰光度法测定,碱解氮用碱解扩散法测定,磷酸酶采用改进的Taba-tabai和Brimner方法[10]测定,球囊霉素依据Wright等[11]方法测定。
1.7数据处理与分析
所得数据经Excel 2003整理后,利用SPSS 19.0生物统计软件进行单因素方差分析(one-way ANOVA),采用Canoco 4.5软件进行PCA和RDA分析。
2结果与分析
2.1土壤总DNA提取与巢式PCR检测
共提取20个土壤样品DNA,经3轮PCR扩增后进行琼脂糖凝胶电泳检测。前2轮扩增后,由于DNA拷贝数低,在琼脂糖凝胶上无法观察到目标条带,第3轮PCR扩增(图1)能得到大量目标产物(270 bp),可用于DGGE电泳分析。
2.2DGGE图谱分析
对DGGE图谱分析可知,不同样品DGGE指纹图谱和灰度存在差异,泳道结构和条带数量有所不同。由图2-A可知,从霸王根围土壤共分离出20个条带,其中与第3泳道结构相似度较高的是第1、4泳道,相似度分别为64.4%和51.0%。条带2、4、5和20是所有样品的共有条带,条带10是乌海特有条带,15条带是乌拉特后旗40~50 cm土层的特有条带。从图2-B可知,在白刺根围土壤中共分离出21个条带,其中与第2泳道结构相似度较高的是第4、1、7泳道,分别为63.3%、62.2%和61.6%,条带2、3、16、21是10个样品的共有条带,条带7、8和12是磴口特有条带,条带10和19为磴口30~40 cm土层特有条带(图2-B)。

图1 第3轮PCR扩增产物的检测

图内数字表示分离的条带编号Figures showed that the separation of stripe number in picture;图2-A为霸王根围土壤样品电泳图Showed the electrophoresis figure of rhizosphere soil sample inZ.xanthoxylum,下面数字中1~5依次为乌海0~50 cm 5个土层样品0-50 cm soil layers in Wuhai,6~10是乌拉特后旗0~50 cm 5个土层样品0-50 cm soil layers in Wulate;图2-B为白刺根围土壤样品电泳图Showed the electrophoresis figure of rhizosphere soil sample inN.tangutorum,1~5依次为磴口0~50 cm 5个土层样品0-50 cm soil layers in Dengkou,6~10是乌拉特后旗0~50 cm 5个土层样品0-50 cm soil layers in Wulate
图2伴生植物根围土壤样品AM真菌DGGE泳道比较
Fig.2DGGE profile amplified from AM fungi of different soil samples under associated plants
2.3AM真菌群落结构相似性比较
利用Quantity One软件处理DGGE电泳图谱,得到各样品间AM真菌群落结构相似性图谱。由图3-A可知,相似性最高的是样品1和3,相似性指数为9.57;样品2和5,样品6和7的相似性指数分别为3.38和0.75;样品4和样品8相似性较低,指数为0.12。由图3-B可知,样品2和样品4、样品9和样品10、样品6和样品7相似性较高,相似性指数分别为14.58、7.57和3.96。通过分析图3,表明不同样地不同土层AM真菌群落组成具有一定的相似性。

图3-A为霸王根围土壤AM真菌群落相似性图谱Figure 3-A showed the community similarity profiles of AM fungi inZ.xanthoxylum,数字1~5依次为乌海0~50 cm 5个土层样品0-50 cm soil layers in Wuhai,6~10是乌拉特后旗0~50 cm 5个土层样品0-50 cm soil layers in Wulate;图3-B为白刺根围土壤AM真菌群落相似性图谱Showed the community similarity profiles of AM fungi inN.tangutorum,1~5依次为磴口0~50 cm 5个土层样品0-50 cm soil layers in Dengkou,6~10是乌拉特后旗0~50 cm 5个土层样品0-50 cm soil layers in Wulate
图3伴生植物根围土壤AM真菌群落相似性图谱
Fig.3Community similarity profiles of AM fungi under associated plants
2.4DGGE图谱丰度、多样性指数和均匀度
DGGE图谱中条带数量的多少能反映样品中微生物组成的差异[12]。由表2可知,乌海样地丰度随土层加深先增后降,最大值在20~30 cm土层;乌拉特后旗1丰度先减后升,在40~50 cm土层有最大丰度。磴口样地丰度变化呈“M”型,最大值在10~20 cm土层;乌拉特后旗2的丰度变化与乌海样地一致。多样性变化与丰度变化趋势保持一致,多样性指数基本维持为1.5~2.0;样地间和土层间均匀度无明显差异,均匀度指数基本保持为0.7~0.9。
2.5DGGE图谱优势度分析
DGGE图谱中条带的粗亮程度反映样品中微生物的含量,条带清晰粗亮,扩增后条带灰度值高,说明菌种在样品中含量丰富,是样品中的数量优势种[12]。由DGGE图谱分析可知,同一条带在不同样品中的优势度各有差异。由图4可见,条带2、4、5、20是乌海和乌拉特后旗样地共有优势条带。条带12是乌海样地优势带,优势度在10%以上;条带18、19是乌拉特后旗优势带,优势度在10%以上。图5表明,条带2、3、16、21是磴口和乌拉特后旗样地共有优势条带;条带4、6是磴口优势带,优势度在15%以上;乌拉特后旗的优势带还有条带18,优势度相对较高。
2.6条带测序与系统发育分析
由表3可知,在霸王根围土壤样品中选取15个条带进行测序,初步鉴定为3个属,包括球囊霉属(Glomus)、管柄囊霉属(Funneliformis)和根内球囊霉属(Rhizophagus)。在获得序列中有12条为Glomus,其中条带4、5、20为样品共有优势带;条带2也是共有优势带,属于Funneliformis,条带14和18属于Rhizophagus,这一结果表明Glomus是霸王AM真菌优势菌群,Funneliformis和Rhizophagus是常见属。其中,G.coronatum(AJ276086.2)和G. sp.(KC558550.1)为霸王根围优势菌种,G.coronatum(FR773145.1)是乌海特有菌种。系统发育显示(图6),条带4和条带15与G.coronatum(AJ276086.2)的相似度都在95%以上,亲缘关系较近,可以确定为一个菌种,这说明同一菌种在DGGE中会有不同迁移率。

表2 不同样品DGGE条带丰度和多样性指数

图4 霸王根围土壤样品DGGE条带优势度

图5 白刺根围土壤样品DGGE条带优势度

克隆编号Clonenumber条带BandNCBI数据库中最相似菌种登录号ThestrainswhichhavethehighestidentityfromNCBI(Accession)相似性/%SimilarityE值E-value12UnculturedFunneliformis(HG004455.1)974×10-9023UnculturedGlomus(HE614947.1)991×10-8934Glomuscoronatum(AJ276086.2)978×10-8745UnculturedGlomus(HF568595.1)972×10-8757Glomuscoronatum(AJ276086.2)943×10-46610Glomuscoronatum(FR773145.1)992×10-103711Glomussp.(FR715050.1)982×10-99812Glomusclarum(AJ505619.1)915×10-29913UnculturedGlomus(JF719636.1)982×10-991014UnculturedRhizophagus(KF386278.1)929×10-321115Glomuscoronatum(AJ276086.2)954×10-801216UnculturedGlomus(JN153048.1)954×10-151318UnculturedRhizophagus(KF386278.1)942×10-781419UnculturedGlomus(HF567355.1)933×10-311520Glomussp.(KC558550.1)921×10-34

采用Neighbour-joining方法绘制DGGE条带序列与近缘种的系统发育树,分支处的数值为支持强度值,1 000次重复,显示支持强度大于50%的分支强度值,图7同
Neighbour-joining tree showing the phylogenetic positions of the 23 sequences and representatives of some other related taxa,based on 18S rDNA NS31/Glo1 domain sequences.Numbers above nodes indicate the bootstrap values for analysis of 1 000 replicates,bootstrap values greater than 50% are shown,the same as Fig.7
图6霸王DGGE序列系统发育树
Fig.6Phylogenetic tree of separated sequences of DGGE ofZygophyllumxanthoxylon
由表4可知,在白刺根围土壤样品中分离鉴定出Glomus、Funneliformis和Rhizophagus3个属。其中,有9条归为Glomus,3条属于Funneliformis,3条为Rhizophagus,分析表明Glomus是白刺AM真菌的优势属,Funneliformis和Rhizophagus是常见属。其中,UnculturedFunneliformis(HG004455.1)和UnculturedRhizophagus(KC797139.1)是白刺根围优势菌种,G.sp.(AJ315521.1),UnculturedRhizophagus(KF386282.1)和UnculturedGlomus(HG972422.1)为磴口特有菌种。系统发育显示(图7),条带9和条带21与UnculturedRhizophagus(KC797139.1)相似度最高,亲缘关系非常接近,确定为一个菌种;条带18和条带19的亲缘关系非常接近,与UnculturedGlomus(KJ209983.1)相似性大,归为同一个种。
2.7土壤理化性质测定
2.7.1土壤因子空间分布由表5可知,霸王根围土壤因子变化呈现一定规律性。同一土层,乌海样地有机质高于乌拉特后旗;乌拉特后旗速效钾、速效磷、球囊霉素与磷酸酶高于乌海。乌海和乌拉特后旗0~30 cm土层速效钾均高于30~50 cm土层;乌海0~20 cm土层碱解氮显著高于其他土层;乌拉特后旗速效磷和EEG在0~30 cm土层显著高于30~50 cm土层。
白刺根围土壤因子变化也呈现出一定规律性。除0~10 cm土层外,磴口样地有机质高于乌拉特后旗;同一土层,乌拉特后旗速效钾、总球囊霉素(TEG)、磷酸酶、碱解氮均高于磴口样地。磴口酸性磷酸酶在0~20 cm土层显著高于其他土层,0~10 cm土层碱性磷酸酶显著高于其他土层;乌拉特后旗2种磷酸酶均在20~30 cm土层有最大值。
综合分析表明,霸王和白刺0~30 cm土层各项土壤因子均高于30~50 cm土层,其中土壤有机质、速效钾、速效磷、酸性磷酸酶等在霸王根围土壤更高,而球囊霉素、碱性磷酸酶在白刺根围土壤中较高。

表4 白刺根围土壤DGGE条带序列Blast结果
2.7.2土壤因子主成分分析(PCA法)在PCA排序图中,每个样方点投影到某一土壤因子箭头的点,其相对位置代表该土壤因子在这些样方中的排序情况,样方点之间的距离越近表示它们之间关系越密切。由图8可知,速效磷是乌海样地的主要影响因子,碱解氮、磷酸酶是乌拉特后旗样地的重要影响因子。前两轴累积解释信息量达87.9%,其中第1轴解释信息量为76.3%,第2轴排序轴为11.6%。分析可知,土壤速效磷、碱解氮、磷酸酶是3个样地的主要影响因子,反映环境的土壤肥力情况。

图7 白刺DGGE序列系统发育树

样地Samplesite土层/cmSoillayer有机质/(mg/g)Organicmatter速效钾/(μg/g)AvailableK速效磷/(μg/g)AvailableP易球囊霉素/(mg/g)EEG总球囊霉素/(mg/g)TEG酸性磷酸酶/[μg/(g·h)]ACP碱性磷酸酶/[μg/(g·h)]ALP碱解氮/(μg/g)AvailableN乌海Wuhai0~1012.22a7.80a0.22a1.26a1.77a12.58a12.58a12.83a10~2014.44a7.62a0.21a1.20a1.62a8.74a8.74b10.50ab20~3013.33a7.10ab0.24a0.98a1.75a12.70a12.70a9.33b30~4012.22a5.80b0.20a0.77a1.18a11.56a11.56ab10.11b40~5013.33a6.15b0.17a1.12a1.44a10.64a10.64b9.33b乌拉特后旗10~1012.22ab8.40a0.21a1.62a2.02a33.91a31.52a8.94aWulate110~2010.00b7.73ab0.22a1.35ab1.88a29.80b23.39ab12.44a20~3011.11ab7.14ab0.16ab1.14b2.09a20.36c16.19b11.28a30~4012.22ab6.03c0.11b0.87b1.52a14.62d25.47ab7.39a40~5014.44a6.18bc0.11b1.35ab1.75a19.33c29.58ab9.72a磴口Dengkou0~1010.00b9.26a0.19a1.70a1.82a16.79a19.90a10.50ab10~2012.22ab6.53b0.21a1.41a1.75a10.57ab9.08b7.00bc20~3014.44a3.64c0.18a1.34a1.76a6.97b7.11b5.83c30~4014.44a6.06b0.16a1.49a1.96a7.19b4.96b4.67c40~5012.22ab3.80c0.15a1.12a1.81a6.68b15.84ab12.83a乌拉特后旗20~1013.33a9.50a0.15b1.50a2.09a17.29c20.73b14.17abWulate210~2011.11ab7.67ab0.30a1.49a1.85a17.87b23.59a11.00c20~3011.11ab5.62b0.17ab1.42a1.84a23.54a24.64a15.83a30~4011.11ab6.84ab0.14b1.36a1.89a20.83ab23.05ab13.11bc40~5010.00b7.17ab0.19ab1.50a2.30a23.01a22.32ab15.78a
注:同列数据后不同字母表示同一样地不同土层在0.05水平上差异显著。
Note:Data with different superscripts in the diferent soil depth of same site indicate significant difference at 0.05 level.
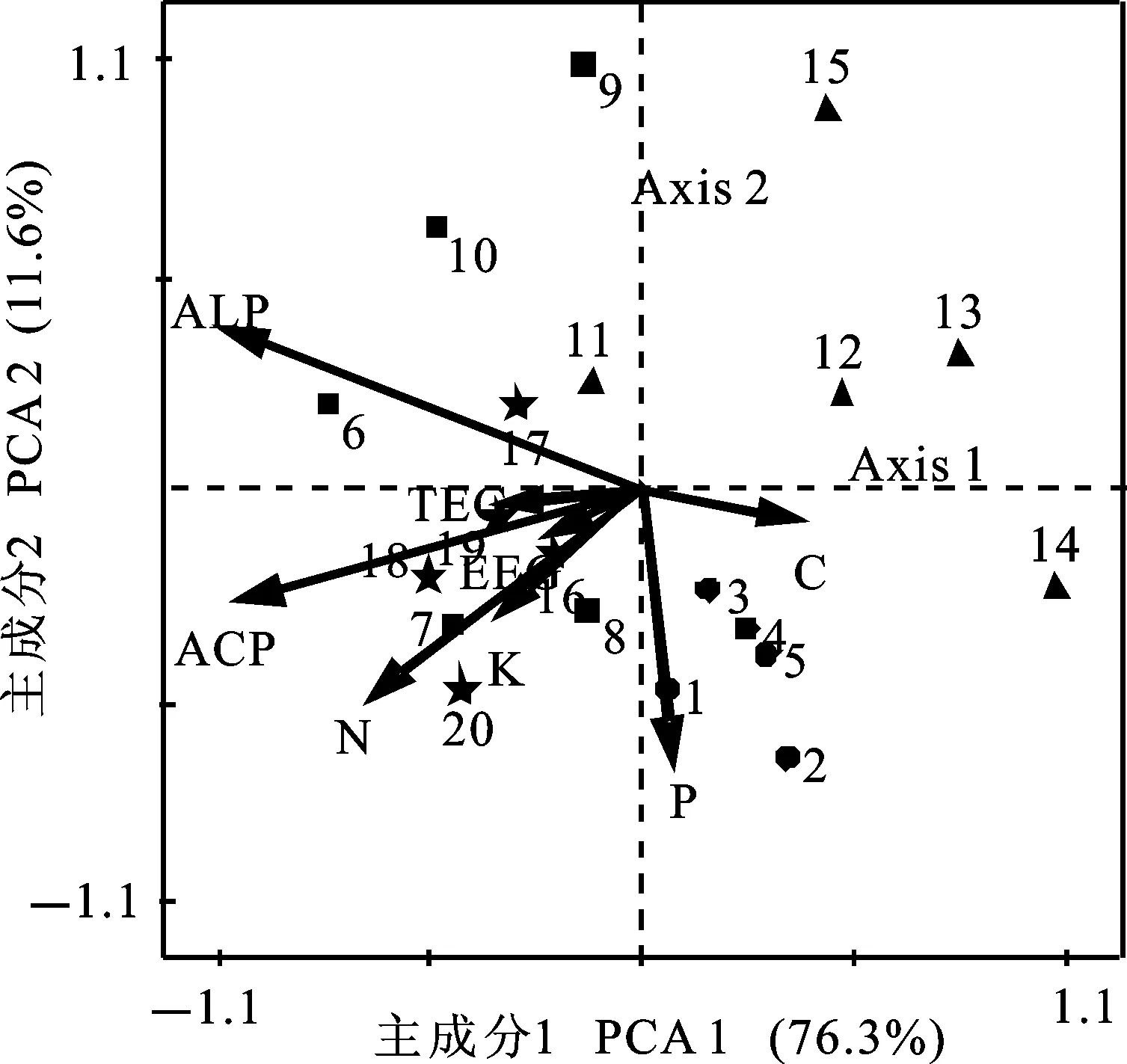
图中各形状代表样地Shape shows sampling site;1~5、6~10、11~15、16~20分别表示乌海、乌拉特后旗1、磴口、乌拉特后旗2样地0~50 cm 5个土层0-50 cm soil layers in Wuhai,Wulate 1,Dengkou,Wulate 2;图9同The same as Fig.9
图8土壤因子PCA排序图
Fig.8Results of PCA analysis of soil factors
2.8AM真菌多样性与土壤因子的冗余分析
在RDA排序图内,土壤因子与物种的箭头方向和夹角表示它们之间的相关性。同向箭头表示正相关,反向表示负相关;夹角越小则相关性越高,夹角越接近直角则相关性越小。由图9可见,前两轴累积解释信息量达100%,其中第1轴排序轴达96.1%,第2轴排序轴为3.9%。土壤速效磷与AM真菌丰度和Shannon-Weiner指数关系显著正相关;碱解氮、碱性磷酸酶与AM真菌群落多样性显著负相关。
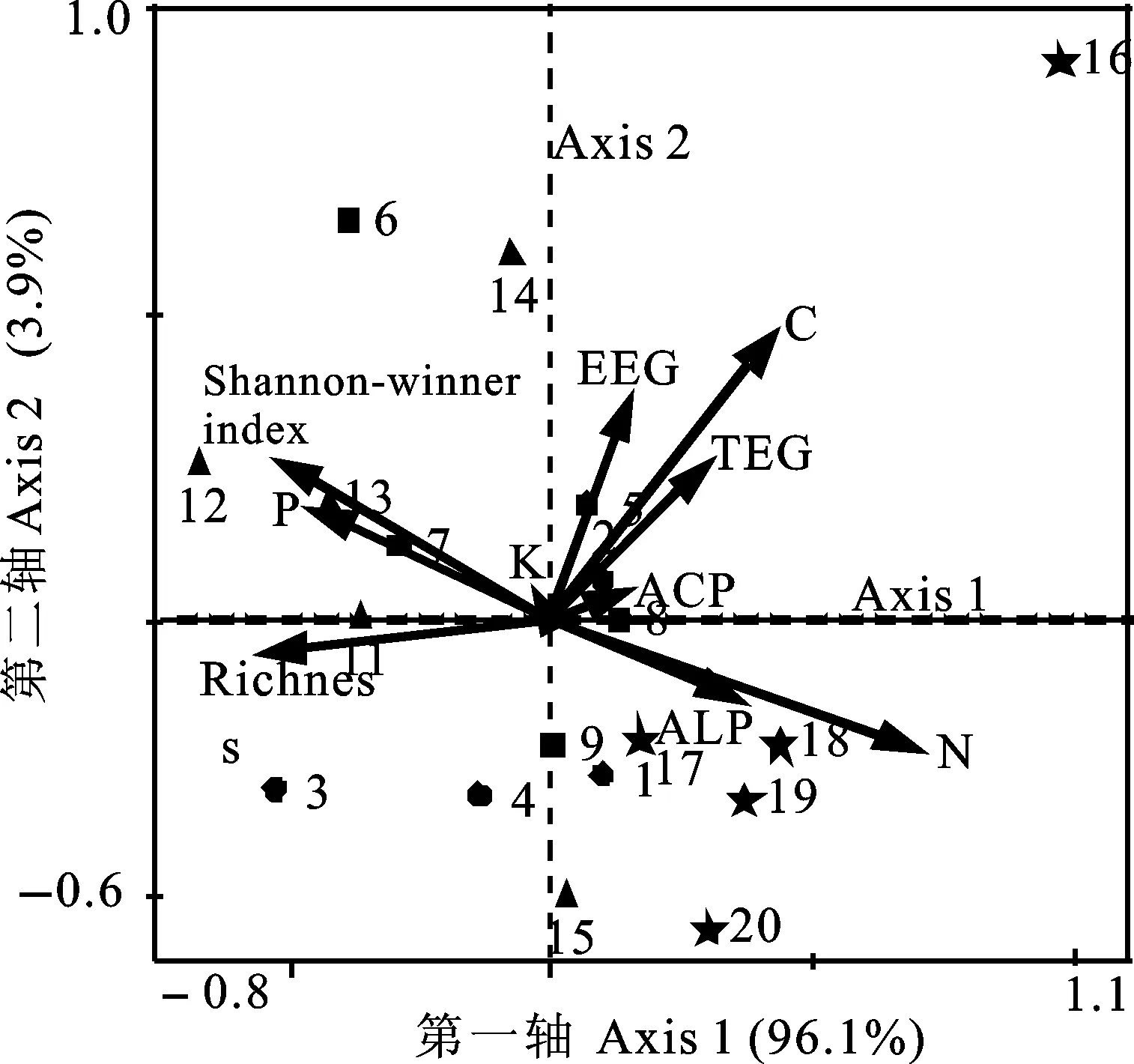
图9 AM真菌多样性与土壤因子的RDA分析
3讨 论
3.1AM真菌遗传多样性特征
研究结果显示,霸王和白刺根围土壤AM真菌群落结构、生态分布具有很高相似度。试验共分离鉴定球囊霉属(Glomus)、管柄囊霉属(Funneliformis)和根内球囊霉属(Rhizophagus)3个属,球囊霉属为3样地共有优势属,在5个土层均有分布,说明Glomus在内蒙古荒漠地区占据重要生态位并广泛分布。由于受GenBank中序列信息不完整的限制,在所测得的30条序列中,只有10个鉴定到种,包括G.coronatum、G. sp.和G.clarum。
未分离出形态学所鉴定的无梗囊霉属、盾巨孢囊霉属和内养囊霉属,这与所选引物特异性有很大关系。AM1/NS31引物对球囊霉属有明显偏扩效应,一些种属AM真菌不能被扩增出来[13-14];NS31/Glo1引物对可以扩增出一些非AM真菌的微生物[15]。也有研究表明,DGGE法只能对微生物群落中数量大于1%的优势种群进行分析[16]。在DGGE中,虽然迁移率不同,但几个条带可能代表同一菌种,一个条带也可能包含几个种的信息,因此不能确定AM真菌的数量和种类等。这些因素都会影响AM真菌种类分离鉴定和对其种群组成分布的研究。
3.2宿主植物与AM真菌遗传多样性的关系
AM真菌属于专性共生真菌,宿主植物能提供AM真菌生长发育所需的碳水化合物,对AM真菌生长发育具有重要影响[17]。试验结果与蒙古沙冬青AM真菌多样性对比发现,霸王、白刺和蒙古沙冬青根围土壤AM菌群的优势属均为球囊霉属,这与荒漠地区AM真菌多样性研究结果一致[18-19]。3种植物AM真菌群落结构具有很高的相似度,但在丰度和多样性指数上,蒙古沙冬青>霸王>白刺。这可能是蒙古沙冬青较伴生植物更早的生活在荒漠生态系统中,与大多数AM真菌长期协同进化和相互选择的结果。对植物根系进行对比发现,沙冬青和霸王的根系比白刺根系更发达粗壮、入土更深,这可能也是造成多样性差异的一个因素。有研究证实,宿主类型和根系特性是影响AM真菌发生和分布的重要因子[20],不同植物根系形态与生理代谢的差异能够影响AM真菌的生长、发育、侵染和繁殖等[21]。
3.3土壤因子与AM真菌遗传多样性的关系
土壤因子对AM真菌的群落组成、生态分布等有重要影响。本研究中乌海和磴口样地AM真菌丰度、Shannon-Wiener多样性指数均高于乌拉特后旗,说明AM真菌多样性与样地土壤环境和土壤肥力有着密切关系。冗余分析结果显示,速效磷是主要土壤因子,对AM真菌的丰度、多样性指数有最大影响;碱解氮、碱性磷酸酶与AM真菌种类呈负相关。Smith等[22]研究发现,在矿质养分中,土壤磷素密切影响着AM真菌的丰度,速效磷过低或过高都会抑制AM真菌生长发育。
有研究表明,土壤深度是AM真菌多样性的驱动力[23],对AM真菌群落结构具有一定影响。本试验中,0~30 cm土层AM真菌丰度和多样性指数高于30~50 cm土层,可能是因为表层土壤的湿度和透气性好,营养物质丰富,为好气性AM真菌的生长发育提供适宜条件[24],这与蒙古沙冬青AM真菌遗传多样性研究结果[25]相同。
综合表明,霸王和白刺根围土壤AM真菌遗传多样性较蒙古沙冬青低,说明蒙古沙冬青与AM真菌适应性更强,更能适应极端荒漠环境。AM真菌能与荒漠植物形成良好的共生关系,增强植物抗逆性,这对荒漠植物生长和生态系统修复具有重要意义。
参考文献Reference:
[1]李昌龙,尉秋实,李爱德.孑遗植物沙冬青的研究进展与展望[J].中国野生植物资源,2004(5):21-23.
LI CH L,WEI Q SH,LI A D.Study and application of the endangered plantAmmopiptanthus[J].ChineseWildPlantResources,2004(5):21-23(in Chinese).
[2]王宇涛,辛国荣,李韶山.丛枝菌根真菌最新分类系统与物种多样性研究概况[J].生态学报,2013,33(3):834-843.
WANG Y T,XIN G R,LI SH SH.An overview of the updated classification system and species diversity of arbuscular mycorrhizal fungi[J].ActaEcologicaSinica,2013,33(3):834-843(in Chinese with English abstract).
[3]贺学礼,王雅丽,赵丽莉.河北安国7种中药材AM真菌遗传多样性研究[J].中国生态农业学报,2012(2):144-150.
HE X L,WANG Y L,ZHAO L L.AM fungal genetic diversity in seven medicinal plant rhizospheres in anguo city of Hebei province[J].ChineseJournalofEco-Agriculture,2012(2):144-150(in Chinese with English abstract).
[4]朱建林,詹鹏,吕文洲.聚合酶链反应-变性梯度凝胶电泳技术研究梯田式人工湿地微生物群落动态变化[J].环境污染与防治,2010(2):46-50.
ZHU J L,ZHAN P,LÜ W ZH.Research on microorganism community dynamics in terrace-type constructed wetland by PCR-DGGE[J].EnvironmentalPollution&Control,2010(2):46-50(in Chinese).
[5]钟凯,袁玉清,赵洪海,等.泰山丛枝菌根真菌群落结构特征[J].菌物学报,2010(1):44-50.
ZHONG K,YUAN Y Q,ZHAO H H,etal.Arbuscular mycorrhizal fungal community structure in rhizospheric soil of Taishan vegetation[J].Mycosystema,2010(1):44-50(in Chinese with English abstract).
[6]杨海霞,郭绍霞,刘润进.盐碱地生境中丛枝菌根真菌多样性与功能变化特征[J].应用生态学报,2015,26(1):311-320.
YANG H X,GUO SH X,LIU R J.Characteristics of arbuscular mycorrhizal fungal diversity and functions in saline-alkali land[J].ChineseJournalofAppliedEcology,2015,26(1):311-320(in Chinese with English abstract).
[7]ZHANG H Q,TANG M,CHEN H,etal.Communities of arbuscular mycorrhizal fungi and bacteria in the rhizosphere ofCaraganakorshinkiiandHippophaerhamnoidesin Zhifanggou watershed[J].PlantandSoil,2010,326:415-424.
[8]YEATES C,GILLING M R.Methods for microbial DNA extraction from soil for PCR amplification[J].BiologicalProceduresOnline,1998(1):40-47.
[9]鲁如坤.土壤农业化学分析方法[M].北京:中国农业科技出版社,2000.
LU R K.Chemical Analysis of Soil Agriculture[M].Beijing:China Agricultural Science and Technology Press,2000(in Chinese).
[10]宋勇春,李晓林,冯固.菌根真菌磷酸酶活性对红三叶草生境中土壤有机磷亏缺的影响[J].生态学报,2001,21(7):1130-1135.
SONG Y CH,LI X L,FENG G.Effect of phosphatase activity on soil organic phosphorus loss in the environment of clover growth[J].ActaEcologicaSinica,2001,21(7):1130-1135(in Chinese with English abstract).
[11]WRIGHT S F,UPADHYAYA A.A survey of soils for aggregate stability and glomalin,a glycoprotein produced by hyphae of arbuscular mycorhizal fungi[J].PlantandSoil,1998,198(1):97-107.
[12]江云飞,蔡柏岩.PCR-DGGE技术在细菌多样性研究中的条件优化[J].生物技术,2009(5):84-87.
JIANG Y F,CAI B Y.Condition optimization of PCR-DGGE in the research of bacterial diversity[J].Biotechnology,2009(5):84-87(in Chinese).
[13]DANIELL T J,HUSBAND R,FITTER A H,etal.Molecular diversity of arbuscular mycorrhizal fungi colonising arable crops[J].FEMSMicrobiologyEcology,2001,36:203-209.
[14]REDECKER D,MORTON J B,BRUNA T D.Molecular phylogeny of the arbuscular mycorrhizal fungiGlomussinuosumandSclerocystiscoremioides[J].Mycologia,2000,92(2):282-285.
[15]王雪,接伟光,蔡柏岩.不同生境黄檗AM真菌菌群结构分析[J].林业科学,2012(9):99-107.
WANG X,JIE W G,CAI B Y.Community composition of the AM fungi ofPhellodendronamurensein different habitats[J].ScientiaSilvaeSinicae,2012(9):99-107(in Chinese).
[16]MUYZER G,DEWAAL E C,UITTERLINDEN A G.Profiling of complex microbial populations by denaturing gradientgel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J].ApplyEnvironmentalMicrobiology,1993,59:695-700.
[17]陈宁,王幼珊,李晓林,等.营养液强度对AM真菌生长发育的影响[J].菌物系统,2003(3):394-401.
CHEN N,WANG Y SH,LI X L,etal.The influence of the intensity of nutrient solution of AM fungi growth[J].Mycosystema,2003(3):394-401(in Chinese).
[18]张玉洁,贺学礼,程春泉,等.新疆沙冬青AM真菌群落结构与遗传多样性分析[J].菌物学报,2015(3):375-385.
ZHANG Y J,HE X L,CHENG CH Q,etal.Community composition and genetic diversity of arbuscular mycorrhizal fungi in the rhizosphere ofAmmopiptanthusnanusin Xinjiang desert area[J].Mycosystema,2015(3):375-385(in Chinese).
[19]冀春花,张淑彬,盖京苹,等.西北干旱区AM真菌多样性研究[J].生物多样性,2007(1):77-83.
JI CH H,ZHANG SH B,GE J P,etal.Arbuscular mycorrhizal fungal diversity in arid zones in northwestern China[J].BiodiversityScience,2007(1):77-83(in Chinese).
[20]ILEANA V G,RODOLFO E M.Arbuscular mycorrhizal fungi and plant symbiosis in a saline-sodic soil[J].Mycorrhiza,2007,17:167-174.
[21]王发园,刘润进.生物因子对AM真菌多样性的影响[J].生态学报,2002,22(3):403-408.
WANG F Y,LIU R J.Effects of biological factors on the diversity of arbuscular mycorrhizal fungi[J].ActaEcologicaSinica,2002,22(3):403-408(in Chinese with English abstract).
[22]SMITH S E,READ D J.Mycorrhizal Symbiosis[M].Cambridge,UK:Academic Press,2008.
[23]PEREZ-NARANJO J C.Dark septate and arbuscular mycorrhizal fungal endophytes in roots of prairie grasses[D].Saskatchewan:University of Saskatchewan,2009.
[24]贺学礼,陈程,何博.北方两省农牧交错带沙棘根围AM真菌与球囊霉素空间分布[J].生态学报,2011,31(6):1653-1661.
HE X L,CHEN CH,HE B.Spatial distribution of arbuscular mycorrhizal fungal and glomalin ofHippophaerhamnoidesL in farming-pastoral zone from the two northern provinces of China[J].ActaEcologicaSinica,2011,31(6):1653-1661(in Chinese with English abstract).
[25]程春泉,贺学礼,赵金莉,等.蒙古沙冬青根围土壤AM真菌PCR-DGGE分析[J].西北农业学报,2014,23(12):175-183.
CHENG CH Q,HE X L,ZHAO J L,etal.PCR-DGGE analysis of AM fungi in the rhizosphere soil ofAmmopiptanthusmongolicus[J].ActaAgriculturaeBoreoli-occidentalisSinica,2014,23(12):175-183(in Chinese with English abstract).
Received 2015-06-27Returned2015-10-15
First authorHU Congcong,female,master.Research area:mycorrhizal biology.E-mail:hcc3004@163.com
CAO Cuilan,female,senior experimentalist.Research area:botany.E-mail:466213016@qq.com
(责任编辑:成敏Responsible editor:CHENG Min)
Diversity of Arbuscular Mycorrhiza Fungi near to the Associated Plants ofAmmopiptanthusmongolicus
HU Congcong1,GUO Qinghua1,HE Xueli1,ZHAO Lili1,LI Yingpeng1and CAO Cuilan2
(1.College of Life Sciences,Hebei University,Baoding Hebei 071002,China;2.College of Life Sciences,Northwest A&F University,Yangling Shaanxi712100,China)
AbstractAmmopiptanthus mongolicus is an excellent sand-fixation and endangered desert ecosystem of leguminous plants.Zygophyllum xanthoxylum and Nitraria tangutorum are associated plants of A.mongolicus that can not only adapt to the desert environment but also repair the ecological system.In order to explore the community composition and genetic diversity of AM fungi in the rhizosphere soil of Z.xanthoxylum and N.tangutorum by PCR-DGGE,in July 2013,the soil samples were collected from three different sites in Wuhai,Dengkou and Wulate of Inner Mongolia,China.Results showed that:(1) AM fungi have abundant genetic diversity.Three genera,including Glomus,Funneliformis and Rhizophagus,were isolated and identified in the rhizosphere soil of Z.xanthoxylum and N.tangutorum.Glomus was dominant genus of all the sample area and soil layer.The superiority strains of Z.xanthoxylum were G.coronatum(AJ276086.2)and G. sp.(KC558550.1).Uncultured Funneliformis(HG004455.1)and Uncultured Rhizophagus(KC797139.1)were the dominant species of N.tangutorum.G.coronatum(FR773145.1)was a special species in Wuhai.G.sp.(AJ315521.1).Uncultured Rhizophagus(KF386282.1)and Uncultured Glomus(HG972422.1)were unique species in Dengkou.(2) The fingerprint characteristics of DGGE,richness,diversity index and dominance of AM fungi varied with host plants,sampling sites and soil depth.Richness and diversity index of AM fungi were higher under A.mongolicus than under the associated plants and in 0-30 cm soil layer than that in 30-50 cm soil layer.For the same soil layer,the richness and diversity index of AM fungi were sequenced in a descending order in Wuhai,Dengkou,and Wulate.The highest richness and diversity index at 20-30 cm soil layer in Wuhai,reaching 16 and 2.06,respectively.(3) Soil available phosphorus had a positive correlation with AM fungi diversity.Available nitrogen,alkaline phosphatase had significantly negative correlation with AM fungi community diversity.The results reflected AM fungi can form good symbiosis with the host plant and have abundant genetic diversity.The host plants and environmental factors significantly influenced the community composition and ecological distribution of AM fungi.
Key wordsAmmopiptanthus mongolicus; Associated plants; AM fungi; PCR- DGGE; Genetic diversity
收稿日期:2015-06-27修回日期:2015-10-15
基金项目:国家自然科学基金(31170488)。
通信作者:贺学礼,男,教授,主要从事植物多样性和菌根生物学研究。E-mail:xuelh1256@aliyun.com
中图分类号Q933
文献标志码A
文章编号1004-1389(2016)06-0921-12
Foundation itemNational Natural Science Foundation of China (No.31170488). HE Xueli,male,professor.Research area:plants diversity and mycorrhizal biology.E-mail:xuelh1256@aliyun.com
网络出版日期:2016-06-01
网络出版地址:http://www.cnki.net/kcms/detail/61.1220.S.20160601.0919.038.html
第一作者:胡从从,女,硕士研究生,从事菌根生物学研究。E-mail:hcc3004@163.com
