文蛤C型凝集素基因(Mm-Lec1)的克隆与表达分析
2016-07-01张晶晶李宏俊秦艳杰刘敏叶晟
张晶晶,李宏俊, 秦艳杰, 刘敏,叶晟
(1. 大连海洋大学 水产与生命学院,辽宁 大连 116023;2. 国家海洋环境监测中心 海洋生态室,辽宁 大连 116023)
文蛤C型凝集素基因(Mm-Lec1)的克隆与表达分析
张晶晶1,2,李宏俊2*, 秦艳杰1, 刘敏1,2,叶晟1,2
(1. 大连海洋大学 水产与生命学院,辽宁 大连 116023;2. 国家海洋环境监测中心 海洋生态室,辽宁 大连 116023)
摘要:文蛤(Meretrix meretrix)是我国重要的滩涂养殖贝类,病害严重影响文蛤增养殖业,研究文蛤的免疫机制有助于解决文蛤的病害问题。C型凝集素(C-type lectin)参与先天免疫,在识别病原相关分子模式和激活体液免疫因子等方面发挥重要作用。本研究检索已构建的文蛤全长cDNA 文库,经过Blast比对得到了文蛤C型凝集素1(Mm-Lec1)基因的全长cDNA序列。Mm-Lec1序列全长586 bp,5`和3`非翻译区(UTR)的长度分别为21 bp和79 bp,开放阅读框长度为486 bp,编码161个氨基酸,分子量为18.65 kD,理论等电点为4.98。预测的氨基酸序列中含有信号肽(Met1-Ser19)、糖识别结构域(CRD)和糖结合位点(QPN)。Mm-Lec1的三级结构是紧凑型,含有β片层结构。同源性分析结果表明,Mm-Lec1与其他物种C型凝集素相似度在20%~32%;邻接法(Neighbor-Joining, NJ)进化树分析结果表明,Mm-Lec1与紫贻贝CTL 6和栉孔扇贝CTL A聚为一支。实时荧光定量分析结果显示,Mm-Lec1在文蛤鳃、肝胰腺、闭壳肌、外套膜、性腺和血细胞中均表达,其中鳃表达量最高,血细胞次之,性腺中表达量最少;在鳗弧菌(Vibrio anguillarum)刺激实验中,6 h时Mm-Lec1在血细胞中的表达量最低,48 h表达量最高,暗示Mm-Lec1参与文蛤抵御细菌入侵的免疫过程。
关键词:文蛤;C型凝集素;表达序列标签;三级结构;实时荧光定量PCR
1引言
文蛤(Meretrixmeretrix)主要分布于广西北部湾、江苏南部、山东蓬莱湾及辽宁辽河口等海区[1],是我国重要的滩涂经济贝类,也是主要的出口鲜活水产品之一。近年来,病害问题严重制约我国文蛤增养殖业发展,文蛤病害暴发具有明显的区域性、季节性和流行性[2],常常导致文蛤大规模死亡,成为文蛤养殖业遇到的最大难题[3—4]。导致贝类病害暴发的原因是多方面的,是外界病原、环境和贝类生物自身共同作用的结果。研究文蛤抵御病害机制,探讨贝类自身免疫防御机理,可以为文蛤抗病选育提供理论基础。
C型凝集素(C-type lectin, CTL)代表一个依赖于钙离子(Ca2+)参与的与糖原结合的蛋白家族,含有1个或多个一级结构和二级结构同源的碳水化合物识别结构域。C型凝集素根据与其结合的配体不同可分为Man型配体和Gal型配体,前者与D-甘露糖、D-葡萄糖结合,在氨基酸序列中对应的三氨基分别是EPN和EPD;后者与D-半乳糖及其衍生物结合,在氨基酸中对应的三氨基分别是QPD和QPN[5—6]。在先天免疫应答中,C型凝集素配体与外来病原菌糖原结合,中和病原菌的致病毒性,通过补体途径或者细胞吞噬途径消灭病原菌[7]。CTL在贝类先天免疫中发挥重要作用,在栉孔扇贝(Chlamysfarreri)[5],合浦珠母贝(Pinctadafucata)[8],海湾扇贝(Argopectenirradias)[9]和偏顶蛤(Modiolusmodiolus)[10]抵御细菌,海湾扇贝损伤愈合[9],凡纳滨对虾(Litopenaeusvannamei)[11]和斑节对虾(Penaeusmonodon)[12]抵御病毒入侵和文蛤(M.meretrix)耐盐和耐高温[13]生理响应中发挥作用。
目前,已报道CTL基因的软体动物包括紫贻贝(Mytilusgalloprovincialis)[14],栉孔扇贝[15—16],海湾扇贝[9,17—18]和合浦珠母贝(P.fucata)[8,19],太平洋牡蛎(Crassostreagigas)[20—21]、美洲牡蛎(Crassostreavirginica)[22]、和皱纹盘鲍(Haliotisdiscus)[23—24]等。本研究克隆了文蛤Mm-Lec1基因的全长cDNA序列,分析了Mm-Lec1基因mRNA在各组织分布情况和病原菌刺激下的表达规律,这将为进一步研究CTL基因在软体动物中的免疫功能提供基础资料。
2材料与方法
2.1材料
试验用文蛤采自辽宁省盘山县辽河口,在室内的60 L的塑料箱中暂养2周后,选取活力较好、壳长均匀(4.0~5.0 cm)的文蛤进行实验。对于组织表达实验,选取3只闭壳肌活力强的文蛤分别取血细胞、鳃、闭壳肌、性腺、外套膜和肝胰腺组织,保存于液氮中备用。对于鳗弧菌(virbrioanguillarum)浸染实验,设置对照组和实验组,实验组海水鳗弧菌终浓度为4×107cfu/mL,在刺激0 h、6 h、12 h、24 h、48 h、72 h和96 h用1 mL注射器采集闭壳肌的血细胞,每个时间点设置3个重复,经4℃,5 500 r/min离心10 min处理后,保存于液氮中备用。
2.2文蛤C 型凝集素基因序列的获得
本课题组已构建了文蛤3个组织的全长cDNA文库,共得到3 129条高质量的EST序列[25—26],在NCBI (http://www.ncbi.nlm.nih.gov/)网站利用tblastx进行同源序列搜索比对,获得文蛤C型凝集素的全长cDNA序列。
2.3文蛤C型凝集素基因序列的生物信息学分析
应用ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)程序确定正确的开放阅读框,并运用BioEdit软件翻译成氨基酸序列;采用InterProScan 5(http://www.ebi.ac.uk/Tools/pfa/iprscan5/)预测氨基酸序列中的疏水区;采用TMHMM在线服务器(http://www.cbs.dtu.dk/services/TMHMM/)预测蛋白跨膜区;用SignalP4.1 Server软件 (http://www.cbs.dtu.dk/services/SignalP/)预测信号肽;采用SMART(http://smart.embl-heidelberg.de/)在线服务软件预测功能域;采用DiANNA(http://clavius.bc.edu/~clotelab/DiANNA/)预测二硫键的个数;采用ExPASy SWISSMODEL软件 (http://swissmodel.expasy.org/interactive)预测空间结构,采用RasTop软件查看模型;运用在线软件TargetP 1.1(http://www.cbs.dtu.dk/services/TargetP/)对蛋白质进行定位;通过NCBI (http://www.ncbi.nlm.gov)中的tblastx工具进行蛋白质序列相似性检索;用ClustalX 2.1软件与GeneDoc软件联合进行多序列比对;应用ClustalX 2.1软件与MEGA 5.0软件构建NJ系统进化树;用在线ClustalW 2服务器(http://www.ebi.ac.uk/Tools/msa/clustalw2/)分析蛋白质序列的相似性。
2.4文蛤C型凝集素的组织分布表达与时序表达
按照天根的提取RNA的试剂盒说明书提取RNA,采用生工的反转录试剂盒获得cDNA。依照宝生物的SYBR Premix Ex Taq kit试剂盒说明书,以cDNA为模板,配置20 μL的扩增体系,同时设置ABI7500系统的扩增程序。为了确保引物的特异性,我们增加了溶解曲线的扩增。为了减少实验的误差,每个组织或时间点重复3次实验。利用Primer Premier 5.0设计C型凝集素和β-actin的特异性引物(表1)。采用2-△△Ct法计算Mm-Lec1基因的相对表达量,以β-actin为内参基因,使用One way ANOVA分析实验组与对照组的Mm-Lec1基因的相对表达量的显著性差异。
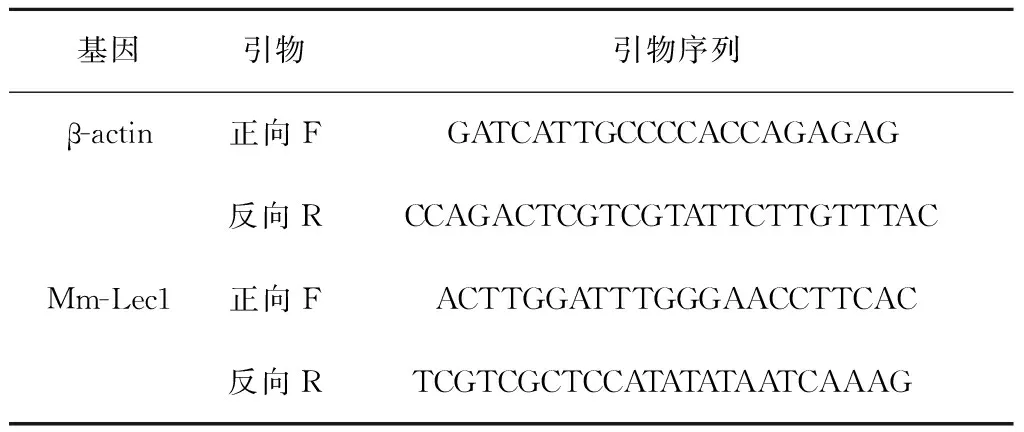
表1 β-actin和Mm-Lec1的特异性引物
3结果
3.1Mm-Lec1序列分析
Mm-Lec1基因的全长cDNA序列为586 bp(GenBank注册号KT276515),3′端有polyA尾巴,5′和3′非翻译区(UTR)的长度分别为21 bp和79 bp,开放阅读框长度为486 bp,编码161个氨基酸。Mm-Lec1氨基酸序列Met1-Ser19为信号肽序列,Ser19和Ala20之间存在剪切位点(图1)。氨基酸序列包含1个糖识别结构域(CRD),CRD包含6个保守的半胱氨酸(Cys21,Cys32,Cys49,Cys125,Cys141和Cyst150)和1个糖结合位点QPN(Gln115,Pro116,Asn117)(图1)。按照标准密码子翻译后的蛋白质推测分子式为C841H1227N217O245S11,分子量计算值为18.65 kD,理论等电点为4.98。极性与极性带电氨基酸共99个(占61.5%),非极性氨基酸62个(占38.5%),极性氨基酸比较大,决定了Mm-Lec1氨基酸具有较好的水溶性。
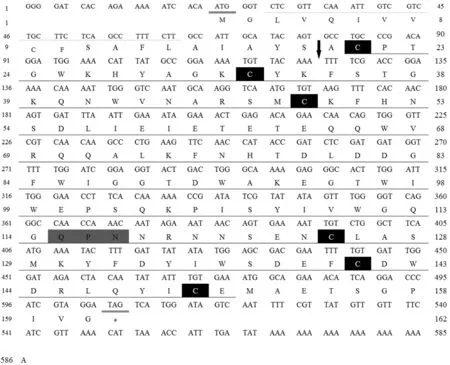
图1 Mm-Lec1基因的核苷酸序列和推测的氨基酸序列Fig.1 The full cDNA and predicted amino acid sequence of Mm-Lec1 gene双下划线代表起始密码子和终止密码子;带有下划线的氨基酸序列是C型凝集素家族糖识别结构域(CRD)序列;带有虚线下划线的氨基酸是疏水氨基酸;黑色背景表示形成二硫键的半胱氨酸;灰色阴影代表糖结合位点;↓代表信号肽剪切位点The start and stop codons are double line underlined; the carbohydrate recognition domain (CRD) is underlined; hydrophobic amino acids are dotted line underlined; cysteines constituting disulfide bonds are in black box; the sugar binding sites are in grey shadow re-gion; ↓ is the token of signal peptide splice site
3.2蛋白亚细胞定位分析
TMHMM预测前60个氨基酸含有疏水氨基酸,存在跨膜区氨基酸的预测值较高(14.5>10),跨膜螺旋结构的预测值低(14.5<18)。InterProScan 5估测Leu3-Leu14为疏水区。TargetP 1.1在线服务器估算Mm-Lec1是分泌蛋白的概率是93.4%。
3.3同源比对、相似度和系统树
从23条C型凝集素基因比对图(图2)发现6个位点的氨基酸是完全一样的,其中5个是半胱氨酸。ClustalW 2在线服务器计算得出文蛤与紫贻贝C-type lectin 6的相似度最大(32%),其次是紫贻贝C-type lectin 4(31%),与斑节对虾的相似度最小(20%)。利用MEGA 5.0软件建立邻接法(Neighbour-Joining,NJ)系统树(图3)。结果显示Mm-Lec1先和紫贻贝(AJQ21497)聚为一支,然后和栉孔扇贝(DQ209289)聚为一大支,和其他的软体动物的C型凝集素的进化距离较远。
3.4蛋白高级结构同源建模分析
运用DiANNA在线软件,得出CRD结构域的6个半胱氨酸形成3个二硫键(分别是1-12:XXXXXCPSGWI-EFNQECFLFGA,29-130:DAEADCRRHSS-RYNYICEXXXX,104-122:NNSANCMSFYM-WRDDRCTARYN),连接方式是第1个和第2个,第3个和第6个,第4个和第5个半胱氨酸形成二硫键。将Mm-Lec1氨基酸序列提交到(http://swissmodel.expasy.org/)网站,在RCSB PCB数据库(http://www.rcsb.org/pdb/home/home.do)中比对,得到1个与之相似度(similarity:37.50%)比较高的氨基酸序列的高级结构模板:小鼠清道夫受体C型凝集素Collectin placenta 1(2ox9.3.A)[27],其全局模型质量评估(Global Model Quality Estimation,GMQE)值为0.63(GMQE值在0~1之间,值越大,模板与目标基因的匹配度越高,得到的蛋白质的三级结构越可信)。根据此模板建立Mm-Lec1高级结构模型(图4A),结果显示蛋白分子空间结构是紧凑型,是由6个Helices,19个Strands,和11个Turns,其中3条β折叠形成1个β片层结构(图4C)。
3.5文蛤Mm-Lec1组织表达规律
3.5.1C型凝集素在文蛤各个组织中的分布情况
采用QRT-PCR,我们分析了Mm-Lec1基因在健康文蛤各个组织中的相对表达量(图5),鳃中Mm-Lec1的mRNA表达量是血细胞的2.6倍(P>0.05),闭壳肌中的表达量是血细胞的0.8倍(P>0.05),外套膜中的表达量是血细胞的0.3倍(P>0.05),肝胰腺中的表达量是血细胞的0.2倍(P<0.05),性腺中的表达量是血细胞0.09倍(P<0.05),所以鳃中表达量最高,血细胞次之,性腺中表达量最低。
3.5.2菌刺激之后Mm-Lec1的时间表达分析
在细菌刺激试验中,我们分析了鳗弧菌(Vibrioanguillarum)刺激后0 h,6 h,12 h,24 h,48 h,72 h和96 h文蛤血细胞C型凝集素基因mRNA的相对表达量(图6),6 h的表达量是0 h的0.4倍(P<0.05),12和24 h的表达量分别是0 h的1.1倍和1.9倍,48 h的相对表达量是0 h的3.3倍(P<0.05),72 h后Mm-Lec1的mRNA表达量恢复到0 h水平。C型凝集素的表达在时间上呈现先降低,再升高,在48 h时达到峰值,而后降低到基础水平的趋势。
4讨论
文蛤是我国沿海常见的一种重要经济贝类,也是出口创汇的重要水产品之一,但是由于病害流行和养殖水域污染,文蛤养殖业受到极大损失,因此研究文蛤免疫机制显得尤为重要。本研究报道了文蛤重要免疫基因C型凝集素Mm-Lec1的cDNA序列,运用InterProScan 5推测Mm-Lec1氨基酸序列Leu3-Leu14为疏水区,猜测这段氨基酸序列存在信号肽或者跨膜区。由TMHMM预测前60个氨基酸含有疏水氨基酸,存在跨膜区的氨基酸预测值相对比较高(14.5>10),跨膜螺旋结构的预测值低(14.5<18),推测Mm-Lec1中不含有跨膜螺旋结构,可能含有1个N端信号肽序列[28]。利用软件SignalP4.0发现文蛤Mm-Lec1氨基酸序列Met1-Ser19是信号肽序列,TargetP 1.1预测得出C型凝集素是分泌蛋白的概率是93.4%,猜测文蛤Mm-Lec1是分泌蛋白。Mm-Lec1氨基酸序列的糖结合位点是QPN,具有D-半乳糖结合特性,说明Mm-Lec1是Gal型配体。从多序列比对图中我们发现,糖结合位点是EPN和QPD的物种数最多,EPD其次,QPN、VPN、YPD和EPS最少。在糖结合位点中,EPN和QPD是高度保守的,其他都是突变型,这种高度保守的碱基被替换说明海洋生物C型凝集素序列的多样性[16]。
用Swissmodel构建的Mm-Lec1模型与小鼠清道夫受体C型凝集素Collectin placenta 1(2ox9.3.A)的同源性较高,序列的氨基酸组成分析得出Mm-Lec1是水溶性蛋白质,推测Mm-Lec1属于Collectin家族中的一员。Mm-Lec1的高级结构是紧凑型,运用DiANNA预测合浦珠母贝[8,19]和海湾扇贝[9]的CRD结构中的二硫键发现,都含有3个二硫键,半胱氨酸的结合方式都是第1个和第2个,第3个和第6个,第4个和第5个结合,但是合浦珠母贝是球形而海湾扇贝是长型(long-form),说明蛋白质的高级结构不仅仅只与二硫键的数量有关,但还需要更多的研究进行佐证。

图2 文蛤C型凝集素与其他已知的C型凝集素的多序列比对Fig.2 Multiple alignments of Mm-Lec1 with other known C-type lectin sequences黑色背景代表同源性,灰色阴影代表相似性;▲表示糖识别结构域中形成二硫键的半胱氨酸。其中文蛤Mm-Lec1 (M. meretrix, KT276515);紫贻贝C-type lectin 8(M. galloprovincialis, AJQ21499);皱纹盘鲍putative perlucin 3 (H. discus discus, ABO26595);紫贻贝C-type lectin 6(M. galloprovincialis, AJQ21497);海湾扇贝 serum C-type lectin (A. irradians, ACE80702);日本鳗鲡 C-type lectin 2(A. japonica, BAC54021);虹鳟 C-type MBL-2 protein(O. mykiss, CAJ14130);长牡蛎 C-type lectin-1(C. gigas, BAF75353);紫贻贝C-type lectin 4(M. galloprovincialis, AJQ21495);海湾扇贝 C-type lectin 4(A. irradians, ACS72239);紫贻贝C-type lectin 5(M. galloprovincialis, AJQ21496);海湾扇贝 C-type lectin 3(A. irradians, ACS72238);栉孔扇贝C-type lectin A(C. farreri, DQ209289);爪蟾 Mrc1-prov protein(X. laevis, AAH77648);大西洋鲑 C type lectin receptor B(S. salar, AAT77221 );松江鲈 C-type lectin(T. fasciatus, AFW17073);紫贻贝C-type lectin 2(M. galloprovincialis, AJQ21493);海湾扇贝 C-type lectin(A. irradians, EU277646);海湾扇贝CLECT(A. irradians, ABZ89710);栉孔扇贝 C-type lectin B(C. farreri, DQ209290);合浦珠母贝C-type lectin 2(P. fucata, ACO36046);大西洋鲑 serum lectin isoform 5 precursor(S. salar, AAO43604);斑节对虾PmAV(P. monodon, AAQ75589)Homology are in the black region; similarity are in the grey region; the acids under ▲ are cysteines constituting disulfide bonds in the structure domain. There include hard clam Mm-Lec1 (M. meretrix, KT276515), blue mussel C-type lectin 8 (M. galloprovincialis, AJQ21499), disk abalone putative perlucin 3 (H. discus discus, ABO26595), blue mussel C-type lectin 6 (M. galloprovincialis, AJQ21497), bay scallop serum C-type lectin (A. irradians, ACE80702), Japanese eel C-type lectin 2 (A. japonica, BAC54021), rainbow trout C-type MBL-2 protein (O. mykiss, CAJ14130), Pacific oyster C-type lectin-1 (C. gigas, BAF75353), blue mussel C-type lectin 4 (M. galloprovincialis, AJQ21495), bay scallop C-type lectin 4 (A. irradians, ACS72239), blue mussel C-type lectin 5 (M. galloprovincialis, AJQ21496), bay scallop C-type lectin 3 (A. irradians, ACS72238), Zhikong scallop C-type lectin A (C. farreri, DQ209289), clawed toad Mrc1-prov protein (X. laevis, AAH77648), Atlantic salmon C type lectin receptor B (S. salar, AAT77221 ), roughskin sculpin C-type lectin (T. fasciatus, AFW17073), blue mussel C-type lectin 2 (M. galloprovincialis, AJQ21493), bay scallop C-type lectin (A. irradians, EU277646), bay scallop CLECT (A. irradians, ABZ89710 ), Zhikong scallop C-type lectin B (C. farreri, DQ209290), Peal oyster C-type lectin 2 (P. fucata, ACO36046), Atlantic salmon serum lectin isoform 5 precursor (S. salar, AAO43604), black tiger shrimp PmAV (P. monodon, AAQ75589)

图3 23条C型凝集素基因构建的NJ进化树以及Mm-Lec1与其他已知CTL基因的相似度Fig.3 The NJ tree of 23 CTL genes and the similarity between other known CTL and Mm-Lec1

图4 文蛤C型凝集素蛋白的高级结构不同角度观测图以及β片层结构展示图Fig.4 The tertiary structure in different views of Mm-Lec1 and the show of β lamellar structureA.正视图;B.仰视图; C.在B基础上Z轴旋转逆时针旋转60°得到的侧视图。蓝色部分是β转角,绿色部分是无规卷曲,黄色部分是β折叠,红色部分是α螺旋A. Front view; B. Vertical view; C. Side view generated after B rotates 60 degrees CCW on Z axis. The blue, green, yellow and red is the token of β-turn, random coil, β folding and α helix, respectively

图5 文蛤C型凝集素在组织中的表达量Fig.5 Relative expression of the Mm-Lec1 mRNA in tissues. Bars display mean ± S. D.*代表显著性差异(P<0.05),柱状图代表的是相对表达量的平均值±方差* is the token of significance difference (P<0.05)
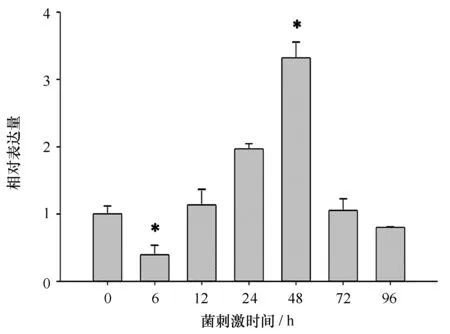
图6 菌刺激后文蛤血细胞中C型凝集素基因mRNA的表达量Fig.6 Relative expression of the Mm-Lec1 gene mRNA in haemocytes after bacteria exposure*代表显著性差异(P<0.05),柱状图代表的是相对表达量的平均值±方差* indicates significance difference (P<0.05). Bars display mean ± S. D.
一般低等生物中的C型凝集素只含有1个CRD,其他物种的则有两个或多个CRD[29]。文蛤Mm-Lec1氨基酸含有1个C型凝集素结构域,这和海湾扇贝(A.irradias)[9,17—18,29]以及合浦珠母贝(P.fucata)[8,19]的C型凝集素结构一致,但和栉孔扇贝(C.farreri)[15—16,30—31]结构不一致,其中Cflec-3[31]和Cflec-4[30]分别含有3个和4个CRD结构域。从23条序列的比对图发现,比软体动物在进化上高级很多的两栖类爪蟾和鱼类,都含有1个CRD结构域的情况。Mm-Lec1与其他物种的CTL的相似度在20%~32%之间(图3),文蛤和紫贻贝 C-type lectin 6的相似度最高(32%),其次是紫贻贝C-type lectin 4(31%),与斑节对虾PmAV的相似度最低(20%),与非同类的日本鳗鲡(28%),松江鲈(27%),爪蟾(27%)和虹鳟C-type MBL-2 protein(27%)却有相对较高的相似度;此外,Mm-Lec1基因和最近发表的Mm-CTL基因碱基组成有很大的差别,相似度仅是36.5%。同时,NJ系统树结果分析显示不同物种及同一物种的CTL基因并没有产生特别明显的系统分化关系,比如,同属于海湾扇贝的CTL基因和同属紫贻贝的CTL基因并没有各自独立分为一支,甚至有一些基因的进化距离相对较远;属于两栖类的爪蟾的C型凝集素基因却与鱼类的聚为一支;与Mm-Lec1同源性相差不多的紫贻贝 C-type lectin 6和紫贻贝C-type lectin 4却与Mm-Lec1有不同进化距离。这些进化关系可能是由于凝集素基因家族成员不同的进化路径和不同的功能造成的,也证实了C型凝集素是一个具有保守序列并且进化较快的基因[8]。
文蛤Mm-Lec1 mRNA在所检测组织中都有表达,其中鳃中表达量最高,血细胞次之,性腺中表达量最少。胡钰婷等[8]发现PoLEC1和PoLEC2在合浦珠母贝肝胰腺中表达量最高;Zhu等[9,29]发现AiCTL1和Ai Lec分别在海湾扇贝的血细胞和肝胰腺中表达量最高。鳃是软体动物免疫系统的“第一道防线”,在贝类免疫方面起到重要的作用;血细胞和肝胰腺是无脊椎动物重要的免疫器官。但是在相同物种或不同物种不同的组织中C型凝集素基因的表达量不同,说明C型凝集素的种类很多,推测是不同C型凝集素基因的具体功能不同造成的[9]。血细胞不仅可以直接隔离并且杀死病原菌而且可以合成和分泌一些在免疫中有作用的活性物质,所以我们选择血细胞为在不同时间的鳗弧菌刺激下的C型凝集素的实验材料。6 h血细胞中的C型凝集素的表达量达到最低值,48 h达到最高值,72 h回到基础水平,暗示C型凝集素在文蛤体内存在诱导表达的恢复模式,这与栉孔扇贝[5]和合浦珠母贝[8]的C型凝集素研究结果一致,说明文蛤的C型凝集素有参与体内免疫和机体防御。
参考文献:
[1]庄启谦. 中国动物志, 软体动物门, 双壳纲, 帘蛤科[M]. 北京: 科学出版社, 2001: 230.
Zhuang Qiqian. Fauna sinica, phylum mollusca, bivalvia, veneridae[M]. Beijing: Science Press, 2001: 230.
[2]王如才, 王昭萍. 海水贝类养殖学[M]. 青岛: 中国海洋大学出版社, 2008: 396-408.
Wang Rucai, Wang Zhaoping. Marine shellfish culture[M]. Qingdao: Ocean University of China Press, 2008: 396-408.
[3]刘连生, 闫茂仓, 林志华, 等. 引起文蛤暴发性死亡病原菌的分离和鉴定[J]. 微生物学通报, 2009, 36(1): 71-77.
Liu Liansheng, Yan Maocang, Lin Zhihua, et al. Pathogens isolation and identification of eruptive epidemic disease inMeretrixmeretrix[J]. Microbiology, 2009, 36(1): 71-77.
[4]刘连生, 闫茂仓, 赵海泉, 等. 文蛤疾病学研究进展[J]. 水产科学, 2009, 28(4): 234-237.
Liu Liansheng, Yan Maocang, Zhao Haiquan, et al. Advancement of pathological researches on hard clamMeretrixmeretrix[J]. Fisheries Science, 2009, 28(4): 234-237.
[5]胥炜, 王昊, 宋林生, 等. 栉孔扇贝C型凝集素基因的克隆与表达研究[J]. 高技术通讯, 2005, 15(1): 83-88.
Xu Wei, Wang Hao, Song Linsheng, et al. cDNA cloning and mRNA expression of C-type lectin from scallopChlamysFarreri[J]. Chinese High Technology Letters, 2005, 15(1): 83-88.
[6]Drickamer K. Engineering galactose-binding activity into a C-type mannose-binding protein[J]. Nature, 1992, 360(6400): 183-186.
[7]Weis W I, Taylor M E, Drickamer K. The C-type lectin superfamily in the immune system[J]. Immunological Reviews, 1998, 163(1): 19-34.
[8]胡钰婷, 张殿昌, 崔淑歌, 等. 合浦珠母贝C-型凝集素基因的序列特征和功能分析[J]. 水产学报, 2011, 35(9): 1327-1336.
Hu Yuting, Zhang Dianchang, Cui Shuge, et al. Sequence features and functional analysis of the C-type lectin gene (PoLEC1) from pearl oysterPinctadafucata[J]. Journal of Fisheries of China, 2011, 35(9): 1327-1336.
[9]Zhu Ling, Song Linsheng, Xu Wei, et al. Molecular cloning and immune responsive expression of a novel C-type lectin gene from bay scallopArgopectenirradians[J]. Fish Shellfish Immunol, 2008, 25(3): 231-238.
[10]Tunkijjanukij S, Olafsen J A. Sialic acid-binding lectin with antibacterial activity from the horse mussel: further characterization and immunolocalization[J]. Dev Comp Immunol, 1998, 22(2): 139-150.
[11]Zhao Zhiying, Yin Zhixin, Xu Xiaopeng, et al. A novel C-type lectin from the shrimpLitopenaeusvannameipossesses anti-white spot syndrome virus activity[J]. J Virol, 2009, 83(1): 347-356.
[12]Luo Tian, Zhang Xiaobo, Shao Zongze, et al.PmAV, a novel gene involved in virus resistance of shrimpPenaeusmonodon[J]. Febs Letters, 2003, 551(1/3): 53-57.
[13]李猛, 周素明, 刘璐, 等. 文蛤(Meretrixmeretrix)C型凝集素基因的分子克隆及表达分析[J]. 海洋与湖沼, 2015, 46(5): 1186-1192.
Li Meng, Zhou Suming, Liu Lu, et al. Molecular clone and expression and expression of C-type lectin inMeretrixmeretrix[J]. Oceanlogia et Limnologia Sinica, 2015, 46(5): 1186-1192.
[14]Gerdol M, Venier P. An updated molecular basis for mussel immunity[J]. Fish Shellfish Immunol, 2015, 46(1): 17-38.
[15]黄海宁. 栉孔扇贝C型凝集素基因的cDNA克隆和序列分析[J]. 福建水产, 2007(4): 6-10.
Huang Haining. cDNA cloning and sequence analysis on C-type lectin gene ofChlamysfarreri[J]. Journal of Fujian Fisheries, 2007(4): 6-10.
[16]Wang Hao, Song Linsheng, Li Chenghua, et al. Cloning and characterization of a novel C-type lectin from Zhikong scallopChlamysfarreri[J]. Mol Immunol, 2007, 44(5): 722-731.
[17]Zhang Huan, Song Xiaoyan, Wang Lingling, et al. AiCTL-6, a novel C-type lectin from bay scallopArgopectenirradianswith a long C-type lectin-like domain[J]. Fish Shellfish Immunol, 2011, 30(1): 17-26.
[18]宋小燕. 海湾扇贝凝集素基因和栉孔扇贝亲环素A基因的克隆与表达[D]. 咸阳: 西北农林科技大学, 2009.
Song Xiaoyan. Molecular cloning, characterization and expression of Lectins fromArgopectenirradiansand cyclophilin a fromChlamysfarreri[D]. Xianyang: Northwest A & F University, 2009.
[19]胡钰婷, 张殿昌, 江世贵, 等. 合浦珠母贝C型凝集素2的序列特征和表达分析[J]. 湖北农业科学, 2011, 50(19): 4026-4030.
Hu Yuting, Zhang Dianchang, Jiang Shigui, et al. Sequence and expression analysis of a novel C-type lectin gene from pearl oysterPinctadafucata[J]. Hubei Agricultural Sciences, 2011, 50(19): 4026-4030.
[20]Yamaura K, Takahashi K G, Suzuki T. Identification and tissue expression analysis of C-type lectin and galectin in the Pacific oyster,Crassostreagigas[J]. Comp Biochem Phys B, 2008, 149(1): 168-175.
[21]Li Hui, Zhang Huan, Jiang Shuai, et al. A single-CRD C-type lectin from oysterCrassostreagigasmediates immune recognition and pathogen elimination with a potential role in the activation of complement system[J]. Fish Shellfish Immunol, 2015, 44(2): 566-575.
[22]Jing Xing, Espinosa E P, Perrigault M, et al. Identification, molecular characterization and expression analysis of a mucosal C-type lectin in the eastern oyster,Crassostreavirginica[J]. Fish Shellfish Immunol, 2011, 30(3): 851-858.
[23]Wang Ning, Whang I, Lee J. A novel C-type lectin from abalone,Haliotisdiscusdiscus, agglutinatesVibrioalginolyticus[J]. Dev Comp Immunol, 2008, 32(9): 1034-1040.
[24]Zhang Jian, Qiu Reng, Hu Yonghua. HdhCTL1 is a novel C-type lectin of abaloneHaliotisdiscushannaithat agglutinates Gram-negative bacterial pathogens[J]. Fish Shellfish Immunol, 2014, 41(2): 466-472.
[25]高祥刚, 李云峰, 宋文涛, 等. 文蛤肠、外套膜和肝胰脏组织CDNA文库构建及其ESTs序列分析[J]. 中国水产科学, 2010, 17(2): 344-350.
Gao Xianggang, Li Yunfeng, Song Wentao, et al. Construction of cDNA libraries from intestine, mantle and hepatopancreas of hard clam (Meretrixmeretrix) and ESTs analysis[J]. Journal of Fishery Sciences of China, 2010, 17(2): 344-350.
[26]Li Hongjun, Liu Weidong, Gao Xianggang, et al. Identification of host-defense genes and development of microsatellite markers from ESTs of hard clamMeretrixmeretrix[J]. Mol Biol Rep, 2011, 38(2): 769-775.
[27]Feinberg H, Taylor M E, Weis W I. Scavenger receptor C-type lectin binds to the leukocyte cell surface glycan Lewisx by a novel mechanism[J]. J Biol Chem, 2007, 282(23): 17250-17258.
[28]Sonnhammer E L L, Von Heijne G, Krogh A. A hidden Markov model for predicting transmembrane helices in protein sequences[C]//Ismb. 1998, 6: 175-182.
[29]Zhu Ling, Song Linsheng, Xu Wei, et al. Identification of a C-type lectin from the bay scallopArgopectenirradians[J]. Mol Biol Rep, 2009, 36(5): 1167-1173.
[30]Zhang Huan, Wang Hao, Wang Lingling et al. Cflec-4, a multidomain C-type lectin involved in immune defense of Zhikong scallopChlamysfarreri[J]. Deve Comp immunol, 2008, 149(1): 168-175.
[31]Zhang Huan, Wang Hao, Wang Lingling, et al. A novel C-type lectin (Cflec-3) fromChlamysfarreriwith three carbohydrate-recognition domains[J]. Fish Shellfish Immunol, 2009, 26(5): 707-715.
Cloning and expression analysis of a C-type lectin gene (Mm-Lec1) in hard clam Meretrix meretrix
Zhang Jingjing1,2, Li Hongjun2, Qin Yanjie1, Liu Min1,2, Ye Sheng1,2
(1.CollegeofFisheriesandLifeScience,DalianOceanUniversity,Dalian116023,China; 2.MarineEcologyDepartment,NationalMarineEnvironmentalMonitoringCenter,Dalian116023,China)
Abstract:The hard clam (Meretrix meretrix) is an economically important bivalve species in China. Aquaculture of M. meretrix is seriously affected by the epidemic diseases. The study of the immune system of M. meretrix is an important approach to solve disease problems. C-type lectins (CTLs) play important roles in the identification of pathogen associated molecular patterns and activation of humoral immunity. In the present study, a C-type lectin gene of M. meretrix(denoted as Mm-Lec1) was obtained through sequencing full-length cDNA library. The full-length cDNA of Mm-Lec1 was 586 bp with a 486 bp open reading frame, encoding 161 amino acids. The length of 5′ and 3′ untranslated region was 21 and 79 bp, respectively. The molecular weight of predicted protein was 18.65 kD, and the theoretical isoelectric point was 4.98. The predicted amino acid sequence had a signal peptide (Met1-Ser19), a sugar-binding site (QPN) and a carbohydrate recognition domain (CRD). The tertiary structure of Mm-Lec1 was predicted as a compact type and had a β sheet structure with three beta sheet layers. The similarity between Mm-Lec1 and the other species mentioned in this study were 20%-32%. Mm-Lec1, Mytilus galloprovincialis CTL 6 and Chlamys farreri CTL A were clustered in one branch in the neighbor-joining (NJ) tree. Mm-Lec1 mRNA was expressed in all tested tissues, including gill, haemocytes, hepatopancreas, mantle, adductor muscle and gonad, with the highest expression level in gill, the second in haemocytes, and the least in gonad. In the bacteria exposure test, the mRNA expression level was lowest at 6 h, and highest at 48 h, suggesting that Mm-Lec1 play a role in defensing against bacterial invasion in M. meretrix.
Key words:Meretrix meretrix; C-type lectin; EST; tertiary structure; real-time PCR
收稿日期:2015-10-15;
修订日期:2016-01-05。
基金项目:国家自然科学基金(31101899,31572595);国家海洋局近岸海域生态环境重点实验室开放基金(201511)。
作者简介:张晶晶(1989—),女,河南省周口市人,研究方向为分子生态学。E-mail:liutian.3090@163.com *通信作者:李宏俊,博士,副研究员,研究方向为分子生态学。E-mail:hjli@nmemc.org.cn
中图分类号:S917.4
文献标志码:A
文章编号:0253-4193(2016)06-0110-09
张晶晶, 李宏俊, 秦艳杰, 等. 文蛤C型凝集素基因(Mm-Lec1)的克隆与表达分析[J]. 海洋学报, 2016, 38(6): 110—118, doi:10.3969/j.issn.0253-4193.2016.06.012
Zhang Jingjing, Li Hongjun, Qin Yanjie, et al. Cloning and expression analysis of a C-type lectin gene (Mm-Lec1) in hard clamMeretrixmeretrix[J]. Haiyang Xuebao, 2016, 38(6): 110—118, doi:10.3969/j.issn.0253-4193.2016.06.012
