热液和冷泉10种无脊椎动物基因组大小测定及比较
2016-07-01郑平王敏晓李超伦孙松
郑平,王敏晓,李超伦,孙松*
(1. 中国科学院海洋研究所 海洋生态与环境科学重点实验室,山东 青岛 266071;2. 中国科学院大学,北京 100049)
热液和冷泉10种无脊椎动物基因组大小测定及比较
郑平1,2,王敏晓1,李超伦1,孙松1*
(1. 中国科学院海洋研究所 海洋生态与环境科学重点实验室,山东 青岛 266071;2. 中国科学院大学,北京 100049)
摘要:基因组大小(或称C值)作为生物单倍体细胞中全套染色体的DNA总量,在一定程度上是恒定的,因而C值可以作为生物物种的一个特定参数。深海热液和冷泉为更好地理解C值与不同环境之间的关系提供了一个特征性的模型。本文采用流式细胞术,测定了来自热液和冷泉环境中的10种深海无脊椎动物的C值,其分布范围从0.87 pg到12.28 pg,其中,相比于软体动物和多毛类,甲壳生物基因组大小及变异均较大。对比热液和冷泉两个群落中共有种(深海偏顶蛤Bathymodiolus platifrons、柯氏潜铠虾Shinkaia crosnieri以及长角阿尔文虾Alvinocaris longirostris)的基因组大小,发现C值差异并不显著。同时,综合已有的数据,对深海化能极端环境与其他环境条件下的物种C值进行对比分析,结果显示深海化能极端环境下生物的基因组大小并没有发现明确的变化趋势。
关键词:热液;冷泉;化能合成生态系统;基因组大小;C值;流式细胞术
1引言
基因组大小(或称C值),指的是生物单倍体细胞中全套染色体的DNA总量。在真核生物中,一般认为,每种生物的DNA含量在一定程度上是恒定的,因而,C值可以作为生物物种的一个特定参数。研究表明,基因组大小及其差异与很多生物特征相关,如光合作用率[1]、发育速度[2]、细胞大小[3]、代谢率[1,4]、濒危速率[5]等。此外,基因组大小还与不同环境的适应性相关[6—10]。尽管对于基因组大小的研究越来越多,但相对其多样性水平,海洋无脊椎动物基因组大小研究却远远不足,现有的基因组大小评估数据是极度缺乏的[11],导致对于海洋无脊椎生物的基因组大小变化的模式只能在较宽泛的水平上进行比较。然而,低等分类阶元内物种之间的比较,更有利于理解基因组大小蕴含的重要生物学意义,因而后续研究需要补充海洋无脊椎动物更多物种基因组大小的基础数据。此外,不同特征生活条件(如深海和浅海,稳定环境和变化环境)之间物种类群基因组大小的比较,也有助于揭示无脊椎动物基因组大小变化的进化过程及其生物学意义,进而推进对特殊环境下海洋无脊椎生物适应性的理解。
深海热液口和冷泉是比较典型的极端环境,为研究极端环境中基因组的大小提供了一个重要模型。深海热液口和冷泉属于化能生态系统,不同于一般的依赖光合作用获取能量的生态系统,其环境严酷,高压、富含甲烷和重金属等有毒有害物质[12—14]。尽管热液口和冷泉都是化能生态系统,但因其成因和分布等不同,环境特征也有较大差别。热液口持续时间短,环境梯度变化非常显著,热液流与周围海水随机混合[15],且相隔几厘米温度可从400℃变到2℃[16],pH可达3~6,重金属种类多且浓度高,富含硫化物是其典型特征。相比之下,冷泉渗出液较为稳定缓慢,存在时间较长,其环境梯度变化更温和[17],因而热液和冷泉为研究化能生态系统中基因组大小的关系提供了可以相互比较的两种环境条件。但对于生命而言,热液和冷泉环境无疑都是不稳定和严酷的,因而研究热液和冷泉无脊椎动物的基因组大小,对于理解深海极端环境尤其是化能合成生态系统内生物的适应性机制具有重要意义。
本文通过流式细胞术,测定了来自热液和冷泉环境中10种常见深海无脊椎动物的C值,并对两种群落中共有种的基因组大小进行比较,同时综合已有的数据,对深海环境与其他环境条件下的物种C值进行对比分析,以阐明不同环境条件下基因组大小变化的关系,分析深海化能生态系统生物的适应性机制。此外,本研究结果也补充了深海无脊椎动物基因组大小的记录,为后期开发利用深海生物基因资源和开展相关研究提供基础数据。
2材料和方法
2.1样品采集和保存
热液和冷泉样品在2014年4月“科学”号热液和冷泉调查航次中获得,由“发现”号ROV采集。热液和冷泉采样点如图1所示,其中热液采样点位于冲绳海槽(27°33′4.157″N,126°58′7.849″E),水深1 361 m;冷泉采样点位于台湾西南的南海冷泉区(22°06′57.144″N,119°17′6.580″E),水深1 113 m。样品取回甲板后,液氮速冻后转移到-80℃超低温冰箱中保存。
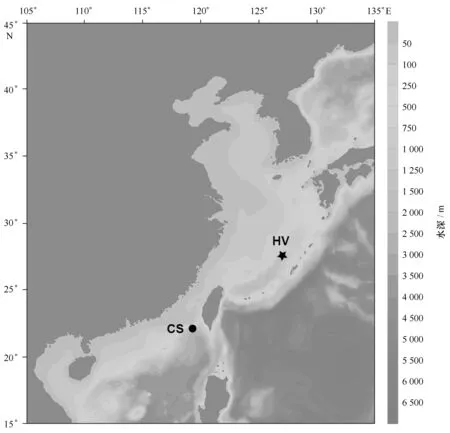
图1 样品采集点示意图,图中“HV”代表热液采样点,“CS”代表冷泉采样点Fig.1 Sampling locations, “HV” represents “hydrothermal vent”, “CS” represents “cold seep”
本研究中共采集到10种深海无脊椎动物,其中3种(深海偏顶蛤Bathymodiolusplatifrons、柯氏潜铠虾Shinkaiacrosnieri以及长角阿尔文虾Alvinocarislongirostris)为热液和冷泉环境共有,其余样品来源及测定样品数见表1。
2.2细胞悬液制备
根据样品类别选择相应的组织制备细胞悬液(表1),细胞悬液制备过程如下:取30~50 mg组织块放入PBS中化冻,并迅速用眼科小剪刀剪成糜状,使用40 μm细胞筛过滤后,滤液 300 g离心6 min后弃上清,然后向细胞片中加入1 mL 70%的酒精溶液冰上固定30 min。最后,将离心收集到的细胞片用PBS洗涤两次之后,加入1 mL PBS重悬得到细胞悬液。将鸡血细胞和组织细胞悬液中加入RNase 试剂(终浓度为125 μg/mL)后用PI冰上染色30 min(终浓度为50 μg/mL),在组织细胞悬液中加入鸡血红细胞,混合均匀,使2种细胞数目大致相等[18]。
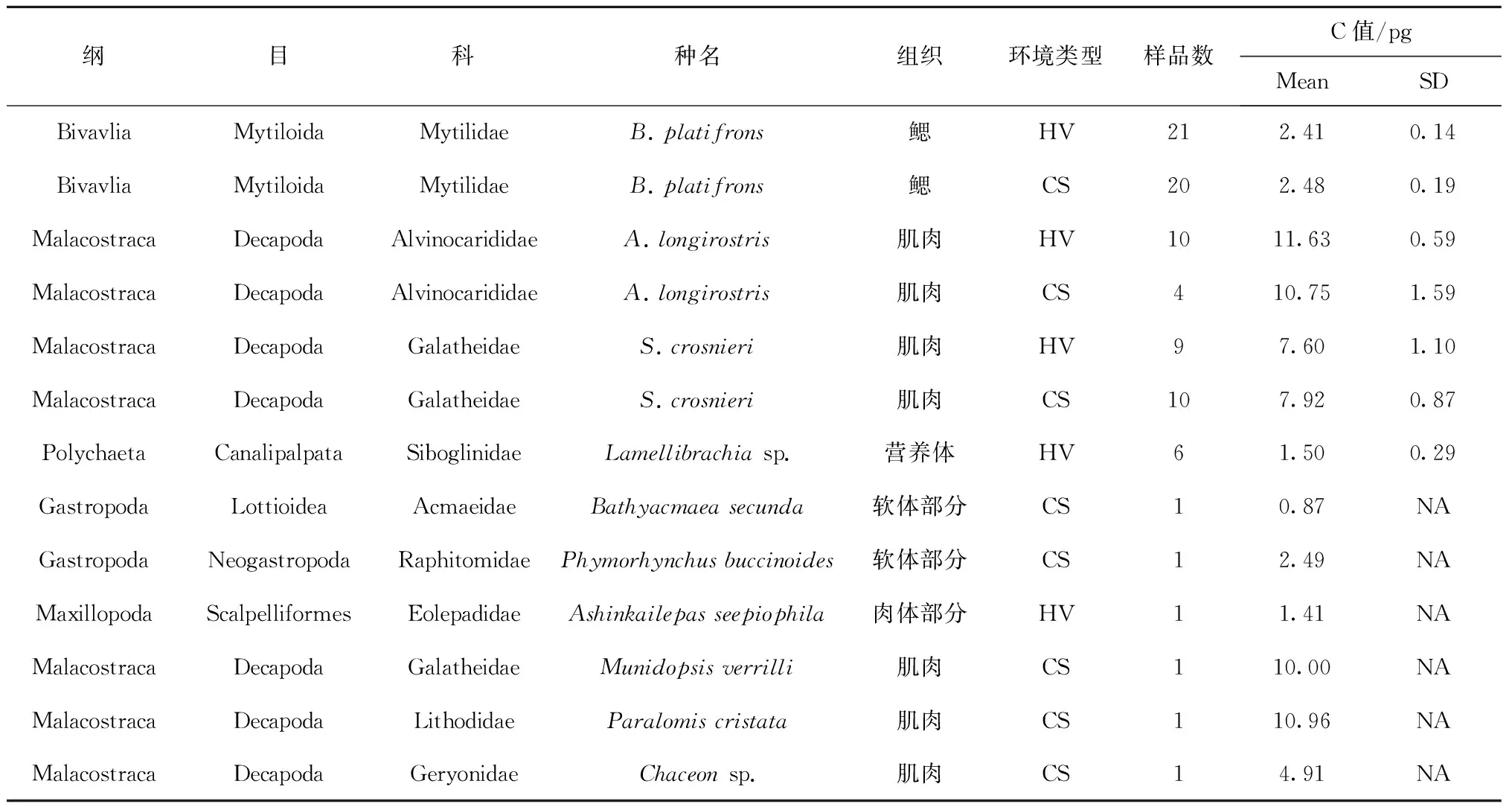
表1 10种深海化能极端环境中无脊椎动物的基因组大小测定结果
注:表中“组织”为细胞悬液制备时采用的组织来源,笠贝科和管螺科采用所有软体部分,希凯茗荷采用去掉外壳之后的所有肉体部分,铠甲虾和长角阿尔文虾采用尾部肌肉,石蟹和查世蟹采用螯足内肌肉。环境类型“HV”表示冲绳热液样品,“CS”表示南海冷泉样品。由于深海样品难以获取,部分样品只有1个个体,无法计算误差,用“NA”表示。
2.3 基因组大小的测定
基因组大小的测定在BD公司C6流式细胞仪上检测,激发光为488 nm,检测的发射波长为(625±10)nm,对应荧光通道为FL2。本文采用成熟大公鸡血样品作为内参,来测定样品的C值(对于与鸡血荧光主峰重叠的样品,采用其他样品混样上机进行辅助计算)。计算公式如下:
(1)
式中,Fsp和Fcd分别表示样品和鸡血的荧光检测峰值,Csp和Ccb分别表示样品和鸡血的C值,其中鸡血C值已知为1.25 pg,1 pg=0.978×109bp。
2.4统计分析
检验热液和冷泉两种环境中同一物种的基因组大小差异时,每组样品量不少于3个,由于样本大小不同,采用独立样本的t检验。仅来自一个群落的生物种,给出平均值和标准误。所有制图和统计使用R(3.01)分析完成。
3结果
3.1基因组大小
深海偏顶蛤B.platifrons、柯氏潜铠虾S.crosnieri、长角阿尔文虾A.longirostris以及管虫Lamellibrachiasp.的荧光分布如图2所示,管虫由于荧光峰值与鸡血重叠太多,换用贻贝样品作为内参。本研究所测定的样品C值见表1,其中,甲壳类生物具有较大C值,其分布范围也比较广泛,从1.41 pg到12.28 pg。其中最小值为铠茗荷目的A.seepiophila,其C值为1.41 pg,其次是十足目的Chaceonsp.为4.91 pg,其余十足目生物C值均较大,其中最大的C值出现在A.longirostris热液种群中,为12.28 pg。相比而言,软体动物类C值较小,为0.87 pg到2.77 pg。其中笠贝科的B.secundaC值较小,为0.87 pg;管螺科的P.buccinoidesC值较大,为2.49 pg。此外,获取的环节动物门中Lamellibrachiasp.的C值也相对较小,为1.50 pg。
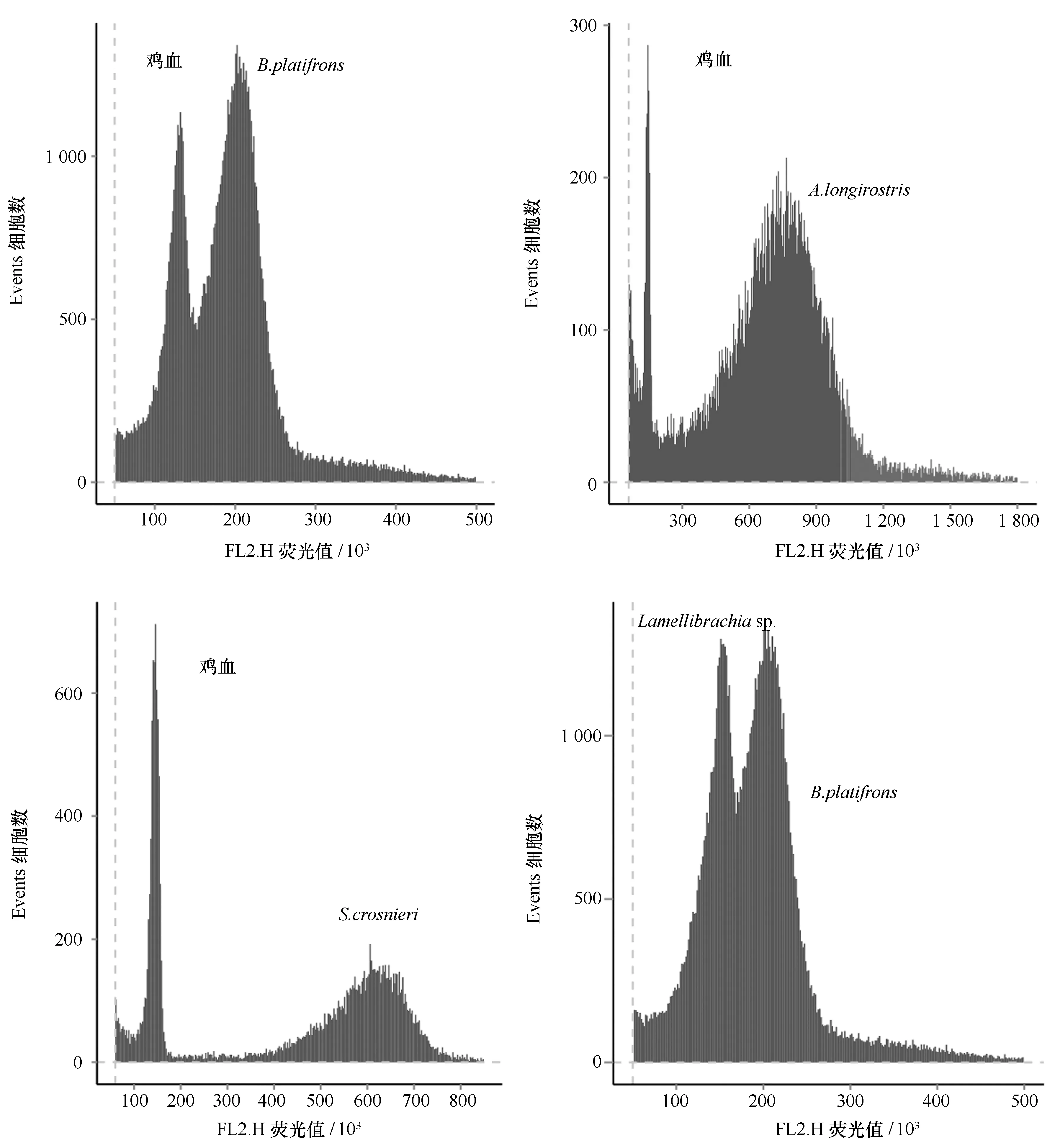
图2 4种样品的流式细胞仪测定结果显示Fig.2 Flow cytometric fluorescent-distributions of 4 invertebrates
对于种群内部基因组大小的变异而言,A.longirostris(冷泉群落)基因组大小SD值最大,为1.59,其次是热液和冷泉的S.crosnieri种群,SD值分别为1.10和0.87,而深海偏顶蛤B.platifrons的SD值相比均较小(热液为0.14,冷泉为0.19)。
3.2热液和冷泉同种生物C值比较
本文对热液和冷泉不同群落中采集到的同一物种比较结果如图3所示,深海偏顶蛤B.platifrons热液和冷泉群落C值分别为(2.41±0.14)pg和(2.48±0.19)pg,A.longirostris热液和冷泉群落C值分别为(11.63±0.59)pg和(10.75±1.59)pg,S.crosnieri热液和冷泉群落C值分别为(7.60±1.10)pg和(7.92±0.87)pg,两个群落中基因组大小的T检验结果均为差异不显著。
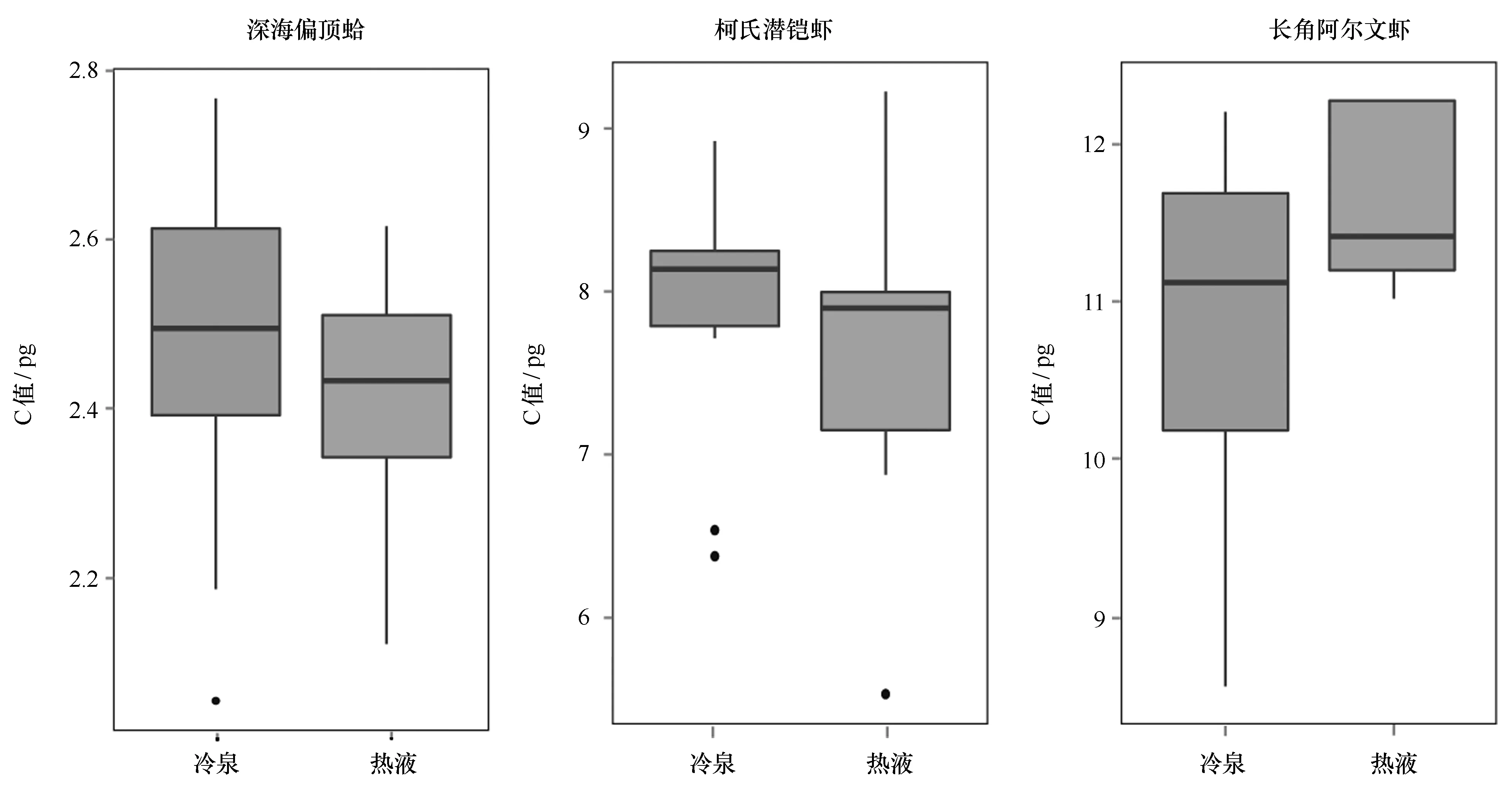
图3 热液和冷泉共有物种C值比较Fig.3 Comparisons of C-value for the common species from hydrothermal vent and cold seep
4讨论
4.1C值测定的影响因素
检测C值首先需要考虑的问题就是避免由于体细胞内复制导致的多倍化影响,体细胞的内复制广泛存在与甲壳类动物中[19]。为了避免体细胞内复制的影响,本研究中同类生物样品优先选择了同源组织来检测C值。
化能合成生态系统中,无脊椎动物与化能共生菌的共生关系普遍存在,外共生可通过PBS冲洗减少影响,而内共生的影响难以避免且很少有评估。为了评估内共生菌对于深海偏顶蛤C值检测造成的潜在影响,同步分析了其内共生菌所在鳃组织的电镜样品,同一细胞内共生菌的数量通常小于5个,而一般细菌的基因组大小小于5 Mb[20—21],因而相对于宿主而言,微生物DNA总量所占的比例不足1%,故影响可以忽略。同时,本研究测定的数值与基因K-mer的评估结果一致,证实了方法的可靠性(未发表数据)。
C值测定面临的另一个问题就是多倍体化。多倍体化是物种形成的一种重要方式,根据已有的研究数据表明,多倍体化在甲壳类中普遍存在,尤其是十足目中[19]。同时,在双壳类以及多毛类某些类群中也存在多倍体化的现象[22—23]。实际上,不能仅仅依据基因组大小来断定多倍体化物种形成是否存在,还需要补充细胞遗传学以及遗传学相关证明。本文检测到的C值,与已有数据相比,检测的深海偏顶蛤贻贝B.platifrons比已知Bathymodiolus属贻贝C值略大,两种铠甲虾均在已有的铠甲虾科C值范围之内,长角阿尔文虾A.longirostris的C值也并未超过已知阿尔文虾科C值分布范围。通过与K-mer预估数据的比较,可以确定深海偏顶蛤B.platifrons没有发生多倍化,而其他生物由于缺少细胞遗传学数据,本文都以二倍体形式来推算其基因组大小。
4.2基因组大小的差异
4.2.1个体间基因组大小的差异
根据流式细胞仪检测结果,柯氏潜铠虾S.crosnieri和长角阿尔文虾A.longirostris均存在个体间差异,而深海偏顶蛤B.platifrons和管虫Lamellibrachiasp.个体间差异较小。本研究所有样品均制备平行样,同一个体进行了多次检测,因此可以排除系统误差造成的影响。一方面,个体大小可能是造成基因组大小在个体间存在差异的原因之一[24],在本研究中,检测的长角阿尔文虾A.longirostris和柯氏潜铠虾S.crosnieri样品体长均较为一致,而深海偏顶蛤样品B.platifrons个体大小变化较大(3~7 cm),但是,深海偏顶蛤B.platifrons种群的基因组大小变异却小于长角阿尔文虾A.longirostris和柯氏潜铠虾S.crosnieri,即表明C值个体间差异与生物个体大小之间并不存在明确的相关关系。
此外,同一物种内基因组大小差异较大,可能是存在不同的亚种[11],为了排除物种鉴定错误及亚种分化造成的影响,我们同步测定了所检测样本的DNA条形码,COI序列的比较结果显示,本研究中个体种内差异均小于1%,未显示物种分化(未发表数据)。根据热液和冷泉不同种群C值的分布图(图4)所示,C值分布也不存在明显的两个峰,结果与COI验证结果一致,所测生物个体均属于同一个种,但受限于样本量大小,研究对应物种基因组大小的变异范围还有待于对更多个体基因组大小的测定。
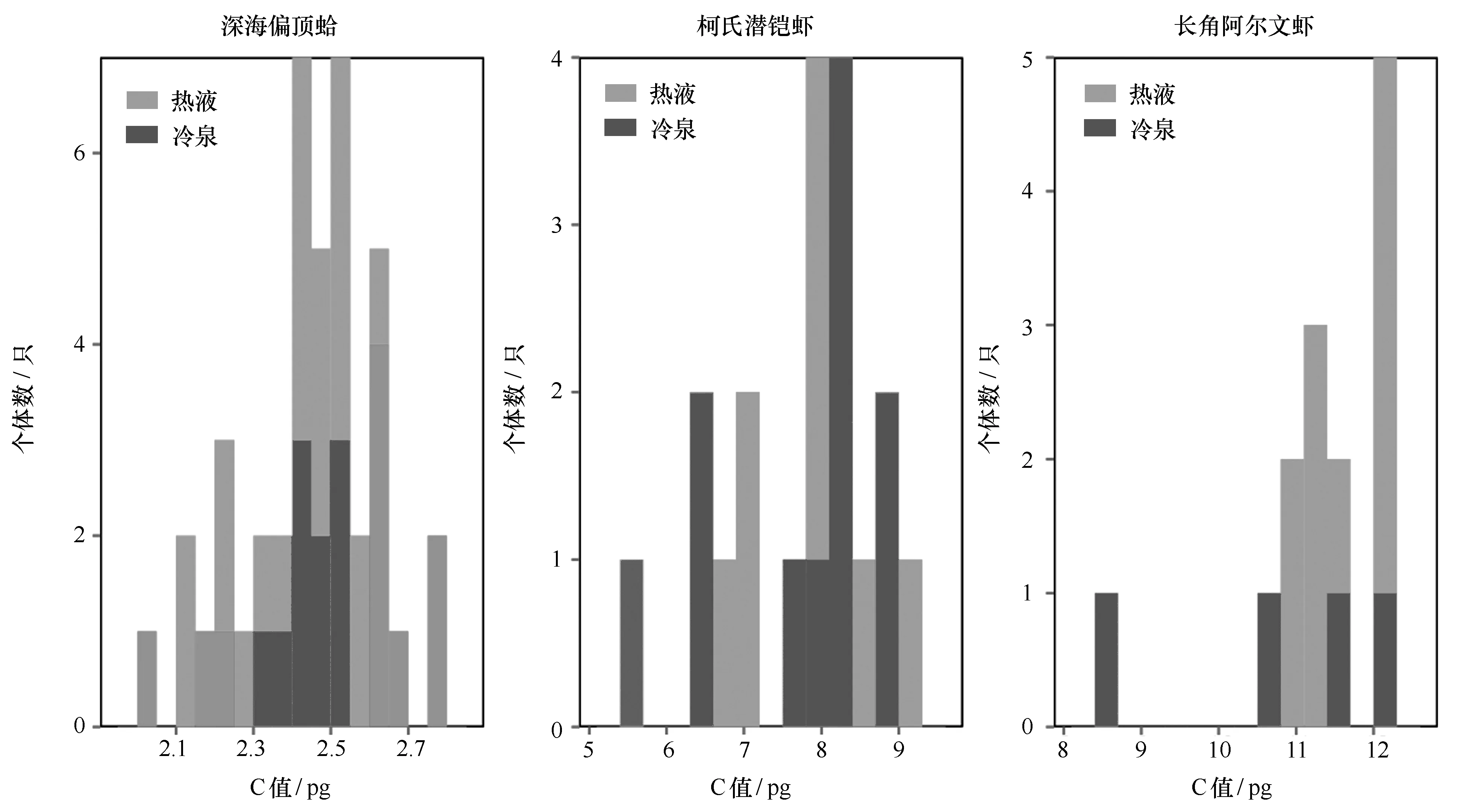
图4 热液和冷泉共有生物种C值个体分布图Fig.4 Distribution of individual C-value for the common species from hydrothermal vent and cold seep
4.2.2种群间基因组大小的差异
关于种群间基因组大小差异的解释,通常与地理分布相关,这些差异可能会来自于生存环境中受到不同刺激,如静水压力[25]、热环境[26]等,而这些刺激因子在深海极端环境中普遍存在。本研究中热液和冷泉共有种的基因组大小比较,为研究不同生境的影响提供了理想的模型。
关于同一个物种在不同环境中C值之间的差异比较,针对植物的研究较多[27—29],也有一些动物相关的研究如竹节虫Bacillusatticus[30]、伪哲水蚤Pseudocalanussp.[31]和果蝇Drosophilamelanogaster[26]。根据已有模型,环境刺激对基因组大小的影响主要有两种,一方面,不同环境刺激可能影响到繁殖,可以导致不育或者单性繁殖从而使染色体减少,这种情况是不同品系间基因组大小减小的主要原因[11]。另一方面,不同刺激下,卫星DNA的变化也会引起基因组大小的差异[32],例如一些甲壳类的渐变群中基因组大小的变化主要是与含有丰富的转座子相关[33]。
然而,不同于已有研究[34],本文中根据热液和冷泉共有种基因组大小差异的检验结果,地理分布上相距甚远的两个群落中深海偏顶蛤B.platifrons、柯氏潜铠虾S.crosnieri以及长角阿尔文虾A.longirostris种群间基因组大小都没有显著性差异(p>0.05)。一般看来,热液和冷泉生态系统中环境差异较大,但是由于采样地点的环境背景数据缺乏,难以更深入的分析两种群落生物基因组大小差异不显著的原因及其与环境因素的关系。
此外,虽然两个群落生物C值差异并不显著,但根据测定结果,明显可见深海偏顶蛤B.platifrons和柯氏潜铠虾S.crosnieri冷泉环境中C值大于热液环境中的C值,而长角阿尔文虾A.longirostris正好相反。根据已有研究显示,环境刺激对基因组大小可产生两种不同的影响,可能在深海极端环境下这两种影响都存在,但不同生物类群对环境的适应机制可能有所差别,导致基因组大小变化模式并不一致,还需要进一步开展基因组相关研究进行验证。
4.3深海化能极端环境对基因组大小的影响
已有研究关于端足目和囊虾总目中最大的基因组大小都出现在极地[35—36],表明在极端环境下基因组大小分布模式可能与其他环境中有所不同。极端环境对基因组大小可能产生两种相反的作用:一方面,根据大基因组不相容原理,即对于含有大量重复序列的大基因组,这些“多余的DNA”会产生额外的消耗[7],热液和冷泉恶劣的环境下,会偏好于选择较小的基因组,这一点由植物基因组大小的分布情况所支持[10,27]。另一方面,热液和冷泉环境梯度多变(冷泉相对更稳定,但会因为渗流变化而变化),当生物受到环境刺激(如压力、重金属、温度等)时,可能通过逆转录转座子扩增或者多倍体形成而导致基因组大小增加[11]。然而,关于深海极端环境下生物基因组大小变化情况,目前研究还比较缺乏,相应变异机制还不明确。
本文将通过深海极端环境中获取的样品C值数据与动物基因组大小数据库(http://www.genomesize.com/)中同一类群生物相比,来探究深海化能极端环境下基因组大小的分布模式。根据已有数据分布情况,本文中深海极端环境和其他环境的比较聚焦在贻贝科和十足目两个分类水平上的物种C值的比较。
对于贻贝科(图5)而言,只有深海偏顶蛤B.platifrons所在的Bathymodiolus属包含深海化能极端环境中的生物,其 C值大小和属内差异与非深海生物相比不存在明显特异性,最大C值和最大差异都出现在Volsella属。
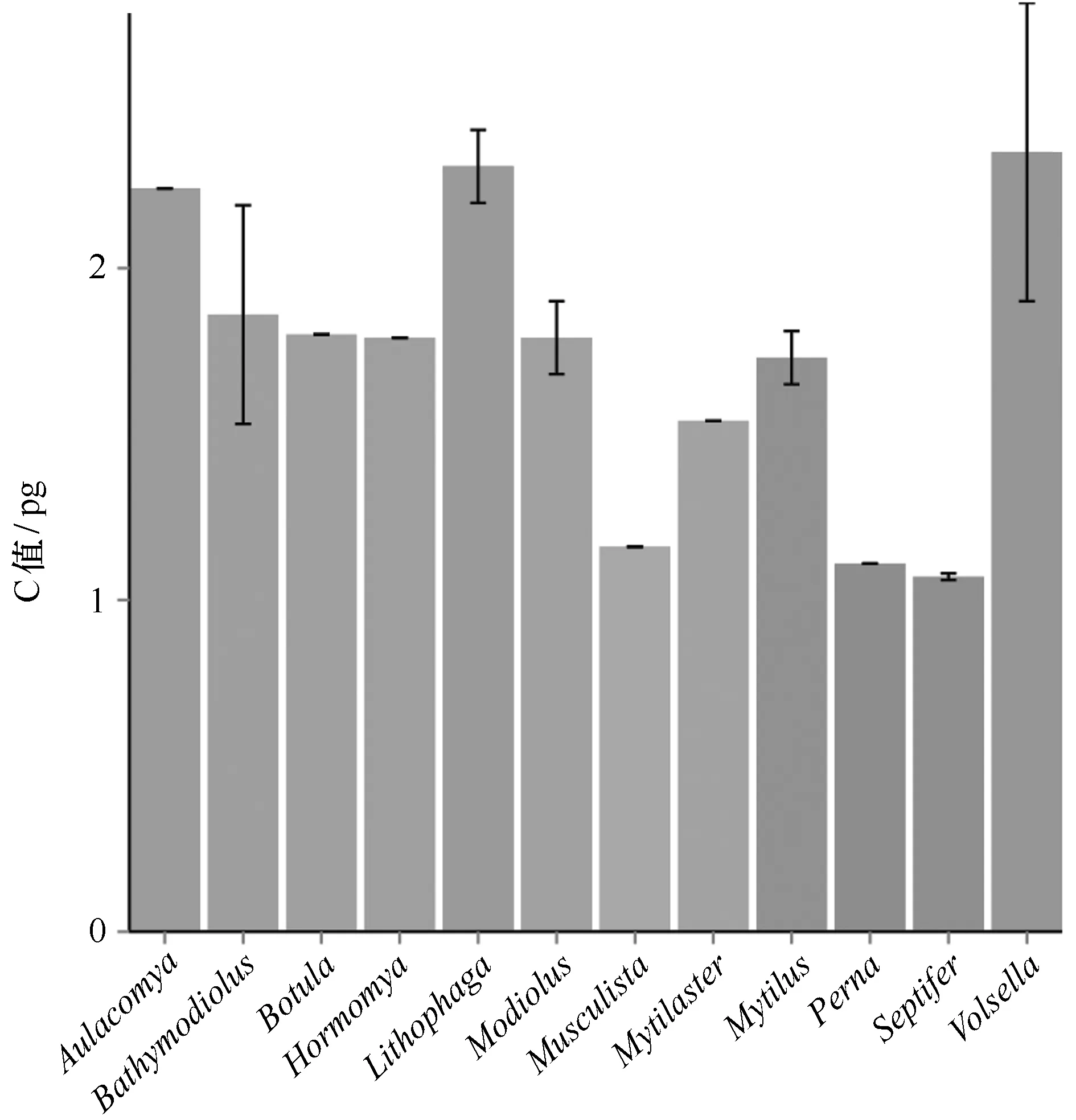
图5 贻贝科不同属生物C值比较Fig.5 Comparisons of C-value in Mytilidae
十足目的比较结果(图6)显示, Alvinocarididae科和Galatheidae科基因组大小平均值分别为11.49 pg和9.65 pg,均值和科内基因组大小差异相比其他科也没有明显特异性。然而,十足目中,极地和深海的一些物种如某极地虾Sclerocrangonferox为40.89 pg、深海虾Bythocarisirene为38.47 pg、某大西洋深海虾Hymenodorasp.为38.00 pg等都具有较高水平的基因组大小,但是对于某淡水虾Macrobrachiumohione也具有较大基因组,C值为22.16 pg。对于极地和深海环境中这些具有较大基因组的生物而言,其C值远远大于同一个科内其他物种,可能与所处的极端环境条件存在某种适应关系,但这种现象在本文研究的深海化能生态系统生物中并不存在。
尽管已有很多研究表明,基因组大小与一些生态因子相关[8,28—29],但对于有些生物而言,C值与环境条件的关系却并不明显[11,37—38]。这种看似相悖的结论其实并不矛盾,基因组大小的多样性涉及到多种因素交互作用,既可以通过突变丢失片段或者异常重组减小基因组大小[39—40],也可以通过逆转录转座子扩增、片段插入、杂交以及多倍体化等增加基因组大小[41—42],很难用某个一维模型来解释,关于基因组大小的统计描述主要是基于现有数据得到的,但是相比于物种多样性而言,现有物种的基因组大小数据可能并不能代表整体上基因组大小的实际情况,因此有待进一步对深海化能生态系统生物开展基因组测序研究。
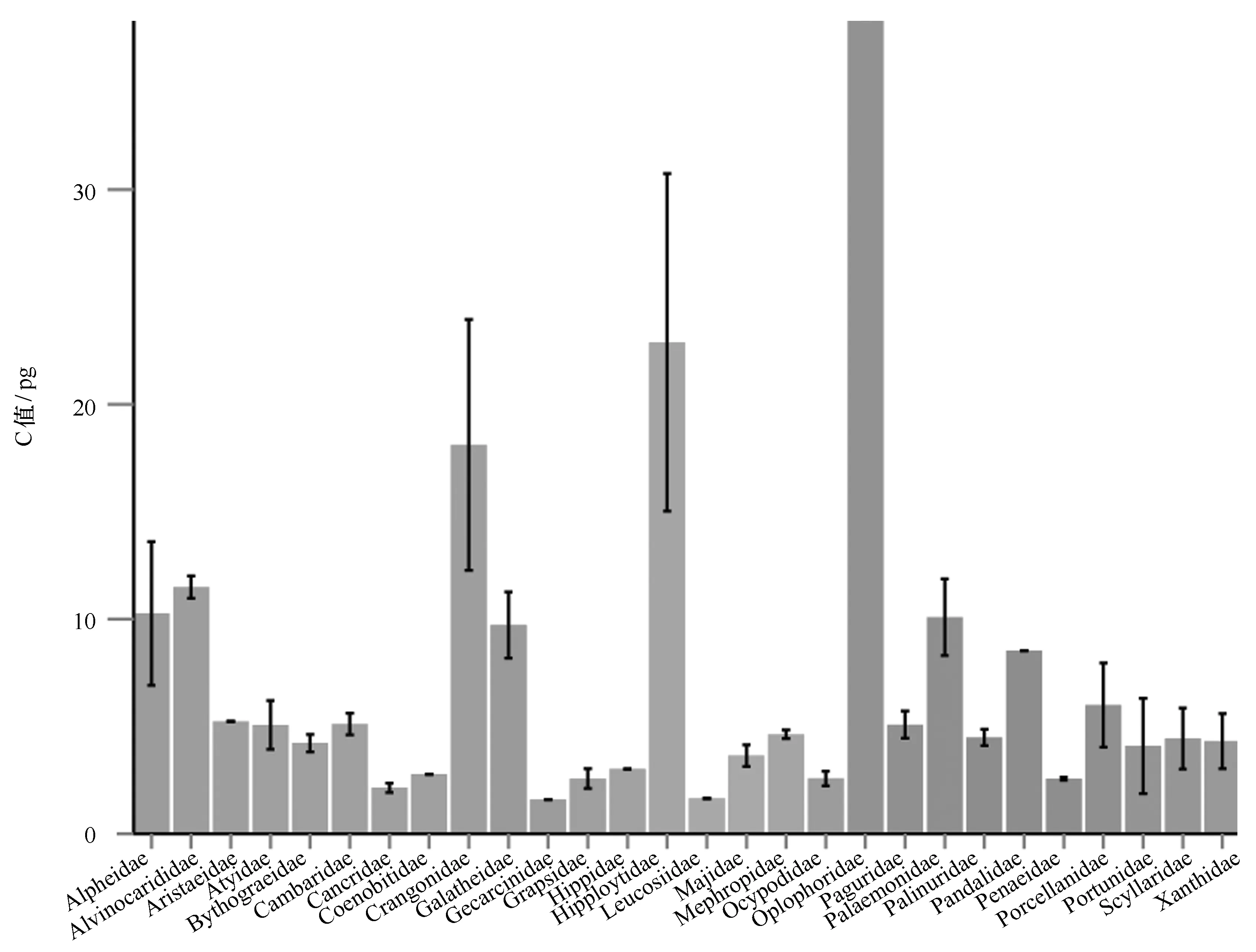
图6 十足目不同科生物C值比较Fig.6 Comparisons of C-value in Decapoda
5结论
综上所述,本研究中个体间基因组大小存在差异的物种中不存在亚种,这种差异与个体大小也不相关。热液和冷泉不同环境条件下的共有种基因组大小差异不显著,深海极端环境下生物的基因组大小,也并没有明确的变化趋势。
研究深海极端环境下无脊椎生物的基因组大小变化模式,在较低等分类阶元上获取更多的深海生物基因组大小基础数据是非常重要的,此外,还应该考虑补充不同环境条件下以及热液和冷泉物种亲缘关系较近物种的C值数据,才能获得更深入的认识。虽然关于十足目基因组大小的分析结果,并不支持极端环境下的大基因组限制理论,但是根据本文检测的结果,深海化能极端环境下基因组大小与其他环境相比差异并不明显,可能预示着极端环境下对基因组大小两种相反的影响机制都存在,还需要进一步研究。
致谢:首先,感谢“科学”号2014年冲绳热液航次和南海冷泉航次的所有工作人员,采集并提供了如此珍贵的深海生物样品。其次,感谢中国科学院海洋研究所沙忠利老师、刘磊老师和赵越同学在实验过程中给与的帮助和指导。
参考文献:
[1]Beaulieu J M, Leitch I J, Knight C A. Genome size evolution in relation to leaf strategy and metabolic rates revisited[J]. Ann Bot, 2007, 99(3): 495-505.
[2]Wyngaard G A, Rasch E M, Manning N M, et al. The relationship between genome size, development rate, and body size in copepods[J]. Hydrobiologia, 2005, 532(1/3): 123-137.
[3]Beaulieu J M, Leitch I J, Patel S, et al. Genome size is a strong predictor of cell size and stomatal density in angiosperms[J]. New Phytol, 2008, 179(4): 975-986.
[4]Gregory T R. The bigger the C-value, the larger the cell: genome size and red blood cell size in vertebrates[J]. Blood Cells Mol Dis, 2001, 27(5): 830-843.
[5]Vinogradov A E. Genome size and extinction risk in vertebrates[J]. Proc Biol Sci, 2004, 271(1549): 1701-1705.
[6]Bottini M C J, Greizerstein E J, Aulicino M B, et al. Relationships among genome size, environmental conditions and geographical distribution in natural populations of NW Patagonian species ofBerberisL. (Berberidaceae)[J]. Annals of Botany, 2000, 86(3): 565-573.
[7]Jordan G J, Carpenter R J, Koutoulis A, et al. Environmental adaptation in stomatal size independent of the effects of genome size[J]. New Phytol, 2015, 205(2): 608-617.
[9]Flavell R B, Bennett M D, Smith J B, et al. Genome size and the proportion of repeated nucleotide sequence DNA in plants[J]. Biochem Genet, 1974, 12(4): 257-269.
[10]Knight C A, Molinari N A, Petrov D A. The large genome constraint hypothesis: evolution, ecology and phenotype[J]. Ann Bot, 2005, 95(1): 177-190.
[11]Bonnivard E, Catrice O, Ravaux J, et al. Survey of genome size in 28 hydrothermal vent species covering 10 families[J]. Genome, 2009, 52(6): 524-536.
[12]Nyholm S V, Robidart J, Girguis P R. Coupling metabolite flux to transcriptomics: insights into the molecular mechanisms underlying primary productivity by the hydrothermal vent tubeworm Ridgeia piscesae[J]. Biol Bull, 2008, 214(3): 255-265.
[13]Jebbar M, Franzetti B, Girard E, et al. Microbial diversity and adaptation to high hydrostatic pressure in deep-sea hydrothermal vents prokaryotes[J]. Extremophiles, 2015, 19(4): 721-740.
[14]Marcon Y, Sahling H, Borowski C, et al. Megafaunal distribution and assessment of total methane and sulfide consumption by mussel beds at Menez Gwen hydrothermal vent, based on geo-referenced photomosaics[J]. Deep-Sea Research Part Ⅰ: Oceanographic Research Papers, 2013, 75: 93-109.
[15]Dziak R P, Johnson H P. Hydrothermal systems. Stirring the oceanic incubator[J]. Science, 2002, 296(5572): 1406-1407.
[16]Van Dover C L, German C R, Speer K G, et al. Evolution and biogeography of deep-sea vent and seep invertebrates[J]. Science, 2002, 295(5558): 1253-1257.
[17]Vanreusel A, De Groote A, Gollner S, et al. Ecology and biogeography of free-living nematodes associated with chemosynthetic environments in the deep sea: a review[J]. PLoS One, 2010, 5(8): e12449.
[18]梁智辉, 朱慧芬, 陈九武. 流式细胞术基本原理与实用技术[M]. 武汉: 华中科技大学出版社, 2008.
Liang Zhihui, Zhu Huifen, Chen Jiuwu. The basic principles and practical techniques of flow cytometry[M]. Wuhan: Huazhong University of Science and Technology Press, 2008.
[19]Bachmann K, Rheinsmith E L. Nuclear DNA amounts in pacificCrustacea[J]. Chromosoma, 1973, 43(3): 225-236.
[20]Ikuta T, Takaki Y, Nagai Y, et al. Heterogeneous composition of key metabolic gene clusters in a vent mussel symbiont population[J]. The ISME Journal, 2016, 10(4): 990-1001.
[21]Kuwahara H, Yoshida T, Takaki Y, et al. Reduced genome of the thioautotrophic intracellular symbiont in a deep-sea clam,Calyptogenaokutanii[J]. Current Biology, 2007, 17(10): 881-886.
[23]Sella G, Redi C A, Ramella L, et al. Genome size and karyotype length in some interstitial polychaete species of the genusOphryotrocha(Dorvilleidae)[J]. Genome, 1993, 36(4): 652-657.
[24]Escribano R, McLaren I A, Breteler W C M K. Innate and acquired variation of nuclear DNA contents of marine copepods[J]. Genome, 1992, 35(4): 602-610.
[25]Zhang Xuelian, Onozato H. Hydrostatic pressure treatment during the first mitosis does not suppress the first cleavage but the second one[J]. Aquaculture, 2004, 240(1/4): 101-113.
[26]Ellis L L, Huang Wen, Quinn A M, et al. Intrapopulation genome size variation in D. melanogaster reflects life history variation and plasticity[J]. PLoS Genet, 2014, 10(7): e1004522.
[28]Rakic T, Siljak-Yakovlev S, Sinzar-Sekulic J, et al. Morphological and genome size variations within populations of Edraianthus graminifolius “Jugoslavicus” (Campanulaceae) from the central Balkan peninsula[J]. Archives of Biological Sciences, 2014, 66(2): 743-763.
[29]Achigan-Dako E G, Fuchs J, Ahanchede A, et al. Flow cytometric analysis inLagenariasiceraria(Cucurbitaceae) indicates correlation of genome size with usage types and growing elevation[J]. Plant Systematics and Evolution, 2008, 276(1/2): 9-19.
[30]Deiana A M, Cau A, Coluccia E, et al. Genome size and AT-DNA content in thirteen species of Decapoda[M]//Schram F R, von Vaupel Klein J C. Crustaceans and the Biodiversity Crisis. Leiden, the Netherlands: Koninklijke Brill NV, 1999: 981-985.
[31]McLaren I A, Sévigny J M, Frost B W. Evolutionary and ecological significance of genome sizes in the copepod genusPseudocalanus[J]. Canadian Journal of Zoology, 1989, 67(3): 565-569.
[32]Lécher P, Defaye D, Noel P. Chromosomes and nuclear DNA of Crustacea[J]. Invertebrate Reproduction & Development, 1995, 27(2): 85-114.
[33]Badaracco G, Baratelli L, Ginelli E, et al. Variations in repetitive DNA and heterochromatin in the genusArtemia[J]. Chromosoma, 1987, 95(1): 71-75.
[34]Bentkowski P, Van Oosterhout C, Mock T. A model of genome size evolution for prokaryotes in stable and fluctuating environments[J]. Genome Biol Evol, 2015, 7(8): 2344-2351.
[35]Traut W, Rees D J, Dufresne F, et al. Amphipod genome sizes: first estimates for Arctic species reveal genomic giants[J]. Genome, 2007, 50(2): 151-158.
[36]Moens P, Rees D J, Belzile C, et al. Large genomes among caridean shrimp[J]. Genome, 2008, 51(2): 159-163.
[37]Nardon C, Weiss M, Vieira C, et al. Variation of the genome size estimate with environmental conditions inDrosophilamelanogaster[J]. Cytometry A, 2003, 55(1): 43-49.
[39]Petrov D A, Sangster T A, Johnston J S, et al. Evidence for DNA loss as a determinant of genome size[J]. Science, 2000, 287(5455): 1060-1062.
[40]Devos K M, Brown J K M, Bennetzen J L. Genome size reduction through illegitimate recombination counteracts genome expansion inArabidopsis[J]. Genome Research, 2002, 12(7): 1075-1079.
[41]Petrov D A. Mutational equilibrium model of genome size evolution[J]. Theor Popul Biol, 2002, 61(4): 531-544.
[42]Baack E J, Whitney K D, Rieseberg L H. Hybridization and genome size evolution: timing and magnitude of nuclear DNA content increases inHelianthushomoploid hybrid species[J]. New Phytol, 2005, 167(2): 623-630.
Survey of genome size in 10 invertebrates from hydrothermal vent and cold seep
Zheng Ping1,2,Wang Minxiao1,Li Chaolun1,Sun Song1
(1.KeyLaboratoryofMarineEcologyandEnvironmentalSciences,InstituteofOceanology,ChineseAcademyofSciences,Qingdao266071,China; 2.UniversityofChineseAcademyofSciences,Beijing100049,China)
Abstract:Knowledge of genome size is an important prerequisite for the development of many genomic resources. To better understand the evolution of genome size and their roles in environmental adaption, it is necessary to collect data covering a broad taxonomic base, especially from particular ecosystems. With high disturbance and unique environmental features, hydrothermal vent and cold seep offer a typical model to investigate the variation of genome size under extreme environments. Here we measured 10 deep-sea invertebrates from hydrothermal vent and cold seep by flow cytometry. The C-value ranged from 0.87 pg to 12.28 pg, and the crustaceans have larger genomes. Then we compared the three common species (Bathymodiolus platifrons, Shinkaia crosnieri and Alvinocaris longirostris) in both environments. Though with high level of intraspecific variations, no significant differences were revealed between two populations. We also compared the obtained genome sizes to their corresponding taxonomic relatives (at family or order level). No evidence of genome size expansion or reduction were found in invertebrates from chemosynthetic environments. In our study, there are no clear trends of the genome size variations under the extreme deep-sea ecological conditions.
Key words:hydrothermal vent; cold seep; chemosynthetic system; genome size; C-value; flow cytometry
收稿日期:2015-12-24;
修订日期:2016-02-17。
基金项目:中国科学院创新国际团队项目(20140491526);中国科学院战略性先导科技专项(A类)资助(XDA11020305);国家基金委-山东省联合基金项目“海洋生态与环境科学”(U1406403);自然科学基金(41106133,41230963)。
作者简介:郑平(1990—),女,湖北省孝感市人,从事深海生物适应性机制研究。E-mail: zhengping13@mails.ucas.ac.cn *通信作者:孙松,男,研究员,主要从事生物海洋学研究。E-mail:sunsong@qdio.ac.cn
中图分类号:Q948.8
文献标志码:A
文章编号:0253-4193(2016)06-0041-10
郑平,王敏晓,李超伦,等. 热液和冷泉无脊椎动物基因组大小测定及比较[J].海洋学报,2016,38(6):41—50, doi:10.3969/j.issn.0253-4193.2016.06.005
Zheng Ping,Wang Minxiao,Li Chaolun, et al. Survey of genome size in 10 invertebrates from hydrothermal vent and cold seep[J]. Haiyang Xuebao,2016,38(6):41—50, doi:10.3969/j.issn.0253-4193.2016.06.005
