白缘5S rDNA的扩增分析及其在染色体上的FISH定位
2016-07-01张伟伟付元帅邵燕王宝琴石东里张兰
张伟伟,付元帅,邵燕,王宝琴,石东里,张兰
(1.滨州学院 山东省黄河三角洲野生植物资源开发利用工程技术研究中心,山东 滨州 256603;2.上海海洋大学 水产与生命学院 ,上海 201306)
张伟伟1,付元帅2,邵燕1,王宝琴1,石东里1,张兰1
(1.滨州学院 山东省黄河三角洲野生植物资源开发利用工程技术研究中心,山东 滨州 256603;2.上海海洋大学 水产与生命学院 ,上海 201306)
摘要:本文分别对雌雄白缘的5S rDNA进行了PCR扩增和序列分析,并采用双色荧光原位杂交(FISH)技术,以白缘的5S rDNA序列和小麦的45S rDNA为探针,对其在白缘雌雄中期染色体上进行了FISH定位研究。结果表明,白缘5S rDNA序列无雌雄差异;5S rDNA的保守区序列为117 bp;5S rDNA和45S rDNA分别被定位于白缘的性染色体和第5号染色体上。同时从GenBank中获得了22种鱼的5S rDNA,运用DNAman软件构建了23种鱼的系统发育树,对白缘的进化地位进行了初步研究。本研究为阐明白缘在鱼类系统进化中的位置、重复序列在脊椎动物性染色体上的分布状况以及与性别决定与分化的关系,提供了资料积累和分析依据。
关键词:Liobagrus marginatus; PCR; 5S rDNA; 系统进化树; 染色体; FISH
1引言

核糖体RNA 基因(rDNA)是真核生物基因组中一类高度重复并有转录活性的基因家族。在细胞遗传学研究中已成为一种非常有用的遗传标记[5]。18S、5.8S、28S rDNA串联在一起形成45S rDNA,排列在某一染色体上形成核糖体RNA基因簇[6]。5S rRNA基因是串联重复的多拷贝基因,它们独立存在,并不与主体 rRNA (18S-5.8S-28S rRNA ) 基因相连,在生物体内含量丰富,广泛分布,而且进化上高度保守,因此常用来研究物种进化和同源性分析,被称为生物体内的活化石[7]。在某些鱼类中,45S rDNA 和 5S rDNA 位于相同的染色体上[8—9],在大部分鱼类中则位于不同的染色体上[10—11]。在某些有异型性染色体的物种中,5S rDNA 位于性染色体上[12—14],有可能参与性别决定和分化。

2材料与方法
2.1材料
根据 5S rDNA 保守序列设计引物:
上游 P1:5′-TACGCCCGATCTCGTCCGATC-3′
下游 P2:5′-CAGGCTGGTATGGCCGTAAGC-3′
由上海生工生物工程公司合成。
2.2方法
2.2.25S rDNA 的扩增、克隆及测序分析
2.2.3序列分析及系统发育树的构建
由 GenBank 获得另外 22 种鱼类的 5S rDNA的保守区序列(表1),用 DNAman 软件进行序列比对分析,并构建鱼类的系统发育树。
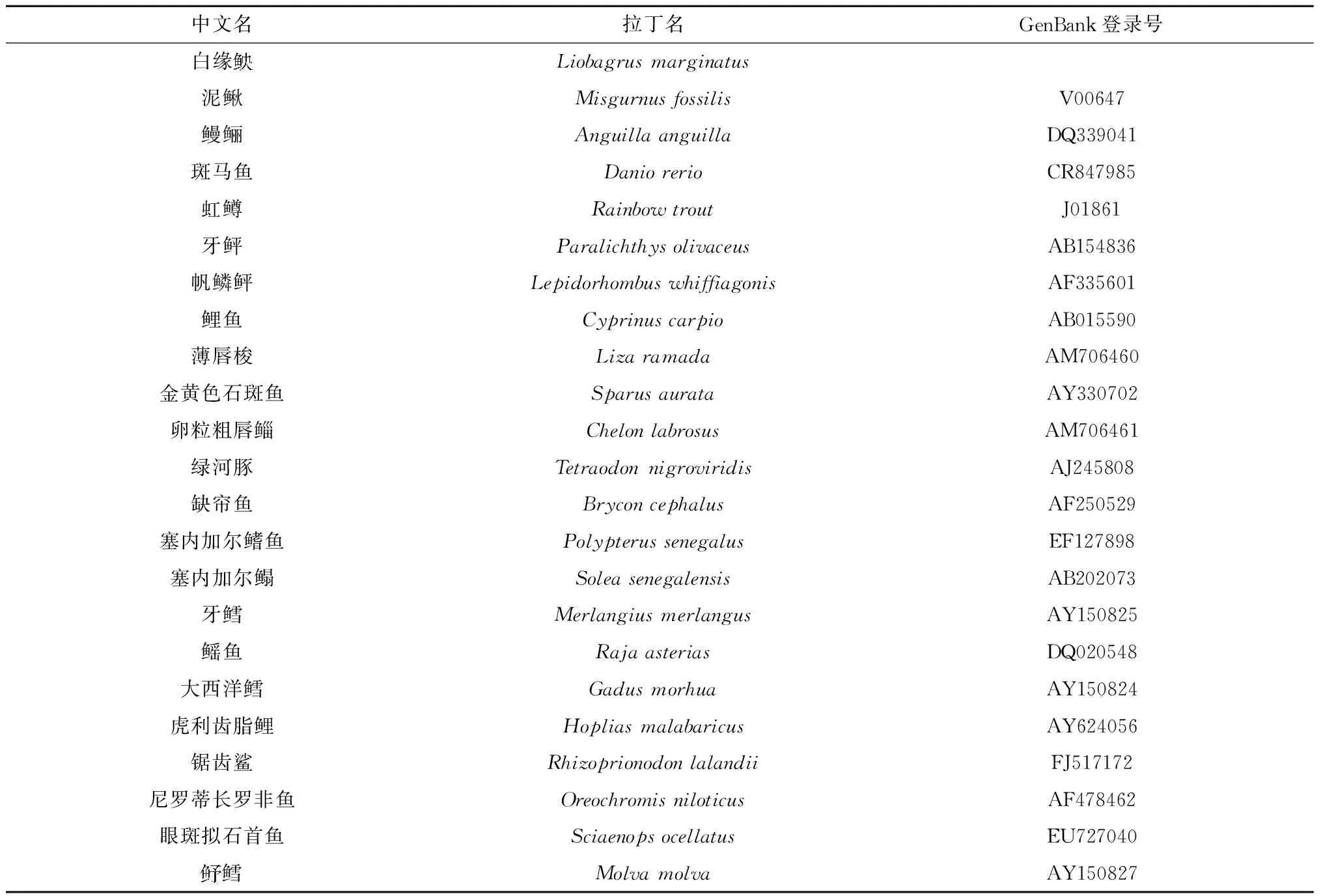
表1 分析用标本名录、GenBank 序列号
2.2.4染色体标本制备
2.2.5FISH 探针的制备

2.2.6荧光原位杂交
从-20℃取出染色体标本,依次进行如下处理:60℃烤片3 h;RNase A(0.1 μg/μL溶于 2×SSC中),37℃处理1 h;2×SSC室温洗涤3×2 min;70%,90%,100%乙醇梯度脱水,各2 min,室温干燥;70%甲酰胺变性液(pH7.0)72℃变性3 min,立即转入-20℃70%乙醇中,5 min后经90%,100%(-20℃)系列乙醇脱水,各5 min,室温干燥。将 5S rDNA和 45S rDNA 探针杂交液(标记探针4 μL、20×SSC 4 μL、去离子甲酰胺 20 μL、20%硫酸葡聚糖4 μL、PBS(pH7.0)/适量水,总体积40 μL),于100℃水浴变性10 min,冰浴5 min,加在处理过的染色体标本上,加盖玻片,橡皮泥封片,置80℃烤箱中3 min,转入湿盒中37℃杂交20 h。
2.2.7荧光原位杂交的信号检测
(1) 洗脱。杂交后的玻片,于2×SSC中除去盖片,按下列程序洗脱:50%甲酰胺/2×SSC 45℃漂洗3×5 min;2×SSC45℃漂洗3×5 min;BUFI室温漂洗5 min。
(2) 封阻。BUFⅡ37℃封阻30 min。
(3) 检测。按下列程序检测杂交信号,所有操作均在暗处进行:链亲和素-Cy3(Streptavidin-Cy3)和抗地高辛(Anti-Dig-Fluorescein Fab fragments)混合液,37℃结合30 min;BUFI漂洗3×5 min;生物素化链抗亲和素(Biotinylated anti-streptavidin)和羊抗地高辛偶联物(Fluorescein Anti-sheep IgG)混合液,37℃结合30 min;BUFI漂洗3×5 min;链亲和素-Cy3 37℃结合30 min;BUF I漂洗3×5 min。70%、90%、100%系列乙醇脱水,各5 min,20 μg/mL DAPI复染,并加入抗荧光萃灭剂PPD(0.5 μg/mL),盖上盖玻片,橡皮泥封片后,用Olympus BX60型荧光显微镜观察杂交结果并照片记录,然后对照片结果进行分析。
3结果与分析
3.1基因组 DNA 提取、PCR 扩增及序列测定
提取的基因组DNA经电泳和分光光度计检测,具有较高的纯度和浓度,将雌雄基因组DNA分别稀释成同一浓度(约 15 ng/uL)。采用 PCR 扩增 5S rDNA 基因序列,得到 250、450 和 1 200 bp 左右的3条扩增带,经克隆测序和GenBank中Blast分析,确定白缘鱼央的5S rDNA保守区序列为 117 bp(图1),并且发现在 5S rDNA 保守区之间存在不同的非转录间隔区(Nontranscribed Spacers,NTS)序列。
3.2序列分析和系统发育树的构建
3.3染色体标本制备
3.45S rDNA 和 45S rDNA 在染色体上的 FISH 定位

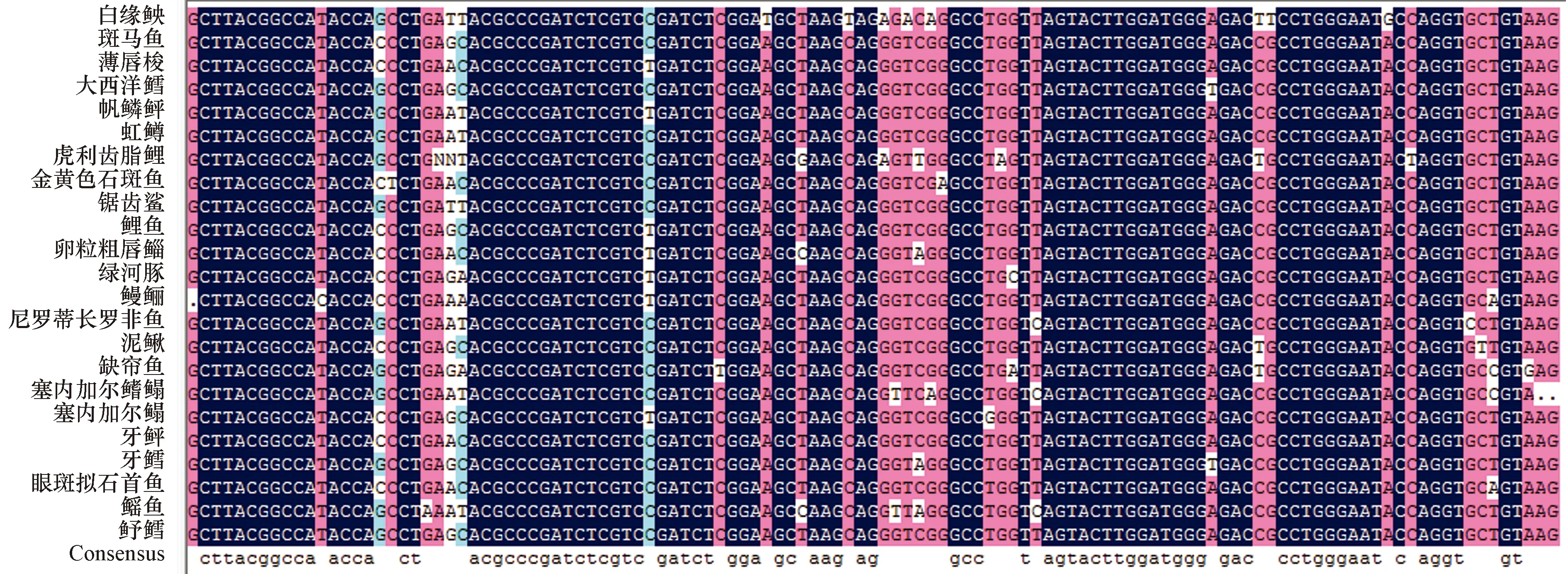
图1 23 种鱼的 5S rDNA 基因的序列比对分析Fig.1 The sequence alignment analysis of 5S rDNA gene of 23 fishes

图2 基于 5S rDNA 序列构建的 23 种鱼类的系统发育树Fig. 2 Phylogenetic tree of 23 fishes based on 5S rDNA sequences
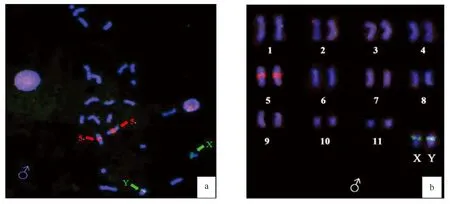
图3 5S rDNA和45S rDNA对白缘雄性中期染色体的FISH分析Fig.3 Localization of 5S rDNA Probe and 45S rDNA by FISH in male samples of Leiobagrus marginatusa.5S rDNA和45S rDNA 在中期染色体上的分布; b.白缘基于5S rDNA和 45S rDNA FISH图像核型图a.Distribution of 5S rDNA and 45S rDNA on metaphase;b.Homologous pairing of chromosomes based on 5S rDNA and 45S rDNA FISH
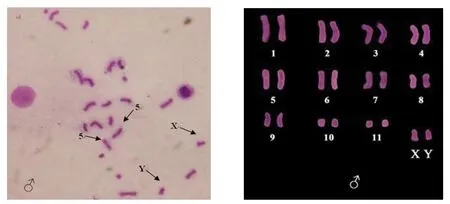
图4 5S rDNA和45S rDNA FISH定位后白缘染色体的Giemsa染色分析Fig.4 Giemsa Karyotype of the male Leiobagrus marginatus after FISH localization of 5S rDNA and 45S rDNA
4讨论

45S rRNA 基因簇在不同的生物中存在于不同的染色体上,该基因簇由高度保守区和可变区组成,在不同的真核生物中含有几百到几千个串联重复序列,每个重复序列由保守的密码序列区和变异较快的非密码间隔区组成,密码序列区(18S、5.8S、28S rRNA结构基因)与非密码间隔区(即转录间隔区)相间排列,在细胞进行转录时整个重复序列均可进行转录。真核生物 45S rRNA 基因簇中的串联重复序列在进化上表现出相当的一致性,并且在种内的独特性序列的同质性相对较高,但其他非独特性序列仍有一定的差异性。因此日益成为近亲分类群间的亲缘关系、系统发育关系和遗传多样性等研究中的优良分子遗传标记。本研究通过 FISH 技术将 45S rDNA 定位于白缘鱼央有丝分裂中期 5 号染色体的中部着丝粒处,但是否与 45S rDNA 位于染色体上核仁组织区(NORs)相一致,还有待进一步研究证实。
5S rDNA在染色体上的位置不像 45S rDNA 那样有与核仁相连系的明显的特征,关于它的定位,在不同物种间差异较大。在一些鱼类中,发现 5S rDNA 分布于性染色体上[12,19—20]。本研究发现,白缘鱼央的 5S rDNA 也位于其性染色体上,并且靠近 X 和 Y 染色体的着丝粒处。综合以前研究,5S rDNA 位于性染色体的这种现象是否就说明其与该物种性染色体相连锁或与性染色体进化有关,或者说与性别决定或性别分化有关,都有待于今后进一步的深入研究和探讨。
参考文献:
[1]成庆泰,郑葆珊. 中国鱼类系统检索(上册)[M]. 北京: 科学出版社,1987: 217-218.
Cheng Qingtai,Zheng Baoshan. The Fish Retrieval System (volume one)[M]. Beijing: Science Press,1987: 217-218.
[2]龙华. 青藏高原淡水鱼类的研究现状[J]. 水利渔业,2004,24(5): 5-7.
Long Hua. Research status and development prospect of freshwater fish in Qingzang Plateau[J]. Reservoir Fisheries,2004,24(5): 5-7.
[3]余先觉,周暾,李康,等. 中国淡水鱼类染色体[M]. 北京: 科学出版社,1989.
Yu Xianjue,Zhou Tun,Li Kang,et al. The Chromosomes of Freshwater Fishes in China[M]. Beijing: Science Press,1989.
Long Hua,Wang Dengqiang. Analysis on karyotype of leiobagrus marginatus(Günther)[J]. Freshwater Fisheries,2004,34(2): 9-10.
[5]Cross I,Merlo A,Manchado M,et al. Cytogenetic characterization of the soleSoleasenegalensis(Teleostei: Pleuronectiformes: Soleidae): Ag-NOR,(GATA)n,(TTAGGG)nand ribosomal genes by one-color and two-color FISH[J]. Genetica,2006,128(1/3): 253-259.
[6]徐延浩,李立家. 花生45S rDNA和5S rDNA的染色体定位研究[J]. 武汉植物学研究,2010,28(6): 649-653.
Xu Yanhao,Li Lijia. Physical mapping of the 45S rDNA and 5S rDNA in the Peanut (ArachishypogaeaL.)[J]. Journal of Wuhan Botanical Research,2010,28(6): 649-653.
[7]屈良鹄,余小强,洪德元,等. 万年青Ls-rRNA 5’端核苷酸序列及进化意义[J]. 植物分类学报,1990,28(6): 425-429.
Qu Lianghu,Yu Xiaoqiang,Hong Deyuan,et al. Ls-rRNA 5’Nucleotide sequence ofRohdeajaponica(Liliaceae) and its evolutionary significance[J]. Acta Phytotaxonomica Sinica,1990,28(6): 425-429.
[8]Almeida-Toledo L F,Ozouf-Costaz C,Foresti F,et al. Conservation of the 5S-bearing chromosome pair and co-localization with major rDNA clusters in five species ofAstyanax(Pisces,Characidae)[J]. Cytogenet Genome Res,2002,97(3/4): 229-233.
[10]Martins C,Galetti P M Jr. Two 5S rDNA arrays in Neotropical fish species: is it a general rule for fishes?[J]. Genetica,2001,111(1/3): 439-446.
[11]Sola L,Rossi A R,Annesi F,et al. Cytogenetic studies inSpratusauratus(Pisces,Perciformes): molecular organization of 5S rDNA and chromosomal mapping of 5S and 45S ribosomal genes and of telomeric repeats[J]. Hereditas,2003,139(3): 232-236.
[12]Ota K,Tateno Y,Gojobori T. Highly differentiated and conserved sex chromosome in fish species (Aulopusjaponicus: Teleostei,Aulopidae)[J]. Gene,2003,317: 187-193.
[13]Pisano E,Ghigliotti L,Mazzei F,et al. Mapping of 5S ribosomal genes revealed independent evolution of sex-linked heterochromosomes in two closely related teleostean species,Pagetopsis macropterus and Chionodraco hamatus (suborder Notothenioidei,family Channichthyidae)[J]. Annales de Génétique,2003,46: 98.
[14]Iturra P,Lam N,de la Fuente M,et al. Characterization of sex chromosomes in rainbow trout and coho salmon using fluorescenceinsituhybridization (FISH)[J]. Genetica,2001,111(1/3): 125-131.
[15]何世斌,柴连琴,谭珺隽,等. 荧光原位杂交技术的研究进展[J]. 植物科学学报,2014,32(2): 199-204.
He Shibin,Chai Lianqin,Tan Junjun,et al. Recent advances in fluorescenceinsituhybridization[J]. Plant Science Journal,2014,32(2): 199-204.
[16]徐延浩,杨飞,程有林,等. 45S rDNA和5S rDNA在南瓜、丝瓜和冬瓜染色体上的比较定位[J]. 遗传,2007,29(5): 614-620.
Xu Yanhao,Yang Fei,Cheng Youlin,et al. Comparative analysis of rDNA distribution in metaphase chromosomes of Cucurbitaceae species[J]. Hereditas,2007,29(5): 614-620.
[17]余先觉,周暾,李渝成,等. 中国淡水鱼类染色体[M]. 北京: 科学出版社,1989: 120-122,135-136,162.
Yu Xianjue,Zhou Tun,Li Yucheng,et al. The Chromosomes of Freshwater Fishes in China[M]. Beijing: Science Press,1989: 120-122,135-136,162.
[18]周爱芬,徐春晖,向凤宁,等. 应用5S rDNA间隔序列分析鉴定体细胞杂种[J]. 生物工程学报,1999,15(4): 529-532.
Zhou Aifen,Xu Chunhui,Xiang Fengning,et al. Study on the identification of somatic hybrids by PCR with 5S rDNA spacer sequence primers[J]. Chinese Journal of Biotechnology,1999,15(4): 529-532.
[19]Moran P,Martinez J L,Garcia-Vazquez E,et al. Sex chromosome linkage of 5S rDNA in rainbow trout (Oncorhynchusmykiss)[J]. Cytogenet Cell Genet,1996,75(2/3): 145-150.
[20]Stein I,Phillips R B,Devlin R H. Identification of the Y chromosome in chinook salmon (Oncorhynchustshawytscha)[J]. Cytogenet Cell Genet,2001,92(1/2): 108-110.
The amplification and FISH analysis of 5S rDNA in Liobagrus marginatus
Zhang Weiwei1,Fu Yuanshuai2,Shao Yan1,Wang Baoqin1,Shi Dongli1,Zhang Lan1
(1.ShandongProvincialEngineeringandTechnologyResearchCenterforWildPlantResourcesDevelopmentandApplicationofYellowRiverDelta,BinzhouUniversity,Binzhou256603,China;2.CollegeofFisheriesandLifeScience,ShanghaiOceanUniversity,Shanghai201306,China)
Abstract:In present research,5S rDNA was amplified by the usual 5S rDNA primers in Genomic DNA of female and male Liobagrus marginatus,and sequenced. The results of amplification showed that there were not different in 5S rDNA between female and male of L.marginatus,and length of 5S rDNA was 117 bp. Using the method of double color Fluorescence In situ Hybridization (FISH),5S rDNA and 45S rDNA on metaphase chromosomes were located in L.marginatus. The results showed that 5S rDNA and 45S rDNA were located on sex chromosome and the 5th chromosome,respectively. Phylogenetic tree of 5S rDNA in L.marginatus and 22 fishes else was constructed by DNAman,so evolution position of L.marginatus in fishes was studied preliminarily. This study offer data accumulating and basis of analysis,to illustrate phyletic evolution of fishes and repeat sequences on sex chromosome,and relation of heterochromatin or repeat sequences and sex determination & differentiation in vertebrates.
Key words:Liobagrus marginatus; PCR; 5S rDNA; phylogenetic tree; chromosome; FISH
收稿日期:2015-06-03;
修订日期:2015-12-24。
基金项目:国家自然科学基金项目(31400401);山东省自然科学基金项目(ZR2013EML002);山东省科技发展计划项目(2011YD21020)。
作者简介:张伟伟(1981—),女,山东省淄博市人,讲师,主要研究方向为动物分子细胞遗传。E-mail:wwzhang1225@163.com
中图分类号:S917.4
文献标志码:A
文章编号:0253-4193(2016)06-0082-07
Zhang Weiwei,Shao Yan,Wang Baoqin,et al. The amplification and FISH analysis of 5S rDNA inLiobagrusmaginatus[J]. Haiyang Xuebao,2016,38(6):82—88,doi:10.3969/j.issn.0253-4193.2016.06.009
