核多元基因选择和极限学习机在微阵列分析中的应用
2016-06-24董洪伟薛燕娜
杨 勤,董洪伟,薛燕娜
(江南大学 物联网工程学院,江苏 无锡 214122)
核多元基因选择和极限学习机在微阵列分析中的应用
杨勤,董洪伟,薛燕娜
(江南大学 物联网工程学院,江苏 无锡 214122)
摘要:针对微阵列数据样本量少、维度高的特点,结合当前数据降维方法中没有考虑特征与特征之间相关性的缺点,提出一种核最小二乘的特征基因选择方法。将解释变量空间通过非线性映射转换到高维空间上,再在高维空间上进行最小二乘回归,并采用极限学习机进行训练和预测。结果表明:对三种经典数据集的分类精度分别达到90.47 %,88.89 %,88.23 %,高于传统的机器学习算法,充分表明本方法的优越性。
关键词:微阵列分类;基因选择;核最小二乘;极限学习机
0引言
随着人类基因组计划的逐步实施和分子生物学迅猛发展,基因数据正在以前所未有的速度增长。微阵列技术就是顺应这一科学发展的产物,并且广泛应用于生物学、医学等领域[1]。微阵列数据是大数据时代的又一突破,但其样本少而维度高的特点极易给数据分析带来"维度灾难"。研究者们在数据降维上提出了多种方法,如典型相关分析(CCA)、主成分分析(PCA)等[2],但是这些降维方法仍达不到高精度生物学的研究目的。普通的单变量法具有低复杂度、高性能的优点,但它忽略了特征之间的联系,会丢弃一些有用的特征。而传统的多变量是基于条件熵之间的相交信息,仍然丢弃了一些被判定为冗余变量的特征,会对结果产生影响[3]。
偏最小二乘(PLS)综合了CCA和PCA的优点,常用于样本值远大于样本数的情况。传统的PLS只是在原始空间利用线性回归捕获基因间的线性关系,在实际应用中,线性方法常常不能捕获所有的基因信息。本文提出的基于核的PLS方法则通过将原始数据隐射到高位空间来揭示原始数据间的内在关系来提取有效的特征基因,并且利用极限学习机(ELM)[4]来对特征基因进行训练,从而对待测数据进行预测。
1微阵列分析概述
随着基因微阵列技术的快速发展,生物学家可以在某一个实验中检测到成千上万的基因表达水平,DNA基因序列通过转录变为mRNA,将细胞中的mRNA定量杂交配种可以得到cDNA或者寡核苷酸阵列,这些体现细胞中相关mRNA平均分子数的数据即为微阵列数据[5]。微阵列数据通常被表示成矩阵形式,m个基因在n个mRNA杂交样本上的基因表达数据以一个m×n矩阵表示,每列表示一个基因,每行表示一个mRNA样本[6]。在微阵列数据的分析中,研究者通常采用有监督的分类方法,比如K近邻算法(KNN)、朴素贝叶斯或者支持向量机(SVM)[7]等方法。本文采用ELM对三个数据集分类,分类精度都高于目前研究中的经典算法。
2核最小二乘和极限学习机
2.1核最小二乘算法
当原始数据和类标签存在线性关系时,可运用偏最小二乘回归(PLS)来分析数据。对于非线性关系和复杂关系,偏最小二乘却难以凑效。将核函数引入到PLS中,将其应用到非线性领域,这样改善了PLS只能用于线性模型分析的局限性,还能在一定程度上提高预测准确率。
在本文中,X∈RN·D表示N维输入数据矩阵(样本),Y∈RN·C表示相应的C维响应矩阵(标签)。假设X和Y都是零均值数据。PLS的目的是针对
avgmax‖v‖=1,‖c‖=1cov(t,u)=cov(Xv,Yc)
(1)
优化问题使用迭代方法计算v(X权重),c(Y权重),t(X贡献值),u(Y贡献值),其中,t=Xv,u=Yc分别是指X和Y的成分。当第一组成分t1和u1给定,第二组成分t2和u2则分别可以通过残差Ex=X-t1pT和EY=Y-t1qT计算出来,p和q指t针对X和Y的荷载,当满足一定条件时,这个过程才停止。本文提出的核最小二乘(KLS)利用非线性变换Φ将基因表达数据映射到高维的核空间K,不需要知道非线性映射的具体数学表达,只需要在满足Merser条件的情况下在原始空间上进行点积运算。为了在算法中实现点积操作,将v约束在这些点的一个线性跨度内,表达为
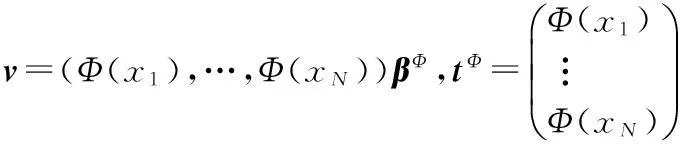
(2)
即
(3)
设Kx(xi,xj)为特征空间的格拉姆矩阵,h为期望的成分数。KLS的第一个成分是平方核矩阵βΦ的特征值,βΦλ=KYKXβΦ,λ为特征值。无论原始矩阵中的X和Y中有多少变量,这些核矩阵不受影响。可见基于核的PLS是一个强大的能迅速解决问题的算法。
2.2极限学习机
针对普通前馈神经网络存在网络结构不稳定、学校效率低、容易产生过拟合等问题,黄广斌提出了极限学习机,已经证明在神经网络中隐含层节点的参数随机赋值能够大大提高网络学习的效率,因而,本文在对微阵列数据分类中直接应用极限学习机,并与先前研究者用的SVM,KNN和ANN等分类算法进行比较。ELM模型表述如下:
普通前馈神经网络中,N个样本(xi,ti)∈Rm×Rn,m和n分别为输入和输出样本的维度,隐含层节点个数为M,M≦N,所选择的激活函数为g(·),因此,普通单层前馈网的模型为
(4)
式中(wi,bi)为隐含层神经元的参数值,βi为输出权值,j=1,2,…,N,则上述式(4)可以写成
Hβ=T
(5)
式中
(6)
为隐含层节点的输出矩阵,其第i列表示第i个隐含层神经元关于输入x1,x2,…,xN的输出值
(7)
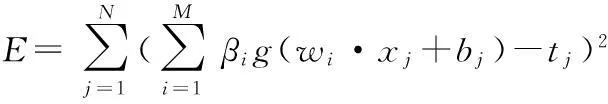
由文献[8]知,当M=N时,一旦给定(wi,bi)的值,输出矩阵H可逆,这时,ELM可以以很小的误差逼近各种学习样本,但是一般情况M远大于N,H为M×N的矩阵,这就要求所求H的广义逆H+,可采用奇异值分析法来求解广义逆矩阵,此时,可由式(3)求解
(8)
使用ELM时一个重要的过程是对激活函数的选取,激活函数有sigmoid,sin,RBF等,文献[8]中将这些激活函数应用于同一非线性样本数据上并作对比,发现sin函数在效率和精确度上最佳,因此,本文也选用sin作为ELM的激活函数。
3实验与结果分析
3.1实验数据准备
在先前的研究中,使用最多的微阵列数据是白血病(Leukemia)、乳腺癌(Breast)和结肠癌(Lung)三个数据集,它们包括二分类和多分类,其相关信息如表1所示。
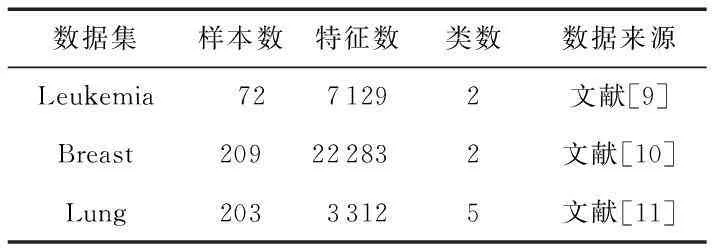
表1 本文使用的微阵列数据
分类器而言分类精度都最大,因此,在基因选择数为25时,对数据集Leukemia中的选择基因与文献[9]进行比较,发现重叠率达到76 %,说明本文提出的特征选择方法是有效的,如表2所示(其中黑体表示本文选取的特征基因与文献[9]重叠的基因名称)。
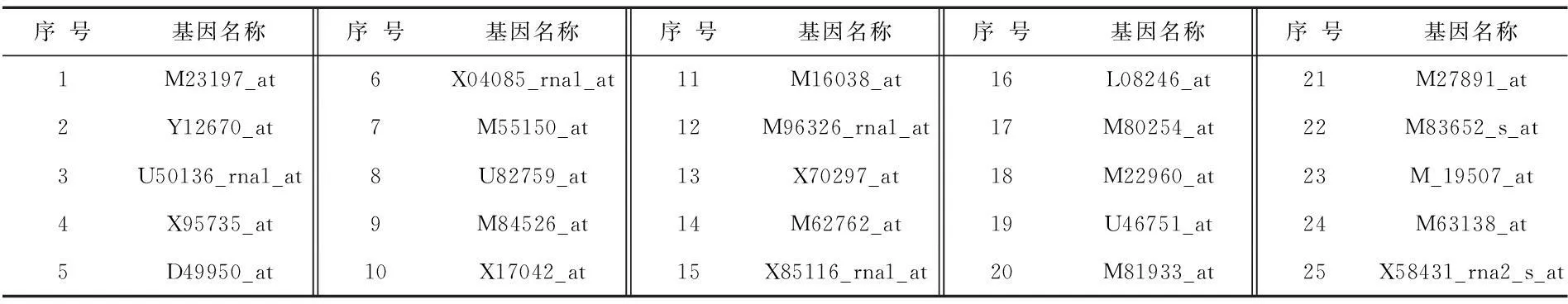
表2 对Leukemia数据集选取的前25个特征基因名称
3.2数据分类
本文对微阵列数据提取特征信息后,应用极限学习机来训练和预测。由于极限学习机参数是随机赋值的,因此其隐含层神经元个数的设置至关重要,本文激活函数为sin函数,隐含层神经元数N由0到2000每间隔11取一次值,对分类精度进行分析,发现随着N值的变大,精度变高,当N=1 800时,精度就达到了88 %,且当N继续增大时,精度变化不大;当N=1 870时,精度最大,达到90.47 %,如图1所示。
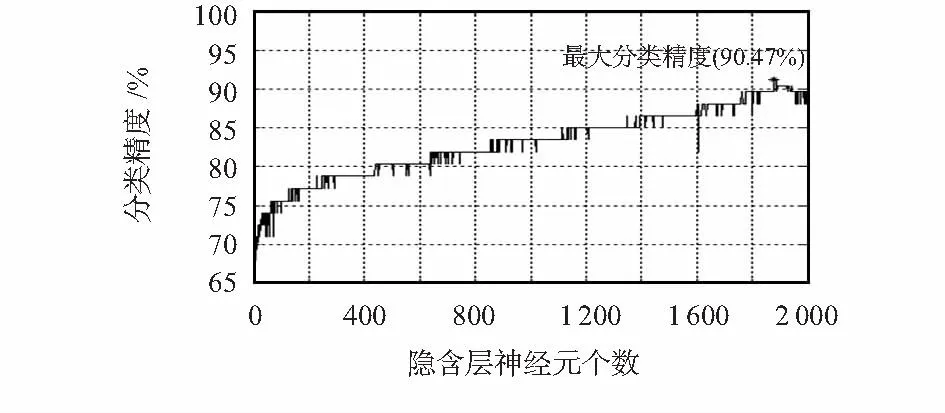
图1 隐含层神经元个数与分类精度效果图Fig 1 Effect figure of numbers of hidden layer neurons and classification percision
对Lymphoma和Lung数据集采用同样的方法进行特征基因选择和隐含层神经元个数设置,最后得到本文方法(KPL_ELM)对其分类的精度,与先前的研究[12]对比情况如表3所示(黑体精度值为几种方法中最大值)。

表3 本文方法与先前经典算法的分类精度比较(%)
从实验结果可以看出:本文提出的KPL_ELM算法对微阵列数据的特征基因选择和分类效果有显著的提高,说明本文方法是有效可行的。
4结束语
本文针对微阵列数据样本量小而维度高的特点,结合当前降维和特征提取方法速度慢、未考虑特征间联系的劣势,提出基于核的最小二乘方法来提取特征基因,并应用极限学习机来对实验数据进行训练和预测,与以往的研究对比,实验精确度得到了很大程度的提高,充分说明本文算法的优越性和实用性,为生物医学中对疾病的诊断和分类开拓了新思路。
参考文献:
[1]于化龙,高尚,赵靖,等.基于过采样技术和随机森林的不平衡微阵列数据分类方法研究[J].计算机科学,2012(5):190-194.
[2]金益,姜真杰.核主成分分析与典型相关分析相融合的人脸识别[J].计算机应用与软件,2014(1):191-193,270.
[3]Sun S,Peng Q,Shakoor A.A kernel-based multivariate feature selection method for micro-array data classification [J].PloS one,2014,9(7):102541.
[4]Huang G B,Zhu Q Y,Siew C K.Extreme learning machine:Theory and applications [J].Neuro-computing,2006,70(1):489-501.
[5]吕娜.极限学习机及其在无线频谱预测中的应用研究[D].兰州:兰州大学,2014.
[6]张丽娟,李舟军.微阵列数据癌症分类问题中的基因选择[J].计算机研究与发展,2009(5):794-802.
[7]李强,石陆魁,刘恩海,等.基于流形学习的基因微阵列数据分类方法[J].郑州大学学报:工学版,2012(5):121-124.
[8]Chacko B P,Krishnan V R V,Raju G,et al.Handwritten character recognition using wavelet energy and extreme learning machin-e[J].International Journal of Machine Learning and Cybernetics,2012,3(2):149-161.
[9]Golub T R,Slonim D K,Tamayo P,et al.Molecular classification of cancer:Class discovery and class prediction by gene expression monitoring [J].Science,1999,286(5439):531-537.
[10] Wang Y,Klijn J G M,Zhang Y,et al.Gene-expression profiles to predict distant metastasis of lymph-node-negative primary breast cancer[J].The Lancet,2005,365(9460):671-679.
[11] Bhattacharjee A,Richards W G,Staunton J,et al.Classification of human lung carcinomas by mRNA expression profiling reveals distinct adenocarcinoma subclasses [C]∥Proceedings of the National Academy of Sciences,2001:13790-13795.
[12] 王刚,张禹瑄,李颖,等.一种微阵列数据降维新方法[J].吉林大学学报:工学版,2014(5):1429-1434.
Application of kernel-based multiple gene selection method and extreme learning machine in microarray analysis
YANG Qin,DONG Hong-wei,XUE Yan-na
(College of The Internet of Things Engineering,Jiangnan University,Wuxi 214122,China)
Abstract:As quantity of microarray data sample is little and dimension of each sample is high,combined with disadvantages that in current data dimension reduction methods,correlation between features is not considered,put forward a kind of kernel-based least squares method for feature gene selection.Map explaining variable space to high dimension space,via nonlinear mapping transformation,and then carry out least-squares regression in high dimensional space;use extreme learning machine for training and predicting.The results show that classification precision of the three kinds of classic data set is 90.47 %,88.89 %,88.23 %,which is higher than traditional machine learning algorithms,which fully demonstrates superiority of this method.
Key words:microarray classification;gene selection;kernel least squares;extreme learning machine
DOI:10.13873/J.1000—9787(2016)05—0146—03
收稿日期:2015—08—17
中图分类号:TP 183
文献标识码:A
文章编号:1000—9787(2016)05—0146—03
作者简介:
杨勤(1990-),男,湖北黄冈人,硕士研究生,主要研究方向为模式识别、生物信息学。
