胃癌相关基因的生物信息学分析及蛋白互作网络构建
2016-06-01罗远卫石波云牛秋玲刘兆宇周新科
罗远卫 梁 敏 石波云 牛秋玲 刘兆宇 周新科
胃癌相关基因的生物信息学分析及蛋白互作网络构建
罗远卫 梁 敏 石波云 牛秋玲 刘兆宇 周新科
目的 分析胃癌和癌旁组织间差异表达基因的功能及其编码蛋白的相互作用,筛选出胃癌相关的关键基因。方法 从NCBI(美国国立生物技术信息中心)公共数据平台GEO(Gene Expression Omnibus)下载胃癌基因芯片数据GSE79973,采用R Bioconductor3.2.4软件对数据进行处理和分析,输出差异表达基因,并通过生物信息学工具DAVID、String、Cytoscape对差异表达基因进行生物学功能及其编码蛋白的互作分析。结果 通过分析GSE79973芯片数据,一共获得567个表达差异明显的基因,其中表达上调的有384个,表达下调的有183个,这些基因主要富集于细胞外区、细胞外基质、胶原蛋白、基底膜等,主要参与细胞增殖、周期以及粘附等生物学过程,并且在细胞外基质受体、局部粘附以及细胞色素P450代谢等肿瘤相关通路明显富集。初步鉴定了COL4A1、IL6、IL8、COL1A2、ITGA2、THBS1、COL5A1、COL3A1、ITGA1、COL2A1、COL4A2、BIRC5为胃癌相关的关键基因。结论 基因芯片结合生物信息学方法能够有效分析胃癌和癌旁组织间差异表达基因,并筛选出胃癌相关的关键基因,为进一步研究胃癌发病的分子机制提供指导。
胃癌; 基因芯片; 差异表达基因; 生物信息学
【Author′s address】 The Fifth Affiliated Hospital, Guangzhou Medical University, Guangzhou, 510700, China
胃癌是常见的恶性肿瘤之一,发病率和致死率居各种恶性肿瘤前列[1]。胃癌的发病是个复杂的生物学过程,包含多个基因共同作用、各种肿瘤相关通路的激活,并伴随着复杂的分子机制[2]。传统的针对单个或数个基因与肿瘤相关性及生物学功能的研究虽然能够发现某些基因在肿瘤形成过程中发挥作用,但效率低且不能较全面的探究胃癌形成过程中各种基因和通路的变化。近年来,基因芯片以及高通量测序等生物学技术的发展和应用为全面探究胃癌发病的分子机制创造了条件[3]。
基因芯片能大规模检测肿瘤组织和正常组织的基因表达情况,而通过生物信息学能够高效的对肿瘤和癌旁组织差异基因进行功能分析,并筛选出可能发挥作用的关键基因。如:Liu YZ等[4]通过分析两组非小细胞肺癌芯片数据发现KIAA1522 在肺癌组织中明显高表达且可能发挥重要作用,接着通过实验证明了KIAA1522 在非小细胞肺癌中的调控作用及可作为独立生物标记和新的治疗靶点的可能;Pénzváltó 等[5]通过分析卵巢上皮癌芯片数据以及免疫组化和RT-PCR的验证,证明了MEK1和卡铂耐药的相关性。另外,通过生物信息学还能间接对非编码RNA的功能进行预测,对非编码RNA在肿瘤中发挥的作用的研究具有指导意义。如赖明广等通过生物信息学方法分析miR-34a的靶基因并进一步预测其生物学功能,同时根据生物信息学分析验证了其对肠癌细胞增殖的影响[6]。因此,基因芯片技术和生物信息学分析在肿瘤疾病分子机制的研究中具有广泛的应用前景。
本组采用生物信息学等方法筛选基因芯片GSE79973中胃癌组织和癌旁组织差异表达的基因,分析胃癌相关基因的功能以及构建了这些基因编码蛋白的蛋白互作网络,并初步鉴定了12个胃癌相关的关键基因,为进一步研究胃癌发生、发展的分子机制提供基础。
1 材料与方法
1.1 芯片来源
胃癌芯片数据GSE79973来源于美国国立生物技术信息中心(NCBI)的GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)。该胃癌芯片数据由Qinshu Shao上传,包括10个胃癌组织样本和10个癌旁组织组织样本(表1),均为全基因组的mRNA芯片数据。

表1 胃癌组织和癌旁组织样本数据
1.2 数据处理
下载胃癌组织芯片GSE79973的CEL数据压缩包和探针文件后,通过R Bioconductor3.2.4软件的affyPLM软件包对芯片数据进行质量控制,并用affty软件包的RMA算法对affymetrix平台的原始数据进行背景校正、标准化和log2转换。接着,对数据中每个探针采用成组t检验、贝叶斯检验。最后通过GPL570平台将探针转换成基因名称,并利用R语言的limma包按照|logFC|>1.5,p值<0.05的入选标准筛选出表达差异明显的基因进一步分析。
1.3 GO 分析和KEGG通路富集分析
GO分析和KEGG通路富集分析广泛用于基因芯片数据的研究,DAVID是常用的基因功能分析的在线生物信息学软件,能够对大规模的基因组数据进行功能分析。我们将差异表达基因数据导入DAVID 6.7在线生物信息学网站(https://david.ncifcrf.gov/),以FDR(False Discovery Rate 错误发现率) <0.05作为入选标准,对差异明显的基因进行功能注释, 分析这些基因主要参与的生物学过程以及主要涉及的肿瘤相关通路。
1.4 蛋白互作网络构建
String (http://string-db.org/)是研究蛋白与蛋白相互作用的在线生物信息学网站。我们将表达差异显著的基因导入String 10.0在线分析网站 (http://string-db.org/),将最低互作分值(minemum required interaction score)设置成高度可信(high confidence:0.7)进行分析,获得了蛋白相互作用的数据,然后通过Cytoscape软件对结果进行可视化和进一步分析。
2 结果
2.1 芯片质量
相对对数表达RLE(Relative Log Expression)的箱线图(图1)显示经过背景校正和标准化后,20个胃癌组织芯片样本相对对数表达值均在垂直中央的位置,且基本接近0,表明芯片质量良好,具有可比性。

图1 胃癌芯片数据的质量控制
2.2 胃癌和癌旁组织中差异表达的基因
通过对这些数据分析,我们一共筛选出567个表达差异明显的基因,其中表达上调的有384个,表达下调的有183个,同时我们发现有36个基因的表达差异倍数在10倍以上。表达差异倍数大于10的基因在癌组织和癌旁组织的表达情况如图所示(图2),红色表示表达上调,绿色表示表达下调。
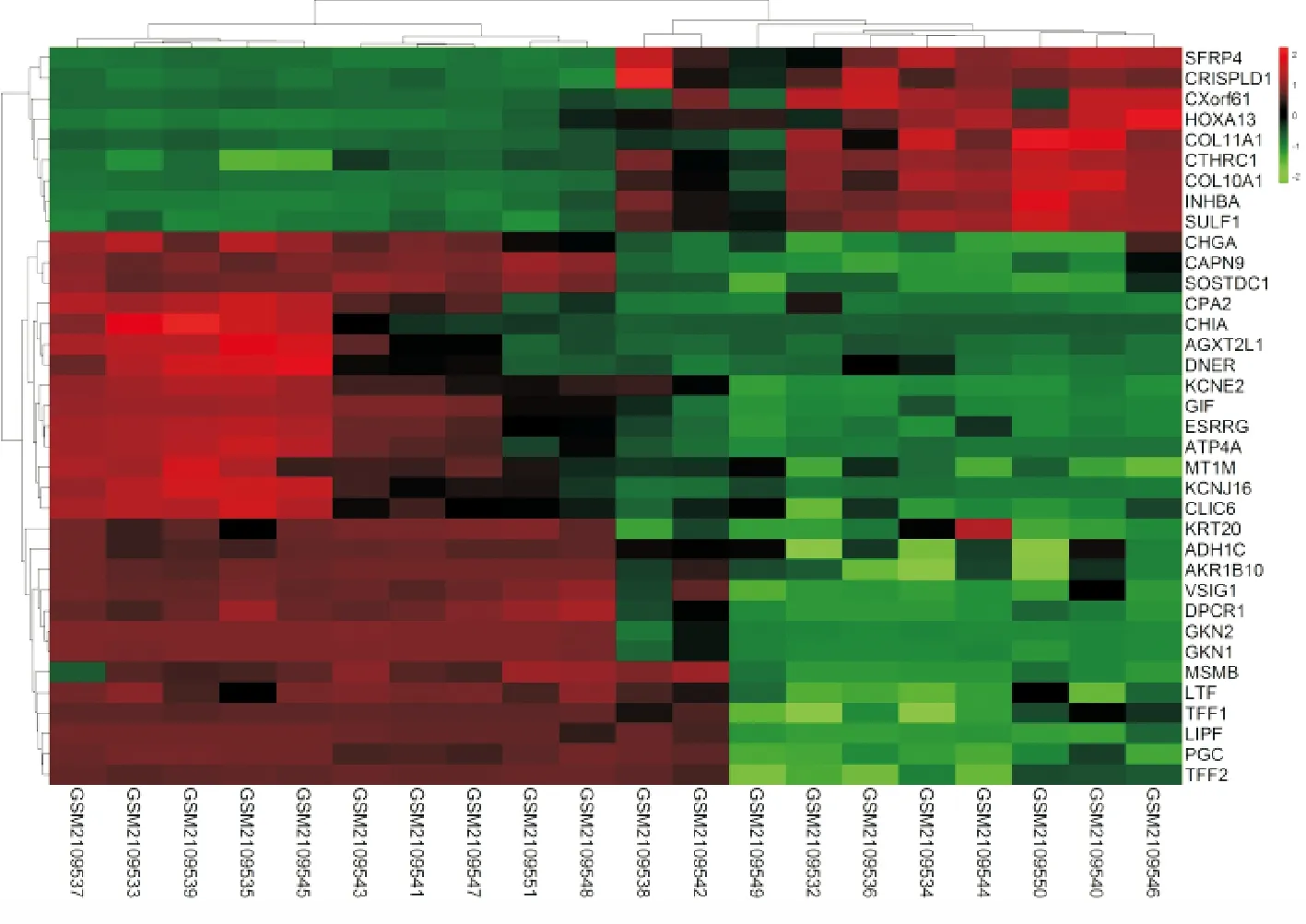
图2 表达倍数超过10倍的差异基因聚类图
2.3 差异表达基因的GO分析和KEGG通路富集分析
接着我们对表达差异的基因进行GO分析和KEGG通路富集分析。首先,通过分析这些差异表达基因的主要分子功能(Molecular Function)、主要参与的生物学过程(Biological Process)以及生物学组成(Cellular Component),发现这些基因主要的分子功能包括细胞外基质的组成以及与糖类、钙、生长因子、胶原蛋白等的结合;主要参与了细胞周期、细胞粘附、细胞周期进程、生物粘附、细胞增殖调节等细胞增殖、细胞周期调节以及细胞粘附能力调控等生物学过程(图3);而细胞组成分析显示这些基因大多参与细胞外区、细胞外基质、胶原蛋白、基底膜等的组成。KEGG通路分析显示,符合FDR(错误发现率)<0.5的标准的信号通路有3条,包括细胞外基质通路(hsa04512:ECM-receptor interaction)、局部粘附(9hsa04510:Focal adhesion)和细胞色素P450代谢(hsa00980:Metabolism of xenobiotics by cytochrome P450)信号通路(表2)。
2.4 蛋白互作网络分析
为了从蛋白水平对胃癌和癌旁组织差异表达基因的生物学功能和调控作用进行研究,找出胃癌相关的关键基因,我们构建了这些差异基因编码蛋白的互作网络。通过在线网站String的分析,我们得到了一份包含226个差异表达基因以及609条互作关系的蛋白互作网络(图3),接着,在Cytoscape软件上将该蛋白互作网络进行可视化处理,然后将互作关系大于10个节点度(Degree>10)的50个蛋白筛选出来进一步分析(图4),按照互作关系从多到少的原则,从中选出了COL4A1、IL6、IL8、COL1A2、ITGA2、THBS1、COL5A1、COL3A1、ITGA1、COL2A1、COL4A2、BIRC5这12个基因,这些基因与其他基因存在较强的相互作用关系,互作的节点度均大于18,可能为胃癌相关的关键基因。
3 讨论
胃癌的形成是一个非常复杂的生物学过程,包括多种肿瘤相关基因的异常表达,抑癌基因的失活,以及各种肿瘤相关通路的激活[7];从分子水平采用基因组学、蛋白质组学方法研究胃癌的分子机制是目前胃癌研究的主要方法之一[8],而基因芯片、高通量测序等现代生物技术的发展以及生物信息学应用为我们从分子水平揭示胃癌的发生、发展机制提供了很好的手段。在本研究中,通过采用生物信息学方法比较分析了胃癌组织与癌旁组织基因的表达差异,筛选出 567个表达差异明显的基因,并采用 DAVID 在线生物信息学分析工具进行对筛选出的差异基因进行 GO 功能注释及 KEGG 通路分析,最后通过蛋白-蛋白互作网络分析胃癌相关的关键基因。
在567个表达差异明显的基因中,表达上调的有384个,表达下调的有183个,其中差异倍数超过10倍的基因有36个。通过行GO 功能注释及 KEGG 通路分析,我们发现这些基因主要参与了细胞增殖和细胞周期调节,和细胞粘附能力调控等生物学过程,而细胞增殖异常和细胞周期调控失常是肿瘤发生的重要机制[9];细胞粘附能力的增强能促进癌细胞的侵袭和转移[10],这些生物学功能异常是肿瘤形成的基本特征。KEGG通路分析显示差异基因主要涉及3条肿瘤相关通路,细胞外基质受体互作通路(hsa04512:ECM-receptor interaction)、和局部粘附通路(hsa04510:Focal adhesion)能够影响细胞的增殖、分化、粘附和转移,主要与肿瘤的侵袭和转移相关[11]。而细胞色素P450代谢通路(hsa00980:Metabolism of xenobiotics by cytochrome P450)在外源物质(包括许多潜在的致癌物质和各种抗癌药物)的代谢中起着至关重要的作用,其中细胞色素P450在化学物质致癌中参与肿瘤启动和促进[12-13]。
接着通过构建蛋白质-蛋白质相互作用网络进行分析,我们发现互作关系大于10条的有50个基因,我们特别注意到了这50个基因中有13个(COL1A2、COL2A1、COL3A1、COL4A1、COL4A2、COL5A1、COL5A2、COL6A3、COL8A1、COL8A2、COL10A1、COL11A1、COL12A1)属于胶原蛋白基因家族,胶原蛋白基因家族是一类能够编码胶原蛋白(Collagens)的基因,主要涉及细胞外基质受体互作和局部粘附这两个通路;而胶原蛋白是细胞外基质的主要成分,在肿瘤发生侵袭和转移过程中,细胞外基质是第一道屏障[14],研究认为胶原蛋白基因与胃癌,特别是与胃癌的侵袭、转移关系密切,有作为胃癌预测标志物的可能[15]。为了进一步筛选出胃癌相关的关键基因,我们按照互作关系条数从多到少的原则从中选出了12个与其他基因存在较强相互作用的基因COL4A1、IL6、IL8、COL1A2、ITGA2、THBS1、COL5A1、COL3A1、ITGA1、COL2A1、COL4A2、BIRC5。其中有些基因在胃癌中的作用已有相关研究, 如COL1A2、COL3A1,、COL4A1、 COL4A2被研究认为与癌细胞的粘附能力和转移相关[16]; Peng L等[17]发现TGA1(integrin alpha1)能够与PRL-3.相互作用,进而促进胃癌的侵袭和转移,Chen J等[18]在 ITGA2的多态性与胃癌风险相关性的研究中发现ITGA2基因C807T多态性可能与胃癌的高风险,分化和胃癌的侵袭相关联,TGA1与TGA2均涉及细胞外基质受体互作通路。Zhao G等[19]报告了IL-6介导的JAK-STAT3-VEGF-C信号通路能够促进胃癌的生长,侵袭和淋巴管生成。尽管还有基因如COL5A1、COL12A1暂时未见与胃癌相关研究的报道,但是这些基因在胃癌组织中明显表达异常,均属于胶原蛋白基因家族,GO分析和KEGG通路分析提示有肿瘤相关生物学功能,因此可以推测很可能在胃癌发病过程中发挥重要作用。

表2 差异表达基因的GO分析
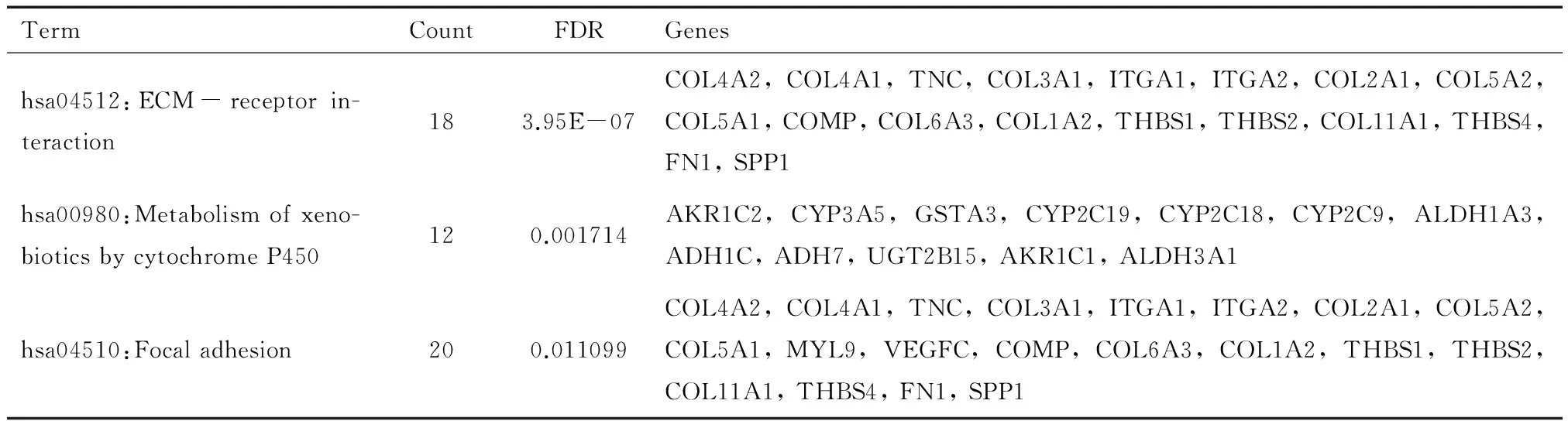
表3 差异表达基因主要涉及的KEGG通路

图3 差异表达基因的蛋白互作网络

图4 与其他基因互作关系较强的差异表达基因
综上所述,本研究采用生物信息学分析的方法对胃癌基因芯片数据GSE79973进行分析,筛选出胃癌组织和癌旁组织差异明显基因。通过对这些基因进行GO分析和KEGG通路分析,揭示这些基因的分子功能和主要涉及的肿瘤相关通路。接着,通过蛋白互作网络构建初步鉴定了COL4A1、IL6、IL8、COL1A2、ITGA2、THBS1、COL5A1、COL3A1、ITGA1、COL2A1、COL4A2、BIRC5为胃癌相关的关键基因,为进一步在细胞和分子水平研究胃癌发生、发展的分子机制提供指导。
[1] Ferro A, Peleteiro B, Malvezzi M, et al. Worldwide trends in gastric cancer mortality (1980-2011), with predictions to 2015, and incidence by subtype[J]. European Journal of Cancer, 2014, 50(7):1330-44.
[2] Wu W K, Lee C W, Cho C H, et al. MicroRNA dysregulation in gastric cancer: a new player enters the game[J]. Oncogene, 2010, 29(43):5761-71.
[3] Zhao X, Cai H, Wang X, et al. Discovery of signature genes in gastric cancer associated with prognosis[J]. Neoplasma, 2016, 53(2):239-245.
[4] Liu Y Z, Yang H, Cao J, et al. KIAA1522 is a novel prognostic biomarker in patients with non-small cell lung cancer[J]. Scientific Reports, 2016, 6:24876.
[5] Pénzváltó Z, Lánczky A, Lénárt J, et al. MEK1 is associated with carboplatin resistance and is a prognostic biomarker in epithelial ovarian cancer[J]. Bmc Cancer, 2014, 14(1):837-837.
[6] 赖明广, 青海涛, 王立生,等. miR-34a抑制SW480细胞增殖的机制研究[J]. 现代医院, 2015(8):7-9.
[7] Nagini S. Carcinoma of the stomach: A review of epidemiology, pathogenesis, molecular genetics and chemoprevention[J]. World Journal of Gastrointestinal Oncology, 2012, 4(7):156-69.
[8] 杨 倩,马 翔,李华驰,等. 基因组学与蛋白质组学在结直肠癌研究中的进展[J]. 中华临床医师杂志(电子版), 2013(03): 137-138.
[9] 沈 翀, 劳 山, 陈 罡. 肿瘤细胞增殖标志物蛋白功能及意义的研究进展[J]. 微创医学, 2009, 4(3):273-2.
[10]周 伟, 李新伟, 张冬云,等. 胃癌转移机制的研究进展[J]. 中国实用医药, 2011, 06(29):260-262.
[11]Bosman F.T. and I. Stamenkovic, Functional structure and composition of the extracellular matrix[J]. Pathol, 2003, 200(4): 423-8.
[12]Rooney P H, Telfer C, Mcfadyen M C, et al. The role of cytochrome P450 in cytotoxic bioactivation: future therapeutic directions[J]. Current Cancer Drug Targets, 2004, 4(3):257-265.
[13]Molina-Ortiz D, Camacho-Carranza R, González-Zamora J F, et al. Differential expression of cytochrome P450 enzymes in normal and tumor tissues from childhood rhabdomyosarcoma[J]. Plos One, 2014, 9(4):e93261.
[14]Gordon M K. and R.A. Hahn, Collagens. Cell Tissue Res, 2010, 339(1): 247-57.
[15]Yin Y, Zhao Y, Li A Q, et al. Collagen: A possible prediction mark for gastric cancer[J]. Medical Hypotheses, 2009, 72(2):163-5.
[16]Jinawath N, Furukawa Y, Hasegawa S, et al. Comparison of gene-expression profiles between diffuse- and intestinal-type gastric cancers using a genome-wide cDNA microarray[J]. Oncogene, 2004, 23(40):6830-6844.
[17]Peng L, Jin G, Wang L, et al. Identification of integrin alpha1 as an interacting protein of protein tyrosine phosphatase PRL-3[J]. Biochemical & Biophysical Research Communications, 2006, 342(1):179-83.
[18]Chen, Nan-Nan, Jia-Qi, et al. Association between ITGA2 C807T polymorphism and gastric cancer risk[J]. World Journal of Gastroenterology Wjg, 2011, 17(23):2860-6.
[19]Zhao G, Zhu G, Huang Y, et al. IL-6 mediates the signal pathway of JAK-STAT3-VEGF-C promoting growth, invasion and lymphangiogenesis in gastric cancer[J]. Oncology Reports, 2016: 1787-1795.
Bioinformatics Analysis of Gastric Cancer Related Gene and Protein-Protein Interaction Network Construction
LUOYuanwei,LIANGMin,SHIBoyun,etal
Objective Analyzing the function of differentially expressed genes between gastric cancer and adjacent tissues and the interactions of their encoded proteins, Selecting key genes associated with gastric cancer. Methods Microarray data of gastric cancer GSE79973 was downloaded from GEO (Gene Expression Omnibus) database of NCBI (National Center for Biotechnology Information). Using R Bioconductor 3.2.4 software for data processing and analysis, and the differentially expressed genes were exported. And bioinformatics tools including DAVID, STRING, Cytoscape was applied to analyze the biological function of differentially expressed genes and their encoded proteins interaction. Results Through the analysis of microarray data GSE79973, a total of 567 significantly differentially expressed genes was found, including 384 upregulated genes and 183 downregulated genes. These genes mainly enriched in the extracellular region, the extracellular matrix, collagen and basement membrane, and involved in the biological processes of cell proliferation, cell adhesion and cell cycle, and also significantly enriched in the KEGG pathway of ECM-receptor interaction, Focal adhesion and Metabolism of xenobiotics by cytochrome P450. Among these genes, 12 genes including COL4A1, IL6, IL8, COL1A2, ITGA2, THBS1, COL5A1, COL3A1, ITGA1, COL2A1, COL4A2 and BIRC5 were identified as key genes associated with gastric cancer. Conclusion Microarray combined with bioinformatics methods can effectively analyze the differentially expressed genes between gastric cancer and adjacent normal tissue, and select key genes related to gastric cancer, which may provide guidance for molecular mechanisms research of gastric cancer pathogenesis.
Gastric Cancer; Microarray; Differentially Expressed Genes; Bioinformatics
广东省教育厅特色创新类项目(编号:2015KTSC0110),广东省教育厅青年创新人才类项目(编号:2015KQNCX127),广州市科创委2014年科技惠民专项项目(编号:2014Y2-00092),广州市健康医疗协同创新重大专项二期拟立项项目(编号:201508020262)
周新科
R735.2; R34
A
10.3969/j.issn.1671-332X.2016.10.004
罗远卫 梁 敏 石波云 牛秋玲 刘兆宇 周新科 : 广州医科大学附属第五医院 广东广州 510700
