石蜡包埋胃镜组织幽门螺杆菌菌体DNA探索性研究
2016-05-14薄威王旭光张忠韩莹
薄威 王旭光 张忠 韩莹
[摘要] 目的 探讨从石蜡包埋胃镜组织中提取幽门螺杆菌菌体DNA的方法和策略。 方法 选取2008年5月~2010年5月中国医科大学263例胃癌术后石蜡包埋标本及2011年6月~2014年6月医学院附属中心医院和沈阳医学院附属第二医院206例幽门螺杆菌阳性石蜡包埋标本,利用试剂盒提取菌体DNA、PCR扩增,对扩增的结果进行统计分析。 结果 263例胃癌大体标本中, 231例检测出随意选取基因片段,阳性率为87.83% ;206例胃镜标本中,194例检测出幽门螺杆菌cagA基因,阳性率为89.81%。胃大体标本和胃镜标本DNA提取阳性率之间比较差异无统计学意义(P>0.05)。 结论 从石蜡包埋胃镜组织中提取幽门螺杆菌菌体DNA是高效可行的。
[关键词] 石蜡包埋组织;幽门螺杆菌;DNA;基因扩增
[中图分类号] R735.2 [文献标识码] B [文章编号] 1673-9701(2016)08-0121-03
从胃癌术后石蜡包埋组织中提取人体基因组DNA已经广泛应用于各种回顾性研究,随着研究的深入和方法的改进,人们从石蜡包埋的少量胃镜组织也可获得较理想的DNA[1-3],但国内尚未见从石蜡包埋胃镜组织提取幽门螺杆菌菌体DNA的报道,主要是由于胃镜组织块本身很小,并且H.pylori主要定殖部分胃小凹和腺体内,数量少之又少,限制了对H.pylori DNA特点的深入研究。目前为分析H.pylori的DNA,人们多采用胃镜获取新鲜胃黏膜,随即在培养基上进行涂抹,于体外进行细菌培养,从而获得菌体DNA,这样改变H.pylori生存的微环境,无形中增加了人为因素。因此,我们在从胃癌术后石蜡包埋组织中提取人体基因组DNA的研究基础上,进行多方面尝试从石蜡包埋的胃镜组织中提取H.pylori DNA。
1 材料与方法
1.1 材料来源
263例胃癌术后石蜡包埋标本来自于中国医科大学(2008~2010年),206例胃镜石蜡包埋标本来自于沈阳医学院附属中心医院和沈阳医学院附属第二医院(2011~2014年)室温保存至今。
1.2 H.pylori的HE染色及特染
206例胃镜标本按常规方法每例标本制作切片3张,厚度为5 μm,分别进行常规HE染色、H.pylori特殊染色——吉姆萨染色(吉姆萨染色液G1010 北京索莱宝科技有限公司)和亚甲蓝(MST-8027 胃幽门螺杆菌染色试剂盒迈新试剂)染色。具体方法见试剂盒。
1.3 DNA提取方法
石蜡组织提取DNA采用的是北京基因公司的试剂盒(GT pureTMFFPE tissue DNA Extraction KIT NO. 56404),胃癌术后标本,每例标本连续切10~12片,每片厚5 μm,胃镜标本每例连续切18~20片,切片厚度为4 μm,放入1.5 mL离心管中,然后按试剂盒操作提取DNA。
1.4 PCR反应
胃癌术后标本随机选取一对基因进行PCR扩增,上游引物5 CAA GCA GTC ATC CTT CTC TC 3; 下游引物5GCA CTT AGG GAT CCC AT GAA 3; 扩增条件94℃ 7min,94℃ 30s,54℃ 20s,72℃ 30s,72℃ 10min,40个循环,4℃保存。
胃镜标本对H.pylori的cagA基因进行巢式PCR扩增。引物序列为cagA 1F 5 CGT TGA TAA CGA TAG GGA TAA C 3;cagA1R 5 GAT CCC CAA ATT TCT GAA AGC TCT T 3;cagA 2F 5 CGA TAG GGA TAA CAG GCA AGC TT 3;cagA1R 5 CTG AAA GCT CTT TGT GGA AGA TTC3两次PCR反应体系均为20 μL,其中含模板DNA 2 μL、10×PCR buffer 2.5 μL、dNTP 2 μL、引物1.25 μL、taq酶0.2 μL,去离子水补足到20 μL。第二次PCR中DNA模版为第一次PCR产物,PCR反应条件为:95℃ 5 min,95℃ 1 min,54℃ 1 min,72℃ 1 min,72℃ 5 min, 两次PCR各进行30个循环。
1.5 统计学方法
采用SPSS16.0统计学软件进行数据分析,计数资料组间比较进行χ2检验,P<0.05为差异具有统计学意义。
2 结果
2.1 H.pylori判定
分别进行HE染色、亚甲蓝染色、吉姆萨染色,三者之一阳性,即为阳性,见封三图10、11、12。
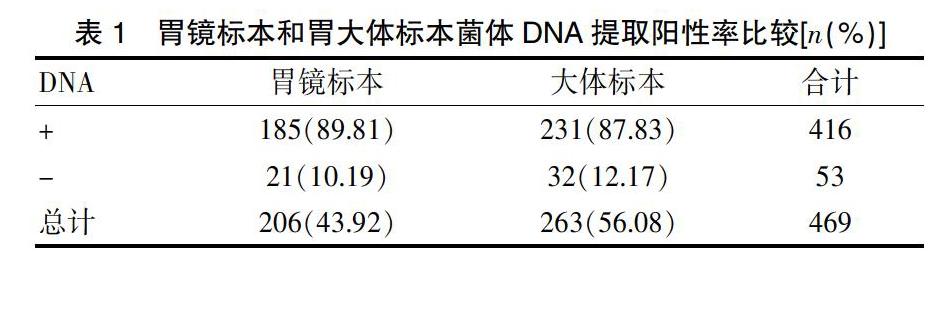
2.2 胃镜标本和大体标本菌体
DNA提取的比较(χ2=0.449,P=0.45>0.05),所以大体和胃镜标本DNA提取阳性率之间无显著性差异。见表1。
2.3 菌体DNA的检测
扩增产物以2%琼脂糖凝胶电泳检测,FlourChem FC2紫外凝胶成像系统观察结果(封三图13)。胃镜206例标本中有194例检测出cagA条带,检出率为89.81%。大体263例标本中有231例检测出随意选取基因条带,检出率为87.83% 。
3 讨论
每年约有900万的胃癌新发病例,其中75%是与幽门螺杆菌(H.pylori)的感染密切相关的[4]。相关领域专家对H.pylori致病性特别是菌体 DNA的特性进行了广泛而深入的研究[5-7]。在对菌体DNA研究时,多采用活检胃黏膜体外菌培养的方法,虽然菌培养可以获得高浓度和纯度的菌体DNA,但菌培养是人为制造的体外环境,所以并不能完全反映H.pylori在人体内微环境的变化。那么有没有更好的方法能替代菌培养呢?人们试想从甲醛固定-石蜡包埋(FFPE)组织中提取菌体DNA,临床上对于石蜡包埋组织的制备多在取得新鲜组织1周内完成,这不但可以较好地表达H.pylori在人体内微环境中的生长和繁殖情况,并且可以解决在短时间内收集到大量新鲜标本的难题。但由于石蜡包埋胃镜组织中菌体量极少,国内尚无人尝试。我们借鉴了大量在FFPE组织中提取人体基因组DNA的经验[8-10],并且采用巢式PCR的方法成功地提取到菌体DNA。结果显示应用FFPE从组织中提取菌体cagA的DNA阳性率为89.81%,这与国内宫月华[11]在体外培养中选取菌株cagA基因检测的阳性率94.4%(101/107)的结果是相似的。在我们前期的实验研究中,对263例福尔马林固定的大材标本进行DNA提取,其中有231例提取得到DNA,阳性率87.83%,低于此次的石蜡包埋标本提取DNA的阳性率,但差异无统计学意义。充分说明从FFPE组织中提取菌体DNA的方法是可行的,可替代体外菌培养。
从FFPE组织中提取菌体DNA有助于追踪长时间的肿瘤分子标志的改变,利于回顾性研究[12]。但我们对菌体DNA提取的阳性率并不是100%的,尽管在提取菌体DNA之前我们对H.pylori进行了特染,选择了H.pylori阳性的标本。考虑的主要原因为经过石蜡包埋容易造成DNA不可逆降解、碱基与组蛋白交联以及苯、寡聚核苷酸片段等一些PCR抑制剂的存在,这使PCR扩增成为难题[13-15]。在应用传统PCR方法无法从FFPE组织中扩增出清晰的电泳条带时,我们选择两对嵌套的引物,即应用巢式PCR后,获得了清晰条带,增加了可重复性,提高了特异性。另外,我们还发现其他一些策略可提高DNA产量:①在熔蜡的次数选择两次效果最好,次数过少蜡不能被完全溶解掉,次数过多易造成DNA的损失,而且第一次根据包埋组织石蜡的熔点的温度不同,选择65℃水浴融蜡2 h比较彻底。②在倾倒离心后的上清液时力度一定要温和,因为组织很轻,力度大易造成菌体DNA的流失。③切片的厚度在4~6 μm较适宜,过厚不利于蛋白酶消化,影响DNA产量,过薄易造成人为DNA损伤。我们认为由于切片的大小不同,以切片的重量计算更加准确,每次抽提25~35 mg切片较适宜,既能充分消化又能提高DNA的产量。尽管我们在石蜡包埋组织中提取菌体DNA取得了一些成效,但是也存在着问题。如果选择H.pylori通用引物可能阳性率会更高些。另外,如何控制菌体过少时的离心时间和转速,避免菌体微滴流失,也尚需解决,下一步我们将扩大样本例数进一步改进方法,总结经验,以提高菌体DNA的阳性率。
[参考文献]
[1] Scholte GH,van Doorn LJ,Quint WG,et al. Polymerase chain reaction for the detection of Helicobacterpylori in formaldehyde-sublimate fixed,paraffin-embedded gastric biopsies[J]. Diagn Mol Pathol,1997,6(4):238-243.
[2] Schmitt BH,Regner M,Mangold KA. PCR detection of clarithromycin-susceptible and -resistant Helicobacter pylori from formalin-fixed,paraffin-embedded gastric biopsies[J]. Mod Pathol,2013,26(9):1222-1227.
[3] Rabelo-Goncalves E,Roesler B,Guardia AC,et al. Evaluation of five DNA extraction methods for detection of H. pylori in formalin-fixed paraffin-embedded(FFPE) liver tissue from patients with hepatocellular carcinoma[J]. Pathol Res Pract,2014,210(3):142-146.
[4] Ameer A,Memon,Nawfal R,et al. Vacuolating cytotoxin genotypes are strong markers of gastric cancer and duodenal ulcer-associated helicobacter pylori strains:A matched case-control study[J]. J Clin Microbiol,2014,52(8):2984-2989.
[5] Heydt C,Fassunke J,Künstlinger H,et al. Comparison of pre-analytical FFPE sample preparation methods and their impact on massively parallel sequencing in routine diagnostics[J]. J Clin Microbiol,2014,9(8):23-29.
[6] Mitui M,Datel A,Leos NK,et al. Novel Helicobacter pylorisequencingtest indentifies high rate of clarithromycin resistance[J]. J Pediatr Gastroenterol Nutr,2014,59(1):6-9.
[7] Shah RH,Scott SN,Brannon AR,et al. Comparison of five protocols to extract DNA from paraffin-embedded tissues for the detection of human papillomavirus[J]. J Pathol Res Pract,2015,211(2):150-155.
[8] F Byeong,Hwa Yun,Lihua Yao,et al. Ormalin-fixed paraffin-embedded tissue as a source for quantitation of carcinogen DNA adducts:Aristolochic acid as a prototype carcinogen[J]. Carcinogenesis,2014,35(9):2055-2061.
[9] Anthony N Snow,Aaron A Stence,Jonathan A Pruessner,et al. A simple and cost-effective method of DNA extraction from small formalin-fixed paraffinembedded tissue for molecular oncologic testing[J]. Carcinogenesis Clinical Pa-thology,2014,30(14):1-10.
[10] Gailey MP,Stence AA,Jensen CS,et al. Multiplatform comparison of molecular oncology tests performed on cytology specimens and formalin-fixed,paraffin-embedded tissue[J]. Cancer Cytopathol,2011,123(1):30-39.
[11] 宫月华,柳云恩,孙丽萍. 中国辽宁地区人群幽门螺杆菌感染菌株与相关性胃疾病的关系[J]. 世界华人消化杂志,2007,15(33):3462-3467.
[12] Kerick M,Isau M,Timmermann B,et al. Targeted high throughput sequencing in clinical cancer settings:Formaldehyde fixed-paraffin embedded(FFPE) tumor tissues,input amount and tumor heterogeneity[J]. BMC Med Genomics,2011,68(4):1775-1779.
[13] Bonin S,Hlubek F,Benhattar J,et al. Multicentre validation study of nucleic acids extraction from FFPE tissues[J].Virchows Arch,2010,457(3):309-317.
[14] Hu YC,Zhang Q,Huang YH,et al. Comparison of two methods to extract DNA from formalin-fixed,paraffin-embedded tissues and their impact on EGFR mutation detection in non-small cell lung carcinoma[J]. Asian Pac J Cancer Prev,2014,15(6):2733-2737.
[15] Huijsmans1 CJ,Damen J,van der Linden JC,et al. Comparative analysis of four methods to extract DNA from paraffin-embedded tissues:Effect on downstream molecular applications[J]. BMC Research Notes,2010,239(3):1756-1761.
(收稿日期:2015-12-21)
