水稻雄性不育突变体gamyb 5的鉴定与基因定位
2016-05-09杨正福张迎信孙廉平张沛沛轩丹丹刘嶺3胡霞李紫荷占小登吴玮勋曹立勇程式华
杨正福张迎信孙廉平张沛沛轩丹丹刘嶺,3胡霞李紫荷占小登吴玮勋曹立勇,*程式华,*
(1中国水稻研究所/国家水稻改良中心/浙江省超级稻研究重点实验室,杭州311401;2沈阳农业大学农学院,沈阳110866;3河南农业大学农学院,郑州450002;#共同第一作者;*通讯联系人,E-mail:caoliyong@caas.cn,shcheng@mail.hz.zj.cn)
水稻雄性不育突变体gamyb 5的鉴定与基因定位
杨正福1,#张迎信1,#孙廉平1,2张沛沛1轩丹丹1刘嶺1,3胡霞1李紫荷1占小登1吴玮勋1曹立勇1,*程式华1,*
(1中国水稻研究所/国家水稻改良中心/浙江省超级稻研究重点实验室,杭州311401;2沈阳农业大学农学院,沈阳110866;3河南农业大学农学院,郑州450002;#共同第一作者;*通讯联系人,E-mail:caoliyong@caas.cn,shcheng@mail.hz.zj.cn)
杨正福,张迎信,孙廉平,等.水稻雄性不育突变体gamyb5的鉴定与基因定位.中国水稻科学,2016,30(2):143-151.
摘 要:在(60)Co-γ射线辐射诱变籼稻中恢8015的突变体库内发现了一个无花粉型雄性不育突变体gamyb5。gamyb5的株型、株高、分蘖数等农艺性状与野生型中恢8015无差异,而其花药细长且呈白色半透明状,花药中无花粉粒。花药半薄切片观察结果表明,gamyb5的小孢子母细胞减数分裂异常,没有形成正常的四分体和小孢子,并且绒毡层异常伸长,细胞程序性死亡延迟。对以gamyb5为母本,与野生型中恢8015和广亲和粳稻品种02428分别配制的杂交组合遗传分析表明,gamyb5突变性状受一个隐性核基因控制。利用gamyb5和02428杂交的F2定位群体,最终将突变基因精细定位于第1染色体长臂的ZF-29和ZF-31两个标记之间,物理距离约16.9kb。对该区域内2个完整的开放阅读框测序分析发现,编码受赤霉素诱导的MYB转录因子基因LOC_Os01g0812000的第2外显子存在8个碱基的缺失,导致翻译提前终止。qRT-PCR检测到影响花药发育的调控因子UDT1、TDR、CYP703A3和CYP704B2的表达量在突变体中比野生型中极显著降低。进一步证明GAMYB在花药减数分裂和绒毡层细胞程序性死亡过程中起关键作用。
关键词:水稻;雄性不育;基因定位;gamyb5
水稻雄性生殖发育是一个复杂的生物学过程,也是水稻繁衍后代和产量形成的关键过程。首先,雄蕊原基的三层细胞经过数轮细胞分裂和分化,形成由四层体细胞包围着花粉母细胞的花药结构。这四层体细胞由外到内依次为表皮层、内皮层、中间层和绒毡层[1-6]。其次花粉母细胞经过减数和有丝分裂最终形成三核花粉粒。最终花药开裂散粉,至此整个雄性生殖发育过程完成。
水稻因其经济上的重要性以及较小的基因组,成为了分子生物学和功能基因组学研究的单子叶模式植物。目前,已克隆了超过20个水稻隐性核雄性不育基因[7]。根据不育基因的功能和表达时期的差异,可将水稻隐性核雄性不育基因大致分为调控小孢子母细胞发育、减数分裂、绒毡层发育、小孢子和花粉外壁发育以及花药开裂等相关的不育基因。MSP1(Multiple Sporocyte 1)是水稻中第1个克隆的早期调控小孢子细胞发育的基因,它编码富含亮氨酸的受体蛋白激酶。msp1突变体产生过量的雌雄孢子母细胞,花粉囊细胞壁和绒毡层发育紊乱,小孢子母细胞发育停留在减数分裂Ⅰ期,而大孢子母细胞的发育不受影响,最终导致花粉彻底败育[8]。PAIR(Pairing Aberration In Rice)系列基因PAIR1、PAIR2和PAIR3参与减数分裂中同源染色体联会配对[9-11]。ZEP1(Zeaxanthin Epoxidase1)[12]、MEL1[13]、PSS1[14]都是调控水稻减数分裂的基因。已克隆的调控水稻花药绒毡层发育的育性基因TDR(Tapetum Degeneration Retardation)[15]、UDT1(Undeveloped Tapetum1)[16]、GAMYB[17]、PTC1(Persistent Tapetal Cell1)[18]和API5(Apoptosis Inhibitor5)[19]等转录因子,主要调控花药绒毡层的PCD过程和花粉粒的形成过程。还有一类基因与小孢子花粉壁蜡质角质等物质形成有关,该类基因突变致使花粉囊和花粉外壁发育受阻,导致花粉败育。例如:WDA1(Wax-Deficient Anther1)[20]和DPW(Defective Pollen Wall)突变体花粉囊蜡质显著减少,花粉粒皱缩退化,导致花粉败育[21]。CYP704B2[22]和CYP703A3[23]都属于细胞色素P450基因家族,在脂肪酸的羟基化过程中起重要作用,表明脂肪酸ω-羟基化在植物雄配子体发育和孢子体发育过程中的角质合成和花粉粒外壁形成中发挥重要作用。AID1(Anther Indehiscence1Gene 1)控制水稻花药的开裂和花粉的育性。aid1突变体中花药不开裂、花粉发育异常,引起雄性不育[24]。OsGAMYB在大麦、黄瓜、拟南芥中的同源基因已克隆。GAMYB是大麦糊粉层中赤霉素依赖的α淀粉酶转录正调控因子。在水稻中,GAMYB除了影响糊粉层和花粉囊发育过程中淀粉酶的表达,还调控绒毡层降解的PCD过程和花粉发育过程,是水稻花粉发育早期所必需的转录因子。但水稻雄配子体发育过程和调控机制并未被完全探明,对控制gamyb5突变体雄性不育基因的定位与克隆有助于更全面地水稻雄配子体发育的分子机制,为雄性不育株系进行育种应用提供理论基础和基因资源[25]。
本研究通过60Co-γ辐射诱变籼稻品种中恢8015,从其后代中分离鉴定出一个无花粉型雄性不育突变体gamyb5,对其进行表型鉴定和基因定位等研究,并筛选出候选基因,为进一步研究该基因功能和探究其育种应用潜力提供理论基础。
1 材料与方法
1.1 试验材料
籼型水稻恢复系中恢8015(ZH8015)经60Co-γ辐射诱变,在其后代中鉴定出一个无花粉型雄性不育突变体gamyb5,通过挖稻桩的方式进行繁殖。所有亲本材料于2013年夏种植于中国水稻研究所实验基地,种植与管理同大田生产。
1.2 遗传分析及定位群体的构建
以突变体gamyb5为母本,分别以野生型中恢8015和广亲和常规粳稻品种02428为父本,配制杂交组合;观察BC1F1和F1的表型,分单株收获种子。在BC1F2和F2群体统计无花粉型与正常野生型的分离比例,用于遗传分析和基因定位。所有材料2014年种植于中国水稻研究所杭州富阳实验基地和海南陵水基地,种植与管理同大田生产。
1.3 花粉育性调查
抽穗期当日开花前,取野生型和突变体的颖花并套自交袋,每株3个重复,挂牌标记,用1%I2-KI染色镜检,拍照并计算典败、圆败、染败、正常花粉粒比率。成熟期摘取自交袋室内考种,计算结实率。
1.4 gamyb5突变体花药的半薄切片观察
取野生型和突变型不同发育时期的小花,于FAA固定液(V38%甲醛∶V冰乙酸∶V50%乙醇=5∶5∶90)中固定,利用真空泵缓慢抽气,近真空状放置,缓慢放气至样品全部沉底,换用新的FAA,固定24h;经梯度酒精脱水后于Technovit7100树脂预渗透液中静置2h,再于树脂渗透液中静置2h;后于包埋板中聚合,60℃恒温箱中烘烤4d左右。Leica RM2265半薄切片机上横切花药,切片厚度为2μm,甲苯胺蓝染色后用Leica DM2000光学显微镜观察拍照。

表1 精细定位和测序引物Table 1.Makers used for fine mapping and sequencing.
1.5 DNA的提取和基因定位
用简易DNA提取法[26]分单株提取组合gamyb5/02428的F2中突变型单株DNA。利用本实验室保存的515对SSR标记[27]和167对InDel标记[28]检测中恢8015和02428之间的多态性,选取平均分布于水稻12条染色体上133对多态引物对8株突变型单株进行染色体连锁分析及初步定位。
根据Gramene(http://www.gramene.org)中公布的粳稻日本晴和籼稻93-11的全基因组序列,用DNASTAR软件对比初定位区间内的基因组序列,寻找插入/缺失(InDel)位点,利用Primer Premiers 5.0软件设计新的InDel分子标记,对突变基因进行精细定位(表1)。所用引物由杭州擎科梓熙生物技术有限公司合成。
PCR扩增采用北京鼎国昌盛生物技术有限责任公司的10μL反应体系:模板DNA 10μL,10× PCR缓冲液1μL,Primer(20μmol/L)1μL,dNTP 0.2μL,Taq酶0.2μL,ddH2O 6.7μL;PCR程序如下:94℃下预变性4min;94℃下变性30s,58℃下退火30s,72℃下延伸45s,共30个循环;最后72℃下延伸5min。PCR扩增产物经8.0%非变性聚丙烯酰胺凝胶电泳,0.1%AgNO3染色、甲醛和NaOH显色。
1.6 候选基因分析
从水稻基因组注释计划数据库(http://rapdb. dna.affrc.go.jp)中查阅位于精细定位区间内的所有开放阅读框(ORF),并分析可能的候选基因。根据候选基因的全长基因组DNA序列,设计测序引物(由上英潍捷基贸易有限公司合成),分别对突变体gamyb5和野生型中恢8015的候选基因进行PCR扩增并测序,PCR扩增采用高保真DNA聚合酶KOD-Plus-Neo,反应体系及反应条件参照说明书。PCR产物送往杭州擎科梓熙生物技术有限公司测序,测序结果由Contig Express软件拼接,并使用DNASTAR中的MegAlign进行序列比对,确定突变位点。
1.7 实时荧光定量PCR分析
用TOYOBO公司包含去基因组酶的逆转录试剂盒(First Strand cDNA Synthesis Kit),提取10 cm长花序时的花药总RNA。以Oligo(dT)18为反转引物进行第1链cDNA合成,反应体系及反应条件参照说明书。利用实时荧光定量PCR分析调控花药育性的关键基因MSP1、TDR、UDT1、CYP703A3、CYP704B2以及突变基因GAMYB在野生型和突变体中的表达量。内参基因为RUBQ (GenBank登录号为NM_001052515)。实时荧光定量PCR体系(20μL)包括蒸馏水6.4μL、2× SYBR qPCR Mix 10μL、上下游引物(10μmol/L)各0.8μL、cDNA模板2μL,每个样品3个重复。PCR扩增程序为95℃下30s;95℃下5s,58℃下30s,72℃下15s,40个循环。
2 结果与分析
2.1 突变体表型分析
突变体gamyb5及其野生型中恢8015在株高、分蘖等株型性状和穗部表型上无明显差异(图1-A~C),但是野生型的花药饱满呈黄色,而突变体的花药瘦弱呈白色(图1-D)。花粉镜检结果显示,野生型中恢8015的花粉粒全部被1%I2-KI染成深色,育性正常,而突变体gamyb5花药经染色压片,并无花粉粒释放出来,完全败育。

A-中恢8015和突变体gamyb5的植株;B-中恢8015和突变体gamyb5的穗部表型;C-中恢8015和突变体gamyb5的颖花形态;D-中恢8015和突变体gamyb5的花药表型。E-中恢8015和突变体gamyb5的花粉育性。A,Plant of Zhonghui 8015and the gamyb5 mutant at the heading stage;B,Panicle of Zhonghui 8015and the gamyb5 mutant;C,Spikelet of the wild type Zhonghui 8015and gamyb5;D,Anther of Zhonghui 8015and gamyb5 mutant;E,Pollen fertility of Zhonghui 8015and gamyb5.图1 野生型中恢8015(WT)与突变体gamyb5抽穗期的表型比较Fig.1.Phenotypic comparison of wild type Zhonghui 8015(WT)and gamyb5.
2.2 突变体遗传分析
以gamyb5突变体为母本,广亲和粳稻品种02428和野生型中恢8015为父本,配制杂交组合,其F1植株花粉育性和小穗育性正常。分别统计BC1F2和F2群体中的野生型和突变型植株数,经卡方检验,均符合3∶1的分离比值(表2),同时还统计了野生型中恢8015和02428杂交F1结实率,在其分离群体中并没有发现无花粉型雄性不育植株,这表明gamyb5突变性状受核隐性单基因控制。
2.3 目的基因的初定位和精细定位

表2 突变体gamyb5的遗传分析Table 2.Genetic analysis of the gamyb5 mutant.
为定位gamyb5雄性不育基因,选取平均分布于12条染色体上的133对多态引物对8株突变型单株进行染色体连锁分析及初步定位。连锁分析发现第1染色体上RD0114和InDel15两个SSR标记与gamyb5连锁。参考Gramene网站(http://www.gramene.org/)上公布的日本晴和93-11全基因组序列,进一步在RD0114和InDel15之间设计InDel标记(表2)。最终将目的基因定位在ZF-29 和ZF-31标记之间,物理距离约16.9kb(图2)。

A-突变基因的初步定位;B-突变基因的精细定位;C-精细基因定位区间中的预测基因;D-候选基因LOC_Os01g0812000的结构及其在突变体中的突变位点(8个碱基缺失)。A,Preliminary mapping of gamyb5;B,Fine mapping of gamyb5;C,Prediction of the candidate gene within the fine-mapped region;D,The structure of candidate gene LOC_Os01g0812000and the mutation site in gamyb5 mutant(8bases deleted).图2 突变基因的图位克隆Fig.2.Positional cloning of the gamyb5 mutated gene.
2.4 候选基因的克隆及序列分析
根据水稻基因组注释数据库(Rice Genome Annotation Project)提供的水稻基因组注释序列,在精细定位的16.9kb的区间内,有两个注释基因(图2-C)。其中,LOC_Os01g0812025是非编码蛋白转录本,LOC_Os01g0812000编码受赤霉素诱导的MYB转录因子,在糊粉层、花序顶部区域、雄蕊原基和花药绒毡层细胞高水平表达,但在营养生长器官中以及伸长的茎中低表达[29]。水稻OsGAMYB由3个外显子和2个内含子组成(图2-D),基因全长4.287kb,编码区长度为1.662kb,编码的MYB转录因子由553个氨基酸组成。将中恢8015和突变体gamyb5中的OsGAMYB基因进行PCR扩增和测序。测序结果显示gamyb5突变体中的OsGAMYB从ATG开始的第446个碱基起,存在8个碱基缺失(图2-D),最终导致翻译提前终止。基于此,gamyb5的候选基因很可能就是OsGAMYB。
2.5 花药半薄切片分析
通过花药半薄横切来观察野生型和突变型在花药发育过程中的差异。根据张大兵等[4]将花药发育划分为14个阶段,在前7个阶段,突变体gamyb5和野生型的花药细胞形态并没有显著差异(图3-A,G),此时小孢子母细胞开始减数分裂。在第8阶段,野生型小孢子母细胞经过连续的两次分裂,分别形成了二分体(图3-B)和四分体。在花药发育的第9阶段,四分体的胼胝质壁降解,小孢子被释放在花粉囊腔中,同时中间层细胞被压缩成带状,并降解(图3-C),此时绒毡层细胞的DNA开始出现片段化,开始其PCD过程,但是在细胞学水平上还看不出明显的降解痕迹。在第10阶段,小孢子液泡化并呈圆形,此时绒毡层细胞呈带状,PCD程度很高,各种细胞器开始降解(图3-D)。在第11阶段,液泡化的小孢子经历一次不对称的有丝分裂,形成一个生殖细胞和一个营养细胞(图3-E)。此后,在第12阶段,生殖细胞再经历一次有丝分裂,形成两个精核,至此由两个精核和一个营养核组成的成熟花粉粒发育成熟。然而,在突变体gamyb5中,小孢子母细胞经历了异常的减数分裂和细胞分裂,没有形成有功能的二价体(图3-H)。此外,在gamyb5中,从第9阶段开始,绒毡层开始不正常伸长,并且中间层并未降解(图3-I),在第12阶段,野生型中已形成成熟的花粉粒,而在gamyb5中,绒毡层仍在异常伸长,几乎占据了整个花粉囊腔,但是没有形成花粉粒(图3-L)。
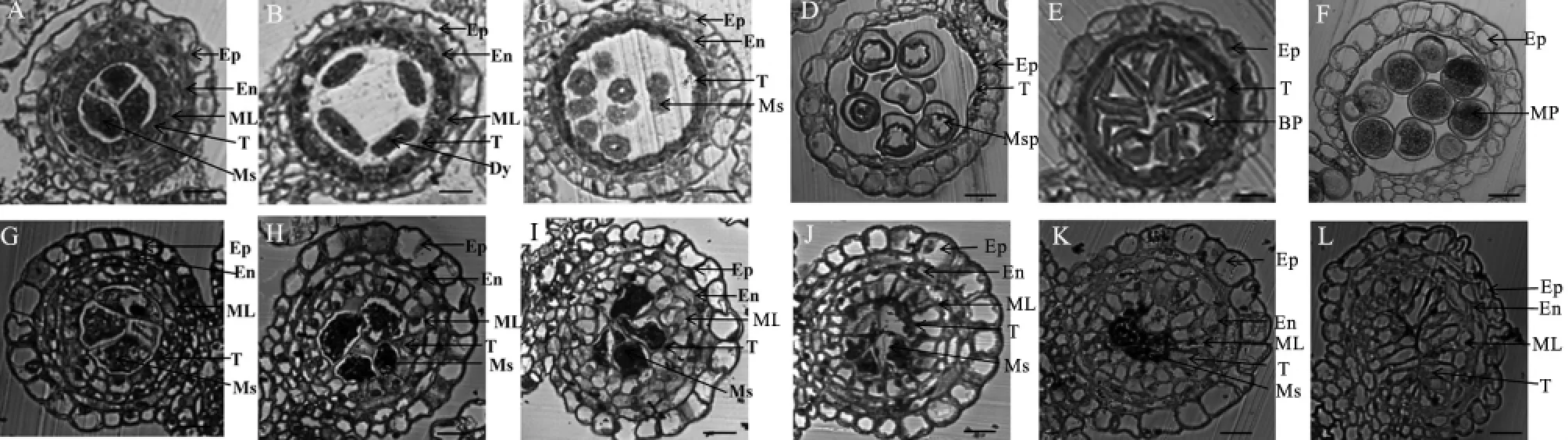
A到F为野生型,G到L为突变体gamyb5。A和G为花药发育第7阶段时的横切;B和H为第8a阶段时花药横切;C和I为第9阶段时花药横切;D和J为第10阶段时花药横切;E和K为第11阶段时花药横切;F和L为第12阶段时花药横切。Ep-表皮层;En-内皮层;ML-中层;T-绒毡层;Ms-小孢子母细胞;Dy-二分体;Msp-小孢子;BP-二孢花粉;MP-成熟花粉。标尺为20μm。A to E,Wild type;G to L,The gamyb5 mutant.A and G,Cross-section of anthers at the stage 7;B and H,Cross-section of anthers at the stage 8;C and I,Cross-section of anthers at the stage 9;D and J,Cross-section of anthers at the stage 10;E and K,Cross-section of anthers at the stage 11;F and L,Cross-section of anthers at the stage 12.Ep,Epidermis;En,Endothecium;ML,Middle layer;T,Tapetum;Ms,Microsporocyte;Dy,Dyad cell;Msp,Microspore;BP,Biceullar pollen;MP,Mature pollen.Bars=20μm.图3 gamyb5突变体和中恢8015野生型不同发育时期花药半薄横切观察Fig.3.Transverse section analyses of the wild type and gamyb5 anthers in various of developmental stages.
2.6 花药发育相关基因的表达分析
利用实时定量PCR比较了gamyb5与野生型中隐性核雄性不育相关基因的表达量。这类基因包括编码LRR类受体激酶的MSP1基因,编码bHLH转录因子的TDR、UDT1基因,细胞色素P450家族基因CYP703A3、CYP704B2,以及本研究中的目的基因GAMYB。如图4所示,与野生型相比,除MSP1基因外,突变体gamyb5的TDR、UDT1、CYP703A3、CYP704B2以及GAMYB基因的表达量均显著降低,说明GAMYB基因突变,导致这些基因表达量下调,即正向调控这些基因。
3 讨论
本研究中的gamyb5突变体表现出花药瘦弱呈白色透明,经I2-KI染色压片后,药室中并未观察到花粉粒,完全败育。花药半薄切片显示,突变体gamyb5减数分裂异常,没有形成有功能的四分体和小孢子。从花药发育的第9阶段起,绒毡层细胞异常伸长,中层并未降解,绒毡层也没有进入PCD过程,绒毡层占据了整个花药腔室最终导致小孢子发育夭亡,形成无花粉型雄性不育。
遗传分析和基因定位表明,gamyb5不育基因是一个单隐性核基因,位于第1染色体长臂的ZF-29和ZF-31两个标记之间,物理距离约16.9kb。经过测序分析发现,该区域中编码受赤霉素诱导的MYB转录因子基因LOC_Os01g0812000的第2外显子有8个碱基缺失,最终导致翻译提前终止,由此导致该基因功能失活。该突变体与之前报道的gamyb-1、gamyb-2、gamyb-3[29]和gamyb-4[17]等位,前3个突变体都是TOS17转座子插入到日本晴中形成的突变体,gamyb-4由辐射诱变所得。gamyb-1、gamyb-3转座子插入到了OsGAMYB的第2外显子中,gamyb-2转座子插入到了第3外显子中;gamyb-4在第2外显子中由单碱基C缺失引起编码框改变,转录提前终止。而gamyb5的突变位点在第2外显子近MYB结构域端,有8个碱基缺失,最终导致翻译提前终止。并且它们的遗传背景也不相同,前四个等位突变体都是来自粳稻品种,而gamyb5是由籼稻品种中恢8015经60Co-γ射线辐射诱变得到。在表型上,gamyb-1、gamyb-2和gamyb-3表型比较强,既有花药皱缩不育,也有内外稃白化畸形,甚至雌雄蕊都不育的强突变体[29];gamyb-4的花药白色,瘦小,没有花粉粒,完全不育[17]。而gamyb5的花药瘦弱,呈白色透明,花药长度并没有发生变化,花粉彻底败育。虽然突变体gamyb5与gamyb-1、gamyb-2、gamyb-3和gamyb-4相比,表型变化较小,但败育彻底。这种差异很可能是由于TOS17转座子插入会引起比较大的表型变化;其次gamyb5的突变位点更接近MYB结构域,影响其空间结构,致使突变更加彻底,但由于是在籼稻遗传背景下,花药表型突变较小。

**在P=0.01水平上差异显著。**significant at P=0.01.图4 实时荧光定量PCR分析野生型与突变体中水稻隐性核雄性不育相关基因的表达Fig.4.Expression analysis of male sterility-associated genes of the wild type and the mutant.
GAMYB含有高度保守的MYB结构域和3个保守域[30]。最早在大麦中报道GAMYB受赤霉素诱导,在糊粉层中正向调控α-淀粉酶的表达,在花药发育、雄蕊生长、花原基诱导和种子萌发等植物生长发育中发挥重要作用[31-33]。Aya等报道GAMYB在绒毡层PCD、花粉外壁以及乌氏体形成中起重要作用[34]。UDT1在花药发育的第6阶段到第10阶段在绒毡层中高表达[16],TDR主要在花药发育的减数分裂和小孢子形成时期在绒毡层中表达[15]。我们通过RT-qPCR分析,在gamyb5中,除了MSP1表达量没有显著性差异外,TDR、UTD1、CYP703A3、CYP704B2以及GAMYB的表达量均极显著降低。已有报道MSP1调控GAMYB和UDT1的表达[35],GAMYB可能与UDT1独立表达,调控花药发育,并且共同正调控TDR的表达[17],而在gamyb5中,UDT1的表达量显著降低,也许GAMYB对UDT1的表达存在某种调节。GAMYB和TDR可以直接调控CYP703A3的表达,CYP703A3突变并不影响绒毡层的细胞程序性死亡,但对花粉壁和角质的形成起关键作用。它催化C12∶0脂肪酸羟基化,形成7-OH-C12∶0脂肪酸[23,34,36]。该物质作为脂肪酸合成的中间产物,最终通过脂肪酸合成途径延长碳链并在CYP704B2的作用下发生脂肪酸ω-羟基化,参与花粉外壁和花药角质的形成[22,37-41]。本研究中,控制gamyb5突变性状基因是1个隐性核基因,是已报道的OsGAMYB的等位基因。GAMYB表达量降低,引起下游调控育性基因TDR、CYP703A3、CYP704B2表达量下调,导致绒毡层异常伸长,花粉壁形成受阻,最终导致突变体gamyb5突变表型的出现。进一步证明了GAMYB在水稻花药小孢子发育和花粉壁形成中起关键作用。
谢辞:感谢上海交通大学植物发育生物学研究室张大兵教授、梁婉琪研究员以及汪洁老师对本实验的大力帮助。
参考文献:
[1] McCormick S.Male gametophyte development.Plant Cell,1993,5:1265-1275.
[2] Scott R J,Spielman M,Dickinson H G.Stamen structure and function.Plant Cell,2004,16(Suppl):46-60.
[3] Ma H.Molecular genetic analyses of microsporogenesis and microgametogenesis in flowering plants.Annu Rev Plant Biol,2005,56:393-434.
[4] Zhang D B,Wilson Z A.Stamen specification and anther development in rice.Chin Sci Bull,2009,54:2342-2353.
[5] Zhang D B,Luo X,Zhu L,et al.Cytological analysis and genetic control of rice anther development.J Genet Genom,2011,38:379-390.
[6] Zhang D B,Yang L.Specification of tapetum and microsporocyte cells within the anther.Curr Opin Plant Biol,2014,17C:49-55.
[7] 马西青,方才臣,邓联武,等.水稻隐性核雄性不育基因研究进展及育种应用探讨.中国水稻科学,2012,26(5):511-520.Ma X Q,Fang C C,Deng L W,et al.Research progress and breeding application of recessive genic male sterility genes in rice.Chin J Rice Sci,2012,26(5):511-520.(in Chinese with English abstract)
[8] Nonomura K,Miyoshi K.The MSP1 gene is necessary to restrict the number of cells entering into male and female sporogenesis and to initiate anther wall formation in rice.Plant Cell,2003,15(8):1728-1739.
[9] Ken-Ichi N,Mutsuko N,Toshiyuki F,et al.The novel gene HOMOLOGOUS PAIRING ABERRATION IN RICE MEIOSIS1of rice encodes a putative coiled-coil protein required for homologous chromosome pairing in meiosis.Plant J,2004,16(4):1008-1020.
[10]Nonomura K I,Nakano M,Murata K,et al.An insertional mutation in the rice PAIR2gene,the ortholog of Arabidopsis ASY1,results in a defect in homologous chromosome pairing during meiosis.Mol Genet Genom,2004,271(2):121-129.
[11]Yuan W Y,Li X W,Chang Y X,et al.Mutation of the rice gene PAIR3results in lack of bivalent formation in meiosis.Plant J,2009,59(2):303-315.
[12]Sandra N Ol,Elizabeth S D,Rudy D.ABA regulates apoplastic sugar transport and is a potential signal for cold-induced pollen sterility in rice.Plant Cell Physiol,2007,48(9):1319-1330.
[13]Ken-Ichi N,Akane M,Mutsuko N,et al.A germ cell-specific gene of the ARGONAUTE family is essential for the progression of premeiotic mitosis and meiosis during sporogenesis in rice.Plant Cell,2007,19(8):2583-2594.
[14]Wang C,Huang W,Ying Y H,et al.Functional characterization of the rice SPX-MFS family reveals a key role of OsSPXMFS1in controlling phosphate homeostasis in leaves.New Phytol,2012,196(1):139-148.
[15]Li L,Li Y X,Song S F.An anther development F-box(ADF)protein regulated by tapetum degeneration retardation(TDR)controls rice anther development.Planta,2015,241(1):157-166.
[16]Jung K H,Han M J,Lee Y S.Rice undeveloped tapetum1is a major regulator of early tapetum development.Plant Cell,2005,17(10):2705-2722.
[17]Liu Z H,Bao W J.Identification of gamyb-4and analysis of the regulatory role of GAMYB in rice anther development.J Integ Plant Biol,2010,52(7):670-678.
[18]Li H,Yuan Z.PERSISTENT TAPETAL CELL1Encodes a PHD-finger protein that is required for tapetal cell death and pollen development in rice.Plant Physiol,2011,156(2):615-630.
[19]Li X W,Gao X Q.Rice APOPTOSIS INHIBITOR5coupled with two DEAD-box adenosine 5′-triphosphate-dependent RNA helicases regulates tapetum degeneration.Plant Cell,2011,23(4):1416-1434.
[20]Jung K H,Han M J.Wax-deficient anther1is involved in cuticle and wax production in rice anther walls and is required for pollen development.Plant Cell,2006,18(11):3015-3032.
[21]Shi J,Tan H X.Defective pollen wall is required for anther and microspore development in rice and encodes a fatty acyl carrier protein reductase.Plant Cell,2011,23(6):2225-2246.
[22]Li H,Pinot F.Cytochrome P450family member CYP704B2 catalyzes theω-Hydroxylation of fatty acids and is required for anther cutin biosynthesis and pollen exine formation in rice.Plant Cell,2010,22(1):173-190.
[23]Yang X J,Wu D.rice CYP703A3,a cytochrome P450 hydroxylase,is essential for development of anther cuticle and pollen exine.J Integ Plant Biol,2014,56(10):979-994.
[24]Zhu Q H,Ramm K,Shivakkumar R,et al.The ANTHER INDEHISCENCE1 gene encoding a single MYB domain protein is involved in anther development in rice.Plant Physiol,2004,135(3):1514-1525.
[25]初明光,李双成,王世全,等.一个水稻雄性不育突变体的遗传分析和基因定位.作物学报,2009,35(6):1151-1155.Chu G M,Li S C,Wang S Q,et al.Genetic analysis and molecular mapping of a male sterile mutant in rice.CropJ,2004,135(3):1514-1525.(in Chinese with English abstract)
[26]卢扬江,郑康乐.提取水稻DNA的一种简易方法.中国水稻科学,1992,6(1):47-48.Lu Y J,Zheng K L.A simple method for isolation of rice DNA.Chin J Rice Sci,1992,6(1):47-48.(in Chinese with English abstract)
[27]Orjuela J,Garavito A,Bouniol M.A universal core genetic map for rice.Theor Appl Genet,2010,120,563-572.
[28]Rychlik W.Oligo primer analysis software version 7.0.2nd ed.Molecular Biology Insights,Inc,Cascade,Co,2008.
[29]Aya K,Miyako Ueguchi-Tanaka M.Gibberellin modulates anther development in rice via the transcriptional regulation of GAMYB.Plant Cell,2009,21(5):1453-1472.
[30]Gocal,Sheldon G F.GAMYB-like genes,flowering and gibberellin signaling in Arabidopsis.Plant Physiol,2001,127,1682-1693.
[31]Gubler F,Kalla R,Roberts J K,et al.Gibberellin-regulated expression of a MYB gene in barley aleurone cells:Evidence for Myb transactivation of a high-pIα-amylase gene promoter.Plant Cell,1995,7:1879-1891.
[32]Fiona J,Woodger A M,Murray F,et al.The role of GAMYB transcription factors in GA-regulated gene expression.J Plant Growth Regul,2003,22,176-184.
[33]Kaneko M,Inukai Y,Ueguchi-Tanaka M.loss-of-function mutations of the rice GAMYB gene impairα-amylase expression in aleurone and flower development.Plant Cell,2004,16:1473-1487.
[34]Aya K,Ueguchi-Tanaka M,Kondo M,et al.Gibberellin modulates anther development in rice via the transcriptional regulation of GAMYB.Plant Cell,2009,21:1453-1472.
[35]Wang Y,Wang Y F,Zhang D B.Identification of the rice (Oryza sativa L.)mutant msp1-4 and expression analysis of its UDT1 and GAMYB genes.J Plant Physiol Mol Biol,2006,32:527-534.
[36]Morant M,Jorgensen K,Schaller H.CYP703is an ancient cytochrome P450in land plants catalyzing in-chain hydroxylation of lauric acid to provide building blocks for sporopollenin synthesis in pollen.Plant Cell,2007,19:1473-1487.
[37]Dobritsa A A,Shrestha J,Morant M.CYP704B1is a longchain fatty acidω-hydroxylase essential for sporopollenin synthesis in pollen of Arabidopsis.Plant Physiol,2009,151:574-589.
[38]Bouquin L,Pinot R,Benveniste F,et al.Cloning and functional characterization of CYP94A2,a medium chain fatty acid hydroxylase from Vicia sativa.Biochem.Biophys.Res Commun,1999,261:156-162.
[39]Kurdyukov S,Faust A,Trenkamp S.Genetic and biochemical evidence for involvement of HOTHEAD in the biosynthesis of long-chain alpha-omega-dicarboxylic fatty acids and formation of extracellular matrix.Planta,2006,224:315-329.
[40]Kandel S,Sauveplane V,Compagnon V.Characterization of a methyl jasmonate and wounding-responsive cytochrome P450 of Arabidopsis thaliana catalyzing dicarboxylic fatty acid formation in vitro.FEBS J,2007,274:5116-5127.
[41]Sauveplane V,Kandel S,Kastner P E.Arabidopsis thaliana CYP77A4is the first cytochrome P450able to catalyze the epoxidation of free fatty acids in plants.FEBS J,2009,276:719-735.
Identification and Gene Mapping of Male Sterile Mutant gamyb 5in Rice
YANGZheng-fu1,#,ZHANGYing-xin1,#,SUNLian-ping1,2,ZHANGPei-pei1,XUANDan-dan1,LIULing1,3,HU Xia1,LI Zi-he1,ZHANXiao-deng1,WU Wei-xun1,CAOLi-yong1,*,CHENGShi-hua1,*
(1National Center for Rice Improvement/Key Laboratory for Zhejiang Super Rice Research,China National Rice Research Institute,Hangzhou 311401,China;2 College of Agronomy,Shenyang Agricultural University,Shenyang 110866,China;3 College of Agronomy,He′nan Agricultural University,Zhengzhou 450002,China;#These authors contributed equally to this work;*Corresponding author,E-mail:caoliyong@caas.cn;shcheng@mail.hz.zj.cn)
YANG Zhengfu,ZHANG Yingxin,SUN Lianping,et al.Identification and gene mapping of male sterile mutant gamyb5 in rice.Chin J Rice Sci,2016,30(2):143-151.
Abstract:The gamyb5,apollen-free male sterile mutant was identified from the mutant library of(60)Co-γ-treated indica cultivar Zhonghui 8015.There was no significant difference in agronomic traits such as plant type,plant height and tiller number between gamyb5 and the wild-type.But gamyb5 exhibited slender and white anthers without mature pollen grains.Observation results of anther cross-sections exhibited that the microspore mother cells failed to form functional tetrads and microspores in gamyb5.Moreover,tapetal cells of gamyb5 abnormally enlarged and the tapetum programmed cell death(PCD)was delayed.gamyb5,as the pollen acceptor,was crossed with the wild type Zhonghui 8015and a japonica cultivar 02428,respectively.Genetic analysis of all hybridization populations indicated that gamyb5 was controlled by a single recessive nuclear gene which was mapped on the long arm of chromosome 1.With developed SSR,Indel markers and F2mapping population of gamyb5/02428,the gene was fine mapped to a region of 16-kb on the long arm of chromosome 1between markers ZF-29and ZF-31.Sequence analysis of the two open reading frames in this region revealed that the LOC_Os01g0812000,which encodes a gibberellin-induced MYB transcription factor,had an 8nucleotides bases deletion in the second extron probably responsible for the male sterility phenotype.Additionally,real-time fluorescent quantitative PCR analysis showed that the expression level of the important regulators UDT1,TDR,CYP703A3 and CYP704B2 in anther decreased significantly in gamyb5.Together,these results suggest GAMYBplays key roles in anther meiosis and tapetum PCD.
Key words:rice;male sterility;gene mapping;gamyb5
中图分类号:Q343.5;S511.032
文献标识码:A
文章编号:1001-7216(2016)02-0143-09
基金项目:农业部农业公益性行业科技专项(20140302,20150308);国家转基因重大专项(2014ZX08001-002);浙江省自然科学基金资助项目(Q13C130009);中国农业科学院创新工程超级稻育种创新团队、水稻杂种优势机理研究创新团队项目(CAAS-ASTIP-2013-CNRRI);国家自然科学基金资助项目(31501290)。
收稿日期:2015-10-19;修改稿收到日期:2015-12-11。
