一个水稻白化致死突变基因的精细定位和遗传研究
2016-05-09初志战刘小林陈远玲刘耀光
初志战刘小林陈远玲刘耀光,*
(1华南农业大学生命科学学院/亚热带农业生物资源保护与利用重点实验室,广州510642;2江西宜春学院,江西宜春336000;*通讯联系人,E-mail:ygliu@scau.edu.cn)
一个水稻白化致死突变基因的精细定位和遗传研究
初志战1刘小林2陈远玲1刘耀光1,*
(1华南农业大学生命科学学院/亚热带农业生物资源保护与利用重点实验室,广州510642;2江西宜春学院,江西宜春336000;
*通讯联系人,E-mail:ygliu@scau.edu.cn)
初志战,刘小林,陈远玲,等.一个水稻白化致死突变基因的精细定位和遗传研究.中国水稻科学,2016,30(2):136-142.
摘 要:从粳稻品种日本晴经(60)Co诱变的M1代材料中发现了一个白化致死突变体,该突变体从萌芽后一直表现白化,3叶期后逐渐衰亡。遗传分析表明,该突变表型受一对隐性核基因控制,将该白化突变体暂定名为al14。与野生型相比,al14突变体的叶绿素含量与类胡萝卜素含量显著降低。电子显微镜观察表明al14突变体不能形成完整的叶绿体,只有原片层体结构。对叶绿体编码基因的表达分析发现,突变体中光系统Ⅰ和光系统Ⅱ基因表达明显下调,核糖体结构基因和质体编码的RNA聚合酶亚基基因表达明显上调,但是PsbN(photosystemⅡprotein N)却上调表达水平最高,达到118.23倍。利用al14突变体与黄华占杂交获得的F2代分离群体进行基因定位,将该基因定位于水稻第6染色体上约40kb的范围。目前,该范围内没有叶色相关基因的报道,可能为一新的调控叶绿体发育的基因。
关键词:水稻;白化致死;基因定位
水稻是世界上三大粮食作物之一,也是研究植物功能基因的模式作物。水稻的物质产量大部分来源于叶片的光合作用,因此叶片的颜色、大小和形状直接影响水稻的产量和品质。水稻的叶色突变较为常见,大多数的叶色突变都会导致叶绿体中叶绿素a,叶绿素b及类胡萝卜素等色素含量改变,引起植株光合作用效率下降,进而导致植株生长缓慢,株型弱小,结实率下降,甚至植株死亡。叶绿体是半自主型细胞器,其生物发生及发育是受核基因组和叶绿体基因组共同调控,所以核基因或叶绿体基因的突变都有可能会影响到叶绿体的正常发育,因此,研究叶色调控不仅可以深入研究叶绿体的形成发育过程,也可以从分子水平对水稻株型改良,尤其是叶色的改良及水稻的高产提供选择。
迄今为止,已报道的水稻叶色突变体超过180个,大多数定位到染色体的特定区间(超过140个),已成功克隆的叶色相关基因有37个。从叶色突变基因的分布来看,突变基因在12条染色体上均有发现,第3染色体上最多。在已报道的水稻叶色突变体中,大多数为单基因控制的隐性突变体;黄化突变基因,返绿型突变基因和白化突变基因较多,其中白化突变基因目前已报道的超过28个。引起叶色变异的机理较多,其中参与叶绿素、类胡萝卜素等色素合成过程中的酶发生突变,导致叶绿素合成缺陷,是目前发现的最多的一类。OsCAO1(水稻叶绿素a加氧酶)是催化叶绿素a合成叶绿素b的关键酶,该酶失活会导致水稻叶片黄化、植株矮化[1]。YGL1[2]编码叶绿素合酶,催化叶绿素酸酯a形成叶绿素a,突变体表型为黄绿。OsDVR(Oryza sativa divinyl reductase,水稻二乙烯还原酶),催化二乙烯叶绿素(酸酯)a转化为单乙烯叶绿素(酸酯)a,突变后水稻叶色黄绿,植株矮小[3]。OsPDS(水稻八氢番茄红素脱氢酶)、OsZDS(水稻ζ-胡萝卜素脱氢酶)、OsCRTISO(水稻类胡萝卜素异构酶)、β-OsLCY(水稻番茄红素β-羟化酶)这4个酶都参与类胡萝卜素前体-ABA的合成,任何一个发生突变均可导致突变体中ABA含量下降,引起穗萌和白化[4]。叶绿体自身结构发育异常也常常导致叶色的突变,Kusumi等[5]在转绿型白叶突变体v1中发现,核基因组编码的叶绿体RNA聚合酶蛋白OsRpoTp表达量急剧减少,导致叶绿体发育不良,是造成早期叶色白化的主要原因。Sumimoto等[6]发现叶绿体不良的转绿型白叶突变体v2也属于此类型。Gothandam等[7]发现叶绿体发育相关的基因OsPPR1表达水平降低会导致水稻叶片白化。Zhao等[8]发现OsCHR4参与了水稻近轴叶肉细胞叶绿体早期分化,其功能的丧失阻碍近轴叶肉细胞叶绿体的发育,所以OsCHR4突变体表现叶片近轴部白化。叶绿素分解代谢障碍也可以导致叶色突变,大多突变表型为叶色常绿。Jiang等[9-11]发现基因Sgr可能参与调控PaO脱镁叶绿酸a加氧酶活性,Sgr可与LHCPⅡ(捕光叶绿素a/b结合蛋白Ⅱ)结合,在类囊体膜上形成Sgr-LHCPⅡ复合物,诱导LHCPⅡ降解,促使叶绿素的降解,其突变体表现出叶片滞绿。NYC1(Non-yellow coloring)[12]编码叶绿素b还原酶,该酶是叶绿素降解过程中的第1个酶,突变可引起叶绿体基粒和捕光系统Ⅱ退化减慢,叶片常绿。
本实验室利用60Co诱变日本晴,M1代获得一个水稻白化致死突变体,将该白化突变体暂命名为al14。遗传分析表明,该性状是由单个隐性基因控制的。本研究对该突变体进行生理生化分析和显微观察,并对该突变基因进行了精细定位,以期为该基因的克隆及对叶绿体发育过程的功能研究提供参考。
1 材料与方法
1.1 突变体材料
白化致死突变体来源于水稻日本晴(粳稻品种)的60Co辐射诱变M1代。由于突变体苗期白化致死,因此M1代表型正常植株单株挂牌,单株收种保存。播种M2代种子,可以分离出白化苗的株系,单株收种,田间材料按常规管理。
1.2 叶绿素含量测定
分别取3叶期前期突变体和野生型材料的叶片,测定其叶绿素a、叶绿素b及类胡萝卜素含量,重复3次,取平均值。测定方法参照文献[13],并略加修改。
1.3 叶绿体显微结构观察
取播种12d的野生型及突变体叶片,用4%戊二醛(用pH7.2的磷酸缓冲溶液配置)4℃固定过夜,磷酸缓冲溶液冲洗3次,1%锇酸固定1h,磷酸缓冲溶液冲洗3次,用30%、50%、70%、80%、95%、100%的乙醇和丙酮逐级脱水5min,最后用树脂包埋样品,切片后用醋酸铀染色,再用透射电镜进行观察。
1.4 定位群体的构建
选取苗期能够分离出白化突变体的M2株系(上一代为杂合单株),随机选取6株生长正常的单株,分别与籼稻品种黄华占杂交,获得F1种子。播种F1后,自交繁殖获得F2种子,单株收种。分株系种植F2代,以能分离出白化苗的F2群体为定位群体。
1.5 基因定位
基因初步定位:采用SDS法[14]提取20株白化苗叶片总DNA用于基因定位。基因精细定位:根据初步定位结果,采用快速打叶法[15]对定位群体植株进行两侧标记检测,并利用内部InDel引物进一步缩小定位区间。

A-秧苗;B-叶片。A-Seedling;B-Leaf.图1 突变体al14及其野生型(WT)的表型Fig.1.Phenotypes of al14 and its wild type(WT).
本研究用于基因定位的分子标记为InDel标记:一部分为本实验室已有的,另一部分为根据已公布的水稻品种93-11和日本晴全基因组序列自行开发。这些标记均匀分布于水稻12条染色体,共147对。
1.6 叶绿体基因表达分析
取2叶期的叶片提取RNA,反转录成cDNA,利用β-Actin(Os03g0718100)(F:CACATTCCAGC AGATGTGGA;R:ACCACAGGTAGCAATAGG TA)作为内参基因,64对引物检测突变体中叶绿体基因组基因的表达量。根据BioRad公司的《荧光定量PCR应用指南》所述,进行数据处理,通过计算2-△△Ct值来确定每个基因在野生型和突变体中的相对表达量。
2 结果与分析
2.1 突变体表型鉴定
正常的自然条件下,al14种子萌发后,子叶为白色,从真叶长出至3叶期,叶片完全为白色,3叶期后,逐渐死亡(图1)。

表1 突变体al14及其野生型中色素含量Table 1.Photosynthetic pigments contents in al14 and its wild type.

1-叶绿体;2-类囊体;3-原片层体。1,Chloroplast;2,Thylakoid;3,Prolamellar body.图2 野生型(A,C)和al14突变体(B,D)的超微结构Fig.2.Transmission electron microscopy observation of wild type(A,C)and al14 mutant(B,D).
2.2 突变体的叶绿素含量变化
通过测定突变体al14及其野生型叶片的叶绿素、类胡萝卜素的含量,发现al14的叶绿素a、叶绿素b和类胡萝卜含量明显下降,与野生型相比均差异显著,表明al14的叶绿素合成受到明显抑制(表1)。
2.3 叶绿体的显微结构
利用透射电镜观察al14及其野生型的叶绿体结构,野生型细胞中叶绿体含量丰富,基粒结构清晰,而al14中没有明显的叶绿体结构,没有类囊体结构,仅有原片层体结构(图2)。
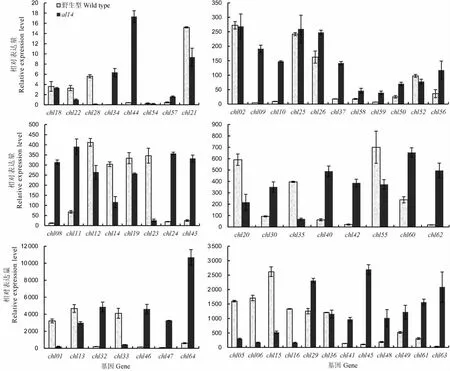
图3 部分叶绿体编码基因在野生型和al14突变体的表达比较Fig.3.Expression analysis of some chloroplast-encoded genes in wild type and al14 mutant.
2.4 叶绿体编码基因的定量表达分析
通过定量结果可以看出,该叶色突变对叶绿体编码基因的表达影响是全面的,共有29个基因表达明显上调(上调倍数>2),上调基因主要为核糖体结构基因(chl63,chl44,chl46,chl47,chl62,chl45,chl42)和RNA聚合酶亚基基因(chl09,chl08),但是上调表达幅度最大的基因为chl34,上调了118.23倍,明显高出其他上调基因;表达量明显降低的基因多为光系统Ⅰ和光系统Ⅱ基因(chl28,chl01,chl06,chl33,chl16,chl35,chl05)及Rubisco大亚基基因(图3)。
2.5 突变体的遗传分析及定位结果

选取均匀分布于水稻12条染色体上在两亲本间有多态性的InDel标记147个。用20株白化苗进行初定位,将白化基因初步定位在第6染色体短臂上的InDel标记606949与608312之间,物理距离约为1400kb的范围。
为了进一步缩小定位区间,选取了F2群体中2012株白化植株和5865株正常表型植株作为精细定位群体。根据公布的粳稻日本晴与籼稻9311序列,在InDel标记606949与607551之间,找到了8对在两个亲本有差异的新标记(表2),并最终将白化基因al14定位在InDel标记607511与607551之间,物理距离约40kb的范围(图4)。

图4 al14在水稻第6染色体的定位Fig.4.Location of al14 on rice chromosome 6.
2.6 候选基因分析
通过作物和模式植物基因组数据库Gramene (http://www.gramene.org/)预测该区域的基因组序列,得到5个ORF,其中有2个假定的表达蛋白(expressed protein),一个为假定的热重复家族蛋白,一个PE-PGRS家族蛋白和1个假定的转座子蛋白(表3)。根据突变体表型分析,突变基因很可能带有叶绿体定位信号。通过PSORT(http://psort.hgc.jp/form.html)预测亚细胞定位结果发现,这5个候选基因中只有基因4具有叶绿体定位信号,但通过测序排除了基因4的可能,因而需要对其余几个候选基因进行测序分析,以确定该白化基因。
3 讨论

表2 用于精细定位的新InDel标记Table 2.New InDel markers for mapping.
水稻叶色白化致死突变是一种比较极端,也比较常见的叶色突变,本研究的突变体中看不到清晰的叶绿体结构,更看不到类囊体垛叠成基粒的结构;叶绿素、类胡萝卜素含量明显下降,这与大部分的白化突变体研究结果相似[16-17]。水稻(Oryza sativa)叶绿体基因组测序工作在1989年已经完成[18],叶绿体编码蛋白预测超过100个[19]。通过对64个叶绿体基因组基因表达的定量分析发现,光系统Ⅰ和光系统Ⅱ基因表达下调明显,这可能与光系统Ⅰ和光系统Ⅱ位于类囊体膜有关。叶绿体基因组转录需要至少两种RNA聚合酶。核基因组编码的RNA聚合酶(NEP),主要转录叶绿体内的转录/翻译相关基因,如rpoA和rpoB,而质体基因组编码的RNA聚合酶(PEP),主要转录叶绿体内光合作用相关基因[20-21]。PEP由rpoA、rpoB、rpoC1和rpoC2编码的4个亚基组成,一旦这些亚基发生突变就会引起PEP的功能受损,从而产生白化表型[22-24]。在al14突变体中,核糖体结构基因和质体编码的RNA聚合酶亚基基因表达上调明显,目前没发现有这方面的报道,猜测可能是植物的应激反应。在上调基因中,基因chl34(PsbN,photosystemⅡprotein N)上调了118.23倍,上调幅度最高,该基因位于psbB操纵子的互补链上,psbT和psbH之间。实际上目前关于PsbN的编码框也一直存在争议,Kashino[25]和Gomez等[26]通过基因组学研究没有发现PsbN蛋白,所以Kashino认为PsbN应为PsbTc[25];Zghidi等[27]证实了拟南芥的PsbN反义RNA不仅仅局限于psbN和psbH之间的区间,甚至包含了psbT的整个编码区。我们通过定量分析发现PsbN在野生型材料中表达量很低,这可能是为什么Kashino等和Gomez等难以用基因组学检测到PsbN蛋白的原因。对于PsbN蛋白的功能研究,Mayes[28]发现在胞藻的psbN和psbH双突变和psbH的单突变表型没有明显差别,因此认为psbN对光合自养生物的生长不是必需的,但是Torabi S等[29]发现,烟草的psbN突变体对光很敏感,光抑制后不能修复,只能在低光强下生活。Zghidi等在sig3突变体中发现psbN的转录水平急剧下降(几乎检测不到),也表明psbN的表达受到sig3的调控,SIG3(sigma3)作为一个辅助因子与PEP (plastid encoded RNA polymerase)共同起作用。al14突变体材料中,PsbN表达量急剧上升,RNA聚合酶多个亚基的表达量也显著增加,PsbN基因表达量的上升与RNA聚合酶亚基表达量的提高是否有关?是否受到sig3的调控?这些都需要进一步的分析。
通过图位克隆将目标基因al14定位在第6染色体40kb的范围。目前,在水稻第6染色体上已报道了16个与叶色发育相关的基因,其中白化表型的有3个,分别为AL1,V3和GWS/ST1。在al14目标区域40kb范围内没有相关叶色基因报道,可能为一个新基因。

表3 定位区间基因功能注释Table 3.Gene annotated in mapping region.
谢辞:感谢华南农业大学张群宇老师提供叶绿体基因组定量分析引物。
参考文献:
[1] Lee S C,Kim J H,Yoo E S,et al.Differential regulation of chlorophyll a oxygenase genes in rice.Plant Mol Biol,2005,57(6):805-818.
[2] Wu Z M,Zhang X,He B,et al.A Chlorophyll-deficient rice mutant with impaired chlorophy-llide esterification in chlorophyll biosynthesis.Plant Physiol,2007,145(1):29-40.
[3] Wang P R,Gao J X,Wan C M,et al.Divinyl chlorophyll(ide)a can be converted to monovi-nyl chlorophyll(ide)a by a divinyl reductase in rice.Plant Physiol,2010,153(3):994-1003.
[4] Fang J,Chai C,Qian Q,et al.Mutations of genes in synthesis of the carotenoid precursors of ABA lead to preharvest sprouting and photo-oxidation in rice.Plant J,2008,54(2):177-189.
[5] Kusumi K,Yara A,Mitsui N,et al.Characterization of a rice nuclear-encoded plastid RNA polymerase gene OsRpoTp.Plant Cell Physiol,2004,45(9):1194-1201.
[6] Sugimoto H,Kusumi K,Tozawa Y,et al.The virescent-2 mutation inhibits translation of plastid transcripts for the plastid genetic system at an early stage of chloroplast differentiation.Plant Cell Physiol,2004,45(8):985-996.
[7] Gothandam K M,Kim E S,Cho H,et al.OsPPR1,apentatricopeptide repeat protein of rice is essential for the chloro-plast biogenesis.Plant Mol Biol,2005,58(3):421-433.
[8] Zhao C F,Xu J M,Chen Y,et al.Molecular cloning and characterization of OsCHR4,a rice chromatin-remodeling factor required for early chloroplast development in adaxial mesophyll.Planta,2012,236(4):1165-1176.
[9] Jiang H W,Li M R,Liang N T,et al.Molecular cloning and function analysis of the stay green gene in rice.Plant J,2007,52(2):197-209.
[10]Sakuraba Y,Park S Y,Paek N C.The divergent roles of STAYGREEN(SGR)homologs in chlorophyll degradation.Mol Cells,2015,38(5):390-395.
[11]Park S Y,Yu J W,Park J S,et al.The senescence-induced stay-green protein regulates chlorophyll degradation.Plant Cell,2007,19(5):1649-1664.
[12]Kusaba M,Ito H,Morita R,et al.Rice NON-YELLOW COLORING1is involved in light-harvesting complex II and grana degradation during leaf senescence.Plant Cell,2007,19 (4):1362-1375.
[13]Wellburn A R.The spectral determination of chlorophylls a and b,as well as total carotenoids,using various solvents with spectrophotometers of different resolution.Plant Physiol,1994,144(3):307-313.
[14]Guillemaut P,Maréchal-Drouard L.Isolation of plant DNA:A fast,inexpensive,and reliable method.Plant Mol Biol Rep,1992,10(1):60-65.
[15]王慧娜,初志战,马兴亮,等.高通量PCR模板植物基因组DNA制备方法.作物学报,2013,39(7):1200-1205.Wang H N,Chu Z Z,Ma X L,et al.A high through-put protocol of plant genomic DNA preparation for PCR.Act Agron Sin,2013,39(7):1200-1205.(in Chinese with English abstract)
[16]程世超,刘合芹,翟国伟,等.水稻白化致死突变体abl4的鉴定和基因定位.中国水稻科学,2013,27(3):240-246.Cheng S C,Liu H Q,Zhai G W,et al.Genetic analysis and gene mapping of an albino lethal mutant in rice.Chin J Rice Sci,2013,27(3):240-246.(in Chinese with English abstract)
[17]魏祥进,宋建,刘胜,等.水稻苗期低温白叶突变体ced2的鉴定和基因定位.中国水稻科学,2014,28(2):111-118.Wei X J,Song J,Liu S,et al.Identification and genetic mapping of a thermo-sensitive white leaf mutant in rice.Chin J Rice Sci,2014,28(2):111-118.(in Chinese with English abstract)
[18]Hiratsuka J,Shimada H,Whittier R,et al.The complete sequence of the rice(Oryza sativa)chloroplast genome:Intermolecular recombination between distinct tRNA genes accounts for a major plastid DNA inversion during the evolution of the cereals.Mol Gen Genet,1989,217(2):185-194.
[19]Cui L,Veeraraghavan N,Richter A,et al.Chloroplast DB:The chloroplast genome database.Nucleic Acids Res,2006,34(1):692-696.
[20]Hajdukiewicz P T J,Allison L A,Maliga P.The two RNA polymerases encoded by the nuclear and the plastid compartments transcribe distinct groups of genes in tobacco plastids.EMBO J,1997,16(13):4041-4048.
[21]Maliga P.Two plastid RNA polymerases of higher plants:An evolving story.Trends Plant Sci,1998,3(1):4-6.
[22]Serino G,Maliga P.RNA polymerase subunits encoded by the plastid rpo genes are not shared with the nucleus-encoded plastid enzyme.Plant Physiol,1998,117(4):1165-1170.
[23]Krause K,Maier R M,Kofer W,et al.Disruption of plastidencoded RNA polymerase genes in tobacco:Expression of only a distinct set of genes is not based on selective transcription of the plastid chromosome.Mol Gene Genet,2000,263(6):1022-1030.
[24]Legen J,Kemp S,Krause K,et al.Comparative analysis of plastid transcription profiles of entire plastid chromosomes from tobacco attributed to wild-type and PEP-deficient transcription machineries.Plant J,2002,31(7):171-188.
[25]Kashino Y,Lauber W M,Carroll J A,et al.Proteomic analysis of a highly active photosystem II preparation from the cyanobacterium Synechocystis sp.PCC 6803reveals the presence of novel polypeptides.Biochemistry,2002,41(25):8004-8012.
[26]Gomez S M,Nishio J N,Faull K F,et al.The chloroplast grana proteome defined by intact mass measurements from liquid chromatography mass spectrometry.Mol Cell Proteom,2002,1(1):46-59.
[27]Zghidi W,Merendino L,Cottet A,et al.Nucleus-encoded plastid sigma factor SIG3transcribes specifically the psbN gene in plastids.Nucl Acids Res,2007,35(2):455-464.
[28]Mayes S R,Dubbs J M,Vass I,et al.Further characterization of the psbH locus of Synechocystis sp.PCC 6803:inactivation of psbH impairs QA to QB electron transport in photosystem 2.Biochemistry,1993,32(6):1454-1465.
[29]Torabi S and Umate P.PsbN Is Required for Assembly of the Photosystem II Reaction.Plant Cell,2014,26(3):1183-1199.
Genetic Analysis and Gene Mapping of an Albino Lethal Mutant in Rice
CHUZhi-zhan1,LIUXiao-lin2,CHENYuan-ling1,LIUYao-guang1,*
(1College of Life Sciences,South China Agricultural University/State Key Laboratory for Conservation and Utilization of Subtropical Agro-bioresources,Guangzhou 510642,China;2 Yichun University,Yichun 336000,China;*Corresponding author,E-mail:ygliu@scau.edu.cn)
CHU Zhizhan,LIU Xiaolin,CHEN Yuanling,et al.Genetic analysis and gene mapping of an albino lethal mutant in rice.Chin J Rice Sci,2016,30(2):136-142.
Abstract:An albino lethal mutant,temporarily termed as al14(albino 14),was obtained from(60)Coγ-ray radiated mutant pool of japonica rice variety Nipponbare.The mutant showed albino phenotype from germination,and died after 3-leaf stage.Genetic analysis revealed that the mutant phenotype was controlled by a single recessive nuclear gene.The chlorophyll and carotenoid contents in al14 declined dramatically.Transmission electron microscopy examination showed there were no obvious chloroplasts except prolamellar bodies in al14.The expression level of most genes of photosystemⅠand photosystemⅡdecreased dramatically,but the ribosomal genes and RNA polymerase genes in chloroplast increased notably in al14 mutant.In al14,the expression amount of PsbN,aphotosystemⅡprotein N gene,was 118.23times as high as that in wild type.By genetic mapping with an F2population generated by crossing the heterozygous al14 plants with indica variety Huanghuazhan,the al14locus was mapped within 40kb on chromosome 6.So far there is no reported gene relative to leaf color,so it is a novel gene controlling chloroplast development.
Key words:rice;albino;gene mapping
中图分类号:Q755;S511.01
文献标识码:A
文章编号:1001-7216(2016)02-0136-07
基金项目:亚热带农业生物资源保护与利用国家重点实验室开放课题资助项目(SKL-CUSAb-2013-04);江西省教育厅科技计划资助项目(GJJ14707);广东省自然科学基金-博士启动项目(2015A030310485)。
收稿日期:2015-09-09;修改稿收到日期:2015-12-05。
