基于语义资源的生物医学文献知识发现研究
2016-05-03李宗耀杨志豪吴晓芳林鸿飞宫本东
李宗耀,杨志豪,吴晓芳,林鸿飞,宫本东,王 健
(1.大连理工大学 计算机科学与技术学院,辽宁 大连 116024;2. 中国烟草总公司大连市公司,辽宁 大连 116010)
基于语义资源的生物医学文献知识发现研究
李宗耀1,杨志豪1,吴晓芳1,林鸿飞1,宫本东2,王 健1
(1.大连理工大学 计算机科学与技术学院,辽宁 大连 116024;2. 中国烟草总公司大连市公司,辽宁 大连 116010)
目前,生物医学文献的数量正在呈指数的方式快速增长,这些文献中隐含着大量有用的信息,挖掘这些文献可以形成医学假设。但传统的基于简单共现的方法会产生大量的目标词,导致很难发现有用的假设。该文提出了一种基于语义资源的方法,利用SemRep工具抽取句子内实体之间的关系,结合语义类型、概念的信息量以及关联规则对连接词、目标词进行过滤,并根据统计量信息对目标词进行排序。通过对Swanson发现的经典病例进行验证,实验结果表明该方法取得很好的效果。
隐含知识发现;共现;语义关系
1 引言
目前,生物医学文献的数量正在呈指数的方式快速增长,例如,生物文献的数据库PUBMED中的文献已经超过两千万。如此海量的数据给研究者们带来丰富的信息,但阅读如此大量的文献对于医学研究者来说是相当困难的。因此,利用数据挖掘的方法自动地从生物医学文献中提取和组织有用的信息变得尤为迫切。
1986年美国芝加哥大学Swanson教授提出了基于非相关文献的知识发现[1]。当将逻辑上相关但相互独立的两个知识片段放到一起加以考虑,并且能够进行合理解释的时候,可能会有新的发现,并通过实例验证了其非相关文献知识发现的思想。发现过程如下:考察两个不相关的文献集CL(C Literature)和AL(A Literature),通过挖掘文献的信息,发现文献集CL中提到雷诺氏病患者(C)存在血液和血管相关的生理改变(B),例如,血黏度和血小板凝集度升高,血管收缩等;而在文献集AL中提到鱼油及其活性成分(A)可降低血黏度和血小板凝集度,并引起血管舒张。由此Swanson 得出,鱼油可能对雷诺病有治疗作用的假设,得到了经典的ABC发现模型,并在之后的医学临床实验得到了验证。在Swanson发现这种假设以前,从未有文章论述鱼油对雷诺病有治疗作用,甚至两类文献很少被共同引用或相互引用,这是两类非直接相关文献,Swanson定义类似的研究为基于非相关文献的知识发现LBD(Literature-based Discovery)。
许多学者重现了Swanson的研究工作,并发现了许多新的假设。早期的LBD研究大都采用信息检索技术,并作如下假定:如果概念A与概念B的共现次数越高,则概念A与概念B有关联的可能性越大。通过使用统计特征,自动化地实现ABC模型,如Yetisgen-Yildiz和Pratt开发了LitLinker系统[2],使用Z-Score、TFIDF、PMI等计算方法,并且使用信息检索中的准确率、召回率和MAP等方法评测知识发现的结果。Srinivasan使用医学主题词MeSH进行研究[3],减小了检索的时空复杂度,并引入了语义类型过滤,取得了不错的效果。上述方法普遍存在的问题是无法对得出的新发现做合理的解释,D.Hristovski[4]利用自然语言处理技术对生物文献进行处理,对每个句子抽取语义关系,并定义了关联规则,得到了可解释的发现。Trevor Cohen等人[5]使用语义索引预测模型发现了许多有效的关联规则,发展了D.Hristovski的方法。Delroy Cameron等人[6]在语义关系的基础上建立了图模型,拓展了ABC模型,并对雷诺氏病进行验证,从语义角度再现了Swanson的发现。但以上基于语义关系的方法都是进行闭合式发现,没有提出一个有效的对目标词排序的算法。本文通过充分挖掘语义资源,利用关联规则、语义类型对连接词、目标词进行过滤,并结合统计量信息,提出了一种有效的目标词排序方法,能够使有效的目标词尽可能排名靠前;同时相较于以前简单MeSH共现的方法,通过使用SemRep语料,根据实体之间的关系能够生成可解释的假设,并大幅度减少了无关的目标词数量,提高目标词的准确率。
2 基于生物医学文献的知识发现方法
2.1 基于生物医学文献的知识发现方法
Weeber等人[7]对Swanson的研究工作进行了总结,将知识发现定义为开放式发现和闭合式发现两个过程。
开放式算法(Open discovery):是一个产生假设的过程。在一种疾病C的治疗方法未知的时候,找到可能治愈或改善C的方法,发现过程如图1所示。
闭合式算法(Closed discovery):已知两个概念C和A,当研究者去寻找两者之间的关系、阐明A对C有效的病理时,可以采用闭合式发现,发现过程如图2所示。

图1 开放式发现

图2 闭合式发现
2.2 生物医学资源SemRep
SemRep是一个能够自动从文献中识别关系预测的自然语言处理系统,例如,SemRep能够从句子“In humans, ACh evoked a dose-dependent increase of NO levels in exhaled air”中抽取出“Acetylcholine | STIMULATES | Nitric Oxide”(乙酰胆碱 | 刺激 | 一氧化氮)。UMLS中定义了54种语义关系,在实验中我们剔除了一些包含信息量比较少的关系,如ISA、PROCESS_OF、PROCESS_OF (SPEC)、ADMINISTERED_TO、ADMINISTERED_TO (SPEC)、 PART_OF等。
2.3 医学主题词MeSH
医学主题词(Medical Subject Headings,MeSH)是美国国家医学图书馆开发和维护的综合性受控词汇表,用来描述生物医学主题或特性。MeSH由主题词变更表、字母顺序表、副主题词和树形结构表组成,其中字母顺序表和树形结构表是MeSH的主要部分,本文主要利用了MeSH的树形结构表,将表中所有的主题词和非主题词按照学科性质和语义关系进行层次分类,表示概念之间的隶属关系。越底层的概念越具体,所包含的信息量越大。
3 方法
3.1 语义类型过滤
UMLS(Unified Medical Language System)本体定义了135种语义类型,每个MeSH词都有固定的语义类型,通过选择合适的连接词、目标词的语义类型,可以对二者进行有效地过滤。连接词主要选取一些能够表示生理状况的语义类型,目标词作为我们要找的能够治疗疾病的物质,其语义类型一般为化学物质。本文主要参考Weeber实验中选择的连接词、目标词的语义类型[7],如表1所示。

表1 实验所选取的语义类型
3.2 发现模板
基于MeSH词简单共现的方法并没有考虑概念之间的关系,D. Hristovski等人[4]提出了发现模板(Discovery Patterns),例如,一个药物C与一个实体B的关系是( INTERACTS_WITH、 INHIBITS、STIMULATES、COEXISTS_WITH),而实体B与疾病A的关系是ASSOCIATED_WITH,我们便假定药物C与疾病A存在关系的可能性比较大。Trevor Cohen等人[5]使用语义索引预测模型发现了许多有效的发现模板,但是这些模板数量的数量有限,会将许多有用的目标词都给过滤掉。在Trevor Cohen等人选择模板的基础上,我们对以前病例进行分析,新加入了一些模板,得到了如表2的模板集合。

表2 实验所选取的发现模板
3.3 MeSH宽泛含义概念过滤
MeSH树状结构表能够表示MeSH上位词与下位词之间的关系。例如,MeSH“羊水”(Amniotic fluid,编号:A16.254.72)是MeSH“胚胎”(Embryo,编号:A16.254)的一个下位词。Seco等人[8]认为在本体中,一个概念包含的下位词越多,那么其包含的信息量越少。所以,一个概念C表达的信息量由下位词数量函数来表示,其计算公式如式(1)所示。
(1)
其中hypc(c)为概念c的下位词数量,Ns为概念c所处大类中概念的数量,在上面的例子中即为MeSH“胚胎结构”(Embryonic Structures)所包含下位词的数量。通过实验分析,我们保留了信息量大于0.6的MeSH词,阈值太小会导致过滤效果不明显,阈值太大会使得一些重要概念被过滤掉。
3.4 目标词排序
通过开放式发现算法,得到目标词的集合,但目标词数量在经过两次共现之后变得非常大,如何对目标词排序是开放式发现的难点。Pratt和Yetisgen-Yildiz采用连接词的数量(Linking Term Count,LTC)对目标词排序[9]。他们基于这样的假定:如果一个目标词可以通过多个连接词得到,就认为这个目标词与初始词的关系越密切。在他们基础之上,我们假定同一个SemRep关系出现的次数越多,这个关系越可信;同时如果一个目标词可以由多个连接词得到,则其越可信。但有的连接词在多篇文献中出现,属于比较泛化的概念,提供的信息相对较少,我们利用信息检索中常用特征DF对其进行平滑,减小其影响因子。我们提出的量化目标词公式如式(2)所示。
(2)
其中,S(A)为概念A的得分,f(Bi,C)为概念C与概念Bi在SemRep中的关系频次,f(Bi,A)为概念Bi与概念A在SemRep中的关系频次,df(Bi)为概念Bi在Medline出现的文档频次,N为Medline文档集中包含的文档个数。
4 实验与评价
实验使用的语料为Semantic MEDLINE Database,这是一个已处理好的SemRep数据库,其下载地址为:http://skr3.nlm.nih.gov/SemMedDB/。实验过程如下,将要查询的疾病设为初始词,利用开放式算法检索数据库,得到连接词集合,对连接词进行语义类型以及MeSH宽泛含义概念过滤;利用过滤后的连接词检索数据库,得到目标词集合,对目标词进行语义类型以及MeSH宽泛含义概念过滤,并剔除与初始词共现过的概念;然后通过发现模板寻找可能的关联,最后利用目标词排序算法对目标词排序。由于目前没有统一的评测标准,重现Swanson发现的三种经典病例(雷诺氏病与鱼油、老年痴呆症与镁、偏头痛与消炎痛)成为了生物医学领域文本知识发现评测的标准答案,以后的学者都重复Swanson的发现来验证他们方法的有效性,以下分别介绍三种疾病的发现结果及结果分析。
4.1 雷诺氏病与鱼油
由于Swanson在1986年发现了两者的关系,我们使用1986年以前的SemRep语料进行实验。为了验证我们提出方法的有效性,我们分步使用不同的过滤方法,来观察对目标词排名的影响。在实验过程中,将初始词设定为Raynaud Disease(雷诺氏病)和Raynaud Phenomenon(雷诺氏现象),在分步使用语义类型过滤、MeSH宽泛含义概念过滤、发现模板过滤后,鱼油的排名依次为274、262、38,可以看到通过模板过滤后,鱼油的排名有大幅提升,主要原因是过滤掉了许多不符合发现模板的概念。鱼油在表1所示的目标词语义类型的排名结果为38,而在油脂语义类型(Lipid)下的排名结果为3(表3)。
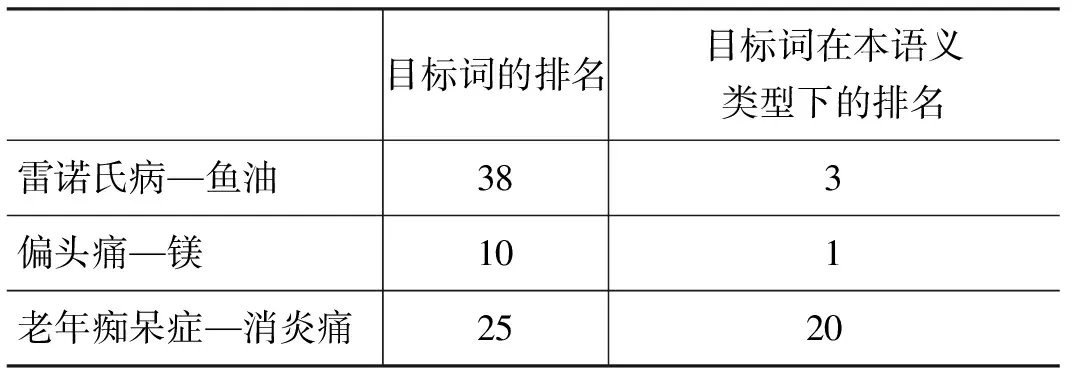
表3 三种疾病目标词的排名
我们依据找到的关系可以从数据库中抽得关系所在的句子,通过这些句子可以进行药理分析,同时十二碳五烯酸(Eicosapentaenoic Acid,EA)作为鱼油的主要成分,我们也考察了其与雷诺氏病的关系,具体的病理分析如表4所示。我们可以看到在PMID为458185的MEDLINE文摘中提到在雷诺氏病中血液粘稠度增加,而在PMID为14851220的MEDLINE文摘中提到鱼油对血液粘稠度有影响;在PMID为1069430的MEDLINE文摘中提到雷诺氏病会伴随动脉障碍,而在PMID的6320945的MEDLINE文摘中提到EA有助于改善动脉障碍。
4.2 偏头痛与镁
Swanson在1988年发现了偏头痛(Migraine)和镁(Magnesium)可能存在关联,我们选取1987年以前的语料进行实验。我们将初始词设为偏头痛,进行开放式发现。再经过语义类型过滤,过滤掉了一些不相关语义类型的概念;通过MeSH宽泛含义概念过滤剔除了一些比较宽泛的概念,诸如,“Anions”、“Elements”等。通过目标词排序算法,最终得到的镁的排名为10(表3),在Element,Ion,or Isotope(元素,离子或同位素)这一种的语义类型下排名为1,而在Srinivasan实验中排名为5。

表4 雷诺氏病与鱼油的病理分析
利用SemRep语料对偏头痛与镁的病理分析,得到的部分关系组合,如表5所示。可以看到在PMID为1164613的MEDLINE文摘中提到偏头痛患者体内的γ-氨基丁酸失常,而PMID为1143360的MEDLINE文摘中提到镁可以提高γ-氨基丁酸的含量;在PMID为239368的MEDLINE文摘中提到偏头痛与组胺有关联,而镁离子对组胺的含量有影响。
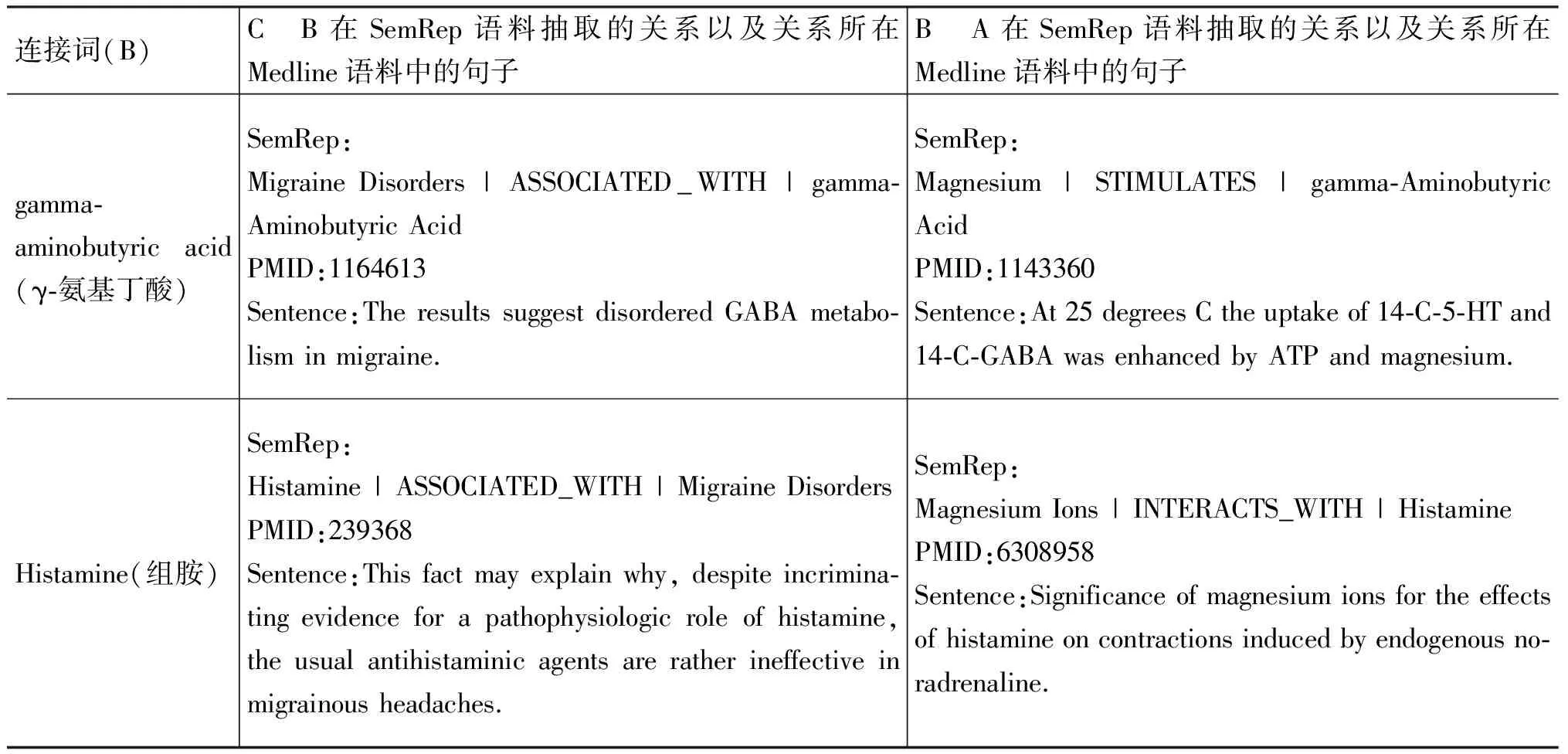
表5 偏头痛与镁的病理分析
4.3 老年痴呆症与消炎痛
在实验过程中,我们发现在1993年之后有多篇文献提及了老年痴呆症(Alzheimer’s Disease)与消炎痛(Indomethacin),为了重现二者的关系,我们选择1993年之前的语料进行实验。初始词设为老年痴呆症,经过开放式发现以及过滤处理后,消炎痛的排名如表3所示。
利用SemRep语料对老年痴呆症与消炎痛的病理分析,得到了一系列的关系组合,如表6所示。我们可以看到在PMID为2187699的MEDLINE文摘中提到老年痴呆症与血酸过多症有关联,而PMID为6276989的MEDLINE文摘中提到消炎痛能改变血酸过多症的影响;在PMID为1946550的MEDLINE文摘中提到老年痴呆症与类脂代谢作用有关联,而PMID为6809337的MEDLINE文摘中提到消炎痛对类脂代谢作用有影响。
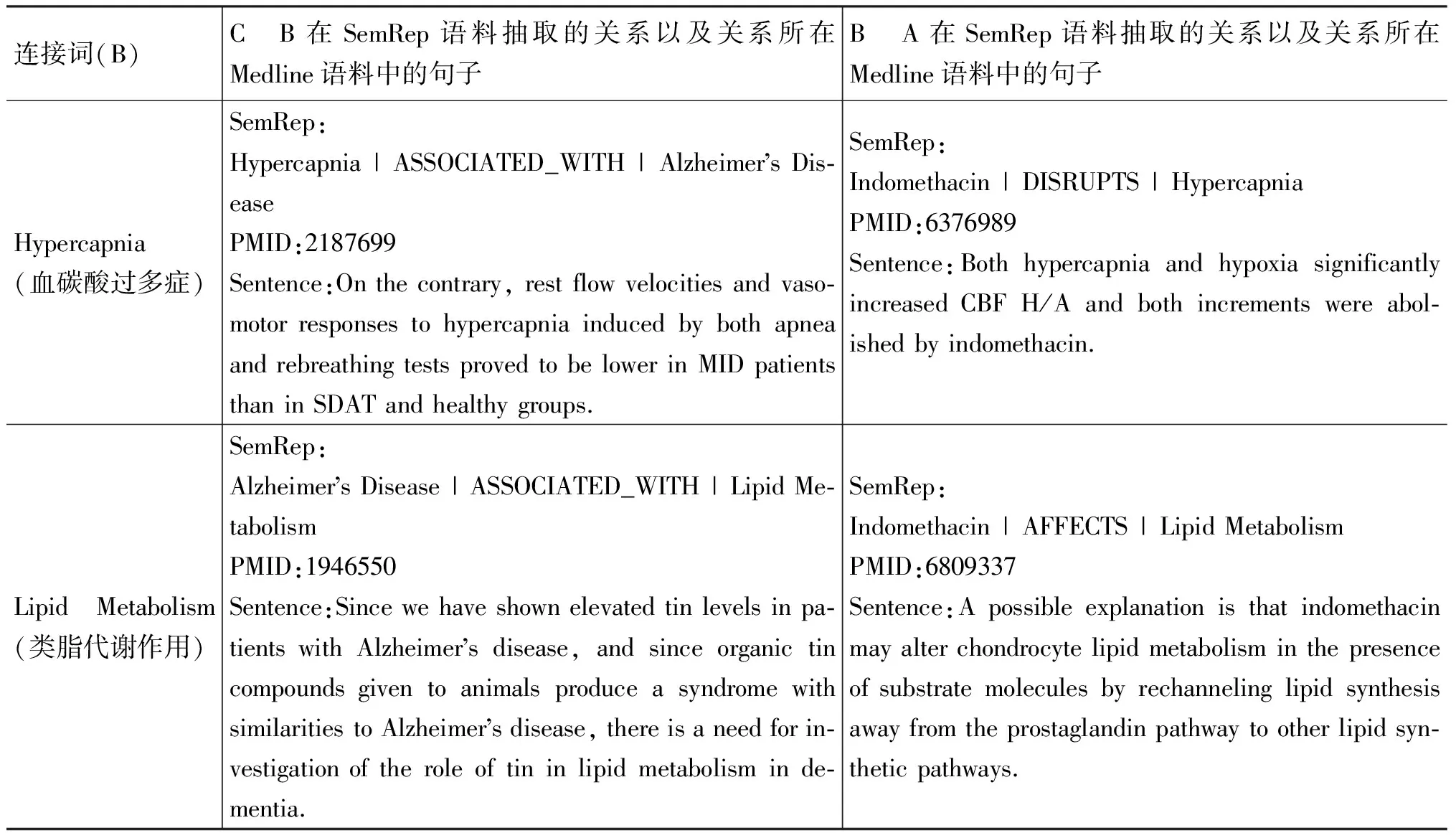
表6 老年痴呆症与消炎痛的病理分析
5 总结和展望
本文提出了一种基于语义关系的非相关文献知识发现方法,旨在帮助生物医学人员发现潜在的知识。相较于以前简单MeSH共现的方法,通过使用SemRep语料,根据实体之间的关系能够生成可解释的假设,并大幅度减少了无关的目标词数量;同时本文综合了前人在连接词和目标词过滤方面的工作,取得了不错的过滤效果;最后根据经过开放式发现后形成的概念之间的网络特点,提出了一种目标词排序算法,得到的目标词排名都相对靠前,验证了方法的有效性。同时SemRep语料中抽取的实体之间的关系准确率还有进一步提升的空间(准确率为0.73,召回率为0.55,综合分类率F值为0.63)[10]。我们下一步的计划是将命名实体识别和事件抽取方面的知识融合到实验中,以提高实体间关系的准确率、召回率,进而改善知识发现的性能。
[1] Swanson D R. Two medical literatures that are logically but not bibliographically connected [J]. Journal of the American Society for Information Retrieval, 1987, 38(4): 228-233.
[2] Yetisgen-Yildiz M, Pratt W. Using statistical and knowledge-based approaches for literature based discovery [J]. Journal of Biomedical Informatics, 2006, 39:600-11.
[3] Srinivasan P. Text mining: Generating hypotheses from MEDLINE [J]. Journal of the American Society for Information Science, 2004, 55(4):396-413.
[4] D.Hristovski, D., Friedman, C., Rindflesch, T.C., et al. Exploiting semantic relations for literature-based discovery[C]//Proceedings of the AMIA Annu. 2006: 349-353.
[5] Cohen T, Widdows D. Discovery at a Distance: Farther Journeys in Predication Space[C]//Proceedings of the 2012 IEEE International Conference on Bioinformatics and Biomedicine Workshops (BIBMW), Bioinformatics and Biomedicine, 2012:218-225.
[6] Cameron D, et al. A graph-based recovery and decomposition of Swanson’s hypothesis using semantic predications. J Biomed Inform (2012)[OL], http://dx.doi.org/10.1016/j.jbi.2012.09.004
[7] Weeber M, Klein H, Berg L, et al. Using concepts in literature based discovery: simulating Swanson’s Raynaud-fish oil and migraine-magnesium examples [J]. Journal of the American Society for Information Science and Technology, 2001, 52(7):548-57.
[8] Seco N, Veale T, Hayes T. An intrinsic information content metric for semantic similarity in wordnet[C]//Proceedings of European Conference on Artificial Intelligence, Valencia,Spain,2004:1089-1090.
[9] Pratt W, Yetisgen-Yildiz M. LitLinker. capturing connections across the biomedical literature [C]//Proceedings of the International Conference on Knowledge Capture (K-CAP’03),Florida,2003:105-112.
[10] Ahlers CB, Fiszman M, Demner-Fushman D, et al. Extracting semantic predications from MEDLINE citations for pharmacogenomics[C]//Proceedings of the Pac Symp Biocomput 2006:209-20.
Using Semantic Relations for Biomedical Literature-Based Discovery
LI Zongyao1, YANG Zhihao1, WU Xiaofang1, LIN Hongfei1, GONG Bendong2, WANG Jian1
(1. School of Computer Science and Technology, Dalian University of Technology, Dalian, Liaoning 116024, China;2. Dalian Branch of China National Tobacco Corporation, Dalian, Liaoning 116010,China)
Nowadays, the amount of biomedical literatures is growing at an explosive speed, and there is a lot of useful information undiscovered in these literatures, e.g. researchers can form biomedical hypotheses through mining these literatures. However, the popular mining solution based on co-occurrence produce too many target useless concepts. This paper presents a mining method based on semantic resources, i.e. using SemRep system to extract relationships between entities within the sentence. By employing a combination of the semantic type, concept information amount and association rules, we can effectively filter the linking and target concepts and sort the target concepts with the statistical information. The experimental results demonstrate that our method works well on the classic cases found by Swanson.
literature-based discovery; co-occur; semantic relation

李宗耀(1990-),硕士,主要研究领域为基于生物医学文献的知识发现。E⁃mail:lizongyao@mail.dlut.edu.cn杨志豪(1973-),教授,博士生导师,主要研究领域为文本挖掘、信息检索。E⁃mail:yangzh@dlut.edu.cn吴晓芳(1989-),硕士,主要研究领域为文本挖掘。E⁃mail:xfwu@mail.dlut.edu.cn
1003-0077(2016)01-0176-07
2014-01-08 定稿日期: 2014-05-07
国家自然科学基金(61070098,61272373);国家社科基金(08BTQ025);中央高校基本科研业务费专项资金(DUT13JB09)
TP391
A
