PRRSV广西分离株GXYL-1403全基因克隆与序列分析
2016-03-30林思远洪绍锋黄加滨黄伟坚韦祖樟
林思远,洪绍锋,黄加滨,韦 莹,陈 樱,黄伟坚,韦祖樟
(广西大学动物科学技术学院动物传染病研究室,广西南宁 530005)
PRRSV广西分离株GXYL-1403全基因克隆与序列分析
林思远,洪绍锋,黄加滨,韦莹,陈樱,黄伟坚,韦祖樟*
(广西大学动物科学技术学院动物传染病研究室,广西南宁 530005)
摘要:对本实验室所分离的1株猪繁殖与呼吸综合征病毒(PRRSV)毒株GXYL-1403进行全基因组克隆和序列分析。参考已发表的扩增全长PRRSV序列的13对特异性引物,用RT-PCR方法对病毒基因组进行分段扩增。将扩增得到的PCR片段克隆到pMD18-T载体上,获得的重组质粒经PCR鉴定后进行序列测定。通过软件对所扩增序列进行拼接,获得GXYL-1403的全长序列,并对GXYL-1403进行比较和遗传进化分析。结果表明GXYL1403株基因序列全长为15 018 bp,与国内外多株PRRSV毒株进行同源性比较发现,与VR-2332的相似性为89.1%,与TJ、JXA1、HEB1、HUB1、HUB2、TP株同源性达到了97.9%~98.1%。另外,GXYL-1403分离株nsp2蛋白含有高致病性PRRSV特异的1+29个氨基酸的缺失。特别重要的是,在缺失29个氨基酸区域的上游,含有连续20个氨基酸缺失,在下游出现了连续74个氨基酸的缺失。遗传进化分析发现美洲型PRRSV主要分为3个亚群,GXYL-1403跟高致病性PRRSV毒株属于同一分支,证实了GXYL-1403株为美洲型PRRSV毒株。
关键词:猪繁殖与呼吸综合征病毒;克隆;序列分析;遗传进化分析
猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV)是猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)的病原。不同年龄、品种和性别的猪均可感染,以妊娠母猪和1月龄以内的仔猪最易感,主要特征为母猪流产、死胎、弱胎、木乃伊胎及仔猪呼吸困难、败血症、发育迟缓和高死亡率等[1]。PRRSV是一种单股、正链、不分节段的RNA病毒,归属于动脉炎病毒科动脉炎病毒属。该病毒具有至少9个开放阅读框架(ORF), 其中ORF1 包括ORF1a 和ORF1b , 主要编码病毒的13个非结构蛋白(nsp), 约占病毒基因组的80%。ORF2a、2b和3-5 编码病毒的囊膜糖蛋白(GP2、E、GP3、GP4和GP5),ORF6编码基质蛋白(M),ORF7编码病毒的核衣壳蛋白(N)。5′端有非编码区, 其作用是作为5′ 前导序列调节PRRSV 基因组RNA 的复制、转录与翻译, 从而介导各结构蛋白的表达。在病毒基因组的3′末端也有一个非编码区, 其功能是作为病毒复制酶识别并结合的区域,以启动负链RNA的合成[2-3]。该病毒的历史是由鼠的动脉炎病毒(乳酸脱氢酶增高病毒-LDV)感染了中欧的野猪后产生的变异病毒,而后这些野猪被引进美国。这些病毒在这两大洲内独立变异和进化,通过家猪和野猪的接触,病毒进入家猪群中,形成了后来的两大独立的猪蓝耳病病毒原型,即欧洲型和美洲型。2个基因型的同源性在60%左右[3-5]。高致病性PRRSV目前已成为国内的主要流行毒株。更多的研究表明PRRSV基因组在持续变异,很有必要对现行流行的PRRSV基因组进行比较和遗传进化分析。通过对流行毒株的基因组特性和遗传进化进行分析,为实施有针对性的免疫预防PRSSV提供科学依据。
1材料与方法
1.1材料
1.1.1病毒、细胞和菌株PRRSV毒株GXYL-1403为本实验室接收、分离、鉴定并保存。大肠埃希菌Dpα、Marc-l45细胞由广西大学动物科学技术学院家畜传染病与分子免疫学实验室保存。
1.1.2主要试剂新生胎牛血清、MEM为GIBCO公司产品;pMD18-T 载体、TaqMix、dNTP、Mixture、Trizol(RNA isolater)、RNase Inhibitor、M-MLV、LATaq酶等为宝生物工程(大连)有限公司产品;DNA 凝胶回收试剂盒为Axyden Scientific公司产品。
1.2方法
1.2.1引物的设计与合成本研究使用的引物参考已发表文献引物[6](表1)。
1.2.2病毒基因组RNA的提取、RT-PCR扩增和克隆收取第3代病毒感染细胞后的上清液,参照说明书使用Trizol抽提病毒基因组RNA,所提取基因组RNA用20 μL 1 g/L DEPC水溶解。

表1 RT-PCR特异性引物
以病毒RNA作为模板,以12.5 μL的体系进行反转录:8 μL RNA作为模板,dNTP(2.5 mmol/μL) 1 μL,反转录酶M-MLV(200 U/μL) 0.25 μL,5×反转录酶M-MLV buffer 2.5 μL,RNA酶抑制剂(40 U/μL) 0.25 μL,随机引物(10 pmol/μL) 0.5 μL。反应条件:42 ℃ 1 h。将得到的cDNA用于PCR扩增。
用cDNA 2 μL、10×LATaqPCR buffer(Mg2+free)2.5 μL、dNTP(2.5 mmol/μL)4 μL、LATaq酶(5 U/μL)0.25 μL的作用下,以各引物对(PF1/PR1203、PF1154/PR2379、PF2108/PR4238、PF4070/PR5373、PF5349/PR6663、PF6574/PR7976、PF7894/PR9232、PF9122/PR10481、PF10378/PR11569、PF11322/PR12186、PF11759/PR13401、PF13138/PR14371、PF14231/PR15300)(10 pmol/μL)(表1)为引物,加双蒸水补至25 μL的体系,进行PCR反应。PCR扩增条件:94 ℃ 1 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 1.5 min,30个循环;72 ℃ 10 min。用12 g/L琼脂糖凝胶电泳检测PCR产物。对所获得正确的DNA条带进行胶回收,再用12 g/L琼脂糖凝胶电泳检测胶回收产物,看其是否与PCR产物条带一致。
将PCR产物与pMD18-T 载体4 ℃连接过夜,连接产物转化到大肠埃希菌Dpα感受态细胞,在37 ℃摇床3 h~4 h。之后在LA培养基中加入100 μL的菌液和40 μL的Amp+涂板,37 ℃培养箱过夜。挑取5个单个菌落放入加有40 μL Amp+的5 mL LB试管中,在37 ℃摇床12 h,最后菌液PCR对电泳检测的产物与引物长度进行比对,条带正确的抽取1 mL送上海杰李生物有限公司测序。
1.2.3生物信息分析 对测序结果用Lasergene软件进行拼接,获得GXYL-1403全基因组核苷酸序列。用DNA Star对毒株基因组进行核苷酸序列及其推导氨基酸序列的比对分析。用MEGA5软件对病毒进行遗传进化分析。
2结果
2.1序列测定和分析比较
收集GXYL-1403感染的Marc-145细胞上清液,用Trizol抽提病毒基因组RNA,对GXYL-1403的基因组进行分段扩增和鉴定。接着克隆到载体,提取阳性结果进行测序。用Seq Man对片段进行拼接,得PRRSV毒株GXYL-1403全长为15 018 bp。将GXYL-1403与其他13株PRRSV毒株(表2)序列相比(BJ-4、VR2332、LV、CH-1a、GD、HEB1、HN1、HUB1、HUB2、JXA1、S1、TJ和TP),基因组全长、各ORFs长度结果见表3。GXYL-1403基因与其他代表株同源性比较见图1。
如表3所示,GXYL1403的5′端UTR 含有189 bp,同其他高致病性PRRSV毒株一样。所有毒株的3′端UTR除S1株为188 bp其余均为150 bp或151 bp。在参考的PRRSV毒株中,ORF1a的长度变化是最大的,经典毒株VR2332、S1、HN1和CH-1a 的ORF1a为7 512 bp,BJ-4 的ORF1a为7 509 bp。而高致病性PRRSV(HP-PRRSV)毒株在ORF1a比VR2332少了90 bp,这是HP-PRRSV的遗传特征即nsp2不连续缺失29+1个氨基酸。GXYL-1403株基因全长为15 018 bp,比其他的参考对比毒株序列都要短,说明该毒株的基因组其他位点也存在缺失。而GXYL-1403株的ORF1a却比VR2332少372 bp,也就是说缺失了124个氨基酸。从图2可以看出GXYL-1403在nsp2高变区不连续缺失了123+1个氨基酸。其中,缺失的“1”同为图中480位,HP-PRRSV毒株缺失的“29”为532-560位,而GXYL-1403缺失的“123”为512-634位,分别在“29”的上游有20个氨基酸缺失,“29”的下游有74个氨基酸缺失。由此可见,GXYL-1403株nsp2高变区的缺失部分相比于HP-PRRSV的经典缺失部分有所扩大。之后对其他的ORF进行分析发现,GXYL-1403株在ORF1b、2~7的长度与其他毒株一致,分别为4 383、771、765、537、603、525、372 bp。
对该毒株与其他13株经典毒株进行同源性比较(图2),发现GXYL-1403与美洲经典株VR-2332同源性为89.1%,高于与欧洲经典株LV的同源性60.9%,与TJ株同源性最高,达到了98.1%;与JXA1、HEB1、HUB1、HUB2、TP株同源性也高达98%,与GD株的同源性也达到了97.9%。由此可见GXYL-1403株为HP-PRRSV毒株。
2.2遗传进化树分析
为了解GXYL-1403株与其他PRRSV毒株之间基因的关系,基于病毒全基因组核苷酸序列绘制遗传进化树(图3),可将GXYL-1403株与国内外PRRSV美洲型参考株分为3个亚群,GXYL-1403株和HP-PRRSV毒株如JXA1、HUB1、HUB2、TJ等在一个亚群中;经典株VR-2332、BJ-4、S1等在1个亚群;在经典毒株与高致病性毒株之间,又有1个亚群以CH-1a为代表。
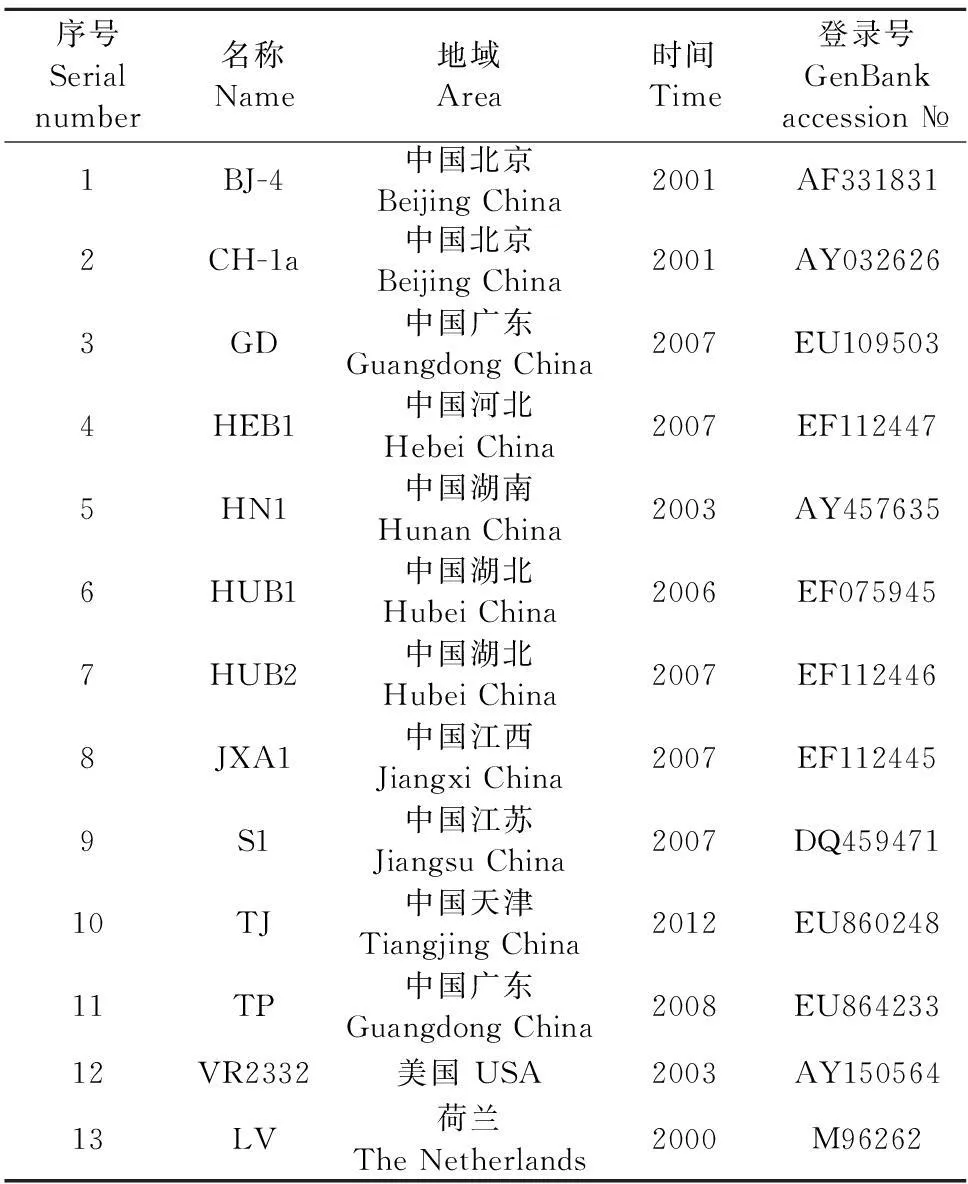
表2 PRRSV毒株名称及其来源
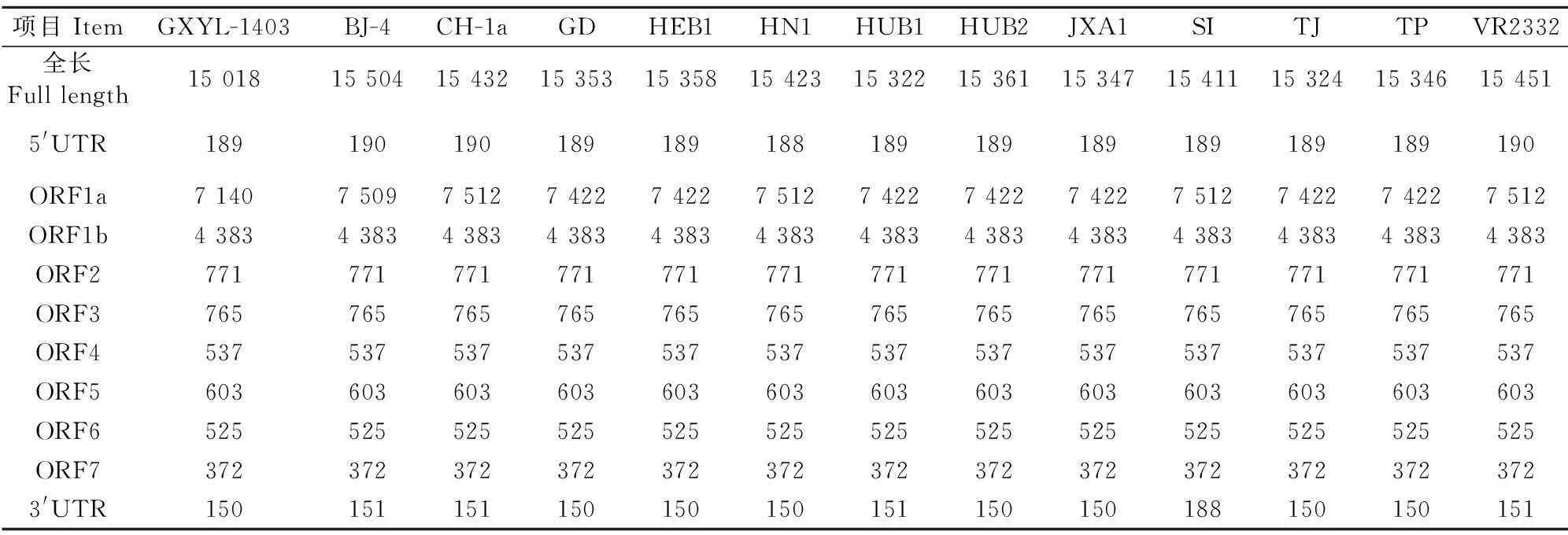
表3 GXYL-1403株与12株PRRSV毒株全基因组的长度比较

图1 GXYL-1403株与12株PRRSV毒株在nsp2高变区氨基酸序比较:
3讨论
PRRSV有2个基因型,即VR-2332株为代表的美洲型Ⅱ型和以LV株为代表欧洲Ⅰ型,这两者的基因序列的同源性仅为60%左右[7]。目前为止,我国分离鉴定的毒株多为Ⅱ型的PRRSV毒株。而Ⅱ型的PRRSV毒株又可分为3个亚群,第1个亚群以VR-2 332位代表,第2个亚群以CH-1a为代表,通过序列比对和遗传进化分析,确定我们分离的毒株是Ⅱ型PRRSV毒株,且是处于第3个亚群即高致病性PRRSV毒株(JXA1、TJ),同亚群毒株全基因组对比同源性高达97.9%~98.1%。
PRRSV各个毒株基因组存在着广泛的变异,特别是nsp2的基因变异最大,并且能够耐受碱基的插入或缺失[8]。在2006年暴发的“高热综合征”后出现的在nsp2蛋白的29+1氨基酸缺失已经成为了我国HP-PRRSV的标志性特征。邓雨修等人于2010年7月研究发现广东PRRSV TM株除经典缺失外,在22-42位有其他21位氨基酸的缺失[9]。刘飞等人的研究显示贵州PRRSV GZZB株除经典缺失外在395位还有1个氨基酸缺失,同时还发现以GZMN株为代表的3株PRRSV序列除经典缺失外在481位氨基酸的两侧还有29个氨基酸的缺失[10]。这些新型的缺失正在提示我们,在我国的南方地区PRRSV病毒基因组缺失有进一步扩大和加强的趋势。然而也有研究表明,HP-PRRSV毒株29+1个氨基酸的缺失与病毒毒力强弱没有关系[11]。在此次研究中发现,GXYL1403株唯一只有ORF1a基因全长与对比参考毒株不同,其余片段则是一致的(表3)。重点分析该毒株nsp2片段(图3)发现,PRRSV GXYL1403株的nsp2蛋白中不仅仅是29+1的氨基酸缺失了,而是123+1的氨基酸缺失,在缺失29个氨基酸区域的上游,含有连续20个氨基酸缺失,在下游出现了连续74个氨基酸的缺失,此缺失是在29个氨基酸缺失的基础上进一步扩大形成的,这是否说明该毒株有进一步的致病性变化,扩大的缺失部分是否对病毒生物学特性有影响,均有待于进一步的试验证实。
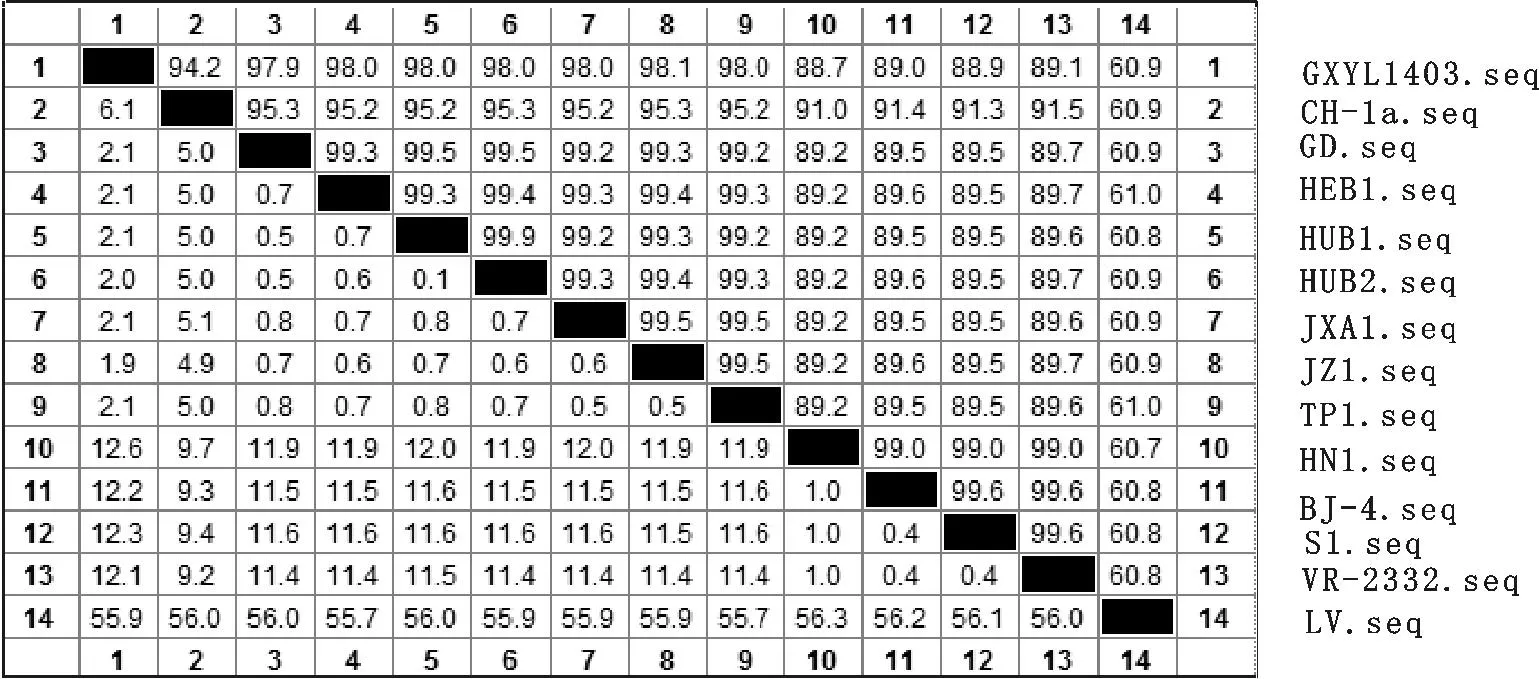
图2 GXYL-1403株基因与其他代表株的同源性比较 (%)
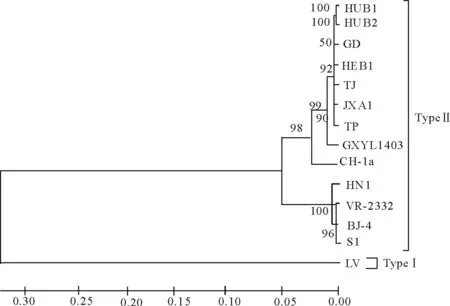
图3 GXYL-1403株与部分PRRSV流行毒株的全基因遗传进化树
在本研究中,对实验室分离到的PRRSV毒株GXYL-1403进行了全基因序列分析、同源性比较和遗传进化分析,确定了该毒株的来源与遗传变异情况,同时也丰富了PRRSV分离株的全基因组序列数据库,这对日后该病的流行病学调查提供了必要的数据,为进一步研究nsp2氨基酸缺失对病毒生物学特性的影响奠定了基础。
参考文献:
[1]罗成波,崔敏,孔令萍,等. 2株高致病性猪繁殖与呼吸综合征病毒分子特征分析[J]. 中国畜牧兽医, 2014,41(8):50-56.
[2]刘光清,薛强,仇华吉,等. 猪繁殖与呼吸综合征病毒CH-1a株非结构基因的分子克隆及其基因特征的研究[J]. 中国预防兽医学报,2002,24(2):81-87.
[3]Leng Chao-liang, Tian Zhi-jun, Zhang Wu-chao, et al. Characterization of two newly emerged isolates of porcine reproductive and respiratory syndrome virus from Northeast China in 2013[J]. Vet Microbiol, 2014,171(1-2):41-52.
[4]刘永宏,赵丽,刘俊峰,等. 从猪繁殖与呼吸综合征病毒的起源与进化谈猪繁殖与呼吸综合征的防制[J]. 中国兽医杂志, 2012,48(3):56-58.
[5]Plagemann G W. Porcine reproductive and respiratory syndrome vVirus: origin hypothesis[J]. Emerg Infect Dis, 2003,9(8):903-908.
[6]Xue Leng, Zhen G L, Ming Q X,et al. Mutations in the genome of the highly pathogenic porcine reproductive and respiratory syndrome virus potentially related to attenuation[J]. Vet Microl,2012,157(1-2):50-60.
[7]Nelsen C J, Murtaugh M P, Faaberg K S. Porcine reproductive and respiratory syndrome virus compareison: divergent evolution on two continents[J].J Virol, 1999,73(1):270-280.
[8]廖承球.广西PRRSV GXA株全基因序列测定、致病性实验及PRRSV分子流行病学调查研究[D]. 广西南宁:广西大学,2011.
[9]邓雨修,招丽婵,苏润环,等. 猪繁殖与呼吸道综合征病毒江苏TM株的分离鉴定及其NSP2序列分析[J] .中国兽医学报, 2010,30(7):878-882.
[10]刘飞,王开功,周壁君,等. 贵州省PRRSV分离株NSP2基因的克隆及序列分析[J] 中国预防兽医学报, 2010,32(12):924-928.
[11]Zhou L, Zhang J L, Zeng J W, et al. The 30-aminoacid deletion in the nsp2 of highly pathogenic porcine reproductive and respiratory syndrome virus emerging in China is not related to its virulence[J]. J Virol, 2009,83(10): 5156-5167.
Cloning and Sequence Analysis Full-length Genome of PRRSV Isolate GXYL1403 in Guangxi
LIN Si-yuan,HONG Shao-feng, HUANG Jia-bin,WEI Ying,CHEN Ying,HUANG Wei-jian,WEI Zu-zhang
(AnimalInfectiousDiseaseLaboratory,CollegeofAnimalScienceandTechnology,GuangxiUniversity,Nanning,Guangxi,530005,China)
Abstract:To clone and analyse the full-length genome of a PRRSV strain GXYL-1403 isolated in our lab, 13 pairs of primers published were used for RT-PCR to amplify the full-length genome of GXYL-1409. The PCR fragments were cloned into pMD18-T vector. The amplicons were identified by emzyme digestion and further sequenced. The full-length genome was obtained through DNA splicing by DNAstar software. The phylogenic tree of GXYL1403 and the reference strains was constructed based on the full-length genome. The results showed that the complete genome of GXYL1403 consisted of 15 018 bp. The complete genome of GXYL-1403 shared 89.1% identity with VR-2332 and shared 97.9%-98.1% identity with HP-PRRSV strains TJ,JXA1,HEB1,HUB1,HUB2,TP. The phylogenic analysis based on the full-length genome revealed that PRRSV strains were classified into 3 subgroups and GXYL-1403 was confirmed as HP-PRRSV. It was suggested that GXYL1403 belongs to type II PRRSV. GXYL-1403 strain contained a deletion of 1+29 aa in nsp2 coding region. More importantly, GXYL-1403 strain contained a deletion of 20 aa in nsp2 coding region, which was located upstream of 29 aa deletion. Beside, a continuous deletion of 74 aa emerged following the 29 aa deletion in nsp2 region.
Key words:Porcine reproductive and respiratory syndrome virus;clone; sequence analysis; phylogenetic analysis
文章编号:1007-5038(2016)02-0044-06
中图分类号:S852.659.6;Q785
文献标识码:A
作者简介:林思远(1992-),男,四川自贡人,硕士研究生,主要从事动物传染病与分子免疫学研究。*通讯作者
基金项目:国家自然科学基金项目(31372444);广西大学科研基金项目(XGZ130959)
收稿日期:2015-04-14
