火炬松生长和松脂性状相关候选基因的单核苷酸多样性分析
2016-02-23杨会肖刘天颐刘纯鑫王金榜黄少伟
杨会肖, 刘天颐, 徐 斌, 刘纯鑫, 王金榜, 黄少伟
(1 广东省林业科学研究院, 广东 广州 510520; 2 广东省森林植物种质创新与利用重点实验室/华南农业大学 林学与风景园林学院, 广东 广州 510642; 3 英德市林业科学研究所, 广东 英德 513000)
火炬松生长和松脂性状相关候选基因的单核苷酸多样性分析
杨会肖1,2, 刘天颐2, 徐斌1, 刘纯鑫2, 王金榜3, 黄少伟2
(1 广东省林业科学研究院, 广东 广州 510520; 2 广东省森林植物种质创新与利用重点实验室/华南农业大学 林学与风景园林学院, 广东 广州 510642; 3 英德市林业科学研究所, 广东 英德 513000)
摘要:【目的】对火炬松Pinustaeda生长和松脂产量相关功能基因单核苷酸多态性(Single nucleotide polymorphism,SNP)位点进行筛选与分析,为分子标记辅助育种提供技术基础。【方法】利用生物信息学对火炬松cDNA文库进行整理、比对、剪接、注释,挑选出与生长素、赤霉素、细胞分裂素及蒎烯合成相关的非重复序列基因(Unigene)作为候选基因片段,利用MEGA5.0和DnaSP4.0软件对火炬松36个单株的8个候选基因片段进行序列比对和分析。【结果】 所测序列总长为5 177 bp,检测到184个SNP位点,平均36.9 bp的基因序列中出现1个SNP位点,其中123个为非同义突变、61个为同义突变SNP位点。核苷酸多态性πa和θw分别为0.020和0.016。对8个候选基因片段内SNP位点进行的连锁不平衡分析显示,随着核苷酸序列长度的延伸,SNP位点的连锁不平衡在基因内部迅速衰退(R2≤0.2)。【结论】在火炬松中,基于候选基因内SNP位点间的连锁不平衡作图是可行的。
关键词:火炬松; 候选基因; 单核苷酸多态性; 连锁不平衡
林木的重要性状大多属于数量性状,由许多基因和环境共同作用来调控其生长与发育。传统的数量遗传学不能检测控制数量性状的基因的数量、基因在染色体上的位置、每个位点的贡献大小以及基因间的关系。在数量性状的多基因体系中,尽管可能存在效应较大的主效基因,但多数基因对目标性状可能只表现微效作用,而且表型受环境影响很大,因而采用传统的育种途径对这些性状进行选择有很大的局限性[1-2]。近年来,连锁分析和连锁不平衡作图已成为许多树种检测复杂性状的手段。关联分析是对控制数量性状的多基因系统进行遗传剖析的有效手段,而关联作图则是植物遗传学研究最重要的工具之一[3]。对某一植物种而言,筛选出的单核苷酸多态性(Single nucleotide polymorphism,SNP)位点在进行关联分析之前,有必要对目标基因的群体遗传学特征如核苷酸多样性、基因进化动力及连锁不平衡特征进行分析,以便为SNP基因型分析位点的确定提供参考[4]。
火炬松Pinustaeda材性偏软,具有粗糙的质感,适用于船舶楼宇及一般建筑。生长素(IAA)、细胞分裂素与赤霉素在树木生长过程中起到重要的调节作用。火炬松含有较丰富的松脂,可为制药和化工等行业提供原料。当松树受到甲虫危害时,能在伤口处产生组成型或诱导型的松脂[5],松脂是一种复杂的混合物,它包含单萜烯(松节油),二萜类树脂酸(松香)和较少量的倍半萜烯,而松节油的主要成分为蒎烯[6]。目前,关于松树SNP标记的研究较多,主要集中在生长[7]、材性[8-9]、代谢[10]、抗旱抗性[4,11]等方面,鲜见火炬松激素和蒎烯相关候选基因SNP标记研究的报道。本研究以生长和松脂产量为目标,从引自美国的火炬松基因资源试验林中选出36个单株为材料,并选择8个相应的候选基因片段进行研究。通过PCR扩增、纯化回收后连接于T载体上,对目的片段的SNP位点进行了筛选,分析了目标基因核苷酸多样性,连锁不平衡特征。研究结果为后续SNP标记与表型性状的关联分析提供参考。
1材料与方法
1.1植物材料
通过美国北卡罗莱纳州立大学林木改良工业协作组织引进的258个不同改良程度的火炬松家系,营造了基因资源林,以生长(树高和胸径)和松脂产量为目标性状,利用ASReml软件估算了258个家系的生长和松脂产量育种值,选取能够最大程度地反映基因资源变异水平的36个家系,每家系1株,共36个单株(36个基因型),采集嫩芽或1年生新鲜松针,-80 ℃冰箱中保存备用。
1.2基因组DNA提取
取出保存的松针或嫩芽,称取0.3 g,放入2 mL离心管中,加入小钢珠,利用Qiagen的MM301磨样仪将其磨碎,期间用液氮浸泡数次。基因组DNA的提取按照新型植物基因组DNA提取试剂盒(天根生化科技有限公司)描述的方法进行。
1.3引物设计和PCR扩增
从公共数据库(GenBank和http://pinetree.ccgb.umn.edu)下载火炬松cDNA文库的所有记录,约32万条,利用生物信息学方法进行序列的比对、剪接、组装、注释,获得非重复序列基因(Unigene)。选取与生长、松脂蒎烯合成相关的8个候选基因片段,采用Primer6和Oligo7软件设计和评估正反向PCR扩增引物[12]。
以选取的36个单株样本为材料,对8个候选基因片段进行PCR扩增。PCR反应体系为25 μL,含30~60 ng模板DNA,每种dNTP 0.2 mmol·L-1,正、反向引物各0.2 mmol·L-1,1×PCR Buffer,1 UTaq聚合酶和体积分数为1%的DMSO。采用PTC-200型PCR仪运行PCR反应,反应程序为:94 ℃预变性5 min后进入循环;每个循环94 ℃变性30 s,45~62 ℃退火30 s,72 ℃延伸60 s,反应共36个循环;72 ℃延伸7 min。
1.4PCR产物的克隆、测序及计算机分析
依据所选的8个候选基因片段的核苷酸序列设计基因特异的引物,以36个样本的总DNA为模板进行PCR扩增,将扩增得到的目的基因片段纯化回收后连接于pMD19-T载体上。连接产物转化大肠埃希菌EscherichiacoliDH5α,筛选阳性克隆,送华大基因公司测序。应用DNAMAN 6.0软件和CLUSTALX软件包推导出氨基酸序列并在NCBI进行检索分析,初步判断候选序列位于编码区还是非编码区。将去除载体后的序列采用BioEdit的ClustalW2程序进行联配分析,获得基因片段的一致序列,结合每条序列测序图,在BioEdit中确定SNP位点并做记录。
1.5基因的SNP多样性分析和连锁不平衡检测
利用MEGA5.0和DnaSP4.0软件对每个基因的36个序列进行比对分析,计算SNP频率、颠换和转换的SNP数量,估算各基因的核苷酸多样性水平[13]。分别用参数θw和πa表示核苷酸多样性水平,其中θw的估算以序列间每位点差异核苷酸的平均数为基础[14],πa的估算以分离位点的数量(S)为基础[15]。单倍型多样性(He)按照Nei[14]的公式计算。除以上定义外,核苷酸多样性的描述还包括:静默位点多样性(θwsil)、非同义突变多样性(πnsyn)和同义突变多样性(πsyn)。将所有位点依次对应的物理距离(bp)及其R2进行组合,利用非线性回归模型估算8个候选基因片段整体水平上的连锁不平衡(LD)延伸。
2结果与分析
2.1候选基因片段的序列分析
8个候选基因片段序列分析结果见表1。每一候选基因片段的扩增长度为431~1 007 bp(不包括插入或缺失位点),8个候选基因片段的测序总长度为5 177 bp,其中,编码区的序列长度为3 322 bp,非编码区的序列长度为1 855 bp(5′-UTR,3′-UTR 和内含子)。共有24个样本发生插入或缺失突变,总的插入片段长度为18 bp,缺失片段长度为80 bp。
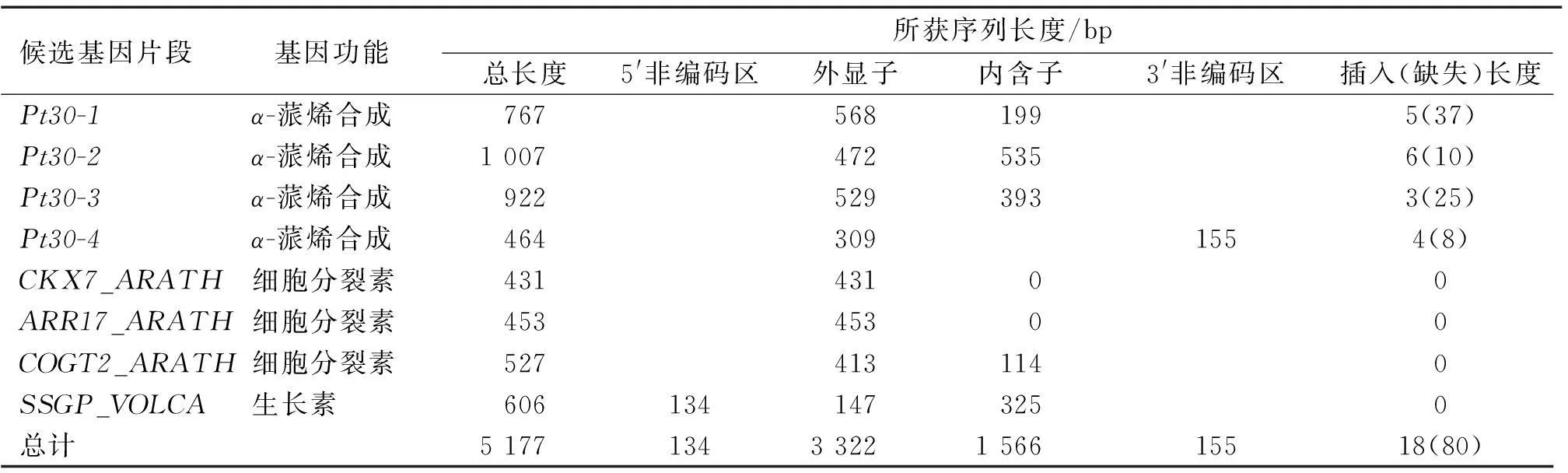
表1 火炬松生长和松脂性状候选基因片段序列分析
2.2SNP位点筛选
SNP位点筛选和分析结果见表2,对8个候选基因片段的序列进行整合后共检测到184个SNP位点,平均36.9 bp的基因序列中出现1个SNP位点。SSGP_VOLCA基因片段上检测了41个SNP位点,平均14.8 bp的基因序列中出现1个SNP位点,而在Pt30-2基因片段上检测了18个SNP位点,平均55.9 bp的基因序列中出现1个SNP位点。非同义突变的位点有123个,静默突变(同义突变)有61个SNP 位点。
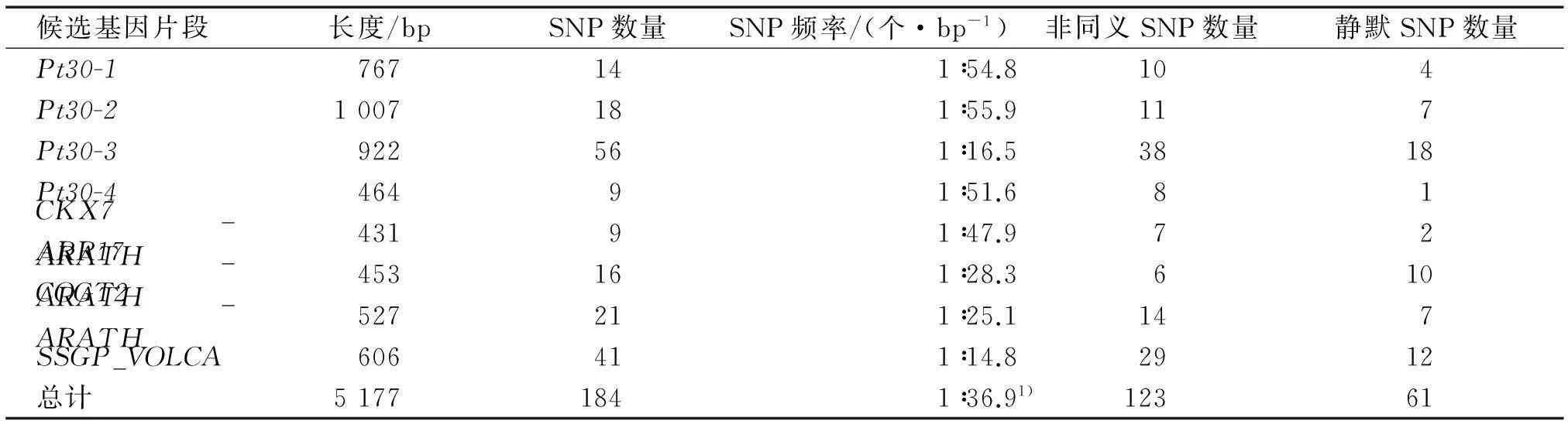
表2 火炬松生长和松脂候选基因片段序列核苷酸变异
1)平均值。
2.3核苷酸多样性
采用MEGA5.0和DnaSP4.0软件对各候选基因36个样本序列进行分析,计算8个候选基因的核苷酸多样性系数(表3)。8个候选基因平均核苷酸多态性πa和θw分别为0.020和0.016,处于较高水平,其中核苷酸多态性πa的变化范围介于0.006~0.034,核苷酸多态性θw的变化范围介于0.003~0.030。这比先前报道的相同树种其他性状或不同树种同类性状相关基因的核苷酸多态性高(表4)。有报道显示,火炬松的材性和生长性状相关基因的核苷酸多态性平均为0.005 9[4,8,16],是本研究的0.3倍。Pot等[17]报道,海岸松P.pinaster和辐射松P.radiata材性方面的核苷酸多态性是0.002 41和0.001 86,分别是本研究的0.12和0.09倍。核苷酸多态性的研究也在其他针叶树种中有所报道,例如樟子松P.sylvestrisvar.monglica[18-19],花旗松Pseudotsugamenziesii[9,16,20]和柳杉Cryptomeriajaponica[21]。
在8个候选基因片段中,具有最高核苷酸多态性πa和θw的基因片段分别是ARR17_ARATH(πa=0.034)和SSGP_VOLCA(θw=0.030),具有最低核苷酸多态性πa和θw的基因片段是Pt30-4和CKX7_ARATH(πa=0.006,θw=0.003)。静默位点的平均核苷酸多样性(θwsil)为0.022,该值与非同义突变位点多样性(πnsyn=0.014)基本相同。本研究与同一树种的其他性状如抗旱性、材性和抗性的候选基因相比,核苷酸多样性θw高出其他相关研究3.2和4.0倍[4,8]。基因片段COGT2_ARATH的非同义突变多样性(πnsyn)是基因片段CKX7_ARATH的14.5倍,非同义突变位点多样性在0.029~0.002之间变化(表3),这表明基因间在适应性进化历史中变化较大[22]。基因片段Pt30-2、Pt30-4和CKX7_ARATH的非同义分歧很低,即0.002~0.004,表明这些基因具有很强的保守核苷酸序列。8个候选基因片段中的2个基因片段(Pt30-1和Pt30-4)的Ka/Ks比值大于1,说明这些基因受正向选择的影响较大。其余基因片段的Ka/Ks比值都小于1,说明他们受负向选择的影响较大,核苷酸序列相对保守。

表3 火炬松生长和松脂性状候选基因片段核苷酸多样性分析1)
1)S为分离位点的数量,θw、πa为总核苷酸多样性,πsyn、πnsyn分别为同义和非同义突变多样性,θwsil为静默位点多样性,Ks为编码区同义突变平均数,Ka为编码区非同义突变平均数,Nh为单倍型个数,He为单倍型多样性,Rm为历史重组事件最小数。
单倍型的平均数(Nh)和单倍型多样性(He)平均值分别为11.25和0.909。在8个候选基因片段中发现核苷酸数和核苷酸多样性有较大的变化,其中单倍型个数的变化范围介于6~21,单倍型多样性变化范围介于0.625~1.000。采用四配子检测进行最小历史重组事件(Rm)估算,结果表明8个候选基因片段的Rm为1~7。
2.4连锁不平衡和重组
将8个候选基因片段的位点间连锁不平衡数据(R2)汇总后进行综合分析,期望从总体上了解火炬松基因组中连锁不平衡情况。对R2值的非线性回归分析结果见图1,随着候选基因片段核苷酸序列长度的增加,SNPs的连锁不平衡程度迅速削弱,当长度达400 bp左右时,R2<0.2,连锁不平衡迅速衰退。这一结果表明,在火炬松中,SNP的连锁不平衡在候选基因片段内部就已衰退。这与其他的针叶树种的报道一致[8-9]。连锁不平衡衰退率在不同的植物中的变化较大,玉米Zeamays的候选基因片段长度为1 500 bp,当在200 bp左右时,R2降低到0.2以下;拟南芥Arabidopsisthaliana的候选基因片段长度为100 000 bp,当在50 000 bp左右时,R2降低到0.2以下[25]。

表4 不同性状的候选基因核苷酸多样性在其他林木中的应用1)
1)θw为总核苷酸多态性,πnsyn和πsyn分别为非同义和同义突变多样性,He为单倍型多样性。
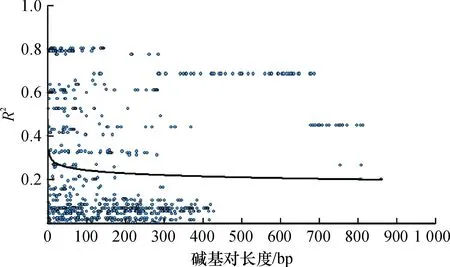
图18个候选基因片段总体连锁不平衡分析的R2随碱基对长度衰减情况
Fig.1Scatter plot of distances in base pairs and linkage disequilibrium estimates (R2) between SNP sites in eight candidate genes in Pinus teada
3讨论与结论
火炬松是南方商品林的主要树种之一,适宜在粤北和粤东北低山种植。火炬松的遗传改良时间较长,目前正处在第2代向第3代过渡阶段。在火炬松第1、2代育种过程中,生长量获得显著提高,但忽略了对产脂量的选择。为了适应林业生产对松树多用途的需求,本研究以高产脂和速生为目标,选择了8个候选基因片段,其中4个基因片段与松脂合成有关,4个基因片段与火炬松生长(包括生长素和细胞分裂素)有关,将8个候选基因片段进行PCR扩增测序,共得到5 177 bp的基因片段序列,其中编码区的序列长度为3 322 bp,非编码区的序列长度为1 855 bp。非同义突变SNP位点(123)多于静默SNP位点(61)。8个候选基因片段的SNP筛选分析研究表明,编码区的SNP数量高于非编码区,与其他松树的SNP分析结果一致,表明火炬松的编码区比其他部位具有更高的保守性。但是不同候选基因的 SNP 频率不一样,说明各个基因的保守性不同,可能是由于在进化过程中受到的选择压力不同所致。
8个候选基因片段的平均核苷酸多样性πa和θw分别为0.020和0.016,与先前报道的核苷酸多样性相比,本研究8个候选基因片段的核苷酸多样性较高,这可能是因为材料具独立的变迁历史和地理分布,不同的遗传基础造成了核苷酸多样性的差异,今后应更大范围取样以便更全面地了解该树种的遗传多样性水平。另一部分原因可能是本研究所用的材料为针叶部位,这个部位的基因组比大配子体复杂。8个候选基因片段的单倍型个数(Nh)和单倍型多样性(He)平均值分别为11.25和0.909,单倍型多样性的平均标准误为0.065,Rm为1~7。本文检测到了较高程度的核苷酸多样性,表明利用SNP标记对火炬松进行分子标记辅助选择(MAS)需要较多的SNP 标记。
本研究发现,随着LD衰退距离的增加,总体趋势基本平稳,但火炬松8个候选基因片段总体的LD水平稍高,这可能是由于所选样品数量较少。Kim等[26]提出当个体遗传背景差异较小时,较大的样本量一般具有较低的LD水平。大量研究表明林木具有较低的LD水平,适宜基于候选基因片段的连锁不平衡作图研究。在杨树中,基于SNP的连锁不平衡检测发现,杨属Populus近缘种的候选基因片段内LD衰退距离为300~1 700 bp不等[22,27-29]。Thumma等[30-31]发现亮果桉EucalyptusnitensLD水平在基因CCR和COBRA-like内已衰退至不明显。在针叶树种中,基于SNP标记的LD水平在几千个碱基长度内已衰退至不明显[8-10,32],这些研究结果与本研究的LD检测基本一致,揭示了基于候选基因片段的关联遗传研究是可行的。本研究进行的连锁不平衡分析仅限于候选基因片段内部,火炬松基因间隔区域、不同染色体及全基因组水平的LD需要更多的标记去验证。随着火炬松全基因组测序、拼接、注释完成及重测序研究的开展,该物种基因组水平的LD衰退趋势将会得到更深入全面的检测。
参考文献:
[1]黄少伟,钟伟华,陈炳铨.火炬松半同胞子代配合选择的遗传增益估算[J].林业科学,2006,42(4):33-37.
[2]汤在祥,陈志军,王学枫,等.标记辅助选择育种中QTL基因型的多点联合推断[J].分子植物育种,2006,4(2):293-298.
[3]SIMKO I, HAYNES K G, JONES R W. Assessment of linkage disequilibrium in potato genome with single nucleotide polymorphism markers[J]. Genetics, 2006,173(4): 2237-2245.
[4]GONZA′LEZ-MARTI′NEZ S C, ERSON E, BROWN G R, et al. DNA sequence variation and selection of tag single-nucleotide polymorphisms at candidate genes for drought-stress response inPinustaedaL[J]. Genetics, 2006, 172(3):1915-1926.
[5]ROBERDS J H, STROM B L, HAIN F P, et al. Estimates of genetic parameters for oleoresin and growth traits in juvenile loblolly pine[J]. Can J Forest Res, 2003, 33(12): 2469-2476.
[6]PHILLIPSM A, WILDUNG M R, WILIAMS D C, et al. cDNA isolation, functional expression, and characterization of (+) -α-pinene synthase from loblolly pine (Pinustaeda): Stereocontrol in pinene biosynthesis [J]. Arch Biochem Biophys, 2003, 411(2): 267-276.
[7]CUMBIEL W P, ECKERT A, WEGRZYN J, et al. Association genetics of carbon isotope discrimination, height and foliar nitrogenin a natural population ofPinustaedaL.[J].Heredity, 2011, 107(1):105-114.
[8]BROWN G R, GILL G P, KUNTZ R J, et al. Nucleotide diversity and linkage disequilibrium in loblolly pine[J]. Proc Natl Acad Sci USA,2004, 101(42): 15255-15260.
[9]KRUTOVSKY K V, NEALE D B. Nucleotide diversity and linkage disequilibrium in cold-hardiness and wood quality-related candidate genes inDouglasfir[J].Genetics,2005, 171(4):2029-2041.
[10]GONZA′LEZ-MARTNEZ S C, WHEELER N C, ERSOZ E, et al. Association genetics inPinustaedaL.:I: Wood property traits[J]. Tree Genet Genom, 2007, 175(1):399-409.
[11]ECKERTA J,WEGRZYN J L, CUMBIE W P, et al. Association genetics of the loblolly pine (Pinustaeda, Pinaceae)metabolome[J]. New Phytol, 2012, 193(4): 890-902.
[12]杨会肖.火炬松基因资源的遗传分析及SNP位点筛选[D].广州:华南农业大学,2013.
[13]ROZAS J, SA′NCHEZ-DEL-BARRIO J C, MESSEGUER X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics, 2003, 19(18): 2496-2497.
[14]NEI M. Molecular evolutionary genetics[M]. New York:Columbia University Press, 1987.
[15]WATTERSON G A. On the number of segregating sites in genetical models without recombination[J]. Theor Popul Biol, 1975, 7(2): 256-276.
[16]NEALE D B, SAVOLAINEN O. Association genetics of complex traits in conifers[J]. Trends Plant Sci, 2004, 9(7): 325-330.
[17]POT D, MCMILLAN L, ECHT C, et al. Nucleotide variation in genes involved in wood formation in two pine species[J]. New Phytol, 2005,167(1): 101-112.
[19]DVORNYK V, SIRVIÖ A, MIKKONEN M, et al. Low nucleotide diversity at the pal1 locus in the widely distributedPinussylvestris[J]. Mol Biol Evol, 2002, 19(2): 179-188.
[20]ECKERT A J, BOWER A D, WEGRZYN J L, et al. Association genetics of coastal douglas fir (Pseudotsugamenziesiivar.menziesii, Pinaceae) :I: Cold-hardiness related traits[J]. Genetics, 2009, 182(4): 1289-1302.
[21]KADO T, MATSUMOTO A, UJINO-IHARA T, et al. Amounts and patterns of nucleotide variation within and between two Japanese conifers, sugi (Cryptomeriajaponica) and hinoki (Chamaecyparisobtusa) (Cupressaceaesensulato)[J]. Tree Genet Genom, 2008, 4(1): 133-141.
[22]INGVARSSON P K. Nucleotide polymorphism and linkage disequilibrium within and among natural populations of European aspen (PopulustremulaL.,Salicaceae)[J]. Genetics,2005,169(2): 945-953.
[23]褚延广. 欧洲黑杨(PopulusnigraL.)水、光资源高效利用相关单核苷酸多态性(SNP)研究 [D]. 北京:中国林业科学研究院, 2009.
[24]KUSUMI J, LI Z D, KADO T, et al. Multilocus patterns of nucleotide polymorphism and demographic change inTaxodiumdistichum(Cupressaceae) in the lower Mississippi River alluvial valley[J]. Am J Bot, 2010, 97(11): 1848-1857.
[25]RAFALSKI A, MORGANTE M. Corn and humans: Recombination and linkage disequilibrium in two genomes of similar size[J]. Trends Genet, 2004, 20(2): 103-111.
[26]KIM S, PLAGNOL V,HU T T, et al. Recombination and linkage disequilibrium inArabidopsisthaliana[J]. Nat Genet, 2007, 39(8): 1151-1155.
[27]TNGVARSSON P K, GARCIA M V, LUQUEZ V, et al. Nucleotide polymorphism and phenotypic associations within and around the phytochrome B2 locus in European aspen (Populustremiila, Salicaceae) [J]. Genetics, 2008, 178(4): 2217-2226.
[28]WEGRZYN J L, ECKERT A J, CHOI M, et al. Association genetics of traits controlling lignin and cellulose biosynthesis in black cottonwood (Populustrichocarpa,Saiicaceae) secondary xylem [J]. New Phytol, 2010, 188(2): 515-532.
[29]XU B, YANG X,LI B, et al. Isolation, expression and single nucleotide polymorphisms analysis of cellulose synthase gene (PtCesA4) fromPopulustomentosa[J]. Scientia Silvae Sinicae, 2009,45(5): 1-10.
[30]THUMMA B R, MATHESON B A,ZHANG D Q, et al. Identification of aCis-acting regulatory polymorphism in a eucalyptCOBRA-like gene affecting cellulose content [J]. Genetics, 2009,183(3):1153-1164.
[31]THUMMA B R, NOLAN M F, EVANS R, et al. Polymorphisms in cinnamoyl CoA reductase (CCR) are associated with variation in micro fibril angle inEucalyptusspp. [J]. Genetics, 2005, 171(3): 1257-1265.
[32]DVOMYK V,SIRVIO A,MIKKONENE M,et al. Low nucleotide diversity at two phytochrome loci along a latitudinal dine inPinussylvestris[J]. Mol Biol Evol, 2002, 19:179-199.
【责任编辑李晓卉】
Analysis of single nucleotide polymorphisms within candidate genes
involved in growth and resin biosynthesis of loblolly pine
YANG Huixiao1,2, LIU Tianyi2, XU Bin1, LIU Chunxin2, WANG Jinbang3, HUANG Shaowei2
(1 Guangdong Academy of Forestry, Guangzhou 510520, China; 2 Guangdong Key Laboratory for Innovative Development and
Utilization of Forest Plant Germplasm/College of Forestry and Landscape Architecture, South China
Agricultural University, Guangzhou 510642, China; 3 Yingde Institute of Forestry, Yingde 513000, China)
Abstract:【Objective】 Single nucleotide polymorphisms (SNPs) within candidate genes involved in growth and resin biosynthesis of loblolly pine,Pinustaeda, were screened and analyzed, preparing technical basis for later marker-assisted breeding.【Method】The candidate unigenes related to auxin, gibberellins, cytokinins or pinene synthesis were selected from loblolly pine cDNA library using bioinformatics. The sequences of eight candidate genes from 36 loblolly pine individuals were aligned and compared using MEGA5.0 and DnaSP4.0 softwares.【Result】 The total length of measured sequences was 5 177 bp. There were 184 SNPs to be detected, and in average there was one SNP site in every 36.9 bp sequence. Among them, 123 were nonsynonymous SNPs and 61 were silent SNPs. The levels of nucleotide diversity for sequenced regions represented byπaandθwwere 0.020 and 0.016, respectively. The linkage disequilibrium(LD) of SNPs in the eight candidate genes declined rapidly (R2≤0.2) with the nucleotide sequences increased in length. 【Conclusion】 LD mapping of SNPs in the five candidate genes are useful for loblolly pine breeding.
Key words:loblolly pine; candidate gene; single nucleotide polymorphism (SNP); linkage disequilibrium (LD)
中图分类号:S722.3
文献标志码:A
文章编号:1001-411X(2016)01-0075-07
基金项目:广东省林业科技创新专项资金(2011KJCX013-02);“十二五”农村领域国家科技计划专题(2012BAD01B0203);联合国发展计划署援建的火炬松改良种子园项目(CPR/91/153,1992-1996)
作者简介:杨会肖(1981—),女,助理研究员,博士研究生,E-mail: hxyang@sinogaf.cn;通信作者:黄少伟(1964—),男,教授,博士,E-mail: shwhuang@scau.edu.cn
收稿日期:2015-04-07优先出版时间:2015-12-07
优先出版网址:http://www.cnki.net/kcms/detail/44.1110.s.20151207.1133.028.html
杨会肖, 刘天颐, 徐斌,等.火炬松生长和松脂性状相关候选基因的单核苷酸多样性分析[J].华南农业大学学报,2016,37(1):75-81.
