高邮鸭群体 MSTN、MyoD1和 MyoG基因外显子中SNP位点的分析
2016-01-15刘宏祥,徐文娟,宋卫涛等
高邮鸭群体MSTN、MyoD1和MyoG基因外显子中SNP位点的分析
刘宏祥,徐文娟,宋卫涛,朱文奇,胡艳,姬改革,李慧芳
(江苏省家禽科学研究所,江苏扬州225125)

摘要:MSTN、MyoD1和MyoG基因与禽类骨骼肌的生长发育密切相关。本试验以高邮鸭为素材,对MSTN、MyoD1和MyoG 3个基因的外显子单核苷酸多态性(SNP)位点进行了鉴定,并对这些位点对蛋白质的影响及群体遗传信息进行了分析。结果显示,MSTN、MyoD1和MyoG 3个基因的外显子分别有9个、4个和7个SNP位点,其中具有中度多态的位点分别为8个、1个和5个,有义突变各1个,这些有义突变均改变了相应蛋白质的理化性质以及二级结构。
关键词:MSTN;MyoD1;MyoG;外显子;单核苷酸多态性;鸭
doi:10.3969/j.issn.1000-4440.2015.03.023
收稿日期:2014-12-30
基金项目:国家自然科学基金项目(31172194);江苏省科技支撑计划项目(BE2011329、BE2012460);现代农业品种创新项目[CX(11)1030]
作者简介:刘宏祥(1985-),男,江苏扬州人,硕士,主要从事家禽遗传育种与资源保护研究。(Tel)13665203762;(E-mail)liuhongxiangzai@163.com
中图分类号:S834`+.89
文献标识码:A
文章编号:1000-4440(2015)03-0604-09
Abstract:There are strong relations between MSTN, MyoD1, MyoG genes and development of skeletal muscle. The single nucleotide polymorphisms (SNPs) in the exons of MSTN, MyoD1 and MyoG genes of Gaoyou duck were detected, and their influences on proteins and population genetics information were studied. There were nine, four SNPs and seven SNPs in the exons of MSTN, MyoD1 and MyoG genes, respectively, among which, eight, one and five SNPs were moderate polymorphic. One sense mutation detected in each gene changed the protein physico-chemical characteristics and second structure.
Analysis of single nucleotide polymorphisms (SNPs) in exons ofMSTN, MyoD1 andMyoGgenes of Gaoyou duck
LIU Hong-xiang,XU Wen-juan,SONG Wei-tao,ZHU Wen-qi,HU Yan,JI Gai-ge,LI Hui-fang
(JiangsuInstituteofPoultryScience,Yangzhou225125,China)
Key words:MSTN;MyoD1;MyoG;exon;single nucleotide polymorphism (SNP);duck
动物机体的生长是一个复杂的生理过程,对肌肉生长具有调节作用的因子很多,其中重要的有MRFs(Muscle regulation factors)家族的MyoD1基因、MyoG基因,它们对肌肉生长具有正向调节功能;而TGFβ家族中的MSTN基因对肌肉生长具有负调控作用,其功能的缺失在哺乳动物中能够引起肌肉的显著增大[2-4]。
具有编码蛋白质功能的基因在DNA水平上的碱基序列变化将传递给蛋白质,而蛋白质序列的改变将影响蛋白质二级结构以及高级结构,甚至改变蛋白质的功能。基因组水平上的单个核苷酸的变异(SNP)在染色体上数量多、分布广,而且能够稳定遗传。因此在动物遗传育种中,对功能基因,尤其直接编码蛋白质的外显子部分的SNP研究具有重要意义。
已有多个研究报道了哺乳动物(比如牛[3, 5]、小鼠、绵羊[7-8]、狗)MSTN基因外显子的错义突变导致了机体肌肉的显著增长。张跟喜等[10]在2011年对边鸡的MSTN基因外显子1进行了SNP筛查,发现一个对边鸡生长有显著效应的SNP位点。
高邮鸭属于肉蛋兼用型地方品种,经过多年选育,目前高邮鸭群体性能已基本稳定。本试验运用单重PCR方法对高邮鸭群体的MSTN、MyoD1和MyoG3个基因的外显子分别进行了SNP筛查,并分析筛查到有义突变导致氨基酸变化以及对蛋白质二级结构的影响,为高邮鸭群体后期的分群以及开发利用提供理论依据。
1材料与方法
1.1 试验动物
高邮鸭试验群体来源于江苏省高邮鸭集团,性成熟时从群体中随机选择30个体质量差异较小的个体进行试验。
1.2 主要仪器和试剂
主要仪器有:PCR仪(型号:Gene Amp PCR,厂家:Norwalk,CT.06859 USA)、电泳仪(型号:JY600+,厂家:北京君意东方电泳设备有限公司)、全自动紫外与可见分析装置(型号:FR-200A,厂家:上海复日科技有限公司)、生物电泳图像分析系统(厂家:上海复日科技有限公司)。
主要试剂有:PCR引物(由上海瀚宇生物工程有限公司合成,PAGE级纯化)、PCR反应试剂(上海有渔生物工程有限公司提供)。
1.3 DNA的提取
对30个高邮鸭个体翅静脉采血,用酚-氯仿法抽提DNA并溶于超纯水中。将DNA原液 1∶100稀释并进行分光光度计检测,OD值为 1.85~1.94。
1.4 引物设计
分别在MSTN基因3个外显子、MyoD1基因4个外显子、MyoG基因5个外显子区域附近设计合适的引物,其中MSTN、MyoD1和MyoG基因分别设计了4、4和3对引物。具体信息见表 1。
表1针对MSTN、MyoD1和MyoG基因外显子区域设计的引物
Table 1The primers for the exons ofMSTN, MyoD1 andMyoGgenes

基因外显子引物名称 引物序列(5'→3')退火温度(℃)产物长度(bp)MSTN外显子1P1-1F:CCTTGGAATATATAAGGTACAC-CAG56600R:CCTGAGCAGGAGTTGTGTG外显子2P1-2F:TTTGCCGTAGAGCGTAAAGC56691R:GCAAAGAGCGAAAGAATTGG外显子3P1-3F:TGATTTGATAGGTTTGTGT-CAGAGA56816R:TGTTGAAACCTGCGTATGGAP1-4F:TGTCAACCAGTGGGAAAAGA56828R:AACATCATTCTTTTCTTGTGAT-GAAMyoD1外显子1P2-1F:CATCGTATTCCCCATTTGACA56385R:AACAGTGACTCCTCCCTAACCA外显子2P2-2F:GGGTCTGTAGGTTGGGAATG59838R:GAGGACGTACGCCAGCAG外显子3P2-3F:GGAGGGGAGGCAAGTTAAGA56474R:GCTGTAACGCATCCCCATAC外显子4P2-4F:TGTCATAATTGGCATCCTTCA56922R:GCACAACAAACCAAGCAACAMyoG外显子1P3-1F:GATGCGAGGAAGCAGCTTAG62973R:GGGAGGGGCACACTTTTATT外显子2、3P3-2F:ATGGCAGTGCCCAGCTAC56454R:AGCTGAGCTGCCAAACCTC外显子4、5P3-3F:TGGGCATTGTGCTGCTGTGA591084R:CGCCGGAGGGATAAATAGGA
1.5 PCR产物扩增与测序
PCR反应体系为20 μl,其中包括:1.0 μl模板(50 ng)、2.0 μl Buffer缓冲液(1×)、0.6 μl镁离子(2 mmol/L)、2.0 μl dNTP(2 mmol/L)、0.2 μlTaq酶(1 U)、2.0 μl引物混合液(0.5 pmol/L),余下用H2O补足。PCR反应条件为:95 ℃预变性2 min;94 ℃ 30 s,按照表 1中的退火温度退火1 min 30 s,72 ℃延伸30 s,共40个循环;最后72 ℃后延伸10 min。PCR产物用3%的琼脂糖凝胶电泳进行检测扩增效果。最后对产物进行纯化,并送由上海翼和应用生物技术有限公司测序。
1.6 统计分析
1.6.3Hardy-Weinberg平衡检验假设某群体的某一位点等位基因有A、B两种,其等位基因频率分别为p、q。该群体基因型有AA、AB、BB 3种,基因型频率分别记为D、H和R。根据观测到的基因型频率,可以计算出等位基因频率:
p=D+H/2,q=R+H/2
根据等位基因频率,可以计算出理论的基因型频率:
D′=p2,H′=2pq,R′=q2
根据样本含量、观测基因型频率和理论基因型频率,可以分别计算出观测基因型频数和理论基因型频数。通过卡方检验:
χ2=(D-D′)2/D′+(H-H′)2/H′+(R-R′)2/R′
计算出卡方值。如果卡方值对应的概率大于0.05,那么可以认为该等位基因处于Hardy-Weinberg平衡状态。
1.6.4蛋白质理化性质分析利用ExPASy网站中的ProtParam服务器(http://web.expasy.org/cgi-bin/protparam/)在线计算MSTN、MyoD1和MyoG蛋白质的理论等电点、分子量、正负电位氨基酸数目、不稳定系数、蛋白质亲水性及脂肪族氨基酸指数等理化性质。
1.6.5蛋白质二级结构的预测蛋白质二级结构是一级结构和三级结构之间的纽带,也是通过一级结构预测三级结构的关键步骤[11]。对MSTN、MyoD1和MyoG基因的外显子突变情况进行了筛查,发现3个基因各有1个有义突变导致了氨基酸的变化。SSPro 4.0(http://scratch.proteomics.ics.uci.edu)可以使用神经网络与同源分析混合进行蛋白质二级结构预测[12],预测的结果较为准确可靠,因此本试验使用该服务器作为蛋白质二级结构预测的工具。
2结果与分析
2.1 MSTN、MyoD1和 MyoG 3个基因外显子突变位点分析
扩增产物经测序、对比,MSTN外显子中发现9个SNP位点,编号为SNP1-1~SNP1-9,具体信息见表2。SNP1-1~SNP1-5位于CDS区,其中SNP1-1导致了甘氨酸(G)到丝氨酸(S)的变化,其余为沉默突变。SNP1-6~SNP1-9位于非CDS区。
MyoD1外显子中共发现4个SNP位点,编号为SNP2-1~SNP2-4。这4个SNP位点均处于CDS区,其中SNP2-1导致了蛋氨酸(M)向亮氨酸(L)的转变,其他3个SNP位点均为沉默突变。
MyoG外显子中共发现了7个SNP位点,编号为SNP3-1~SNP3-7。这7个SNP位点均处于CDS区,其中SNP3-7为有义突变,导致谷氨酸(E)到天冬氨酸(D)的变化,其余均为沉默突变。
2.2 MSTN、MyoD1和 MyoG 3个基因外显子的遗传多样性
对30个高邮鸭个体的MSTN、MyoD1和MyoG基因的基因型频率以及基因频率分别进行了统计,统计结果分别见表3、表4和表5。
根据各个SNP位点的基因型分布情况,对各个位点进行了Hardy-Weinberg平衡检验,结果见表6。MSTN基因外显子所有突变位点均处于Hardy-Weinberg平衡状态;MyoD1基因突变位点SNP2-2、SNP2-3和SNP2-4均不处于Hardy-Weinberg平衡状态;MyoG基因突变位点SNP3-2、SNP3-3和SNP3-5均不处于Hardy-Weinberg平衡状态。
表2MSTN、MyoD1和MyoG基因突变位点
Table 2SNPs ofMSTN, MyoD1 andMyoGgenes
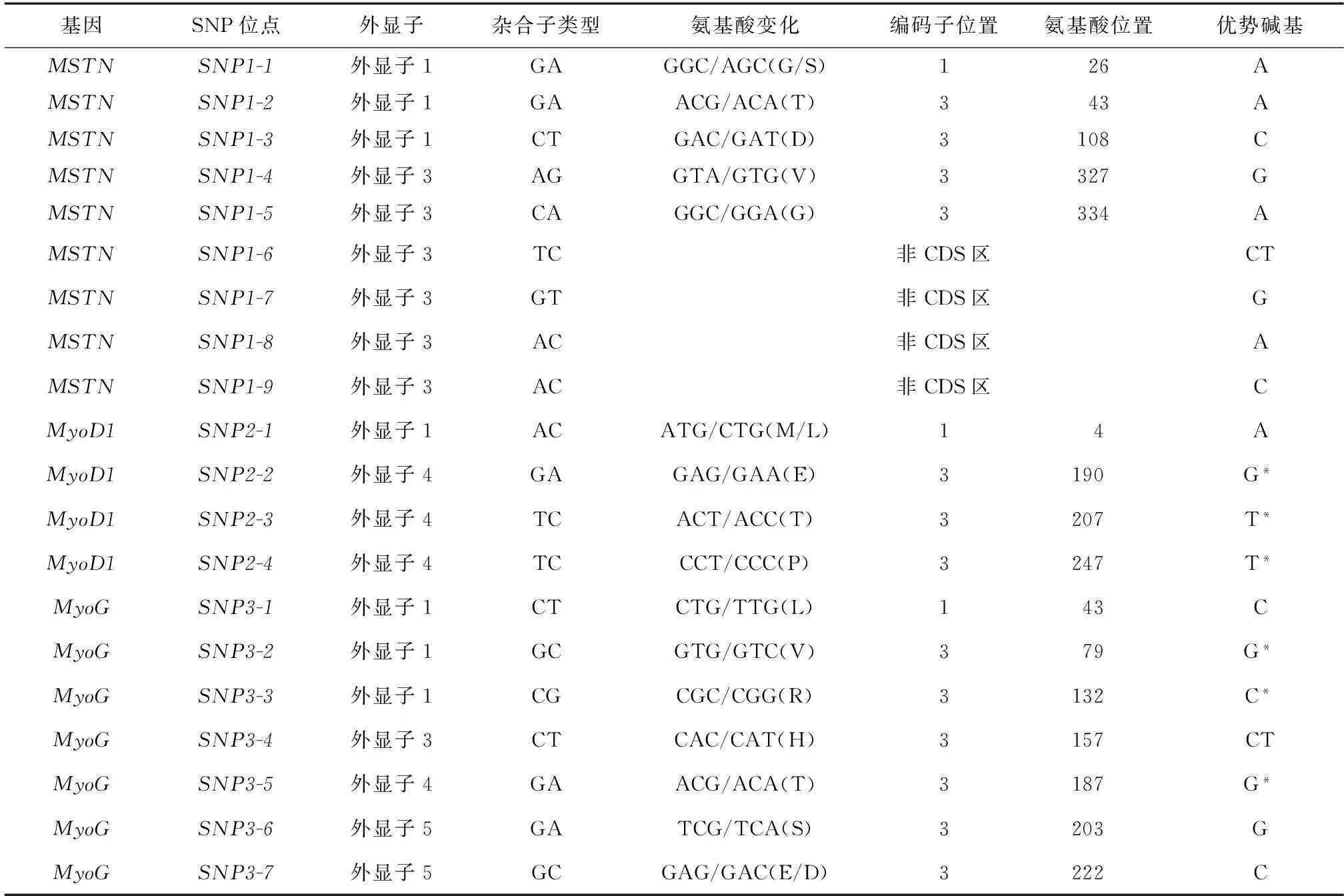
基因SNP位点外显子杂合子类型氨基酸变化编码子位置氨基酸位置优势碱基MSTNSNP1-1外显子1GAGGC/AGC(G/S)126AMSTNSNP1-2外显子1GAACG/ACA(T)343AMSTNSNP1-3外显子1CTGAC/GAT(D)3108CMSTNSNP1-4外显子3AGGTA/GTG(V)3327GMSTNSNP1-5外显子3CAGGC/GGA(G)3334AMSTNSNP1-6外显子3TC 非CDS区CTMSTNSNP1-7外显子3GT 非CDS区GMSTNSNP1-8外显子3AC 非CDS区AMSTNSNP1-9外显子3AC 非CDS区CMyoD1SNP2-1外显子1ACATG/CTG(M/L)14AMyoD1SNP2-2外显子4GAGAG/GAA(E)3190G*MyoD1SNP2-3外显子4TCACT/ACC(T)3207T*MyoD1SNP2-4外显子4TCCCT/CCC(P)3247T*MyoGSNP3-1外显子1CTCTG/TTG(L)143CMyoGSNP3-2外显子1GCGTG/GTC(V)379G*MyoGSNP3-3外显子1CGCGC/CGG(R)3132C*MyoGSNP3-4外显子3CTCAC/CAT(H)3157CTMyoGSNP3-5外显子4GAACG/ACA(T)3187G*MyoGSNP3-6外显子5GATCG/TCA(S)3203GMyoGSNP3-7外显子5GCGAG/GAC(E/D)3222C
氨基酸变化栏中括号前的字母表示三联密码子的变化情况,括号中的字母表示氨基酸的变化情况;编码子位置栏中数字表示三联密码子中发生突变的碱基在密码子中的位置,非CDS区表示该突变不在CDS区中;优势碱基表示在突变位点的2个等位碱基在群体中出现频率较大的碱基;*表示该位点的2个碱基在群体中的分布不符合Hardy-Weinberg平衡。
表3MSTN基因SNP位点的群体信息
Table 3The population information about SNPs ofMSTNgene

SNP位点等位基因型频率等位基因频率SNP1-1AAAGGGAG0.63(17)0.33(9)0.04(1)0.800.20SNP1-2AAAGGGAG0.67(18)0.30(8)0.04(1)0.810.19SNP1-3CCCTTTCT0.96(26)0.04(1)0(0)0.980.02SNP1-4AAAGGGAG0.14(4)0.43(12)0.43(12)0.360.64SNP1-5AAACCCAC0.43(12)0.43(12)0.14(4)0.640.36SNP1-6CCCTTTCT0.29(8)0.43(12)0.29(8)0.500.50SNP1-7GGGTTTGT0.46(13)0.43(12)0.11(3)0.680.32SNP1-8AAACCCAC0.46(13)0.43(12)0.11(3)0.680.32SNP1-9AAACCCAC0.21(6)0.46(13)0.32(9)0.450.55
括号内数字表示该等位基因型在检测群体中的观测个数。粗体表示有义突变。
表4MyoD1基因SNP位点的群体信息
Table 4The population information about SNPs of MyoD1 gene

SNP位点等位基因型频率等位基因频率SNP2-1AAACCCAC0.90(27)0.10(3)0(0)0.950.05SNP2-2AAAGGGAG0.29(7)0.04(1)0.67(16)0.310.69SNP2-3CCCTTTCT0.08(2)0.13(3)0.79(19)0.150.85SNP2-4CCCTTTCT0.13(3)0(0)0.88(21)0.130.88
括号内数字表示该基因型在检测群体中的观测个数。粗体表示有义突变。
表5MyoG基因SNP位点的群体信息
Table 5The population information about SNPs ofMyoGgene

SNP位点等位基因型频率等位基因频率SNP3-1CCCTTTCT0.86(25)0.14(4)0(0)0.930.07SNP3-2CCGCGGCG0.10(3)0.34(10)0.55(16)0.280.72SNP3-3CCGCGGCG0.45(13)0.34(10)0.21(6)0.620.38SNP3-4CCCTTTCT0.33(7)0.33(7)0.33(7)0.500.50SNP3-5GGAGAAGA0.87(13)0.13(2)0(0)0.930.07SNP3-6AAAGGGAG0.07(1)0.40(6)0.53(8)0.270.73SNP3-7CCGCGGCG0.40(6)0.27(4)0.33(5)0.530.47
括号内数字表示该等位基因型在检测群体中的观测个数。粗体表示有义突变。
根据等位基因频率计算出群体各个基因突变位点的多态信息含量(PIC)以及有效等位基因数(表 6)。PIC值介于0.25和0.50之间为中度多态,可知MSTN基因外显子除了SNP1-3外,其他位点均为中度多态;MyoD1基因第4外显子的1个突变位点SNP2-2为中度多态,MyoG基因突变位点SNP3-2、SNP3-3、SNP3-4、SNP3-6和SNP3-7为中度多态;所有其他突变位点均为低度多态。对于有效等位基因数,MSTN基因SNP1-3位点,MyoD1基因SNP2-1位点、SNP2-3位点和SNP2-4位点,MyoG基因SNP3-1位点和SNP3-5位点有效等位基因数均不超过1.4,与理论等位基因数2.0有较大差异,即这些位点遗传变异较小。有效等位基因数的分析结果与多态信息含量的结果相似。
表6MSTN、MyoD1和MyoG3个基因的遗传多样性
Table 6Genetic diversity ofMSTN, MyoD1 andMyoGgenes

基因SNP位点多态信息含量(PIC)有效等位基因数(Ne)哈代-温伯平衡(Hardy-Weinberg)MSTNSNP1-10.27181.48020.99MSTNSNP1-20.25621.43221.00MSTNSNP1-30.03571.03771.00MSTNSNP1-40.35381.84910.94MSTNSNP1-50.35381.84910.94MSTNSNP1-60.37502.00000.75MSTNSNP1-70.34111.77381.00MSTNSNP1-80.34111.77381.00MSTNSNP1-90.37211.97730.95MyoD1SNP2-10.09051.10500.96MyoD1SNP2-20.33741.75340*MyoD1SNP2-30.21811.33180.05*MyoD1SNP2-40.19481.28000*MyoGSNP3-10.12021.14730.92MyoGSNP3-20.31971.66530*MyoGSNP3-30.36001.88990*MyoGSNP3-40.37502.00000.31MyoGSNP3-50.11671.14210*MyoGSNP3-60.31461.64231.00MyoGSNP3-70.37391.99120.13
粗体表示有义突变。
2.3 突变导致的蛋白质理化性质的改变
MSTN、MyoD1和MyoG野生型蛋白质及突变型蛋白质的理化性质分析结果见表7。MSTN突变型蛋白质与野生型蛋白质相比,除了分子量变化之外,其不稳定系数变大(由44.80变为45.83),亲水性变强(疏水性指数由-0.405变为-0.406),其他如理论等电点、正负电位点数以及脂肪族系数均没有变化。MyoD1突变型蛋白质与野生型蛋白质相比,分子量变小,亲水性变弱(疏水性指数由-0.917变为-0.910),脂肪族系数变大。MyoG突变型蛋白质与野生型蛋白质相比,除了分子量变小之外,其理论等电点与不稳定系数均变小,疏水性没有变化。
表7蛋白质理化性质
Table 7Protein physico-chemical properties
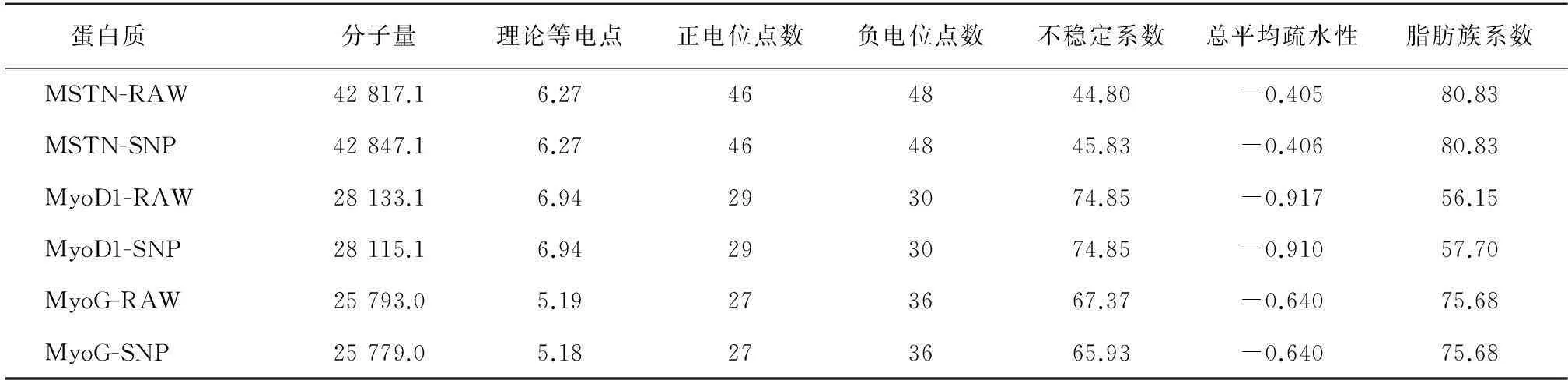
蛋白质分子量理论等电点正电位点数负电位点数不稳定系数总平均疏水性脂肪族系数MSTN-RAW42817.16.27464844.80-0.40580.83MSTN-SNP42847.16.27464845.83-0.40680.83MyoD1-RAW28133.16.94293074.85-0.91756.15MyoD1-SNP28115.16.94293074.85-0.91057.70MyoG-RAW25793.05.19273667.37-0.64075.68MyoG-SNP25779.05.18273665.93-0.64075.68
RAW表示野生型,SNP表示突变型。
2.4 突变导致蛋白质二级结构的变化
2.4.1MSTN蛋白质二级结构的变化MSTN基因外显子1中筛查到的有义突变导致了第26位氨基酸由甘氨酸到丝氨酸的变化。通过软件分析,MSTN蛋白质在5~8位置由折叠变成了螺旋,在17~18位置、19~22位置由卷曲变成了螺旋,在136、190位置由卷曲变成了折叠(图1)。折叠、螺旋、卷曲的比例由野生型的26.13%、15.73%、58.14%分别变成突变型的25.87%、18.13%、56.00%。

RAW表示野生型;SNP表示突变型。黑色背景表示突变型与野生型蛋白质二级结构间有变化。H表示螺旋,E表示拉长的折叠股,C表示卷曲。括号中的数字表示每一行最后的氨基酸在蛋白质中的位置。 图1 MSTN野生型与突变型蛋白质二级结构的比较 Fig.1 The alignment of wild-type and mutated MSTN protein secondary structures
2.4.2MyoD1蛋白质二级结构的变化MyoD1基因外显子1中的有义突变导致了第4位氨基酸从蛋氨酸到亮氨酸的变化。通过软件分析,MyoD1蛋白质在4~5位置由野生型的卷曲变成突变型的螺旋,在30位置由野生型的折叠变成突变型的卷曲(图2)。折叠、螺旋、卷曲的比例由野生型的2.78%、26.98%、70.24%分别变成突变型的2.38%、27.78%、69.84%。
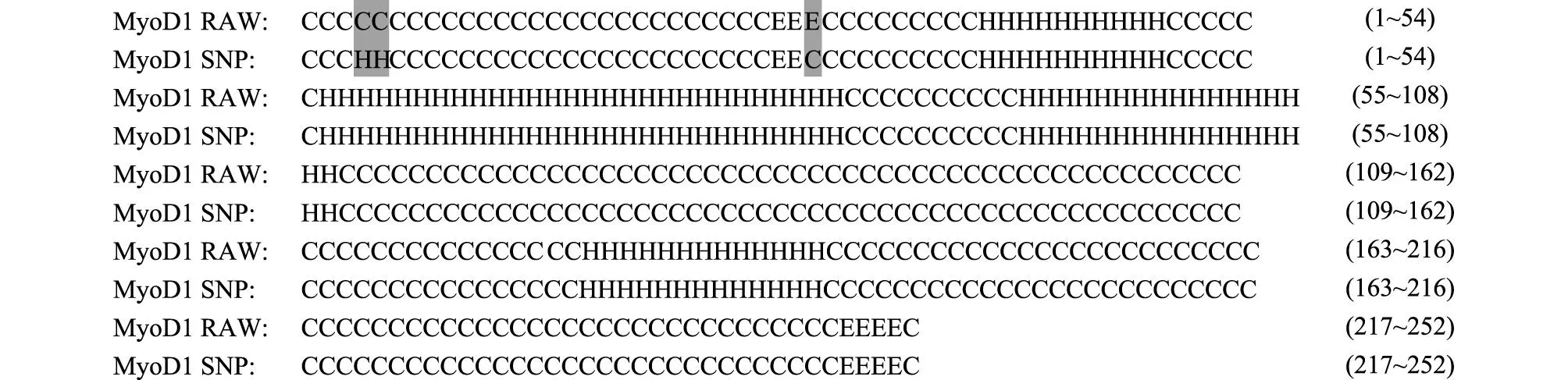
RAW表示野生型;SNP表示突变型。黑色背景表示突变型与野生型蛋白质二级结构间有变化。H表示螺旋,E表示拉长的折叠股,C表示卷曲。括号中的数字表示每一行最后的氨基酸在蛋白质中的位置。 图2 MyoD1野生型与突变型蛋白质二级结构的比较 Fig.2 The alignment of wild-type and mutated MyoD1 protein secondary structures
2.4.3MyoG蛋白质二级结构的变化MyoG基因的第5外显子的一处突变导致了第222位氨基酸从谷氨酸到天冬氨酸的改变。通过软件分析,MyoG蛋白质在149、200、211位置由野生型的螺旋变成突变型的卷曲(图3)。突变型的折叠所占比例0.88%没有变化,螺旋、卷曲的比例分别从野生型的30.40%、68.72%变成突变型的29.07%、70.04%。
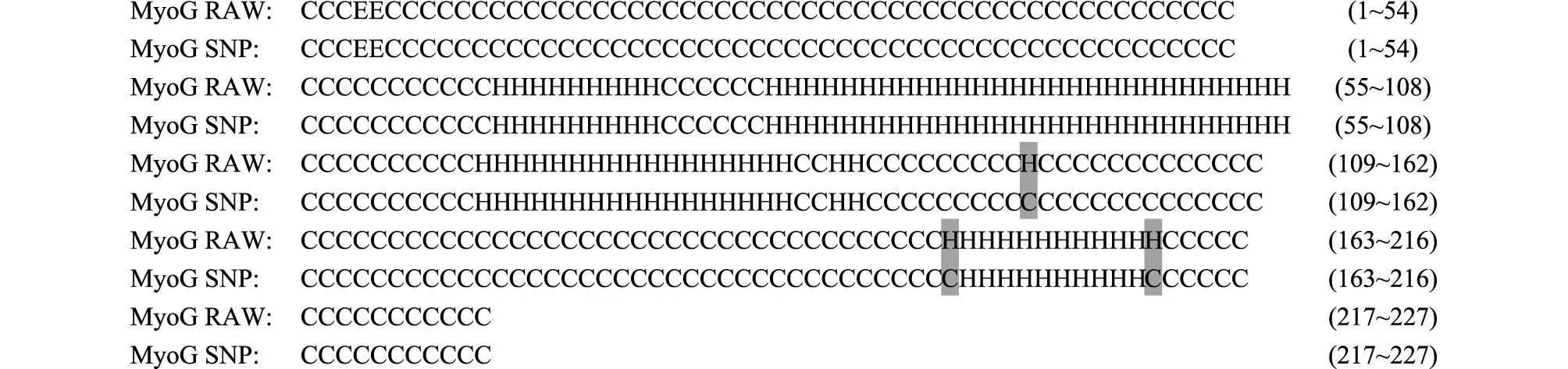
RAW表示野生型;SNP表示突变型。黑色背景表示突变型与野生型蛋白质二级结构间有变化。H表示螺旋,E表示拉长的折叠股,C表示卷曲。括号中的数字表示每一行最后的氨基酸在蛋白质中的位置。 图3 MyoG野生型与突变型蛋白质二级结构的比较 Fig.3 The alignment of wild-type and mutated MyoG protein secondary structures
3讨论
禽类肌肉组织肌纤维的数量在胚胎期即已确定[13],出壳后主要是通过已有肌纤维的卫星细胞相互融合而变得粗大[14]。肌纤维的形成与发育受到MRF家族基因的正向调控,MyoD1基因能够将多种类型的细胞转换为成肌细胞并促进肌细胞融合成肌管[15],MyoG基因的表达可终止成肌细胞的增殖,促进单核成肌细胞融合成多核成肌细胞[16-17]。作为肌肉的负调控因子,MSTN可以抑制成肌细胞的增殖和分化[18]。MSTN不仅可以下调MyoD1基因的表达[19-20],而且还是MyoD1的下游靶基因[21]。MSTN、MyoD1和MyoG基因三者对肌肉的调节是相互协调保持相对的平衡,因此开展MSTN、MyoD1和MyoG3个基因的SNP位点筛查对于生长发育有重要的科学意义。
本研究发现高邮鸭群体MSTN、MyoD1和MyoG基因外显子中分别有9、4和7个SNP位点。其中MSTN基因的所有7个SNP位点均处于Hardy-Weinberg平衡状态。鸭MyoD1基因和MyoG基因的多态性报道较少。Xu等在北京鸭群体中发现MSTN基因CDS区129位置有1个T/C突变[22],与本试验发现的SNP1-2位点(CDS区第129位置)A/G突变一致。该突变位点2个等位基因在北京鸭上频率接近,而在高邮鸭中A等位基因为优势基因,这可能与鸭不同遗传背景有关。除了本试验筛查到的MSTN基因外显子中的7个SNP位点,其他研究者也发现了其他多个SNP位点[23-25],说明MSTN基因的多态性位点在鸭上非常丰富。
寇洁等[26]对鸭MyoD1基因外显子1进行多态性检测,发现1处C/G的突变引起了氨基酸的变化,并且导致鸭屠体腿肌率的显著增加。王琼等[27]对优质肉鸡研究结果表明,MyoG基因5′调控区存在影响肌纤维生长发育的SNP位点。这些说明MSTN、MyoD1和MyoG基因是影响禽类肌肉生长的潜在候选基因。
氨基酸的总平均疏水性可以体现蛋白质的亲疏水性质,数值越高代表疏水能力越强,数值为负值表明该蛋白质为亲水性蛋白质[28]。本研究发现,MyoD1突变蛋白质相比原始蛋白质,总平均亲水性变小,即疏水性变强,这可能对蛋白质的折叠以及功能的发挥有一定的影响。蛋白质的不稳定系数小于40,该蛋白质可认为是稳定的;不稳定系数大于40,那么该蛋白质为不稳定蛋白质[29]。MSTN、MyoD1和MyoG野生型蛋白质和突变型蛋白质不稳定系数均大于40,说明这些蛋白质均为不稳定蛋白质,在动物机体内的半衰期时间较短。
基因外显子水平上的有义突变可以影响蛋白质的二级结构。本试验涉及的3个基因各有1处有义突变,通过蛋白质二级结构预测发现,这些有义突变导致的氨基酸变化均影响了蛋白质的二级结构。其中MSTN蛋白质2处连续的折叠和无规则卷曲均变成了螺旋结构;MyoD1蛋白质1处无规则卷曲变成了螺旋结构,1处折叠变成了无规则卷曲;MyoG蛋白质3处螺旋结构均变成了无规则卷曲。
参考文献:
[1]RUDNICKI M, SCHNEGELSBERG P, STEAD R, et al. MyoD or Myf-5 is required for the formation of skeletal muscle. Cell, 1993, 75(7): 1351-1359.
[2]GROBET L, MARTIN L, PONCELET D, et al. A deletion in the bovine myostatin gene causes the double-muscled phenotype in cattle. Nature Genetics,1997, 17(1): 71-74.
[3]KAMBADUR R, SHARMA M, SMITH T. Mutations in myostatin (GDF8) in double muscled Belgian blue and Piedmon tese cattle. Genome Research, 1997(7): 910-916.
[4]WHITTEMORE L, SONG K, LI X, et al. Inhibition of myostatin in adult mice increases skeletal muscle mass and strength. Biochemical and Biophysical Research Communications, 2003, 300(4): 965-971.
[5]MCPHERRON A C, LEE S J. Double muscling in cattle due to mutations in the myostatin gene. Proc Natl Acad Sci USA, 1997(94): 12457-12461.
[6]NISHI M, YASUE A, NISHIMATU S, et al. A missense mutant myostatin causes hyperplasia without hypertrophy in the mouse muscle. Biochemical and Biophysical Research Communications, 2002, 293(1): 247-251.
[7]CLOP A, MARCQ F, TAKEDA H, et al. A mutation creating a potential illegitimate microRNA target site in the myostatin gene affects muscularity in sheep. Nature Genetics, 2006, 38: 813-818.
[8]BOMAN I A, KLEMETSDAL G, NAFSTAD O, et al. Impact of two myostatin (MSTN) mutations on weight gain and lamb carcass classification in Norwegian white sheep (Ovisaries). Genetics Selection Evolution, 2010, 42(1): 4.
[9]MOSHER D S, QUIGNON P, BUSTAMANTE C D, et al. A mutation in the myostatin gene increases muscle mass and enhancesracing performance in heterozygote dogs. Plos Genetics, 2007, 3(5): 779-785.
[10]张跟喜,丁馥香,是燕萍,等. 肌肉生长抑制素基因 (MSTN) 外显子1的多态性及其与边鸡生长性状的关联分析. 农业生物技术学报, 2011, 19(1): 122-127.
[11]王志新. 蛋白质结构预测的现状与展望. 生命的化学, 1998, 18(6): 19-22.
[12]吴祖建,高芳銮,沈建国. 生物信息学分析实践. 北京: 科学出版社, 2010.
[13]REMIGNON H, GARDAHAUT M F, MARCHE G, et al. Selection for rapid growth increases the number and the size of muscle fibres without changing their typing in chickens. J Muscle Res Cell Motil, 1995, 16(2): 95-102.
[14]GROUNDS M D. Towards understanding skeletal muscle regeneration. Pathol Res Pract, 1991, 187(1): 1-22.
[15]WEINTRAUB H, TAPSCOTT S J, DAVIS R L, et al. Activation of muscle-specific genes in pigment, nerve, fat, liver, and fibroblast cell lines by forced expression of MyoD. PNAS,1989, 86(14): 5434-5438.
[16]NAIDU P S, LUDOLPH D C, TO R Q, et al. Myogenin and MEF2 function synergistically to activate the MRF4 promoter during myogenesis. Mol Cell Biol,1995, 15(5): 2707-2718.
[17]OLSON E N, BRENNAN T J, CHAKRABORTY T, et al. Molecular control of myogenesis: antagonism between growth and differentiation. Mol Cell Biochem, 1991, 104(1-2): 7-13.
[18]JOULIA D, BERNARDI H, GARANDEL V, et al. Mechanisms involved in the inhibition of myoblast proliferation and differentiation by myostatin. Experimental Cell Research, 2003, 286(2): 263-275.
[19]LANGLEY B, THOMAS M, BISHOP A. Myostatin inhibits myoblast differentiation by down-regulating MyoD expression. Journal of Biological Chemistry, 2002, 277(51): 49831-49840.
[20]阮井玲,甄鑫,刘娣,等. Myostatin通过Smad3下调MyoD的表达来抑制骨骼肌卫星细胞的分化. 中国生物工程杂志, 2008, 28(5): 99-103.
[21]SPILLER M P, KAMBADUR R, JEANPLONG F, et al. The myostatin gene is a downstream target gene of basic helix-loop-helix transcription factorMyoD. Molecular and Cellular Biology, 2002, 22(20): 7066-7082.
[22]XU T S, GU L H, ZHANG X H, et al. Characterization of myostatin gene (MSTN) of Pekin duck and the association of its polymorphism with breast muscle traits. Genet Mol Res, 2013, 12(3): 3166-3177.
[23]LU J, HOU S, HUANG W, et al. Polymorphisms in the myostatin gene and their association with growth and carcass traits in duck. African Journal of Biotechnology, 2011, 10(54): 11309-11312.
[24]卢俊清. 北京鸭肌肉生长抑制素基因(MSTN)多态性与骨骼肌和脂肪生长发育性状的关联性分析. 中国: 中国农业科学院, 2008.
[25]刘倩,陈永华,蔡凤仙,等. 高邮鸭生长抑制素基因外显子3多态性与腹脂率的关联分析. 中国家禽, 2012, 34(23): 27-30.
[26]寇洁,李亮,王继文. 5个鸭品种MyoD基因外显子1的多态性及其与屠宰性状的关联分析. 中国畜牧杂志, 2011, 47(19): 10-12.
[27]王琼,刘益平,蒋小松,等.MyoG基因多态性与优质肉鸡屠宰性状和肉质性状的相关性分析. 遗传, 2007, 29(9): 1089-1096.
[28]薛庆中. DNA和蛋白质序列数据分析工具. 3版. 北京: 科学出版社, 2012.
[29]WALKER J M. The proteomics protocols handbook.Totowar: Humana Press, 2005.
(责任编辑:袁伟)
胡成,朱善元,左伟勇,等. 鸭I型肝炎病毒一步法RT-LAMP可视化快速检测方法的建立[J].江苏农业学报,2015,31(3):613-618.
