3MBA类FtsZ蛋白抑制剂的分子动力学模拟及抗菌作用机制
2015-12-29卢俊瑞穆江蓓刘金彪杨旭云王美君张瑞波天津理工大学化学化工学院天津300384
张 贺 卢俊瑞 穆江蓓 刘金彪 杨旭云 王美君 张瑞波(天津理工大学化学化工学院,天津300384)
3MBA类FtsZ蛋白抑制剂的分子动力学模拟及抗菌作用机制
张 贺 卢俊瑞*穆江蓓 刘金彪 杨旭云 王美君 张瑞波
(天津理工大学化学化工学院,天津300384)
采用分子动力学模拟、蛋白质二级结构测定(DSSP)、口袋体积测量(POVME)以及MM-PBSA (molecular mechanics Poisson-Boltzmann surface area)方法,系统研究了金黄色葡萄球菌丝状温度敏感性蛋白Z(SaFtsZ)-二磷酸鸟苷(GDP)二元复合物和SaFtsZ-GDP-3MBA(3-甲氧基苯甲酰胺)类衍生物三元复合物体系的稳定性、蛋白质二级结构、蛋白质构象、关键残基质心距、活性口袋体积以及相对结合自由能的变化规律.研究表明:当不含抑制剂存在时SaFtsZ-GDP二元复合物体系稳定性较差,其T7Loop区域残基(203-209)波动较大,且蛋白二级结构发生明显变化,活性口袋体积急剧减小,底物通道显著变窄且不稳定.而含有抑制剂PC190723、Compound1的类衍生物三元复合物体系的表现截然不同,这主要是由于它们均能和活性口袋T7Loop区周围残基形成关键性的氢键以及疏水作用,与FtsZ蛋白紧密结合.在SaFtsZ-GDP-3MBA三元复合物体系中,3MBA仅能与活性口袋中部分残基形成疏水作用,与FtsZ蛋白亲和力较弱,使其不能稳定地存在于活性口袋中,进一步导致它的抗菌活性明显低于PC190723、Compound1.这些发现深入揭示了3MBA类衍生物对FtsZ蛋白的作用机制和影响规律,为该类FtsZ蛋白抑制剂的结构优化和产品开发应用提供了重要的理论依据.
3MBA类衍生物;FtsZ;分子动力学模拟;抗菌作用机制;结合自由能
1 引言
近年来,抗生素滥用导致了严重的细菌耐药性问题,而已有抗菌药物主要是针对细菌细胞壁的合成、细菌蛋白质的合成、核苷酸的合成、叶酸的合成等四类靶标,致使现行抗菌药物较易产生细菌耐药性.解决上述问题,迫切需要开发具有新的作用靶点或新的作用机制的抗菌药物.
近年来,细菌分裂蛋白逐渐成为抗菌药物研究的热点.1-3细菌分裂蛋白在形态上高度保守、不易变异.细菌细胞分裂过程中,FtsZ蛋白首尾相接,在细胞中央区域组装聚合,形成与细胞质膜内表面镶嵌的Z环,最终以Z环为平面分裂成两个子细胞.FtsZ作为起始蛋白,具有三磷酸鸟苷酶(GTPase)活性,能够催化三磷酸鸟苷(GTP)水解生成二磷酸鸟苷(GDP),从而为Z环形成收缩和解聚提供能量.4-7研究表明,T7Loop附近区域是启动FtsZ蛋白组装的关键开关,而组装开关的启动必须要求C端序列区域残基(223-310)向N端序列区域残基(12-171)移动,并关闭两区域之间的活性口袋通道.8如果能够找到合适的化合物,有效阻断FtsZ蛋白的组装或抑制GTP酶的活性,就能够导致细菌的裂解死亡.6,9
目前已经发现了多种不同骨架结构的FtsZ抑制剂,按照其来源可分为天然化合物和全合成化合物两类.天然化合物主要有绿垂曲霉(viriditoxin)、苯并啡啶碱类生物碱血根碱(sanguinarine)、二萜酚(totarol)、白花丹素(plumbagin)、GTP类似物Br-GTP等.10-16天然化合物往往因其生物利用度较差或体内毒副作用较强等限制了它们的应用.如绿垂毒素虽然对多重耐药菌,如葡萄球菌属、肠球菌属、肺炎链球菌等具有较好的抗菌活性(MIC=2-32µg·mL-1),但其生物利用度存在不足,必须对其结构进行优化,以提高其抗菌活性和靶向性.10全合成化合物主要通过高通量筛选、分子对接等方法寻找得到. 2005年Stokes等17从大约105000个化合物中筛选出活性化合物PC58538,并通过结构优化得到了代表性的化合物PC170942,其对金黄色葡萄球菌的MIC为64µg·mL-1.2006年Ito等18从大约95000个化合物中筛选出活性化合物A-189,其对GTP酶的抑制活性(IC50)为80µg·mL-1,对金黄色葡萄球菌的MIC为16µg·mL-1,研究表明其主要通过抑制GTP酶的活性来干扰FtsZ蛋白的组装,从而抑制了细菌生长.2009年Beuria等19研究了29种骨架结构的81个化合物对FtsZ蛋白组装的影响,发现其中绕丹宁衍生物(OTBA)对FtsZ蛋白的特异性较强,且对哺乳动物的微管蛋白几乎无抑制作用,其抑制枯草芽孢杆菌细胞增殖的MIC约为1µg·mL-1,是一种有潜力的先导化合物.迄今为止,在全合成化合物中3-甲氧基苯甲酰胺(3MBA)是公认的最具开发前景的先导化合物.早在1999年Ohashi等20便发现了3MBA能够抑制枯草芽孢杆菌的二分裂,但抗菌活性较低,对金黄色葡萄球菌属的MIC为4000µg· mL-1.2008年Haydon等21通过对3MBA衍生物修饰,得到了优异的FtsZ靶向抑制剂PC190723 (9PC).研究表明,9PC能显著抑制FtsZ蛋白的GTP酶活性,对GTP酶的IC50达到0.55µg·mL-1,对枯草芽孢杆菌、金黄色葡萄球菌的MIC达到0.5-1µg· mL-1.为了验证其体内活性,Haydon等21对腹腔接种潜在致死量金黄色葡萄球菌的小鼠样本进行皮下或静脉注射30 mg·kg-1剂量的9PC溶液,结果发现小鼠存活率达到100%.2012年Stokes等22对3MBA衍生物进行修饰,得到一种具有优异体内、外活性的化合物Compound 1(Com1),其在小鼠体内的生物利用率达到82%.Com1对所有葡萄球菌属的平均MIC达到0.12µg·mL-1,对枯草芽孢杆菌的MIC达到0.03µg·mL-1,抗菌活性与抗菌药物三氯生相近.23,24上述研究表明,3MBA作为FtsZ蛋白抑制剂先导化合物具有突出优势和巨大潜力.图1给出了3MBA及其典型衍生物的化学结构.
研究表明,抑制剂与FtsZ蛋白主要有两个结合位点,即GTPase活性区域和T7Loop区的疏水性活性口袋.如天然产物绿垂霉素、Br-GTP均作用于GTPase活性区域,是GTP的竞争性抑制剂,但因抑制能力较弱,导致其抗菌活性相对较差.其余多数抑制剂,如3MBA、9PC、Com1等均作用于T7Loop的活性口袋区域.9PC具有2个独特化学基团,即苯甲酰胺和噻唑并吡啶,能够与FtsZ蛋白形成氢键及疏水等作用,而抑制剂在活性口袋中结合的稳定性是决定其亲和力以及靶向选择性的关键因素.25在SaFtsZ-GDP-9PC三元复合物体系中,9PC的酰胺基团主要与FtsZ蛋白活性口袋上Val207、Asn263的羰基形成氢键,2-Cl基团处于疏水残基Leu200、Leu209附近,6-CL基团处于疏水残基Val203、Leu297附近.噻唑并吡啶能够与H7螺旋区域和C端区域之间的残基(Gly193,Gly196,Ile197,Met226, Ile228,Thr309等)形成疏水作用.这些结合模式均有利于三元复合物体系的形成和稳定,构成了9PC作为抑制剂的分子生物学基础.

图1 3MBA及其衍生物的化学结构Fig.1 Chemical structures of the 3MBAand its derivatives
尽管FtsZ蛋白已被公认为是一种较好的抗菌药物作用靶点,而且已经发现合成了一批FtsZ抑制剂,但因其体内抗菌活性、作用机制等研究尚不够深入,迄今,FtsZ蛋白抑制剂仍未进入临床应用.因此,利用FtsZ蛋白晶体结构,研究抑制剂与FtsZ蛋白的结合模式和作用机制,对FtsZ蛋白抑制剂的开发应用具有重要意义.本文以FtsZ蛋白晶体结构为基础,应用分子动力学模拟、蛋白质二级结构测定(DSSP)、口袋体积测量(POVME)以及MM-PBSA方法,对SaFtsZ-GDP二元复合物体系及SaFtsZ-GDP-3MBA类抑制剂三元复合物体系进行研究,系统研究了T7Loop的疏水性活性口袋区域的稳定性、蛋白二级结构、蛋白质构象、关键残基质心距离,活性口袋体积以及相对结合自由能的特征和变化规律,揭示了3MBA类抑制剂对细胞分裂蛋白FtsZ的内在作用机制,为针对FtsZ靶点的药物设计、筛选和开发应用提供了重要的理论依据.
2 实验部分
2.1 蛋白质结构准备
从PDB结构数据库获得代号为4DXD26的金黄色葡萄球菌FtsZ蛋白晶体结构,删除晶体结构中的水分子,建立SaFtsZ-GDP-9PC三元复合物体系,删除其中9PC的结构信息建立SaFtsZ-GDP二元复合物体系.通过AutoDock27分子对接获得SaFtsZGDP-3MBA以及SaFtsZ-GDP-Com1三元复合物体系.在AutoDock对接中,盒子中心坐标定义为晶体配体中醚氧原子的空间坐标(x-center:-14.018;ycenter:42.480;z-center:18.071),格点间距为3.75× 10-2nm,盒子大小为50个格点组成的正方体区域.采用拉马克遗传算法进行计算,其运行次数设置为200次,最大能量评估次数设置为2.5×106,最多迭代次数设置为2.7×105,其他参数为默认值.使用acpype程序28生成配体及GDP的拓扑文件,用于构建分子动力学模拟中的复合物拓扑文件.
为了深入认识抑制剂对FtsZ蛋白的作用机制,通过ZDOCK 3.0.2程序29进行蛋白与蛋白全局刚性对接采集SaFtsZ蛋白二聚体结构,对接产生2000个二聚体结构.之后对所有结构进行打分排序,从中挑选出打分较好并且满足结合位点信息的结构用于结果分析,并与晶体结构中詹氏甲烷球菌MjFtsZ-GTP二聚体(PDB代号为1W5A)的构象对比.
2.2 分子动力学模拟
运用Gromacs 4.6.4分子动力学模拟软件,30在Amber ff99SB力场31下,分别对上述四种复合物体系进行分子动力学模拟.体系质心均位于截角八面体盒子中心,溶质外围加上1.0 nm的TIP3P模型32的水分子层.首先用最陡下降法对复合物进行50000步的能量优化,以消除分子间的高能碰撞,然后分别进行100 ps的NVT和100 ps的NPT模拟,使体系温度保持在300 K、压强保持在105Pa左右的平衡状态,最后分别对四种复合物体系进行100 ns无约束的分子动力学模拟.采用周期性边界条件和PME(Particle Mesh Ewald)方法处理长程静电作用.通过V-rescale和Parrinello-Rahman控制系统的温度和压力.33,34使用LINCS算法35固定所有成键原子之间的相对距离.模拟过程中采用的积分步长为2 fs,每5步更新一次非键连接表,每1 ps保存一次轨迹结构用于后续数据的分析.36
所有计算机模拟工作均在HP Z820工作站上完成,主要配置:CPU为Intel(R)Xeon(R)E5-2680@ 2.7 GHz,内存为64 GB,硬盘为2 T,配有GPU (NVIDIATesla C2075)加速计算显卡.
2.3 T7Loop区活性口袋体积测定
分别从四种体系100 ns的分子动力学轨迹文件中每隔50 ps提取一帧复合物坐标文件.将提取的2000个坐标文件与初始坐标文件进行叠加,采用POVME程序37对每帧叠加之后的蛋白进行活性口袋体积计算.以9PC中的醚氧原子的空间位置(-14.018,42.480,18.071)为中心坐标,以1.0 nm为半径构建球体,使球体包含整个活性口袋.使用POVME程序生成半径为0.1 nm的体积-格点文件,删除与氨基酸残基相叠合的格点,同时保留以中心坐标为球心,半径0.4 nm范围内缺失不多于2个的连续格点.38最后根据格点的数量计算得到活性口袋体积随时间变化的曲线图.
2.4 结合自由能计算
从分子动力学模拟过程中得到的配体与受体蛋白的相对结合自由能通常比分子对接得分具有更高的实验结果相关性以及筛选能力.通过g_mmpbsa工具运用MM-PBSA方法计算得到配体与FtsZ蛋白之间的相对结合自由能G.G=EMM+Gsolv,其中EMM=Gele+Gvdw,Gsolv=Gpolar+Gnon-polar.相对结合自由能计算中忽略了熵的变化.其中Gele为静电作用, Gvdw为范德华力,Gsolv为溶剂化作用,Gpolar为极性溶剂化作用,Gnon-polar为非极性溶剂化作用.极性和非极性项的计算则采用APBS软件,39电性项的参数gridspacing取0.05 nm,探针半径0.14 nm,复合物介电常数设为1,溶剂的介电常数设为80.非极性项Gnon-polar采用溶剂可及表面(SASA)模型计算:Gnon-polar= γSSASA+β.其中γ为表面张力参数,β为拟合修正参数, γ=2.27 kJ·mol-1·nm-2,β=3.85 kJ·mol-1.34,40相对结合自由能值为从分子动力学模拟最后10 ns轨迹中每隔100 ps获得构象的平均结合自由能.配体与FtsZ蛋白的相对结合自由能反映了配体与蛋白结合能力的强弱,决定了配体能否稳定存在于活性口袋中.而配体与活性位点结合的稳定性往往是决定抑制剂亲和力以及靶向选择性的关键因素.
3 结果分析与讨论
3.1 MjFtsZ二聚体和SaFtsZ二聚体结合模式
如图2所示,A代表詹氏甲烷球菌MjFtsZ-GTP二聚体构象,B代表金黄色葡萄球菌SaFtsZ-GDP-9PC复合物体系二聚体构象.在MjFtsZ-GTP二聚体构象中,FtsZ蛋白首尾相接,FtsZ蛋白的Loop区嵌入到另一个FtsZ蛋白的核苷酸结合区域,行使催化功能,水解GTP转化为GDP,为细菌分裂提供能量.在SaFtsZ-GDP-9PC复合物体系二聚体构象中,蛋白构象与图2A相似,T7Loop区与另一个FtsZ蛋白的核苷酸结合区域靠近,配体9PC存在于T7Loop区周围的活性口袋,能够稳定FtsZ蛋白构象.

图2 MjFtsZ蛋白二聚体和SaFtsZ蛋白二聚体构象Fig.2 Conformations of MjFtsZ protein dimer and SaFtsZ protein dimer
3.2 四种体系的稳定性与残基波动情况
对四种体系分别进行100 ns的分子动力学模拟.以晶体初始蛋白构象为参照,分析蛋白中Cα原子的均方根偏差(RMSD)随模拟时间变化趋势,考察四种体系蛋白构象的稳定性以及残基波动情况.从图3中可知,二元复合物的RMSD值在30 ns左右达最大值后逐渐平衡,平衡后RMSD值在0.180-0.297 nm之间,RMSD平均值为0.223 nm,SaFtsZGDP-3MBA体系在前60 ns其RMSD值不断升高,随后逐渐平衡,平衡后RMSD值为0.190-0.300 nm, RMSD平均值为0.238 nm,两种体系与初始蛋白晶体构象相比变化幅度较大.SaFtsZ-GDP-9PC三元复合物体系在10 ns后已达到平衡,平衡后RMSD值在0.098-0.209 nm区间内变化,RMSD平均值仅为0.149 nm.SaFtsZ-GDP-Com1三元复合物体系在30 ns左右达到平衡,平衡后的RMSD值为0.120-0.227 nm,RMSD平均值仅为0.163 nm.配体9PC、Com1的存在使蛋白构象变化与整体变化幅度均较小.这主要是因为9PC或Com1能够稳定FtsZ蛋白的整体构象,使其能够较好地保持初始蛋白构象.
为研究FtsZ蛋白氨基酸残基的波动情况,分别计算四种体系在分子动力学模拟过程中氨基酸残基的均方根涨落值(RMSF).如图4所示,二元复合物中所有残基的平均RMSF值为0.111 nm,在3MBA、9PC、Com1存在的三元复合物中所有残基的平均RMSF值分别为0.123、0.110、0.110 nm,四种体系氨基酸残基RMSF值较为相近,氨基酸残基整体波动情况较为一致,但在SaFtsZ-GDP-3MBA体系中部分氨基酸残基波动明显.为此深入研究四种体系RMSF值差异性较大的区域,对T7Loop区域以及H7螺旋区域(Met179-Ala202)进行进一步分析.发现当9PC、Com1存在时活性口袋T7Loop区残基RMSF值波动较小,平均值分别仅为0.094、0.117 nm,表现出很好的稳定性.而3MBA存在的三元复合物或无配体的二元复合物体系中,T7Loop区残基的RMSF值波动较大,平均值分别为0.243、0.123 nm,相对于其他两个三元复合物体系的T7Loop区残基表现出较大的波动性.对H7螺旋区域残基RMSF值进行对比发现,在配体3MBA、9PC、Com1存在时RMSF平均值分别为0.152、0.129、0.147 nm,而无配体存在时H7螺旋区域残基的RMSF平均值仅为0.102 nm.配体的存在反而使得H7螺旋区域残基有较大的浮动,配体对这一区域残基RMSF值的影响导致三元复合物体系所有残基的RMSF平均值与二元复合物体系所有残基的RMSF平均值较为相近.以上数据表明,当9PC、Com1存在时能够降低T7Loop区残基的柔性,使得靠近苯甲酰胺基团的活性口袋区域构成一个较为稳定的体系.这主要是由于苯甲酰胺基团能够和T7Loop区残基形成较强的氢键以及疏水作用,使其靶向作用于这一活性口袋区域.

图3 四种体系Cα相对于晶体结构位置的均方根偏差随模拟时间的变化Fig.3 Alpha-carbon root mean square deviations(RMSD) of the four models relative to the crystal structure,as a function of time
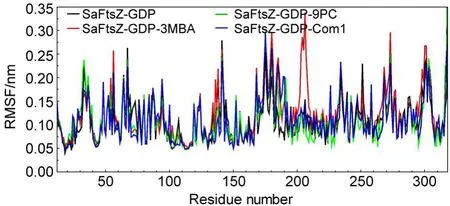
图4 动力学模拟过程中蛋白各个残基的均方根涨落Fig.4 Root mean square fluctuations(RMSF)of residues in the protein during dynamics simulations

图5 Met179-Ala202残基二级结构随模拟时间的变化Fig.5 Secondary structure changes of the Met179-Ala202 residues as a function of time
3.3 四种体系中蛋白二级结构变化情况
通过DSSP程序41计算四种体系蛋白二级结构随时间变化的规律,发现主要变化集中在T7Loop区域以及H7螺旋区域.二元复合物体系T7Loop区中Ser204-Gly205残基的二级结构为coil卷曲,然而随着模拟的进行,主要在coil卷角、bend弯曲之间相互转化,最终以bend弯曲形式稳定存在.三元复合物中除3MBA体系外该区域以coil卷曲形式稳定存在.3MBA体系该区域二级结构随着模拟的进行,在coil卷角、bend弯曲、turn转角之间不断转化.
图5所示为二元复合物体系与典型的SaFtsZGDP-9PC三元复合物体系H7螺旋区域二级结构随模拟时间变化的情况.在模拟初期,SaFtsZ-GDP二元复合物体系中H7螺旋区域中Gln195-Ala202残基的二级结构为α螺旋,随着模拟的进行该区域二级结构在310螺旋、α螺旋、turn转角之间转化,最终二级结构稳定以turn转角形式存在.H7螺旋区域中Ala182-Glu185残基的二级结构在310螺旋、α螺旋、turn转角之间转化,平衡之后以310螺旋稳定存在.而在SaFtsZ-GDP-9PC三元复合物体系中Gln195-Ala202残基的二级结构构象较为单一稳定,主要以α螺旋规则构象存在,仅观察到T7Loop区与H7螺旋区连接处残基Leu200-Ala202二级结构在α螺旋、turn转角之间不断转化,最终主要以turn转角形式存在.H7螺旋区域中Ala182-Asp187残基的二级结构从35 ns左右开始在310螺旋、α螺旋、turn转角之间不断转化.可见,配体的存在能够较好地稳定T7Loop区域残基的二级结构,但同时引起了H7螺旋区域Ala182-Asp187残基二级结构的变化.
3.4 T7Loop区活性口袋与配体的结合模式
为了更清晰地描述3MBA类衍生物与活性口袋关键残基的相互作用,通过对四种体系最后10 ns分子动力学模拟轨迹进行聚类分析,分别得到四种体系具有代表性的构象.在聚类分析过程中,基于所有原子的RMSD,应用gromos计算方法提取构象类似的帧为一类,选取最大类的构象进行结合模式的分析.应用Pymol程序42和LigPlot+软件43和对三元复合物体系进行结合模式的分析,如图6所示, 3MBA并没有与FtsZ蛋白形成氢键作用,仅仅与活性口袋两侧部分残基形成疏水作用.而9PC、Com1不仅能够与T7Loop区Gly205、Val207或Leu209残基形成氢键作用,而且均能与活性口袋一侧Asn263残基形成关键的氢键作用,同时与活性口袋区域中的Gly193、Gly196、Leu200、Thr309等残基形成疏水作用.在最后10 ns代表性构象中三元体系配体3MBA、9PC、Com1与初始配体构象的RMSD值分别为1.03、0.16、0.300 nm.可见配体9PC和Com1均能较好地保持初始构象,而配体3MBA构象变化极大.通过与初始构象结合模式进一步对比,发现在初始构象中9PC、Com1均能与Gly205、Val207或Leu209残基形成氢键作用.其中在初始构象中Com1并没有和Asn263残基形成氢键作用.然而在分子动力学模拟过程中,通过分子构象的不断变化,Com1与Asn263残基形成氢键并稳定存在. 3MBA虽然在初始蛋白构象能够与Gly205、Val207、Leu209、Asn263残基形成氢键作用,但与其形成疏水作用的残基相对较少,导致疏水作用较弱,影响3MBA在活性口袋的稳定性,随着模拟时间的进行,氢键作用逐渐消失,3MBA逐渐远离活性口袋区域.

图6 最后10 ns中三种体系代表构象结合模式图Fig.6 Representative binding modes of the three models during the last 10 ns
3.5 T7Loop区活性口袋关键残基质心距离
T7Loop区活性口袋形状类似于狭长的通道,抑制剂进入活性口袋后占据底物通道,使得活性口袋区域保持较为稳定的构象.为了更清晰地描述底物通道的变化,通过测量四种体系T7Loop区周围Leu200残基与Asn263残基之间的质心距,考察活性口袋通道两侧距离随模拟时间的变化.表1所示为四种体系Leu200残基与Asn263残基质心距的初始距离、最小距离、最大距离以及平均距离.图7所示为SaFtsZ-GDP二元复合物体系以及选取的SaFtsZ-GDP-9PC三元复合物体系中Leu200残基与Asn263残基质心距随模拟时间的变化曲线.在二元复合物体系中,Leu200残基与Asn263残基初始质心距为0.787 nm,在模拟初期0.2 ns左右达最大值(0.951 nm),随着时间的增加质心距逐渐变小,在26 ns左右达到最小值(0.504 nm),之后逐渐稳定,模拟过程中质心距平均值仅为0.651 nm,而在SaFtsZGDP-9PC三元复合物体系中,Leu200残基与Asn263残基初始质心距为0.822 nm,随模拟时间波动较小,在1.2 ns左右达最大值1.037 nm,最小值仅为0.775 nm,模拟过程中质心距平均值为0.853 nm,远远高于二元复合物的质心距相对应值,这些数据表明FtsZ活性口袋区域底物通道残基具有较高的柔性,无配体存在的二元复合物体系中活性口袋通道距离变窄.而配体9PC、Com1的存在能够稳定活性口袋通道,较好地保持初始蛋白构象,Leu200残基与Asn263残基质心距较初始距离稍微增加,导致底物通道距离变宽.SaFtsZ-GDP-Com1三元复合物体系Leu200残基与Asn263残基质心距的相对应值均稍高于SaFtsZ-GDP-9PC三元复合物体系.而SaFtsZ-GDP-3MBA三元复合物体系中配体的存在并没有对质心距造成很大的影响.在分子动力学模拟初期,配体3MBA的存在一定程度上稳定了底物通道,但随着模拟时间的进行,可以看出Leu200残基与Asn263残基质心距和二元复合物体系质心距趋势类似,仅仅比二元复合物体系质心距平均值高0.017 nm,可见3MBA并不能很好的稳定存在于活性口袋中.
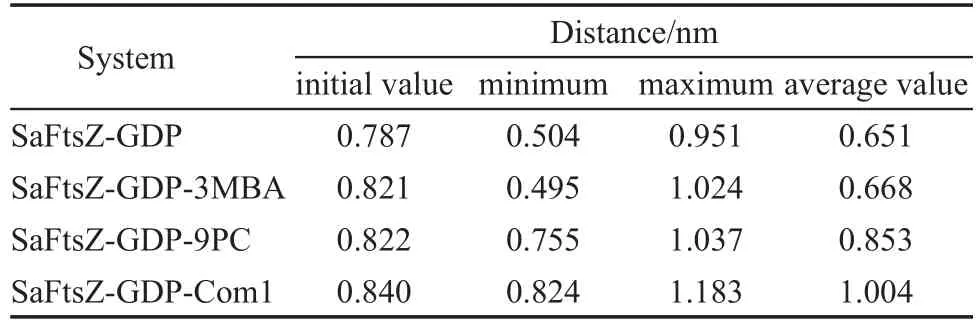
表1 氨基酸Leu200与Asn263质心距特征值Table 1 Eigenvalues of center-of-mass distance between residues Leu200 andAsn263

图7 Leu200残基与Asn263残基之间的质心距离随时间的变化曲线Fig.7 Center-of-mass distance between residues Leu200 andAsn263 as a function of time
3.6 T7Loop区活性口袋体积分析
活性口袋体积随模拟时间变化的规律如图8所示,SaFtsZ-GDP二元复合物体系活性口袋体积随着模拟时间迅速减小,波动范围较广,体积在0-0.197 nm3区间上下波动,后60 ns体积平均仅为0.016 nm3,而三元复合物的体积变化较小,以SaFtsZGDP-9PC三元复合物体系为例,模拟平衡后,活性口袋体积变化较小,以平均体积0.399 nm3保持稳定存在.而在SaFtsZ-GDP二元复合物体系中,底物通道距离变窄,活性口袋体积呈显著性缩小变化.这主要是由于在9PC体系中,9PC既能与Val207、Asn263等残基形成氢键,又能与活性口袋中Gly193、Leu200、Val203、Leu209、Thr309等残基形成疏水作用,从而稳定存在于活性口袋中.与9PC体系比较,Com1体系活性口袋体积变化较为类似,而配体3MBA存在时,体系的活性口袋体积变化明显,平均活性口袋体积则介于9PC体系和二元体系之间.
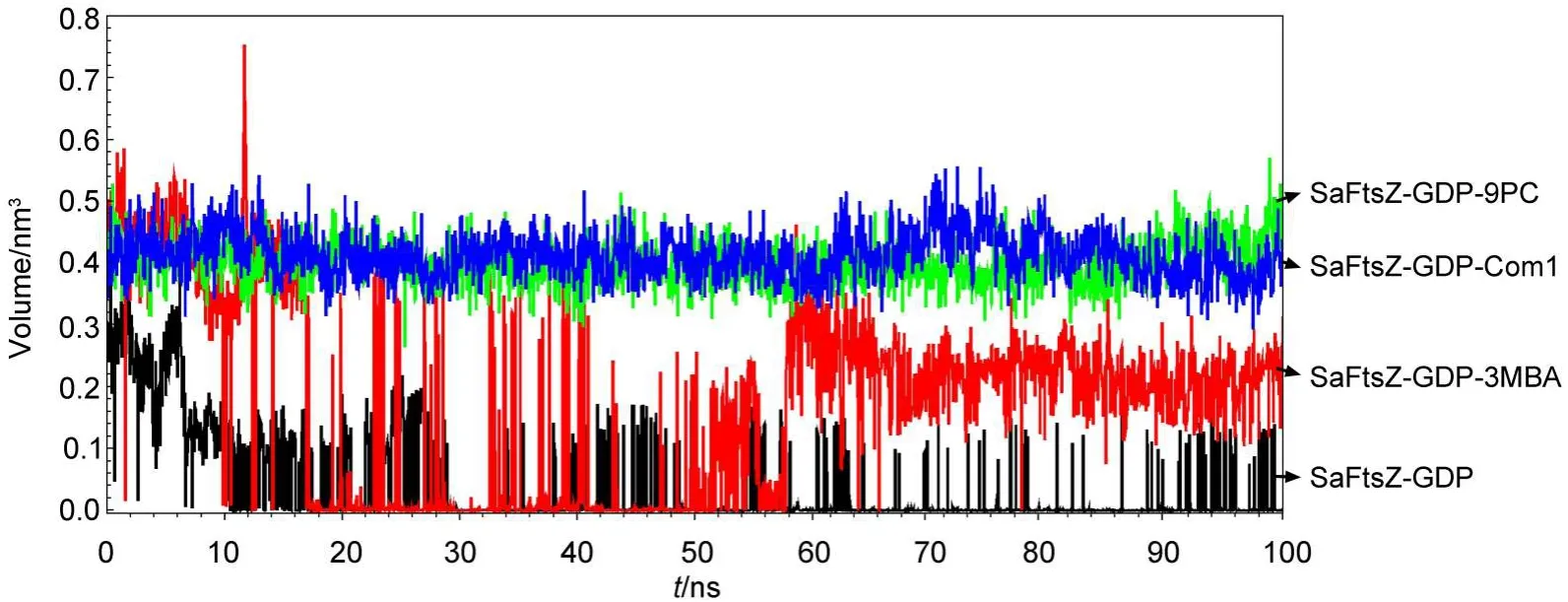
图8 四种体系中活性口袋体积随时间的变化Fig.8 Volumes of the active pockets of the four models as a function of time
由于T7Loop区附近区域是启动FtsZ蛋白组装的开关,而组装开关的启动必须要求C端序列移向N端序列并关闭两个结构域之间的狭长活性口袋区域.在9PC、Com1存在时,两个结构域中Leu200残基与Asn263残基质心距较初始质心距增加,同时活性口袋体积波动较小,能够较好地保持初始蛋白构象,进一步表现为阻碍C端序列移向N端序列,使FtsZ蛋白组装开关呈关闭状态,进而影响细菌分裂.
3.7 MM-PBSA相对结合自由能分析
通过MM-PBSA方法计算SaFtsZ-GDP-3MBA、SaFtsZ-GDP-9PC、SaFtsZ-GDP-Com1体系相对结合自由能Gbinding大小依次为-62.85,-157.19,-199.27 kJ·mol-1.配体9PC、Com1与FtsZ蛋白的相对结合自由能均小于-150 kJ·mol-1,结合能值是衡量配体是否能够稳定存在于活性口袋的重要指标,较低的结合能使得配体与FtsZ蛋白能够较好的结合.与9PC和Com1相比,3MBA与FtsZ蛋白相对结合自由能较高,仅为-62.85 kJ·mol-1,因此3MBA虽然在模拟初期能够与FtsZ蛋白形成氢键以及疏水作用,但由于结合能力相对较弱,随着分子动力学模拟的进行,3MBA并不能很好地稳定存在于活性口袋中.这与以上研究结果中Leu200和Asn263残基质心距变化,T7Loop区域活性口袋体积变化是一致的.同样生物活性测试结果中3MBA、9PC、Com1对金黄色葡萄球菌属的MIC值分别为4000、0.5-1、0.12µg· mL-1.AutoDock分子对接中3MBA、9PC、Com1的结合能分别为-27.00、-42.11、-43.70 kJ·mol-1.可见MM-PBSA相对结合自由能计算结果与生物活性测试结果以及AutoDock分子对接结果反映的规律相一致.
为进一步分析相对结合自由能中不同结合能的贡献,对三种配体与FtsZ蛋白的分项结合自由能进行比较,结果如表2所示.
从表2中可以看出,范德华作用对相对结合自由能的贡献最为突出,然后依次是静电作用和非溶剂化作用.在形成复合物的过程中,范德华作用、非极性溶剂化能和静电作用均有利于配体的结合,而溶剂化能为正值,不利于配体的结合.这主要是由于溶质分子在进入溶剂后要排开一部分溶剂,同时阻断溶剂分子之间的相互作用,这个过程是热力学不利的.
为了更进一步考察配体与FtsZ蛋白分子动力学模拟过程中各部分残基对相对结合自由能的贡献,通过自由能分解得出各个残基对结合自由能的贡献.为方便分析,将结合能分成>-2 kJ·mol-1、-2--4 kJ·mol-1、-4--6 kJ·mol-1、<-6 kJ·mol-1四级.表3列出了三元复合物体系在结合过程中主要残基对结合自由能的贡献.
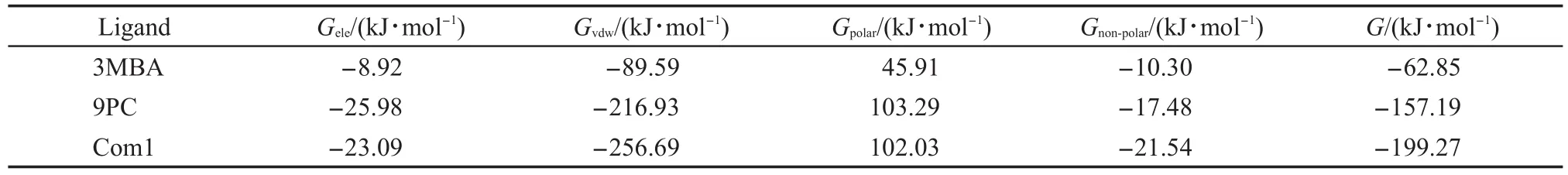
表2 三种配体与FtsZ蛋白的分项结合自由能Table 2 Partial binding energy of three ligands with FtsZ protein

表3 主要残基对结合自由能的贡献Table 3 Binding energy contributions of some main residues
从表3中可以看出,SaFtsZ-GDP-3MBA三元复合物体系中,仅有Glu301、Leu302、Val307三个残基与3MBA形成较强的相互作用能.而9PC、Com1存在的三元体系中,T7Loop区域周围残基如Asp199、Leu200、Leu209均能与配体形成很强的相互作用能.尤其是SaFtsZ-GDP-Com1三元复合物体系,能够与配体形成<-6.0 kJ·mol-1的残基有Ile197、Ile311、Asp199、Leu200.其中Leu200与配体的相互作用能最低,仅为-8.89 kJ·mol-1,这对Com1能够稳定存在于活性口袋的贡献是巨大的.
4 结论
应用分子动力学模拟以及相关计算方法研究了3MBA类衍生物与金黄色葡萄球菌FtsZ蛋白T7Loop区周围活性口袋的作用规律.研究表明,在分子动力学模拟中,SaFtsZ-GDP二元复合物体系和抑制剂存在的三元复合物体系均能形成低能的动态平衡状态.与二元体系和3MBA三元体系相比,配体9PC、Com1存在的三元复合物体系中,9PC、Com1均能够限制FtsZ蛋白的构象,同时对蛋白质二级结构、关键残基质心距、活性口袋体积的变化起到关键性的控制作用,使T7Loop区底物通道保持稳定存在,表现为C端序列不能移向N端序列, FtsZ蛋白组装开关呈关闭状态.主要原因是9PC、 Com1既能与Val207、Leu209、Asn263等残基形成氢键,又能与活性口袋中Gly193、Gly196、Leu200、Thr309等残基形成关键性的疏水作用,从而稳定存在于活性口袋.而3MBA与FtsZ蛋白形成疏水作用的残基相对较少,因此疏水作用较弱,影响3MBA在活性口袋的稳定性,随着模拟时间的延长,3MBA逐渐远离活性口袋区域.对三种配体的生物活性测试结果、分子对接结果以及MM-PBSA相对结合自由能计算结果比较发现,三种实验结果反映的规律相一致.上述研究结果为深入认识抑制剂对FtsZ蛋白的抗菌作用机制,以及以FtsZ蛋白为靶点的药物设计、筛选和产品开发提供了重要的理论依据.
(1)Freiberg,C.;Brotz-Oesterhelt,H.Drug.Discov.Today2005,10 (13),927.doi:10.1016/S1359-6446(05)03474-4
(2)Walsh,C.Nature2000,406(6797),775.doi:10.1038/35021219
(3)Walsh,C.Nat.Rev.Microbiol.2003,1(1),65.doi:10.1038/ nrmicro727
(4)Buddelmeijer,N.;Beckwith,J.Curr.Opin.Microbiol.2002,5 (6),553.doi:10.1016/S1369-5274(02)00374-0
(5)Qi,Y.K.Design,Synthesis andAntibacterial Evaluation of Novel 3-MBADerivatives Targeting FtsZ.Master Thesis, Shangdong University,Jinan,2012.[齐昀坤.以FtsZ为靶点的新型3-MBA衍生物的设计、合成及抗菌活性研究[D].济南:山东大学,2012.]
(6)Kapoor,S.;Panda,D.Expert.Opin.Ther.Targets2009,13(9), 1037.doi:10.1517/14728220903173257
(7)Lu,C.;Reedy,M.;Erickson,H.P.J.Bacteriol.2000,752(1), 164.doi:10.1128/JB.182.1
(8)Matsui,T.;Han,X.;Yu,J.;Yao,M.;Tanaka,I.J.Biol.Chem.2014,289(6),3501.doi:10.1074/jbc.M113.514901.
(9)Dai,K.;Lutkenhaus,J.J.Bacteriol.1991,173(11),3500.
(10)Wang,J.;Galgoci,A.;Kodali,S.;Herath,K.B.;Jayasuriya,H.; Dorso,K.;Vicente,F.;González,A.;Cully,D.;Bramhill,D.; Singh,S.J.Biol.Chem.2003,278(45),44424.doi:10.1074/jbc. M307625200
(11)Beuria,T.K.;Santra,M.K.;Panda,D.Biochemistry2005,44 (50),16584.doi:10.1021/bi050767+
(12)Lopus,M.;Panda,D.FEBS J.2006,273(10),2139.doi: 10.1111/j.1742-4658.2006.05227.x
(13)Jaiswal,R.;Beuria,T.K.;Mohan,R.;Mahajan,S.K.;Panda,D. Biochemistry2007,46(14),4211.doi:10.1021/bi602573e
(14)Constantine,G.H.;Karchesy,J.J.;Franzblau,S.G.;LaFleur,L. E.Fitoterapia2001,72(5),572.doi:10.1016/S0367-326X(01) 00272-6
(15)Bhattacharya,A.;Jindal,B.;Singh,P.;Datta,A.;Panda,D. FEBS J.2013,280(18),4585.doi:10.1111/febs.12429.Epub 2013Aug 2.
(16)Lappchen,T.;Hartog,A.F.;Pinas,V.A.;Koomen,G.J.;den Blaauwen,T.Biochemistry2005,44(2),7879.
(17)Stokes,N.R.;Sievers,J.;Barker,S.;Bennett,J.M.;Brown,D. R.;Collins,I.;Errington,V.M.;Foulger,D.;Hall,M.;Halsey, R.;Johnson,H.;Rose,V.;Thomaides,H.B.;Haydon,D.J.; Czaplewski,L.G.;Errington,J.J.Biol.Chem.2005,280(48), 39709.doi:10.1074/jbc.M506741200
(18)Ito,H.;Ura,A.;Oyamada,Y.;Tanitame,A.;Yoshida,H.; Yamada,S.;Wachi,M.;Yamagishi,J.Microbiol.Immunol.2006,50(10),759.doi:10.1111/j.1348-0421.2006.tb03851.x
(19)Beuria,T.K.;Singh,P.;Surolia,A.;Panda,D.Biochem.J.2009,423(1),61.doi:10.1042/BJ20090817.
(20)Ohashi,Y.;Chijiiwa,Y.;Suzuki,K.;Takahashi,K.;Nanamiya, H.;Sato,T.;Hosoya,Y.;Ochi,K.;Kawamura,F.Journal of Bacteriology1999,181(4),1348.
(21)Haydon,D.J.;Stocks,N.R.;Ure,R.;Galbraith,G.;Bennett,J. M.;Brown,D.R.;Baker,P.J.;Barynin,V.V.;Rice,D.W.; Sedelnikova,S.E.;Heal,J.R.;Sheridan,J.M.;Aiwale,S.T.; Chauhan,P.K.;Srivastava,A.;Taneja,A.;Collins,I.;Errington, J.;Czaplewski,L.G.Science2008,321(5896),1673.doi: 10.1126/science.1159961.
(22)Stokes,N.R.;Baker,N.;Bennett,J.M.;Berry,J.;Collins,I.; Czaplewski,L.G.;Logan,A.;Macdonald,R.;Macleod,L.; Peasley,H.;Mitchell,J.P.;Nayal,N.;Yadav,A.;Srivastava,A.; Haydon,D.J.Antimicrobial Agents Chemotherapy2012,57(1), 317.doi:10.1128/AAC.01580-12
(23)Ji,D.;Lu,J.R.;Xin,C.W.;Peng,C.Y.;Mu,J.B.;Li,J.F. Chin.J.Org.Chem.2012,33,1062.[戢 丹,卢俊瑞,辛春伟,彭春勇,穆江蓓,李建发.有机化学,2012,32,1062.]
(24)Ji,D.;Lu,J.R.;Lu,B.W.;Xin,C.W.;Mu,J.B.;Li,J.F.;Peng, C.Y.;Bao,X.R.Bioorganic&Medicinal Chemistry Letters2013,33(5),1062.
(25)Hu,X.;Compton,J.R.;AbdulHameed,M.D.M.;Marchand,C. L.;Robertson,K.L.;Leary,D.H.;Jadhav,A.;Hershfield,J.R.; Wallqvist,A.;Friedlander,A.M.;Legler,P.M.J.Med.Chem2013,56(13),5275.
(26)Tan,C.M.;Therien,A.G.;Lu,J.;Lee,S.H.;Caron,A.;Gill,C. J.;Lebeau-Jacob,C.;Benton-Perdomo,L.;Monteiro,J.M.; Pereira,P.M.;Elsen,N.L.;Wu,J.;Deschamps,K.;Petcu,M.; Wong,S.;Daigneault,E.;Kramer,S.;Liang,L.Z.;Maxwell, E.;Claveau,D.;Vaillancourt,J.;Skorey,K.;Tam,J.;Wang,H.; Meredith,T.C.;Sillaots,S.;Wang-Jarantow,L.;Ramtohul,Y.; Langlois,E.;Landry,F.;Reid,J.C.;Parthasarathy,G.;Sharma, S.;Baryshnikova,A.;Lumb,K.J.;Pinho,M.G.;Soisson,S. M.;Roemer,T.Sci.Trans.Med.2012,4(126),126ra35.doi: 10.1126/scitranslmed.3003592.
(27)Srinivasan,J.;Cheatham,T.E.;Cieplak,P.J.Am.Chem.Soc1998,120(51),9401.
(28)Sousa da Silva,A.W.;Vranken,W.F.BMC Research Notes2012,5(1),367.
(29)Pierce,B.G.;Wiehe,K.;Hwang,H.;Kim,B.H.;Vreven,T.; Weng,Z.Bioinformatics2014,30(12),1771.doi:10.1093/ bioinformatics/btu097.
(30)Hess,B.;Kutzner,C.;Van Der Spoel,D.;Lindahl,E.J.Chem. Theory Comput.2008,4(3),435.doi:10.1021/ct700301q
(31)Hornak,V.;Abel,R.;Okur,A.;Strockbine,B.;Roitberg,A.; Simmerling,C.Proteins:Structure,Function and Bioinformatics2006,65(3),712.doi:10.1002/prot.v65:3
(32)Jorgensen,W.L.;Chandrasekhar,J.;Madura,J.D.;Madura,J. D.;Impey,R.W.;Klein,M.L.J.Chem.Phys.1983,79,926. doi:10.1063/1.445869
(33)Essmann,U.;Perera,L.;Berkowitz,M.L.;Darden,T.;Lee,H.; Pedersen,L.G.J.Chem.Phys.1995,103(19),8577.doi: 10.1063/1470117
(34)Li,B.;Liu,M.;Hu,W.X.Acta Phys.-Chim.Sin.2010,26(1), 206. [李 博,刘 明,胡文祥.物理化学学报,2010,26(1), 206.]doi:10.3866/PKU.WHXB20100117
(35)Hess,B.;Bekker,H.;Berendsen,H.J.C.;Fraaije,J.G.E.M.J. Comput.Chem.1997,18(12),1463.
(36)Ai,Y.X.Study on Molecular Mechanisms of FabI Inhibitor and Drug Design.Master Thesis,Tianjin University of Technology, Tianjin,2014.[艾义新.FabI抑制剂的分子作用机制及药物设计研究[D].天津:天津理工大学,2014.]
(37)Durrant,J.D.;de Oliveira,C.A.F.;McCammon,J.A.J.Mol. Graph.Model.2011,29(5),773.
(38)Ai,Y.X.;Lu,J.R.;Xin,C.W.;Mu,J.B.;Yang,X.Y.;Zhang, H.Acta Phys.-Chim.Sin.2014,30(3),559.[艾义新,卢俊瑞,辛春伟,穆江蓓,杨旭云,张 贺.物理化学学报,2014,30(3), 559.]doi:10.3866/PKU.WHXB201401132
(39)Chong,L.T.;Duan,Y.;Wang,L.Proc.Natl.Acad.Sci.1999,96,14330.
(40)Kumari,R.;Kumar,R.;Lynn,A.J.Chem.Inf.Model.2014,54 (7),1951.
(41)Kabsch,W.;Sander,C.Biopolymers1983,22(12),2577.
(42)DeLano,W.L.The PyMOL Molecular Graphics System; DeLano Scientific:San Carlos,CA,USA,2002.
(43)Laskowski,R.A.;Swindells,M.B.J.Chem.Inf.Model.2011,51(10),2778.
Molecular Dynamics Simulation and Antibacterial Mechanism of 3MBA Derivatives as FtsZ Protein Inhibitors
ZHANG He LU Jun-Rui*MU Jiang-Bei LIU Jin-Biao YANG Xu-Yun WANG Mei-Jun ZHANG Rui-Bo
(School of Chemistry and Chemical Engineering,Tianjin University of Technology,Tianjin 300384,P.R.China)
In this paper,the complex stability,secondary structure,protein conformation,residue distance, active site volume,and binding free energy of the binary complex of filamentous sensitivity division protein Z of Staphylococcus aureus-guanosine diphosphate(SaFtsZ-GDP)and the ternary complex of SaFtsZ-GDP-3MBA(3-methoxybenzamide)derivatives were studied using molecular dynamics simulations,definition of secondary structure of proteins(DSSP),pocket volume measurer(POVME),and the molecular mechanics Poisson-Boltzmann surface area(MM-PBSA)method.The results show that the SaFtsZ-GDP binary complex was unstable in the absence of inhibitor,and the residues of its T7Loop area(residues 203-209)show obvious fluctuations.The secondary structure of the protein in the T7Loop area also changes significantly,the active pocket volume decreases dramatically and the substrate channel size becomes narrow and unstable.However, the SaFtsZ-GDP-3MBA derivatives ternary complex looks completely different in the presence of inhibitor PC190723 or Compound 1.Both of the inhibitors can form hydrogen bonds and hydrophobic interactions with high affinity to FtsZ.However,the ligand only forms hydrophobic interactions with partial residues of the active site as a function of simulation time in the SaFtsZ-GDP-3MBA ternary complex.This low affinity means that3MBAcannot stably exist in the active site,and so the antibacterial activity of 3MBAis significantly lower than that of PC190723 or Compound 1.The study shows the antibacterial mechanism and effect of 3MBAderivatives on FtsZ,and provides an important theoretical basis for inhibitor structural optimization,development and applications.©Editorial office ofActa Physico-Chimica Sinica
3-MBAderivative;FtsZ;Molecular dynamics simulation;Antibacterial mechanism; Binding free energy
O641
10.3866/PKU.WHXB201501061www.whxb.pku.edu.cn
Received:October 27,2014;Revised:January 6,2015;Published on Web:January 6,2015.
∗Corresponding author.Email:lujunrui@tjut.edu.cn;Tel:+86-22-60216638.
The project was supported by the National Natural Science Foundation of China(21476174,21176194).
国家自然科学基金(21476174,21176194)资助项目
