谷氨酰胺转胺酶基因的结构分析及定点突变
2015-12-28
(中南民族大学生命科学学院,湖北武汉430074)
谷氨酰胺转胺酶(transglutaminase,TG),即蛋白质-谷氨酸-γ-谷氨酰胺基转移酶(EC2.3.2.13),可以催化蛋白质和氨基酸之间交联、蛋白分子内谷氨酰胺基的水解以及蛋白分子内及分子间的连接,可以改善食品结构、提高食品弹性、保持食品营养。TG 可从植物、动物、微生物中获得[1-2],其中微生物来源的谷氨酰胺转胺酶(MTG)是TG 获取的主要渠道。酶修饰法反应条件温和、副作用少、作用快捷安全,广泛应用于食品改造[3]。因此,MTG 作为一种新型的食品添加剂,在改造及生产新型蛋白食品方面具有良好的发展前景[4]。
蛋白质三维结构模型的确定和分析在酶促反应、突变分析、蛋白质复合物和酶活位点的界面相互作用分析等方面有着重要的作用。但蛋白质序列数据库中多肽记录的数量与结构数据库中结构记录的数量都很少,需要发展计算机预测方法来预测蛋白质的三维结构。目前,三维结构的主要预测方法有:比较建模(comparative modeling,CM)、穿线(threading)及自由建模(free modeling)。其中比较建模法是最为成功的方法,它是利用相似性的已知结构来预测未知序列的三维结构,而常用的就是利用网络直接搜索PDB数据库和SWISS-MODEL服务器。
作者在此根据分离得到的吸水链霉菌的MTG 基因,预测其三维结构,确定其酶活中心,并构建定点突变体。
1 实验
1.1 材料
1.1.1 菌株及载体
吸水链霉菌(Streptomycessp.)H197菌株、质粒pET-22b(+)、E.coliBL21(DE3),中南民族大学生命科学学院生物工程实验室;pMD19-T,TaKaRa公司。
1.1.2 培养基
LB液体培养基:NaCl 1%,胰蛋白胨1%,酵母提取物0.5%,pH 值7.0。
高氏一号固体培养基:可溶性淀粉2%,MgSO4·7H2O 0.05%,NaCl 0.05%,KNO30.1%,K2HPO40.05%,琼脂2.5%,pH 值7.2~7.4。
发酵培养基:葡萄糖2.5%,蛋白胨2.5%,酵母膏0.5%,K2HPO40.2%,KH2PO40.2%,MgSO4·7H2O 0.2%,CaCO30.5%,pH 值7.0。
1.1.3 试剂
BamHⅠ、HindⅢ、Amp+、Ex-Taq 酶、pfu酶,TaKaRa 公司;凝胶回收试剂盒,Axygen 公司;DL2000DNA marker、λHindⅢ,BBI公司;其它试剂均为国产分析纯。
1.1.4 引物
引物由上海生工生物工程有限公司合成,测序由擎科生物公司完成。扩增引物f1(正向)、f2(反向)及加酶切位点的扩增引物F1(正向)、F2(反向)由DNAMAN5.0设计,突变引物R1(正向)、R2(反向)为PrimerX 在线设计,R1和R2是两条互补的引物,在互补区域内引入突变位点。
f1:5′-ATCCATGTACAAACGCCGGAGATT-3′f2:5′-CTTACGGCCAGCCCTGCTT-3′
F1:5′-CGAGGATCCATGTACAAACGCCGGAGATT-3′(下划线为BamHⅠ酶切位点)
F2:5′-CGCAAGCTTACGGCCAGCCCTGCTT-3′(下划线为HindⅢ酶切位点)
R1:5′-CAAAACCATATGGACCAAA·GCCAACCACTATCACG-3′(“·”标记为突变位点)
R2:5′-CGTGATAGTGGTTGGCTTTGGTCCATATGGTTTTG-3′(“·”标记为突变位点)
1.2 方法
1.2.1 目的基因片段的获得
将实验室-70 ℃保藏的吸水链霉菌H197 在高氏一号固体培养基上30 ℃培养3~4d;挑取单菌落,接种到5 mL LB 液体培养基中,于30 ℃、200r·min-1培养3~5d;再挑取单菌落,接种到20mL发酵培养基上进行扩大培养,于30℃、200r·min-1培养4~5d[5];采用优化的SDS方法[6]提取链霉菌总DNA,经0.8%琼脂糖凝胶电泳进行鉴定检测;以其为模板,以f1、f2为引物,扩增MTG 基因。
反应体系(25μL):10×PCR buffer(含Mg2+)2.5μL,dNTP(10mmol·L-1)2μL,f1、f2(10mmol·L-1)各1.0μL,Ex-Taq酶(2.5U·μL-1)0.5μL,总DNA 1.0μL,ddH2O 17μL。
反应条件:预变性94 ℃5min;94 ℃40s,58 ℃60s,72℃40s,30个循环;72℃延伸10min,10℃保温20min。PCR 产物经1%琼脂糖凝胶电泳检测,大孔径切胶回收,连接pMD19-T 载体进行测序。
1.2.2 目的基因片段的序列比对及分析
将测序后的序列(完整的ORF)在NCBI上进行序列比对。通过文献找到这个蛋白的活性位点(Cys64-Asp255-His274)[7];通过序列比对,找到目的蛋白的可能的酶活位点Cys151-Asp342-His361。利用ExPASy对序列进行MTG 编码蛋白的理化性质分析,并在线预测MTG 的信号肽序列、糖基化位点。
1.2.3 目的蛋白的三维结构预测
通过SWISS-MODEL获取PDB格式的蛋白三维结构[8-10],根据数据库比对结果,在构建的三维结构中找到可能活性位点。并根据氨基酸的性质,确定突变其中一个位点,研究其对酶活性的影响。
1.2.4 定点突变
采用重叠延伸PCR(SOE-PCR)技术进行定点突变,其原理与过程见图1。
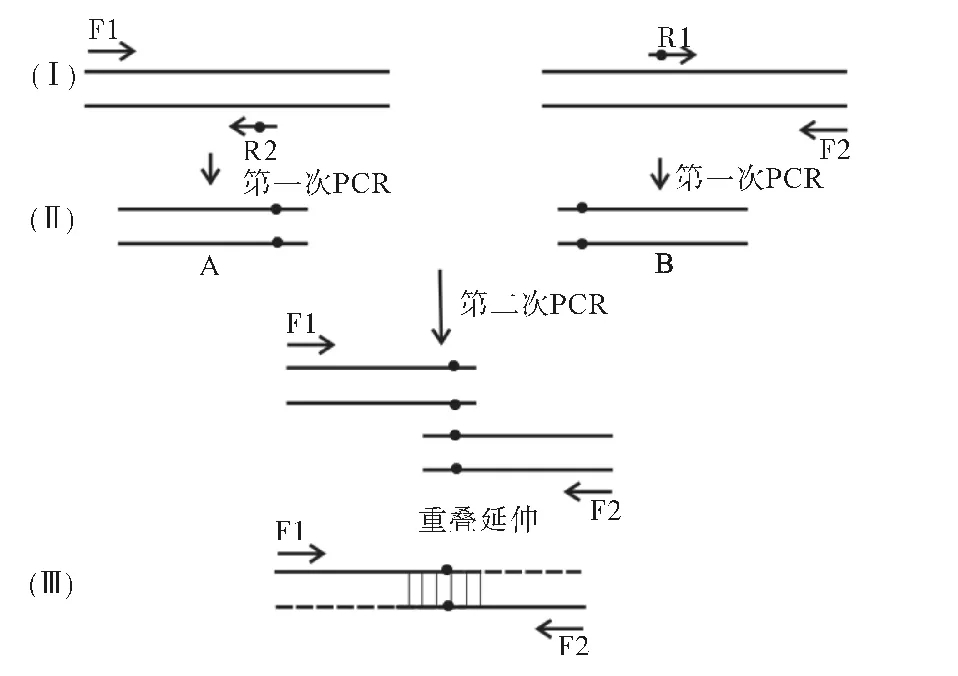
图1 SOE-PCR原理与过程Fig.1 Principle and process of SOE-PCR
1)以构建的pMD19-T-MTG 质粒作为模板,以F1、R2、F2、R1为引物,分别扩增出上下游片段A、B。
反应体系:10×PCR buffer(含Mg2+)2.5μL,dNTP(10 mmol·L-1)2μL,F1、R2(F2、R1)(10 mmol·L-1)各1.0μL,pfu酶(2.5 U·μL-1)0.5 μL,总DNA 1.0μL,ddH2O 17μL。
反应条件:预变性94 ℃5min;94 ℃40s,60 ℃40s,72℃40s,25个循环;72℃延伸10min,10℃保温20min。PCR 产物经1%琼脂糖凝胶电泳检测,然后分别大孔径切胶回收,-20 ℃保存。
2)将所得2份PCR 产物互为模板及引物,进行第二次PCR 扩增。
反应体系:F1、F2(10mmol·L-1)各1.0μL,上游目的片段A 1.0 μL,下游目的片段B 1.0 μL,ddH2O 16μL,其它同第一次PCR。
反应条件:同第一次PCR。PCR 产物经1%琼脂糖凝胶电泳检测,大孔径切胶回收,-20 ℃保存。
1.2.5 突变工程菌的构建
pET-22b(+)质粒及PCR 产物的双酶切:分别在37 ℃下用酶切体系处理pET-22b(+)质粒及PCR 产物6h,取3μL进行1%琼脂糖凝胶电泳检测,大孔径切胶回收,-20 ℃保存。
酶切体系:10×K buffer 5.0μL,BamHⅠ1.0 μL,HindⅢ1.0μL,ddH2O 8.0μL,pET-22b(+)质粒35μL(PCR 产物35μL),37 ℃,6h。
重组质粒的连接:将双酶切后的PCR 产物8.0 μL、线性化pET-22b(+)质粒5.0μL、10×T4buffer 1.5μL、ddH2O 0.5μL、T4DNA ligase 1.0μL 16 ℃过夜,采用CaCl2法将连接产物转化到感受态细胞E.coliBL21(DE3)中,采用蓝白斑法,挑取阳性菌落进行菌落PCR 验证,阳性转化子测序。
2 结果与讨论
2.1 目的基因片段的检测结果
吸水链霉菌H197总DNA 和MTG 基因扩增产物的电泳图谱见图2。
由图2a可以看到,在23kbp处有特异性片段,初步确定DNA 提取成功。由图2b可以看到,MTG 基因扩增产物出现1 200bp左右的目的基因片段。
2.2 目的基因片段的序列比对及检测结果
在NCBI中进行protein blast,跟一株已知结构的茂原链轮丝菌进行比对,根据茂原链轮丝菌基因的结构,找到可能酶活位点Cys151-Asp342-His361(图3中的方框)。图中阴影部分为MTG 的α螺旋和β折叠区域。
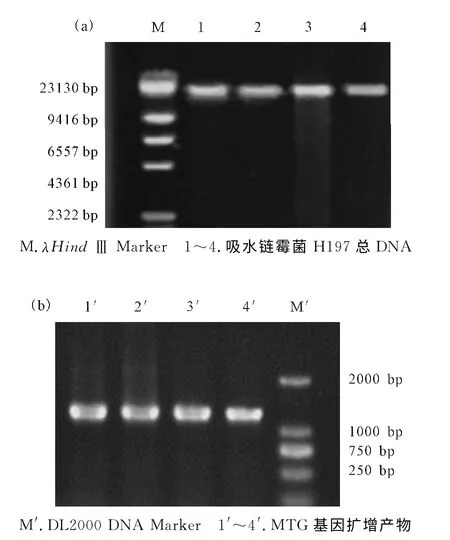
图2 吸水链霉菌H197总DNA(a)和MTG 基因扩增产物(b)的电泳图谱Fig.2 Electrophorogram of Streptomyces sp.H197genomic DNA(a)and PCR product of gene MTG(b)

图3 目的基因片段序列比对Fig.3 Alignment of objective gene fragment sequence
经ExPASy服务器在线分析MTG 的氨基酸组成,结果如表2所示。
MTG 蛋白共418 个氨基酸长度,预期大小为46 635.95Da,PI点为7.08,GC 含量为61.26%,通过http://www.cbs.dtu.dk/services/SignalP-2.0/在线预测蛋白质信号肽,发现MTG 信号肽含有29个氨基酸。通过http://www.cbs.dtu.dk/service/NetOGlyc/预测MTG 蛋白O-糖基化修饰位点时发现,该序列含有苏氨酸糖基化位点,但其G 值低于可被糖基化的最小阈值,所以不被糖基化。通过http://www.cbs.dtu.dk/service/NetNGlyc-1.0/检测也未发现N-糖基化位点。表明表达的蛋白大小不会改变。
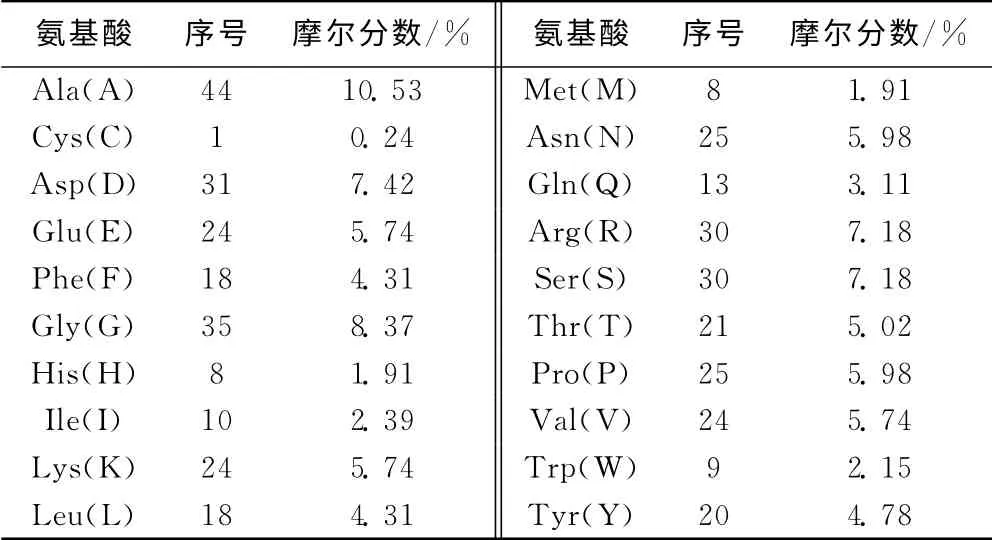
表2 吸水链霉菌H197中MTG 的氨基酸组成Tab.2 Amino acid composition of MTG from Streptomyces sp.H197
2.3 目的蛋白的三维结构预测(图4)
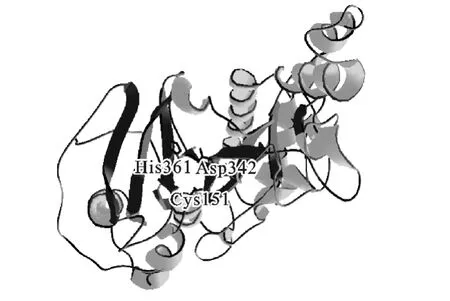
图4 MTG 三维结构预测Fig.4 Three-dimensional structure prediction of MTG
通过SWISS-MODEL构建的三维结构模型分析,MTG 分子主要由α螺旋区(灰色)和β折叠区(黑色)构成,α螺旋区相对分布在分子的外部,而β折叠区相对分布在分子内部,其中两个活性位点都在β折叠区。预测的活性中心在分子内部相对靠拢,形成一个内陷的空间。
2.4 定点突变
以构建的pMD19-T-MTG 质粒为模板,以F1、R2、F2、R1为引物,分别扩增出上下游目的片段A、B,其中上游目的片段A 大小为1 117bp,下游目的片段B大小为186bp,经1%的琼脂糖凝胶电泳检测,在相应大小的位置检测到了特异性片段(图5a、b)。经重叠延伸后,在1 200bp左右的位置找到了单一的目的条带(图5c),与预期目的片段大小相当,初步确定已获得目的突变片段。
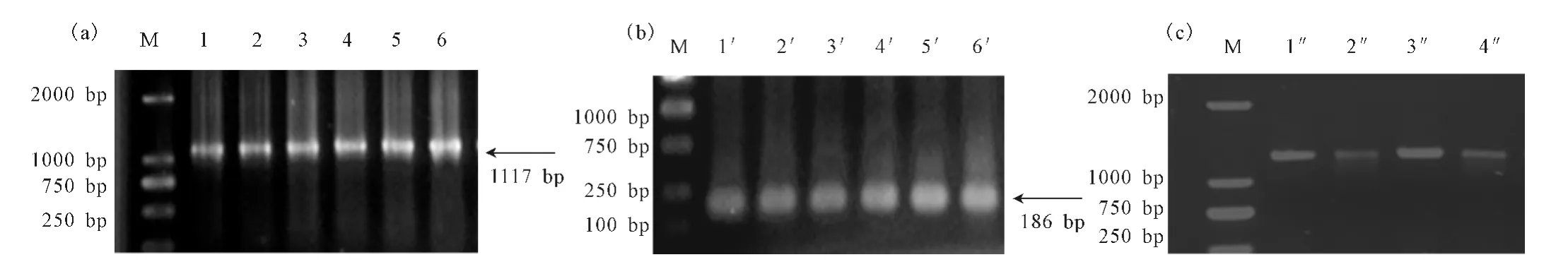
图5 目的片段A(a)、B(b)和SOE-PCR产物(c)的电泳图谱Fig.5 Electrophorogram of objective fragment A(a),objective fragment B(b)and SOE-PCR product(c)
2.5 突变工程菌的构建
将pET-22b(+)质粒和突变后的PCR 基因产物采用BamHⅠ、HindⅢ进行双酶切,回收酶切后的产物,将连接后重组质粒pET-22b(+)-MTG 采用CaCl2法转化感受态细胞E.coliBL21(DE3),使用蓝白斑筛选,提取阳性转化子进行单、双酶切验证,经1%琼脂糖凝胶检测,在相应位置检测到预期大小的目的片段(图6)。
将突变型基因测序结果与野生型基因序列对比,在1 090位和1 092位的C均变成了A(图7),与预期结果相同。表明已成功应用SOE-PCR 技术进行了突变,且突变工程菌pET-22b(+)-MTG 已成功构建。
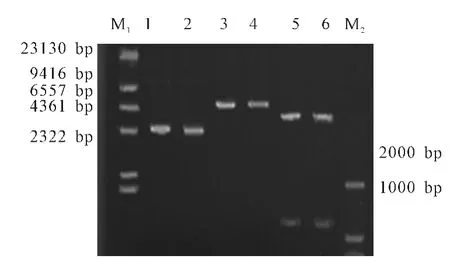
图6 单酶切及双酶切电泳图谱Fig.6 Electrophorogram of single enzyme digestion and double enzyme digestion
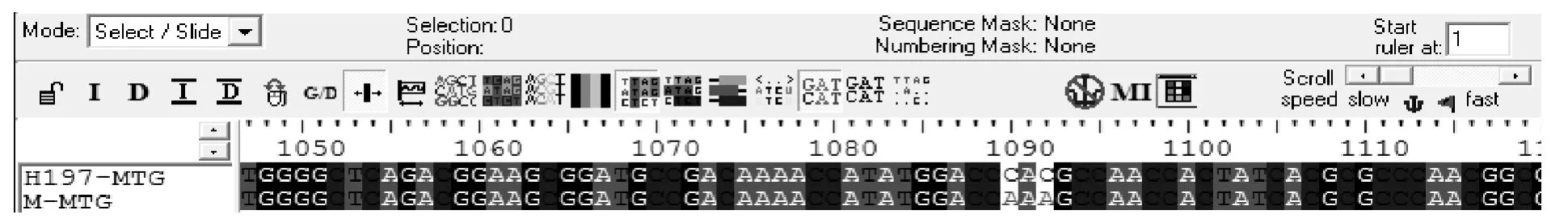
图7 突变基因序列与野生型基因序列比对Fig.7 Alignment of mutant gene sequence and wild gene sequence
2.6 讨论
定点突变可以便捷地改造优化基因,是研究基因与蛋白质结构与功能的重要手段之一。陈熙等[11]利用定点突变技术将人溶菌酶进行定点突变并在真核表达系统中进行体外表达,得到较野生型更高活力的突变体。郝大利等[12]定点突变大肠杆菌aroG基因,并与trpBA基因串联表达,获得了高产色氨酸菌株。
SOE-PCR技术自1989年由Horton等构建以来已成为主要的定点突变技术[13]。孙万菊等[14]利用SOE-PCR 技术成功突变幽门螺杆菌热休克蛋白60基因,在原核表达载体中获得了高效表达的可溶性hsp60蛋白。在使用SOE-PCR 技术的时候应注意DNA 聚合酶的质量,应避免使用普通的Taq酶,而应使用高保真Taq酶,因为普通Taq酶会在PCR 产物的3′末端加上一个A,从而造成移码突变,本实验采用的pfu高保真酶避免了此问题的发生。SOE-PCR 技术因原理简单、操作方便、适用性广而被广泛用于基因克隆的研究。本实验成功地通过SOE-PCR 技术实现了定点突变,构建了突变体。
3 结论
通过预测谷氨酰胺转胺酶基因的三维结构,同源比对确定其可能的酶活中心,利用SOE-PCR 技术定点突变其中一个活性位点,并构建了突变工程菌,为后续研究其活性位点的功能奠定了基础。
[1]FALCONE P,SERAFINI-FACASSINI D,DUCA S D.Comparative studies of transglutaminase activity and substrates in different organs ofHelianthustuberosus[J].Plant Physiol,1993,142(3):265-273.
[2]ANDO H,ADACHI M,UMEDA K,et al.Purification and characterisrics of a novel transgluatminase derived from microorganisms[J].Agriculture Biological Chemistry,1989,53:2163-2617.
[3]ZHU Y,RINZEMA A,TRAMPER J,et al.Microbial transglu-taminase-A review of its production and application in food processing[J].Applied Microbiology and Biotechnology,1995,44(3-4):277-282.
[4]常中义,江波.微生物谷氨酰胺转胺酶的应用进展[J].食品科学,2002,21(9):6-8.
[5]何冬兰,张莹,彭宝玉.重组谷氨酰胺转胺酶基因的原核表达研究[J].中南民族大学学报(自然科学版),2010,29(4):32-36.
[6]刘炳辉,曹远银,闫建芳,等.6种链霉菌基因组DNA 提取方法比较[J].河南农业科学,2008,(10):86-89.
[7]KASHIWAGI T,YOKOYAMA K,ISHIKAWA K,et al.Crystal structure of Microbial transglutaminase fromStreptoverticillium mobaraense[J].J Biol Chem,2002,277(46):44252-44260.
[8]ARNOLD K,BORDOLI L,KOPP J,et al.The SWISS-MODEL workspace:A web-based environment for protein structure homology modeling[J].Bioinformatics,2006,22(2):195-201.
[9]SCHWEDE T,KOPP J,GUEX N,et al.SWISS-MODEL:An automated protein homology-modeling server[J].Nucleic Acids Research,2003,31(13):3381-3385.
[10]GUEX N,PEITSCH M C.SWISS-MODEL and the Swiss-Pdb-Viewer:An environment for comparative protein modeling[J].Electrophoresis,1997,18(15):2714-2723.
[11]陈熙,陈毓,齐小雨,等.人溶菌酶定点突变基因在毕赤酵母中的表达及活性分析[J].江苏农业学报,2014,30(6):1396-1401.
[12]郝大利,诸葛斌,方慧英,等.大肠杆菌aroG基因的定点突变及与trpBA基因的串联表达[J].应用与环境生物学报,2013,19(5):817-821.
[13]WARRENS A N,JONES M D,LECHLER R I.Splicing by overlap extension by PCR using asymmetric amplification:An improved technique for the generation of hybrid proteins of immunological interest[J].Gene,1997,186(1):29-35.
[14]孙万菊,杜东龙,蒋宏,等.幽门螺杆菌热休克蛋白60基因的定点突变及其原核表达[J].中国病原生物学杂志,2013,8(8):695-697.
