花鲈和西伯利亚鲟生长激素/类胰岛素样生长因子-Ⅰ轴相关基因全长cDNA的克隆和序列分析
2015-12-20张志勇吴秀峰郑银桦
张志勇 薛 敏 王 嘉 吴秀峰 郑银桦 韩 芳
(1.中国农业科学院饲料研究所,国家水产饲料安全评价基地,北京 100081;2.中粮营养健康研究院有限公司,动物营养与饲料中心,北京 102209;3.农业部饲料生物技术重点开放实验室,北京 100081)
鱼类的生长是受到生长激素(GH)/类胰岛素样生长因子-Ⅰ(IGF-Ⅰ)轴的相关基因控制的。GH/IGF-Ⅰ轴主要包括GH、生长激素受体(GHR)和类胰岛素样生长因子(IGFs)以及下游IGFs受体等。GH是由脊椎动物腺垂体细胞分泌的蛋白质,是一种肽类激素。它能够促进生长、促进蛋白质和脂肪的代谢、提高食物的转化效率和调节鱼类的渗透压,是动物生长最主要的调控因子。GH与靶细胞膜表面的GHR结合后,开启细胞内的信号传导机制,促进肝脏中IGF-Ⅰ的表达和分泌,通过血液循环到达其他组织,促进细胞的生长分化[1]。
花鲈(Lateolabrax japonicus)和西伯利亚鲟(Acipenser baerii Brandt)均为我国重要的经济鱼类,在 FishBase(www.fishbase.org)中两者均被列为广盐肉食性品种,但在分类学地位上相差较远。花鲈,又称鲈鱼、七星鲈,属鲈形目,鮨科,是我国重要的海水养殖鱼类品种,年产量突破10万t,稳居我国海水养殖鱼类之首。西伯利亚鲟,属鲟型目,鲟科,是一类古老的软骨硬鳞鱼类,在鱼类的进化史上占有重要地位,有“活化石”之称,常常被作为鱼类生物学模型动物。本课题组前期在对2种鱼类利用混合植物蛋白源替代鱼粉的比较研究中发现,高植物蛋白质替代鱼粉会导致花鲈生长性能显著下降,而西伯利亚鲟的生长则不受影响[2]。这种生长性能上的差异,有可能是2种鱼类的GH/IGF-Ⅰ轴应对植物蛋白质不同的响应调控机制所致。为进一步深入研究这2种鱼类的GH/IGF-Ⅰ轴,本研究拟通过简并引物反转录聚合酶链式反应(reverse transcription-polymerase chain reaction,RT-PCR)和 cDNA末端快速扩增技术(rapid-amplification of cDNA ends,RACE)相结合的方法,克隆花鲈和西伯利亚鲟GH和GHR基因的cDNA全长序列,并进行同源性比较和系统进化树分析,为探讨花鲈和西伯利亚鲟的生长调控机理提供分子生物学基础。
1 材料与方法
1.1 试验材料
试验所用花鲈和西伯利亚鲟分别购自山东威海裕隆水产开发有限公司和北京市水产科学研究所房山基地。待试验鱼被麻醉后,在冰盘上分离出垂体、肝脏组织,立即置于液氮中,随后转移至-80℃超低温冰箱中保存。
1.2 引物设计与合成
根据美国国立生物技术信息中心(NCBI)GenBank中已知其他物种的 GH、GHR、GHRⅠ和GHRⅡ的氨基酸序列寻找相应的保守片段,采用CodeHop原理[3]设计简并引物克隆花鲈相应基因的保守片段。根据扩增到的保守片段,使用软件Primer 5.0设计用于3'-和5'-RACE扩增的特异性引物。根据GenBank中西伯利亚鲟GH(Gen-Bank登录号FJ428829)和GHR(GenBank登录号FJ428827)的3'末端部分序列设计用于5'-RACE扩增的特异性引物。所有引物均由生工生物工程(上海)有限公司合成。本研究中所用到的引物序列及相应的退火温度(Tm)见表1。
1.3 总RNA提取
从-80℃冰箱中取出2种试验鱼的垂体和肝脏,经液氮研磨后用RNAiso Plus(TaKaRa,大连)分别抽提总RNA。总RNA用变性琼脂糖凝胶电泳显示28S和18S条带,验证所提取的总RNA的完整性。采用PowerWaveTMXS2全波长酶标仪(BioTek,美国)检测其在260 nm处的吸光度值与在280 nm处的吸光度值的比值(A260/A280),并对所提取的总RNA进行定量。
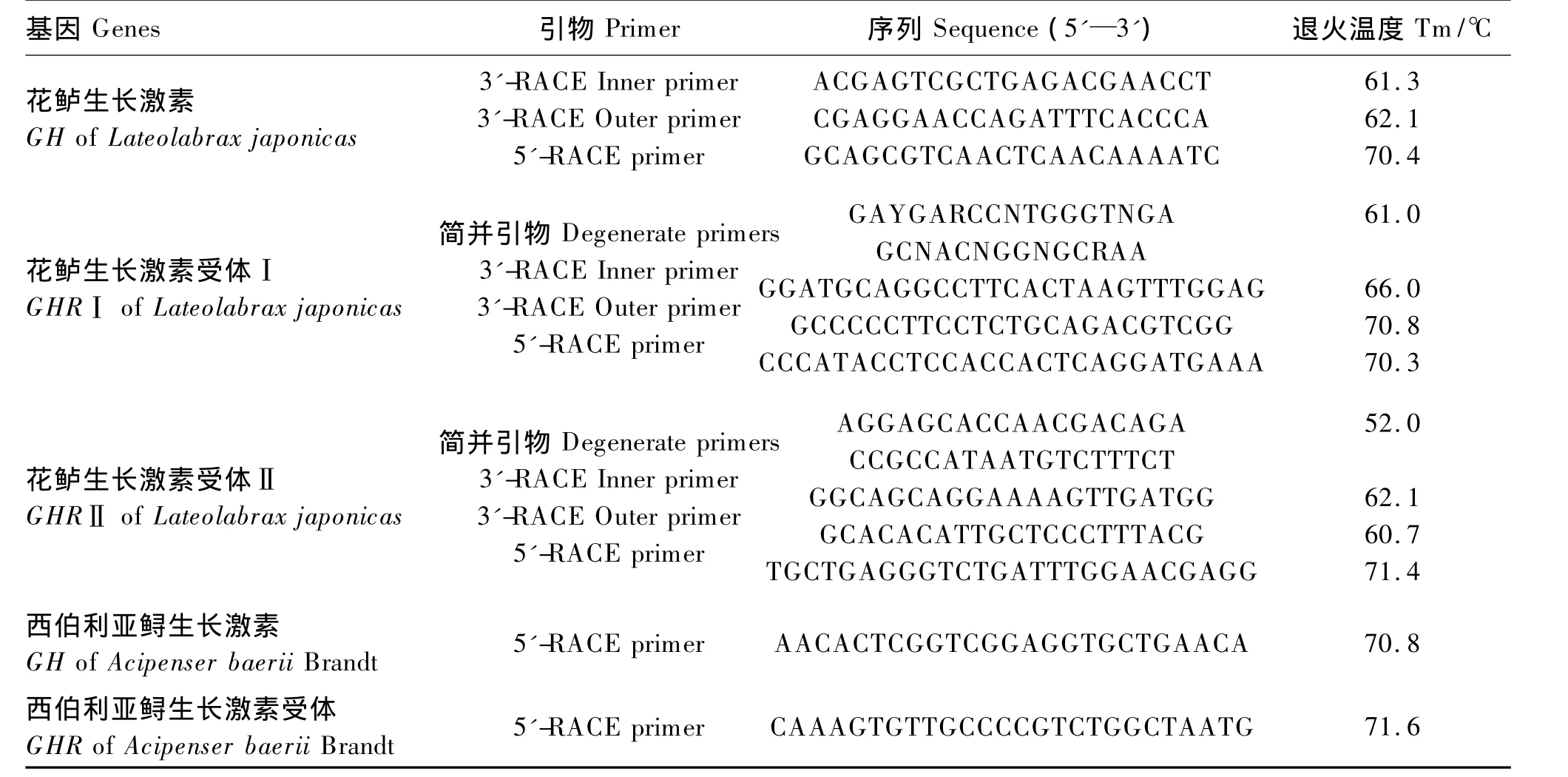
表1 本研究中所用到的引物序列及相应的退火温度Table 1 Primer sequence and Tm used in this study
1.4 保守片段克隆
cDNA第1链的合成采用PrimeScriptⅡ 1stStrand cDNA Synthesis Kit(TaKaRa,大连),每个反转录反应使用约1.0μg相应组织的总RNA,操作步骤参照说明书进行。分别采用各基因的简并引物进行保守片段PCR扩增,PCR反应体系包含合成的cDNA 第1链1μL、10×PCR Buffer 2.5μL、TaKaRa Ex Taq 0.625 U、dNTP 200 μmol/L、上游引物0.4 μmol/L、下游引物 0.4 μmol/L,加灭菌水至总体积25 uL。PCR扩增条件为:94℃,30 s;Tm,30 s;72℃,1 min,共35个循环。PCR产物经电泳后,在紫外灯下切取与目标序列大小一致的片段,使用 Agarose Gel DNA Purification Kit Ver.2.0(TaKaRa,大连)对扩增片段进行纯化。纯化产物与pMD19-T Vctor(TaKaRa,大连)连接,转化入大肠杆菌(E.coli)JM109感受态细胞(TaKa-Ra,大连)中,经蓝白斑筛选后,对有插入片段的阳性克隆进行测序(北京华大基因服务公司)。
1.5 3'-RACE 扩增
3'-RACE扩增采用3'-Full RACE Core Set Ver.2.0(TaKaRa,大连),操作步骤参照说明书进行。采用1.0μg垂体或肝脏总RNA和3'-RACE Adaptor合成cDNA第1链,经过2轮嵌套PCR反应扩增各基因的3'末端。首先以合成的cDNA第1链为模板,分别以试剂盒提供的3'-RACE Outer primer和所设计的基因特异性3'-RACE Outer primer为引物进行第1轮PCR扩增,反应条件为:94 ℃,30 s;Tm,30 s;72 ℃,1.5 min,共 35 个循环。再以100倍稀释的Outer PCR产物为模板,分别以试剂盒提供的3'-RACE Inner primer和所设计的基因特异性3'-RACE Inner primer为引物进行第2轮PCR扩增,反应条件为:94℃,30 s;Tm,30 s;72℃,1.5 min,共30个循环。对 Inner PCR产物进行电泳、切胶纯化、连接、转化和测序。
1.6 5'-RACE 扩增
5'-RACE扩增采用SMARTTMRACE cDNA Amplification Kit(Clontech,美国),操作步骤参照说明书进行。采用1.0μg垂体或肝脏总 RNA、5'-CDS Primer A和SMARTⅡ A Oligo合成cDNA第1链,以Advantage 2 PCR Enzyme System(Clontech,美国)、UPM引物和所设计的基因特异性5'-RACE引物进行PCR扩增。扩增采用Touch-Down PCR程序:94 ℃,30 s;72℃,2.5 min;5个循环;94℃,30 s;70℃,30 s;72℃,2 min;5个循环;94℃,30 s;68℃,30 s;72℃,2 min;30个循环。对PCR产物进行电泳、切胶纯化、连接、转化和测序。
1.7 序列分析、同源性比对及系统进化树构建
将测序所获得的核心片段与3'和5'区域拼接即获得了相关基因的全长序列。将拼接结果与NCBI GenBank中的已知序列作blastx同源性分析,用 DNAMAN(VERSION 5.2.2)软件分析所得cDNA序列及开放阅读框,预测编码氨基酸序列。根据GenBank中已报道的GH/IGF-Ⅰ轴相关基因的蛋白质序列用Lasergene 7.0程序中MegAlign进行多序列比对和一致性数值计算。采用MEGA 5.1 软件,选择邻接(neighbor-joining,NJ)法构建系统进化树[4]。
2 结果
2.1 cDNA全长序列和结构分析
花鲈GH cDNA全长序列及推测编码的氨基酸序列见图1。cDNA全长949 bp,包括72 bp的5'非翻译区,615 bp的开放阅读框及262 bp的3'非翻译区;开放阅读框编码204个氨基酸,预测分子质量为 23.06 ku,等电点为 7.27。经过 blastx比对发现其与其他物种GH具有很高的相似性,提交至GenBank获得登录号为JQ995145。
花鲈GHRⅠcDNA全长序列及推测编码的氨基酸序列见图2。cDNA全长 3 070 bp,包括227 bp的5'非翻译区,1 911 bp的开放阅读框及932 bp的3'非翻译区;开放阅读框编码637个氨基酸,预测分子质量为70.79 ku,等电点为4.37。经过blastx比对发现其与其他物种GHR和GHRⅠ具有很高的相似性,提交至GenBank获得登录号为JX402001。
花鲈GHRⅡcDNA全长序列及推测编码的氨基酸序列见图 3。cDNA全长 2 926 bp,包括252 bp的5'非翻译区,1 749 bp的开放阅读框及925 bp的3'非翻译区;开放阅读框编码582个氨基酸,预测分子质量为64.42 ku。经过blastx比对发现其与其他物种GHR和GHRⅡ具有很高的相似性,提交至GenBank获得登录号为JQ995146。

图1 花鲈GH cDNA全长序列和预测的氨基酸序列(GenBank登录号JQ995145)Fig.1 GH full length cDNA sequence and deduced amino acid sequence of Lateolabrax japonicas(GenBank accession No.JQ995145)
西伯利亚鲟GH cDNA全长序列及推测编码的氨基酸序列见图4。cDNA全长999 bp,包括53 bp的5'非翻译区,645 bp的开放阅读框及301 bp的3'非翻译区;开放阅读框编码214个氨基酸,预测分子质量为 24.14 ku,等电点为 5.76。经过blastx比对发现其与其他物种GH具有很高的相似性,提交至GenBank获得登录号为JX003684。
西伯利亚鲟GHR cDNA全长序列及推测编码的氨基酸序列见图5。cDNA全长2 283 bp,包括377 bp的5'非翻译区,1 716 bp的开放阅读框及190 bp的3'非翻译区;开放阅读框编码571个氨基酸,预测分子质量为63.41 ku,等电点为4.92。经过blastx比对发现其与其他物种GHR和GHRⅡ具有很高的相似性,提交至GenBank获得登录号为JX003685。
2.2 多序列比对
将花鲈和西伯利亚鲟GH的氨基酸序列与其他脊椎动物GH的氨基酸序列进行同源性分析和多序列比对,结果见图6。分析表明,花鲈GH与罗非鱼(Oreochromis niloticus)GH的同源性最高,为90.2%;与斑点叉尾(Ictalurus punctatus)、鸡(Gallus gallus)、非洲爪蜍(Xenopus laevis)、草龟(Mauremys reevesis)、西伯利亚鲟和小鼠(Mus musculus)GH的同源性分别为56.2%、39.3%、38.9% 、38.8% 、38.8% 和 36.0% 。对于西伯利亚鲟而言,其GH与草龟GH的同源性最高,为70.1%;与鸡、小鼠、非洲爪蜍、斑点叉尾、花鲈和罗非鱼 GH 的同源性分别为 66.4%、65.7%、62.6% 、44.2% 、38.8% 和 38.3% 。
将花鲈GHRⅠ的氨基酸序列与其他脊椎动物GHRⅠ或GHR的氨基酸序列进行同源性分析和多序列比对,结果见图7。分析表明,花鲈GHRⅠ与金头鲷(Sparus aurata)和黑棘鲷(Acanthopagrus schlegelii)GHRⅠ的同源性最高,均为86.2%;与西伯利亚鲟、鸡、海龟(Chelonia mydas)、非洲爪蜍及人(Homo sapiens)GHR的同源性分别为43.7% 、35.6% 、34.9% 、34.8% 和 32.2% 。

图2 花鲈GHRⅠcDNA全长序列和预测的氨基酸序列(GenBank登录号JX402001)Fig.2 GHRⅠ full length cDNA sequence and deduced amino acid sequence of Lateolabrax japonicas(GenBank accession No.JX402001)

图3 花鲈GHRⅡcDNA全长序列和预测的氨基酸序列(GenBank登录号JQ995146)Fig.3 GHRⅡ full length cDNA sequence and deduced amino acid sequence of Lateolabrax japonicas(GenBank accession No.JQ995146)

图4 西伯利亚鲟GHR cDNA全长序列和预测的氨基酸序列(GenBank登录号JX003684)Fig.4 GHR full length cDNA sequence and deduced amino acid sequence of Acipenser baerii Brandt(GenBank accession No.JX003684)
将花鲈GHRⅡ的氨基酸序列与其他脊椎动物GHRⅡ或GHR的氨基酸序列进行同源性分析和多序列比对,结果见图8。分析表明,花鲈GHRⅡ与金头鲷GHRⅡ的同源性最高,为81.4%;与罗非鱼 GHRⅡ、西伯利亚鲟 GHR、海龟 GHR、鸡GHR、非洲爪蜍GHR及人GHR的同源性分别为72.7%、39.5%、32.6%、32.1%、31.2% 和 30.8%。
将西伯利亚鲟GHR的氨基酸序列与其他脊椎动物GHR或GHRⅠ的氨基酸序列进行同源性分析和多序列比对,结果见图9。分析表明,西伯利亚鲟GHR与草鱼(Ctenopharyngodon idella)GHRⅠ的同源性最高,为49.2%;与鸡、鲤鱼(Cyprinus carpio)、海龟、小鼠、人和非洲爪蜍GHR的同源性分别为 48.4%、48.4%、47.4%、45.8%、43.4%和 43.4%。
2.3 系统进化树分析
花鲈和西伯利亚鲟GH系统进化树结果见图10。如图所示,花鲈GH与罗非鱼的GH以bootstrap值为100的支持度形成了一小分支,再以bootstrap值为94的支持度与大西洋鲑(Salmo salar)的GH合并为一支,最后以bootstrap值为100的支持度与其他硬骨鱼类形成大支;西伯利亚鲟的GH与虎螈(Ambystoma barbouri)、非洲爪蜍及其他高等动物的GH形成另外一大支。
花鲈和西伯利亚鲟GHR系统进化树结果见图11。如图所示,整个进化树分为两大支,一支为高等硬骨鱼类,一支为西伯利亚鲟、陆生动物及鸟类;花鲈的GHRⅠ与黑棘鲷和金头鲷的GHRⅠ以bootstrap值为82的支持度合并为一支,再与大菱鲆(Scophthalmus maximus)、罗非鱼及红树林鳉(Kryptolebias marmoratus)的GHRⅠ合并为支,再与鲤鱼的 GHR、草鱼的 GHRⅠ、黄颡鱼(Pelteobagrus vachellii)的GHR和南方鲇(Silurus meridionalis)GHRⅠ以bootstrap值为95的支持度合并为一大支;花鲈的GHRⅡ和金头鲷 GHRⅡ以bootstrap值为91的支持度合并为1支,再与罗非鱼、红树林鳉、虹鳟(Oncorhynchus mykiss)、大麻哈鱼(Oncorhynchus keta)、南方鲇的GHRⅡ合并为一大支,然后两大支形成一大支。西伯利亚鲟的GHR与非洲爪蜍、人、小鼠、鸡及海龟的GHR形成另外一大支。

图5 西伯利亚鲟GHR cDNA全长序列和预测的氨基酸序列(GenBank登录号JX003685)Fig.5 GHR full length cDNA sequence and deduced amino acid sequence of Acipenser baerii Brandt(GenBank accession No.JX003685)

图6 花鲈和西伯利亚鲟与其他物种GH氨基酸序列比对结果Fig.6 Alignment results of GH amino acid sequences between Lateolabrax japonicas,Acipenser baerii Brandt and other species
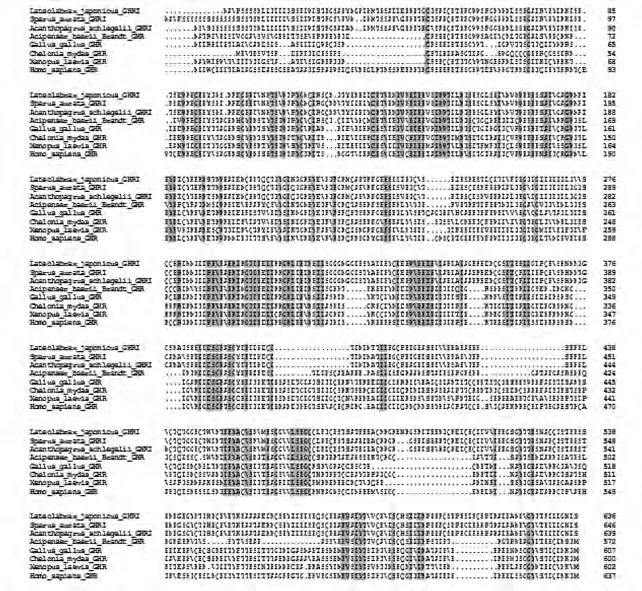
图7 花鲈与其他物种GHR或GHRⅠ氨基酸序列比对结果Fig.7 Alignment results of GHR or GHRⅠ amino acid sequences between Lateolabrax japonicas and other species

图8 花鲈与其他物种GHR或GHRⅡ氨基酸序列比对结果Fig.8 Alignment results of GHR or GHRⅡ amino acid sequences between Lateolabrax japonicas and other species
3 讨论
Pérez-Sánchez 等[5]研究证实鱼类的 GH 与促乳素(PRL)和垂体生长促乳素(SL)一起组成了GH/PRL/SL家族。这类家族具有类似的蛋白质结构,并认为它们起源于共同的祖先。在有关GH/PRL/SL家族各激素的研究中,对GH的研究比较全面和深入。本研究成功克隆了花鲈和西伯利亚鲟GH的cDNA全长序列,并推导出它们的蛋白质一级结构。花鲈GH的成熟肽中有4个半胱氨酸(C),可以形成2个二硫键,二硫键的形成对于GH的二级空间结构的维持、正常折叠和生理功能的有效发挥有着重要的作用[4]。Ayson 等[6]经研究发现鲤科鱼类和鲑鳟鱼类的GH中有2个N-糖基化位点,而花鲈只有1个N-糖基化位点,与尼罗罗非鱼和加州鲈的GH结构相似[7-8],这个糖基化位点是蛋白质转导入膜的信号,是硬骨鱼GH的特征之一。同源性分析发现,花鲈的GH与罗非鱼的相似性最高,与其结构的相似度呈现一致性。西伯利亚鲟的GH成熟肽中含有5个半胱氨酸,也可以形成2个二硫键,且与花鲈的GH类似,只含有1个N-糖基化位点。同源性分析发现,西伯利亚鲟的GH与硬骨鱼类的相似性较低,而与高等脊椎动物的相似性较高;从系统进化树(图10)分析中也发现,其与高等脊椎动物的亲缘关系较近,这可能与鲟鱼在进化中的特殊地位有关[9]。
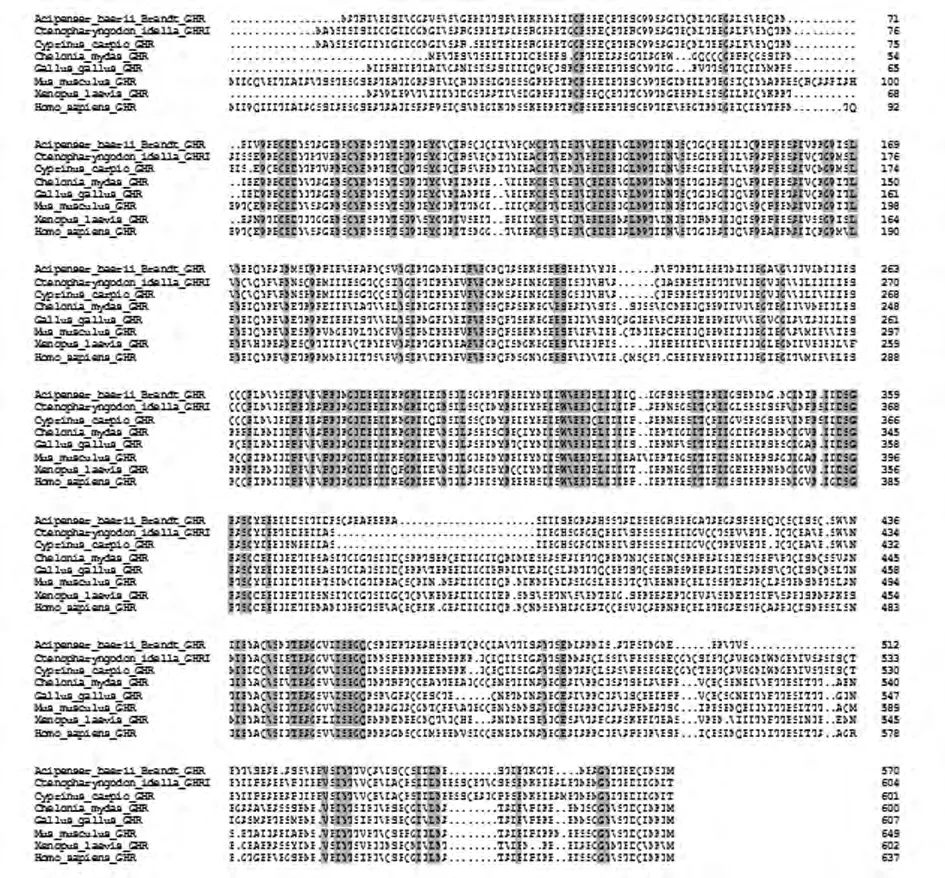
图9 西伯利亚鲟与其他物种GHR或GHRⅠ氨基酸序列比对结果Fig.9 Alignment results of GHR or GHRⅠ amino acid sequences between Acipenser baerii Brandt and other species
花鲈的GHRⅠ胞外区在靠近跨膜区的位置有一个保守的FGEFSmofit(232~235)基序,这是已知鱼类GHR的共性之一,这一结构可能在 GH与GHR结合过程中稳定空间构象,对于GH与GHR结合及其后的信号传导起非常重要的作用[10]。另外,花鲈GHRⅠ胞外区有7个保守的半胱氨酸,其中6个可形成3个二硫键,起着维持GHR胞外区段特定空间结构的作用。大多数硬骨鱼类中均存在2种类型的GHR[11-13],本研究也同样克隆到花鲈2种类型的GHR cDNA全长序列。花鲈的GHRⅡ没有FGEFSmofit基序,说明其与GHRⅠ在结构上存在较大差异,二者的同源性也较低。硬骨鱼类的2种类型的GHR虽然在结构、体内表达位置及表达量存在差异,但二者共同作用维持机体正常的生长及新陈代谢[12]。系统进化树及同源性分析表明,花鲈的GHRⅠ与黑棘鲷和金头鲷的GHRⅠ同源性最高,GHRⅡ与金头鲷的GHRⅡ同源性最高,这与二者的分类地位及食性相一致。而对于西伯利亚鲟而言,其GHR也没有FGEFSmofit基序,说明其与硬骨鱼类的GHRⅠ差异较大。同源性分析发现,西伯利亚鲟的GHR与硬骨鱼类的GHRⅠ和GHRⅡ以及与高等脊椎动物的GHR的同源性均不高(<50%),这也说明西伯利亚鲟不同于硬骨鱼类,可能只存在1种类型的GHR。此外,系统进化树分析显示,西伯利亚鲟的GHR与高等脊椎动物的亲缘关系较硬骨鱼类更近,这与对GH的分析结果一致。
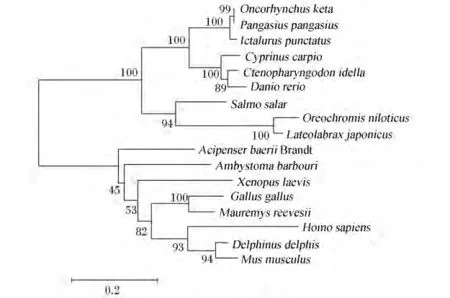
图10 花鲈和西伯利亚鲟与其他物种GH氨基酸序列系统进化树聚类分析Fig.10 Cluster analysis of phylogenetic tree of GH amino acid sequence in Lateolabrax japonicas,Acipenser baerii Brandt and other species
4 结论
①本研究成功克隆到花鲈的GH、GHRⅠ、GHRⅡ和西伯利亚鲟的GH、GHR的cDNA全长序列;多序列比对发现花鲈的GH、GHRⅠ及GHRⅡ和西伯利亚鲟的GH与其他已知物种的相应基因具有很高的相似性,但西伯利亚鲟的GHR与其他已知物种的相应基因相似性均较低。
②系统进化树分析显示,花鲈的GH、GHRⅠ及GHRⅡ与硬骨鱼类的亲缘关系较近,而西伯利亚鲟的GH和GHR与高等脊椎动物的亲缘关系较硬骨鱼类更近,这可能与鲟鱼特殊的进化地位有关。

图11 花鲈和西伯利亚鲟与其他物种GHR氨基酸序列系统进化树聚类分析Fig.11 Cluster analysis of phylogenetic tree of GHR amino acid sequence in Lateolabrax japonicas,Acipenser baerii Brandt and other species
[1] 钟欢.GH/IGF轴相关基因在不同倍性鲫鲤中的表达及与生长的相关性研究[D].博士学位论文.长沙:湖南师范大学,2012.
[2] 张志勇,薛敏,王嘉,等.混合植物蛋白质替代鱼粉对花鲈和西伯利亚鲟生长和肉质影响的比较研究[J].动物营养学报,2013,25(6):1260-1275.
[3] ROSE T M,SCHULTZ E R,HENIKOFF J G,et al.Consensus-degenerate hybrid oligonucleotide primers for amplification of distantly related sequences[J].Nucleic Acids Research,1998,26(7):1628-1635.
[4] KUMAR S,TAMURA K,JAKOBSEN I B,et al.MEGA2:molecular evolutionary genetics analysis software[J].Bioinformatics,2001,17(12):1244-1245.
[5] PÊREZ-SÁNCHEZ J,LE BAIL P Y.Growth hormone axis as marker of nutritional status and growth performance in fish[J].Aquaculture,1999,177(1/2/3/4):117-128.
[6] AYSON F G,DE JESUS E G T,AMEMIYA Y,et al.Isolation,cDNA cloning,and growth promoting activity of rabbitfish(Siganus guttatus)growth hormone[J].General and Comparative Endocrinology,2000,117(2):251-259.
[7] 马细兰,张勇,黄卫人,等.尼罗罗非鱼生长激素及其受体的cDNA克隆与mRNA表达的雌雄差异[J].动物学报,2006,52(5):924-933.
[8] 李胜杰,白俊杰,叶星,等.加州鲈生长激素和胰岛素样生长因子I cDNA的克隆及序列分析[J].广东海洋大学学报,2007,27(3):1-5.
[9] GARDINER B G.Sturgeons as living fossils[M]//ELDREDGE N,STANLEY S M.Living fossils.New York:Springer Verlag Press,1984:148-152.
[10] CALDUCH-GINER JA,MINGARRO M,DE CELISS V R,et al.Molecular cloning and characterization of gilthead sea bream(Sparus aurata)growth hormone receptor(GHR).Assessment of alternative splicing[J].Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,2003,136(1):1-13.
[11] VERY N M,KITTILSON J D,NORBECK L K,et al.Isolation,characterization,and distribution of two cDNAs encoding for growth hormone receptor in rainbow trout(Oncorhynchus mykiss)[J].Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,2005,140(4):615-628.
[12] SAERA-VILA A,CALDUCH-GINER J A,PÉREZSÁNCHEZ J.Duplication of growth hormone receptor(GHR)in fish genome:gene organization and transcriptional regulation of GHR typeⅠandⅡin gilthead sea bream(Sparus aurata)[J].General and Comparative Endocrinology,2005,142(1/2):193-203.
[13] 章力,黄希贵,焦保卫,等.南方鲇两种生长激素受体的结构分析及其组织分布和激素调节[J].动物学报,2006,52(6):1096-1106.
