内蒙古鄂尔多斯地区羊鲜乳与酸乳中乳酸菌的分离鉴定
2015-12-16张冬蕾任艳德亮亮陈红霞杨彦荣刘文俊孙天松
张冬蕾,任艳,德亮亮,陈红霞,杨彦荣,刘文俊,孙天松
(内蒙古农业大学乳品生物技术与工程教育部重点实验室,呼和浩特010018)
0 引言
羊奶营养成分最接近母乳,被称为“奶中之王”[1]。而酸羊奶营养价值更是优于鲜羊奶[2]。
乳酸菌是一群形态、代谢和生理特征不完全相同的革兰氏阳性菌。国内外关于羊奶制品中乳酸菌分离鉴定的报导很多。张文羿等[3]从青海海西州11份酸山羊奶中分离得到33株乳酸菌,Streptococcus thermophilus为优势菌群。Medina等[4]对阿根廷西北部绵羊奶中的乳酸菌分离鉴定结果表明Enterococcus为优势菌群。A Yelnetty等[5]对印度尼西亚酸山羊奶中的乳酸菌分离鉴定结果表明,杆菌数量显著多于球菌数量。
本研究采用纯培养技术对内蒙古鄂尔多斯羊鲜乳与酸乳中的乳酸菌分离、鉴定,以及16S rRNA序列分析,并分析其优势菌群,以获得鄂尔多斯羊奶制品中乳酸菌的基本信息,为羊奶中乳酸菌资源的开发利用奠定基础。
1 实验
1.1 材料和仪器
1.1.1 材料
本研究中21份羊鲜乳与酸乳样品(4份鲜绵羊奶,10份酸绵羊奶,以及7份酸山羊奶,均为鄂尔多斯地区当地土种羊)采集信息如表1所示,由实验室的研究人员于2014年7月14日-2014年7月17日在内蒙古鄂尔多斯市的不同地区采集(其中,羊酸乳为自然发酵乳,采集条件:温度为18~23℃,pH值在4.5以下,1~2 d发酵)。样品采集后低温保存,带回实验室于-80℃超低温保藏,进行后续实验。
1.1.2 常用培养基及试剂
培养基:MRS液体培养基(CM0359),MRS固体培养基(288210),M17液体培养基(CM0817B),M17固体培养基(CM0785B)。

表1 内蒙古鄂尔多斯地区羊乳制品种类及来源
试剂:提取DNA、PCR扩增以及电泳检测所用到的缓冲液和常用试剂根据参考文献[6]。
1.1.3 仪器设备
HWS28型电热恒温水浴锅,AR2202CN型电子天平,KDC-1044型低速离心机,LRH-250生化培养箱,ZHJH-C1214C型超净工作台,BX50型光学显微镜,HA-300M型全自动高压蒸汽灭菌器,SP-650型全自动高压干热灭菌器,WD-9405B型水平摇床,LL-6SFPY型真空冷冻干燥机,DYY-12型电泳仪,GDS-8000型凝胶成像仪,ND-1000型微量紫外分光光度计。
1.2 方法
1.2.1 样品采集
采集乳样时,先将其混匀,再移取1.5 mL至装有灭菌中和剂(内含0.5 g, ∶m淀粉=1∶50,质量比)的2.5 mL无菌螺口冻存管中,混匀并用封口膜封口后标号。最后将采集的乳样置于4℃便携式冰箱中以维持其低温状态,带回实验室尽快进行乳酸菌的分离鉴定。
1.2.2 乳酸菌的活菌计数
乳酸菌的活菌计数根据文献[7]中采用倾注法进行。30℃恒温培养箱中厌氧培养48 h进行乳酸菌的活菌计数。
1.2.3 乳酸菌的分离
纯化及保存:羊乳制品中乳酸菌的分离及保存根据文献[8]中方法进行,简单概括为,将样品梯度稀释后,涂布于MRS和M17两种培养基上随机选择不同菌落形态的单菌落进行乳酸菌的初步判定,然后进行进一步验证和鉴定。
1.2.4 乳酸菌基因组DNA的提取以及纯度检测
采用液氮反复冻融-CTAB法提取乳酸菌基因组DNA[9]。再用ND-1000型微量紫外分光光度计测其浓度以及OD260/280值。根据所测得的DNA原液质量浓度将其稀释至最终质量浓度为100 ng/μL,稀释液置于4℃冰箱中备用。
1.2.5 乳酸菌16S rRNA基因的PCR扩增
将1.2.4中的DNA稀释液作为PCR扩增的模板采用50 μL体系进行扩增。以细菌16S rRNA基因通用引物[10]进行16S rRNA基因的PCR扩增。
16S rRNA基因的PCR扩增引物序列如下:
FA-27F:5'-GCAGAGTTCTCGGAGTCACGAAGAGTTTGATCCTGGCTCAG-3',
RA-1495R:5'-AGCGGATCACTTCACACAGGACTACGGCTACCTTGTTACGA-3'
扩增体系:5 μL10× EasyTaq Buffer(+Mg2+);4 μL High Pure dNTPs(2.5 mmol/L);1.5 μL 引物 FA-27F(10 mmol/L);1.5 μL引 物 RA-1495R(10 mmol/L);0.5 μL EasyTaq DNA polymerase(5 U/μL);2 μL DNA模板(100 ng/μL);35.5 μL ddH2O。16S rRNA基因的PCR扩增循环参数为:94℃预变性5 min;94℃变性1 min,58℃退火1 min,72℃延伸2 min,30个循环;72℃末端延伸10 min。
扩增后,取约2 μL的PCR扩增产物用1.0%的琼脂糖凝胶电泳检测,若在1 500 bp处有清晰的扩增条带且无拖尾、弥散现象,则PCR扩增成功。
1.2.6 乳酸菌16S rDNA序列测定
乳酸菌16S rDNA序列测定由上海美吉生物技术有限公司完成。运用SeqMan(DNAStar 5.01)软件进行序列拼接后,再利用NCBI中的BLAST将拼接好的序列与数据库中已鉴定的乳酸菌16S rDNA序列进行同源性比对,以同源性大于99%为种的分界阈值将待测菌株鉴定到种或亚种。运用MEGA 4.0软件进行乳酸菌模式株和分离株系统发育关系研究和系统进化树构建。
2 结果与分析
2.1 内蒙古鄂尔多斯地区羊乳制品中乳酸菌计数结果
内蒙古鄂尔多斯地区羊乳制品中乳酸菌活菌数见表2。从中可以看出内蒙古鄂尔多斯地区采集的羊乳制品中,鲜奶样品活菌数在5.03~5.88 mL-1(对数值,下同)之间,酸奶样品活菌数在7.78~9.84 mL-1之间。其中,4份样品活菌数大于109mL-1,9份样品活菌数大于108mL-1,4份样品活菌数大于107mL-1,4份样品活菌数小于106mL-1。由乳酸菌活菌计数结果可知,所有酸奶样品活菌数均大于107mL-1,可见内蒙古鄂尔多斯地区的酸羊乳制品新鲜度较高,菌株存活能力强,有利于下一步的分离鉴定。而鲜奶样品活菌数均小于106mL-1,符合国家标准生鲜乳收购标准,说明内蒙古鄂尔多斯地区的鲜绵羊奶样品新鲜度高。
乳酸菌活菌数显示了样品的新鲜度以及在运输、贮藏过程中菌株的存活状况。样品编号为YM39~YM41,YM44~YM46,YM48的样品为酸山羊奶,其活菌数对数值在7.83~9.41 mL-1之间。张文羿等[11]对11份青海海西州酸山羊奶中的乳酸菌进行了计数,结果为8.40~9.48 mL-1(对数值)之间。包秋华等[12]对13份云南酸山羊奶的研究表明,其乳酸菌活菌数为8.32~10.34 mL-1(对数值)之间。由此可见,本研究的7份酸山羊奶中乳酸菌的计数结果与青海以及云南酸山羊奶中乳酸菌活菌数基本一致。

表2 内蒙古鄂尔多斯地区羊乳制品中乳酸菌活菌计数结果
尽管上述研究对我国山羊乳中乳酸菌数量进行了研究,但对同一地区中不同羊种的发酵乳中乳酸菌研究较少,特别是对同一羊种鲜乳和自然发酵乳中乳酸菌的数量研究更少,本研究不仅对鄂尔多斯地区绵羊乳和山羊乳中乳酸菌数量进行了分析,还对鲜绵羊乳和自然发酵绵羊乳中乳酸菌数量进行了分析(图1)。由图1可以看出,酸奶样品的活菌数高于鲜奶样品的活菌数。

图1 不同样品中乳酸菌活菌计数结果
图1中,差异显著性分析显示鲜绵羊奶、酸绵羊奶、酸山羊奶间乳酸菌活菌计数结果差异不显著(P>0.05)。
2.2 乳酸菌分离株系统发育树的构建及16S rDNA序列鉴定结果
为了将待测菌株鉴定到种,登陆NCBI中的Blast分析所有待测菌株的16S rRNA基因的核苷酸序列。再将拼接好的序列上传到GenBank数据库中。利用MEGA 4.0软件[13]进行乳酸菌模式株和分离株系统发育关系研究和系统进化树构建。
选取部分具有代表性的菌株用Mega 4.0构建系统发育树,如图2所示。

图2 部分分离株的16S rRNA基因序列的系统发育树
由图2可以看出,菌株IMAU11214(KP716575)、IMAU11217(KP716576)与标准菌株Enterococcus durans ATCC19432聚为一类,且同源性为100%,故将其鉴定为 Enterococcus durans。菌株IMAU11334(KP716580)、IMAU11335(KP716581)与标准菌株Enterococcus faecalis JCM5803聚为一类,且同源性为100%,因此可将其鉴定为Enterococcusfaecalis。菌株IMAU11225(KP716584)、IMAU11227(KP716585)与标准菌株 Lactococcus lactis subsp.lactis ATCC 19435聚为一类,且同源性为99%,故将其归为Lactococcus lactis subsp.lactis。菌株IMAU11298(KP716597)、IMAU11306(KP716631)与标准菌株Lactobacillus plantarum ATCC 14917聚为一类,且同源性高达100%,因此可将其归为Lactobacillus plantarum。菌株IMAU11215(KP716598)、IMAU11216(KP716599)与标准菌株Lactobacillus helveticus DSM 20075聚为一类,且同源性高达100%,故将其鉴定为Lactobacillus helveticus。菌株IMAU11275(KP716595)、IMAU11292(KP716596)与标准菌株Leuconostoc pseudomesenteroides NRIC 1777聚为一类,且同源性达到99%,因此鉴定为Leuconostoc pseudomesenteroides。菌株IMAU11230(KP716569)、IMAU11359(KP716573)与标准菌株Leuconostoc mesenteroides NCFB 529聚为一类,且同源性高达100%,故将其归为Leuconostoc mesenteroides。根据16S rRNA基因序列分析,21份羊乳制品中共分离得到68株乳酸菌,其中,乳杆菌36株(占总分离株的52.94%),乳球菌32株(占总分离株的47.06%)。这些菌株归为4个属,7个种或亚种。
5个乳球菌属种为:Enterococcus durans,Enterococcus faecalis,Lactococcus lactis subsp.lactis,Leuconostoc mesenteroides,Leuconostoc pseudomesenteroides。
2个乳杆菌属种为:Lactobacillus helveticus,Lactobacillus plantarum。
2.3 羊乳制品中优势菌种分析
本研究中,21份羊乳制品共分离出68株乳酸菌,分别为7个种及亚种。内蒙古鄂尔多斯地区21份羊乳制品样品的菌株分离鉴定结果如表3所示。
由表3可以看出,21份样品中有14份样品能够分离得到Lactobacillus helveticus,且共分离得到31株,占总分离株的45.59%,说明Lactococcus lactis subsp.lactis为鄂尔多斯地区羊乳制品中乳酸菌的优势种。6份样品中能够分离得到Lactococcus lactis subsp.lactis(17.65%),5份样品中能够分离得到Leuconostoc mesenteroides(11.76%),其他菌种,包括:Enterococcus durans,Enterococcusfaecalis,Leuconostocpseudomesenteroides,Lactobacillus plantarum在样品中分离数量相对较少。
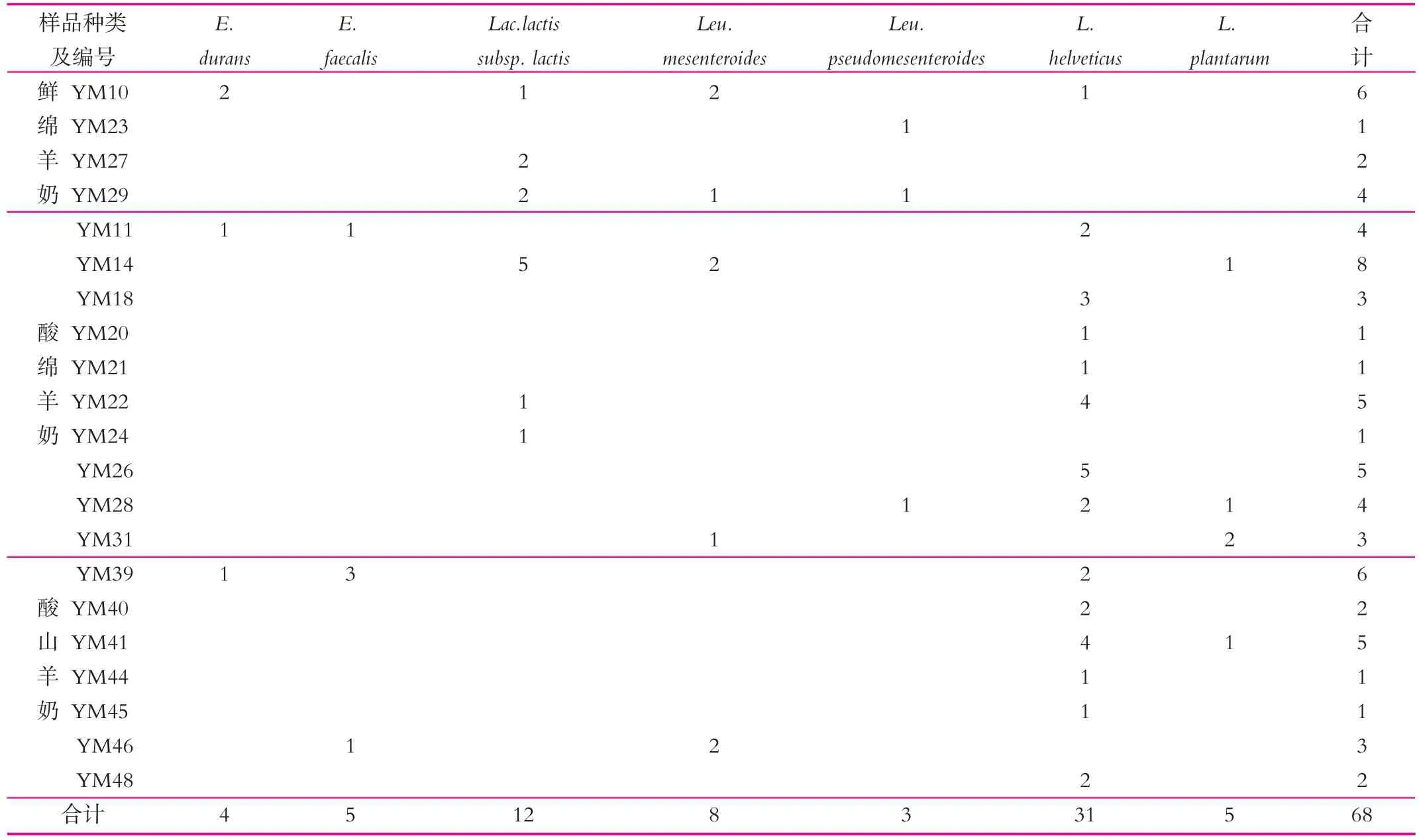
表3 内蒙古鄂尔多斯地区羊鲜乳与酸乳样品的菌株分离鉴定结果
4份鲜奶样品中有3份样品能够分离得到球菌,只有1份样品能够分离得到杆菌,而17份酸奶样品中有15份样品能够分离得到杆菌,只有6份样品能够分离得到球菌,说明鲜奶样品中球菌数量较多,而酸奶样品中杆菌数量较多。这是因为杆菌与球菌的细胞壁或细胞膜对酶活性的保护作用不同,导致杆菌耐酸,适合在酸奶中生长,而球菌不耐酸,在鲜奶中数量较多。Acurcio L B等[14]分析了20份绵羊鲜奶中的微生物组成,发现Enterococcus为其优势菌群。
特别是样品YM14中共分离得到8株乳酸菌,其中5株为Lactococcus lactis subsp.lactis,说明Lactococcus lactis subsp.lactis为样品YM14中的优势菌。样品YM22、YM41中共分离得到5株乳酸菌,其中4株为Lactobacillus helveticus,说明 Lactobacillus helveticus为样品 YM22、YM41中的优势菌。
不同羊乳制品中乳酸菌的分离株数量可比较内蒙古鄂尔多斯地区不同羊乳制品中乳酸菌的优势菌种,结果如图3所示。
由图3可以看出,本研究的内蒙古鄂尔多斯地区绵羊鲜奶中,Lactococcus lactis subsp.lactis为优势菌种,占绵羊鲜奶总分离株的38.46%。而酸羊乳制品中的总体优势菌种为Lactobacillus helveticus,占总分离株的45.59%。其中,酸绵羊奶L.helveticus占总分离株的51.43;酸山羊奶L.helveticus占总分离株的60%。这与刘红霞等[15]对蒙古戈壁地区3份酸山羊奶的分离结果,以及董晓婉等[16]对新疆地区25份酸奶的分离结果相似。

图3 不同羊乳制品中乳酸菌分离株的相对数量比较分析
3 结论
本研究采用传统纯培养方法对采集自鄂尔多斯地区的21份羊鲜乳和酸乳制品中的乳酸菌进行分离和鉴定,21份乳制品中共分离68株乳酸菌。通过16S rRNA基因同源性比较及系统发育树分析,将68株乳酸菌鉴定为4个属,7个种及亚种。其中,L.lactis subsp.lactis为绵羊鲜奶的优势菌种,L.helveticus为鄂尔多斯地区羊酸乳制品中乳酸菌的优势种,分别占总分离株的38.46%和45.59%。乳酸菌活菌计数结果表明,鄂尔多斯地区采集的羊乳制品中,鲜奶样品活菌数在5.03~5.88 mL-1(对数值)之间,酸奶样品活菌数在7.78~9.84 mL-1(对数值)之间,且不同羊乳制品中乳酸菌活菌数不同,其中酸奶样品的活菌数高于鲜奶样品的活菌数,表明内蒙古鄂尔多斯地区鲜羊乳与酸羊乳优势菌群显著不同。
[1]娄新曼,李钰,史永翠,等.羊乳及其制品的脱膻技术与市场现状[J].乳业科学与技术,2014,37(3):23-26.
[2]祝静.羊酸奶发酵工艺及其质构特性和风味成分研究[D].西北农林科技大学,2011.
[3]ZHANG W Y,YUN Y Y,SUN T S,et al.Isolation and identification of dominant microorganisms involved in naturally fermented goat milk in Haixi region of Qinghai,China[J].Annals of Microbiology,2008,58(2):213-217.
[4]MEDINA R,KATZ M,GONZALEZ S,et al.Characterization of the lactic acid bacteria in ewe's milk and cheese from northwest Argentina[J].Journal of Food Protection,2001,64(4):559-563.
[5]YELNETTY A,PURNOMO H,MIRAH A.Biochemical Characteristics of lactic acid bacteria with Proteolytic Activity and Capability as Starter Culture Isolated From Spontaneous Fermented Local Goat Milk[J].Journal of Natural Sciences Research,2014,4(10):137-146.
[6]SAMBROOK J,RUSSELL D W.分子克隆实验指南:上,下册[M].黄培堂等译.北京:科学出版社.2002.
[7]YU J,GAO W,QING M,et al.Identification and characterization of lactic acid bacteria isolated from traditional pickles in Sichuan,China[J].The Journal of General and Applied Microbiology,2012,58(3):163-172.
[8]BAO Q H,LIU W J,YU J,et al.Isolation and identification of cultivable lactic acid bacteria in traditional yak milk products of Gansu Province in China[J].The Journal of General and Applied Microbiology,2012,58(2):95-105.
[9]张春林,刘文俊,张彦斌,等.西藏地区传统发酵乳宏基因组DNA的提取及微生物群落多样性分析[J].食品与发酵工业,2010(4):15-20.
[10]SUN Z,LIU W,GAO W,et al.Identification and characterization of the dominant lactic acid bacteria from kurut:The naturally fermented yak milk in Qinghai,China[J].The Journal of General and Applied Microbiology,2010,56(1):1-10.
[11]ZHANG W Y,YUN Y Y,SUN T S,et al.Isolation and identification of dominant microorganisms involved in naturally fermented goat milk in Haixi region of Qinghai,China[J].Annals of Microbiology,2008,58(2):213-217.
[12]BAO Q H,CHEN X,et al.Isolation and identification of cultivable lactic acid bacteria from traditional goat milk cake in Yunnan province of China[J].African Journal of Microbiology Research,2011,5(29):5284-5291.
[13]TAMURA K,DUDLEY J,NEI M,et al.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Molecular biology and evolution,2007,24(8):1596-1599.
[14]ACURCIO L B,SOUZA M R,NUNES A C,et al.Isolation,enumeration,molecular identification and probiotic potential evaluation of lactic acid bacteria isolated from sheep milk[J].Arquivo Brasileiro de Medicina Veterinária e Zootecnia,2014,66(3):940-948.
[15]刘红霞,徐杰,云月英,等.蒙古戈壁地区自然发酵乳中乳酸菌的分离鉴定[J].微生物学杂志,2007,27(3):42-46.
[16]董晓婉,李宝坤,卢士玲,等.传统分离培养结合PCR-DGGE技术分析传统乳制品中的乳酸菌[J].食品与发酵工业,2014,40(3):97-101.
