世居不同高海拔藏族EGLN1多态与海拔水平的关联研究
2015-11-19王博洋巴桑卓玛崔超英龚嘎兰孜尼玛玉珍王笑峰
王博洋 ,巴桑卓玛,李 蕾,央 拉,刘 兰,崔超英,龚嘎兰孜,尼玛玉珍,多 吉,王笑峰,金 力
(1.复旦大学 生命科学学院 现代人类学教育部重点实验室,上海 200438;2.复旦大学 遗传工程国家重点实验室,上海 200438;3.西藏大学医学院,西藏自治区 拉萨 850002;4.西藏自治区人民医院,西藏自治区 拉萨 850001;5.日喀则疾病预防控制中心,西藏自治区 日喀则 857000)
在高原环境中,氧气浓度随着海拔升高而逐渐降低,成为人类生存的严峻挑战[1-2].高原适应(High Altitude Adaptation)是指高原世居者为了在高原低氧环境中生存而产生的长期的、不可逆的、可遗传的形态结构和生理改变[3].藏族人在青藏高原上已世代居住超过2.5万年[4-5],而长期居住使藏族人的表型适应着高海拔低氧环境的自然选择[6].此外,藏族人与外族通婚少、遗传背景均一[4,7],成为了人类高原适应研究最广泛的人群.
既往研究显示,同低海拔人群相比,世居藏族人群具有较高的肺通气量和较低的血红蛋白(Hemoglobin,Hb)浓度[8-10].另有候选基因关联研究显示,遗传因素与藏族人肺通气量和动脉血氧饱和度(SaO2)等表型相关[11-12].最近,包括我们在内的全基因组研究在Egl-9家族同源物1(EGLN1)中发现世居藏族高原适应的强烈正选择信号[5-6,13-17].同低海拔人群相比,藏族人群的EGLN1 单核苷酸多态(Single Nucleotide Polymorphism,SNP)及构成的单倍型频率高度分化,提示在长期高原生存过程中,获得高海拔低氧适应的功能变异在藏族人中高度富集[6,17-18].此外,全基因组研究还发现EGLN1多态与藏族人较低的Hb浓度存在关联[17].然而,极少有研究报道同样生活在高海拔地区的同一个高原民族随着居住海拔水平的升高,其遗传是否有差异,某基因变异是否随海拔升高进一步被选择[19-20].
Moore等[21]比较了肌红蛋白(Myoglobin,Mb)基因A79G 和T106C多态在世居于3 000m、3 700m和4 500m 的世居藏族人群和低海拔人群的分布,发现这3个藏族人群的Mb79A 频率均高于低海拔人群.他们进一步在世居于3 个不同水平高海拔的藏族人群中比较Mb79A 的频率.虽然Mb79A 在3 000m、3 700m 与4 500m 海拔逐渐升高时分布无显著差异[21],但Moore等的研究方法提示了一个研究高原适应机制的切入点,即在藏族人群中进一步寻找不同的高海拔水平的遗传差异.基于以上考虑,本研究选择既往在高海拔同平原人群对比研究中多次报道的EGLN1 的4 个热点SNPs rs2790882、rs480902、rs2486736和rs2739513[6,13,17],探索它们及其构成的单倍型在世居于2 700 m、3 700 m 和4 700m的3个世居藏族人群中的频率是否有差异,并探索EGLN1多态与藏族人肺功能、SaO2等生理表型的关联.值得一提的是,在藏族人中这4个SNPs在EGLN1第一内含子中构成了一个长度为17.6kb的单倍域(r2位于0.994~0.999之间).同时选择此单倍域中的4个SNPs进行研究,除了它们是高海拔同平原人群差异的热点SNPs外,还为考察本研究结果的重复性及稳定性提供参考.
1 对象与方法
1.1 研究对象
随机选取602名16~26岁健康藏族个体,其中178名从西藏波密县(海拔2 700m)、245名从西藏波密县(海拔3 700m)、179名从西藏安多县(海拔4 700m)募集.所有对象无血缘关系,无异族通婚史,在当地居住至少三代以上.根据居住地的海拔,将研究对象分为低水平高海拔组(LHA),中水平高海拔组(MHA)和高水平高海拔组(HHA).具体的人口学资料见表1.本研究获得复旦大学生命科学学院伦理委员会批准,18岁以上对象由本人、18岁以下对象由监护人签署书面知情同意书.

表1 世居藏族研究对象的人口学和表型特征Tab.1 Demographic and phenotypic characteristics of the studied Tibetan subjects
1.2 研究方法
1.2.1 指标测量
测量体重(kg)和身高(m),并计算BMI.使用标准汞柱式血压计测量收缩压(SBP)和舒张压(DBP).使用SPIDA5型肺功能仪测量用力肺活量(FVC)和一秒用力呼气容积(FEV1).使用Nellcor NPB-40型脉氧仪测量SaO2.具体的生理表型见表1.
1.2.2 位点分型
采集隔夜空腹静脉血,EDTA-Na2抗凝,红细胞裂解液裂解红细胞,蛋白酶K 消化;酚抽提法提取基因组DNA,定量并标化至10ng/μL.使用ABI公司的Taqman 基因分型试剂盒对选取的4个EGLN1SNPs分型.参照制造商的说明对样本DNA进行PCR扩增,并使用ABI公司的7900HT型荧光定量PCR仪检测荧光,并用ABI公司的SDS软件对等位基因打分、聚类.随机选取1%的样本测序验证,一致率为100%.
1.3 统计处理
所有的统计检验均在SPSS 20.0软件上完成.Hardy-Weinberg平衡、两组间等位基因频率及基因型频率比较采用卡方检验.基因型和表型的关联分析采用方差分析和协方差分析.多重校正采用Bonferroni法.
2 结果
2.1 世居藏族EGLN1多态与高海拔水平的关联
世居藏族EGLN1多态与高海拔水平的关联分析结果见表2(第486页).经检验,4个SNPs均符合Hardy-Weinberg平衡.随着海拔从2 700m 升高至3 700m 及4 700m,4个SNPs的常见等位基因频率均显著增加:例如,rs2790882-C 的频率从LHA 组的52.5%,增加至MHA 组的60.0%,进而增加至HHA 组的62.1%(P<0.05).在世居于不同水平高海拔的3个藏族人群中,4个SNPs各常见基因型的分布也均存在显著差异(P<0.05):如rs2790882-CC 的频率从LHA 组的29.4%增加至MHA 组的35.3%,进而增加至HHA 组的36%(P<0.05).此外,随着海拔逐渐升高,4个SNPs构成的常见单倍型CTCC的频率从LHA 组的52.5%,增加至MHA 组的59.8%,进而增加至HHA 组的62.3%(P=0.001 2).以上结果经年龄、性别和BMI调整及Bonferroni校正后仍然显著.
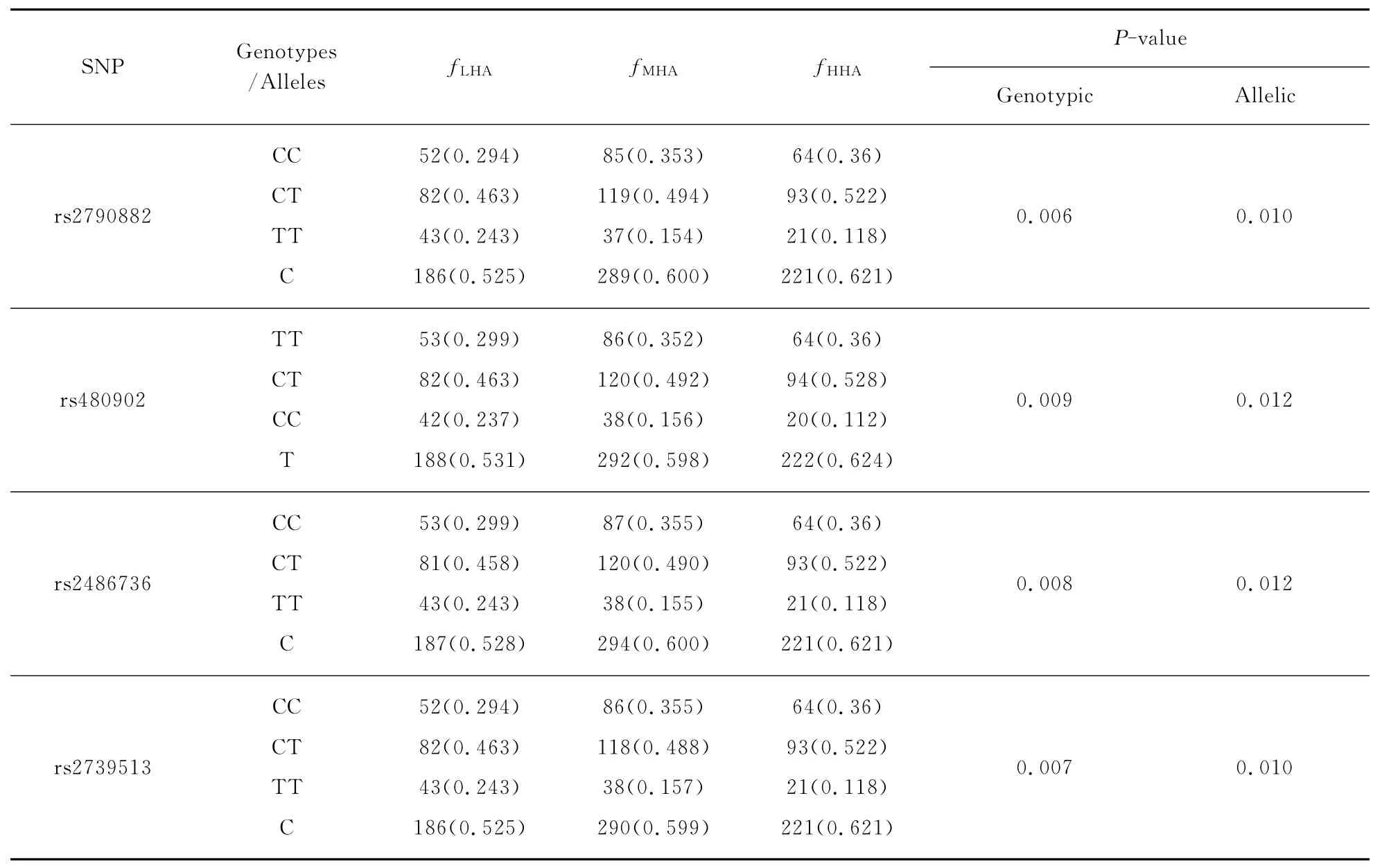
表2 世居藏族EGLN1多态与高海拔水平的关联Tab.2 Association between EGLN1SNPs and level of high altitude among native Tibetans
2.2 世居藏族EGLN1多态与SaO2水平的关联
世居藏族EGLN1多态与SaO2水平的关联分析结果见表3.在HHA 组中,4个SNPs随着少见纯合子、杂合子和常见纯合子其SaO2水平均依次升高,具有统计学差异.如rs2790882-TT 携带者、rs2790882-CT 者和rs2790882-CC者的SaO2水平依次从(81.57±3.93)升高到(82.76±4.43),进而升高到(84.47±4.03)(P=0.008).此外,在HHA 组中,同4个SNPs构成的少见单倍型TCTT 携带者(占62.3%)相比,常见单倍型CTCC携带者(占37.4%)的SaO2水平增加了1.6%(P=0.001 5).以上结果经年龄、性别和BMI调整和Bonferroni校正后仍然显著.除此之外,在LHA 组和MHA 组未发现各多态及单倍型携带者同SaO2相关,也未发现同其他表型相关.
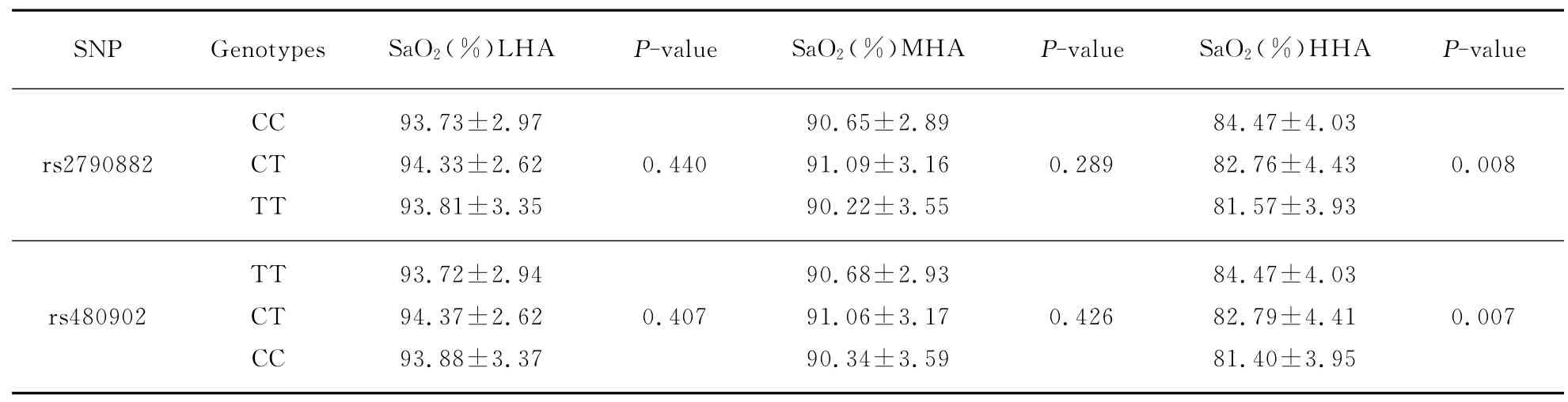
表3 不同水平高海拔世居藏族EGLN1多态与血氧饱和度的关联Tab.3 Association between EGLN1polymorphisms and level of SaO2among native Tibetans from different levels of high altitude
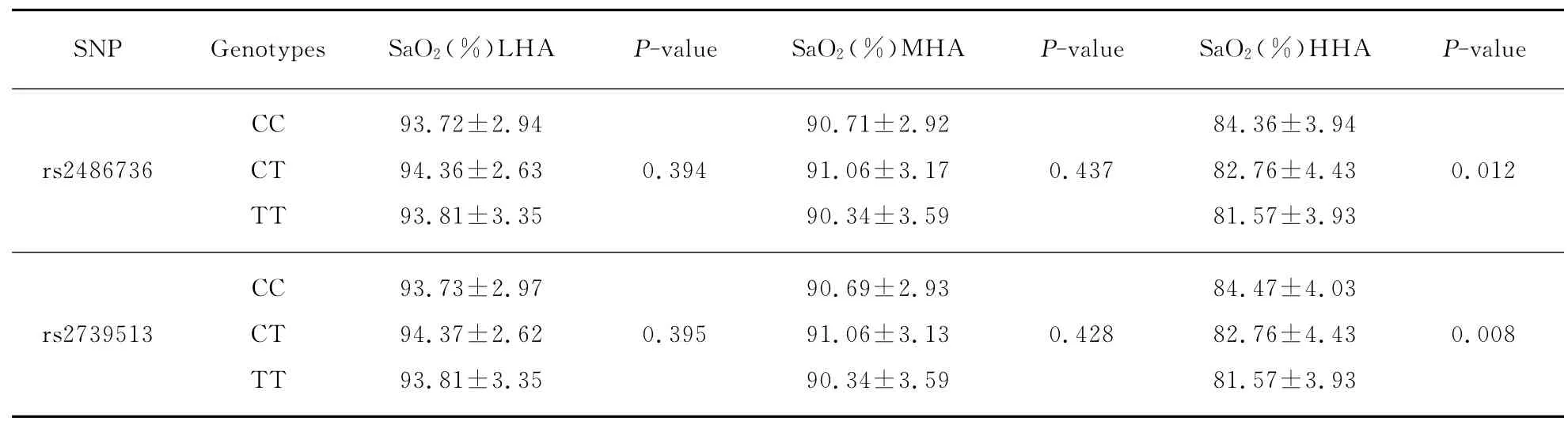
(续表)
3 讨论
EGLN1位于人类1号染色体长臂(1q42.2),长度约60.6kb,含有5个外显子,编码脯氨酸羟化酶2(Prolyl Hydroxylase Domain-containing protein 2,PHD2)[22].PHD2以氧浓度依赖的方式负调控缺氧诱导因子1A 和2A(Hypoxia Inducible Factor-1A/2A,HIF1A/HIF2A).常氧条件下,PHD2使HIFA 特定的脯氨酸残基羟基化,使得HIFA 被VHL 泛素化裂解酶(von Hippel-Lindau ubiquitylation ligase)识别,并被送至蛋白酶体降解;低氧条件下,PHD2 的羟基化活性显著降低,从而稳定了HIF1A 和HIF2A[23].HIFA 与HIFB结合成HIF转录因子进入细胞核,调控糖酵解酶、血管内皮生长因子、血红素氧化酶、促红细胞生成素等数百种基因表达,从而增强机体的低氧适应性[24-25].
本研究首次发现,EGLN1 4个SNPs常见等位基因及常见单倍型CTCC在世居藏族人群中随着海拔进一步升高频率逐渐升高.据此推测,极端高海拔低氧压力对世居于不同水平高海拔藏族人群的EGLN1基因变异具有选择作用.一个可能的解释是,在长期的高海拔低氧压力下,常见单倍型CTCC可能更有利于适应更高的低氧压力,故在世居于较高海拔的藏族人群中得到富集,而且随着海拔逐渐升高进一步增加[26],到4 700m 的极端高度时频率最高(表4).另外,我们在藏族人群中检测出4个SNPs常见等位基因频率在52.5%~62.4%范围波动,同他人在高原全基因组研究[17]的频率范围类似,说明了本研究结果的可靠性.

表4 海拔关联的EGLN1多态在藏族人与低海拔人种中的频率Tab.4 Allele frequencies of the altitude-associated EGLN1SNPs in Tibetans and low altitude populations
全基因组研究发现,相对于低海拔人群,EGLN1在高原藏族人种中受到选择;而我们进一步发现,相对于同样生活在高原的藏族人,随着海拔从2 700m 升高至3 700m 及4 700m,EGLN1基因中至少覆盖了17.6kb的CTCC单倍型进一步被选择.根据Xiang等[17]的假说,HIF2A和ELGN1可能在新石器时代早期一起传入喜马拉雅地区,HIF2A在藏族人进入较低水平的高海拔(2 700~3 000m)时被选择,而当藏族人向更高水平的高海拔移民时,EGLN1可辅助其更顺利地适应更极端的高海拔低氧环境,从而被富集.以上结果提示海拔的升高对EGLN1的选择是一个持久的、长期的过程.
本研究还发现,EGLN1 4个SNPs及单倍型的常见型可升高极端高海拔藏族人(HHA 组)的SaO2水平,而在世居于稍低水平高海拔的藏族人群(LHA 组和MHA 组)中观察不到这一效应.我们推测,在极端高海拔低氧压力下,EGLN1 4个SNPs常见变异可能与升高SaO2并进一步增强机体生理适应性有关.另外,这些常见变异也可降低病理性表型如高原肺水肿(High Altitude Pulmonary Edema,HAPE)的发生.Mishra等[27]发现,同HAPE患者组相比,世居高原人群rs480902位点常见基因型频率及SaO2水平均增加,而少见基因型频率降低.Buroker等[28]对汉族急性高原病(Acute Mountain Sickness,AMS)的病例对照研究也发现,在AMS组中rs480902常见等位基因携带者的SaO2水平显著高于少见型携带者,而对照组中无此变化.
虽然rs2790882、rs480902、rs2486736和rs2739513均位于EGLN1的内含子区,它们可能通过某个连锁位点调控EGLN1来影响机体SaO2水平.第一内含子的变异可能以一种氧依赖的、影响可变剪接的方式影响EGLN1的转录水平[29-31],并进一步影响HIF1A和HIF2A的表达乃至SaO2水平,从而同对极端高海拔低氧压力的适应性相关.但是,可能由于我们检测出来的EGLN1基因变异效应较弱,所以未在稍低海拔或其他表型中检出阳性.
综上,本研究首先发现极端的高海拔低氧压力对藏族人群中的EGLN1基因变异的选择信号.本研究也为EGLN1与高海拔低氧选择压力增加的关联性提供了信息(在极高海拔EGLN1同SaO2水平相关),并佐证了全基因组研究中发现的EGLN1在藏族人群高原适应中的作用.但是,本研究的结果需要进一步的验证.
[1]Ward M P.Everest 1953,first ascent:A clinical record[J].High Alt Med Biol,2003,4(1):27-37.
[2]Cheviron Z A,Brumfield R T.Genomic insights into adaptation to high-altitude environments[J].Heredity(Edinb),2012,108(4):354-361.
[3]Moore L G.Human genetic adaptation to high altitude[J].High Alt Med Biol,2001,2(2):257-279.
[4]Aldenderfer M.Peopling the Tibetan plateau:Insights from archaeology[J].High Alt Med Biol,2011,12(2):141-147.
[5]Wang B,Zhang Y B,Zhang F,et al.On the origin of Tibetans and their genetic basis in adapting highaltitude environments[J].PLoS One,2011,6(2):e17002.
[6]Xu S,Li S,Yang Y,et al.A genome-wide search for signals of high-altitude adaptation in Tibetans[J].Mol Biol Evol,2011,28(2):1003-1011.
[7]Wu T.The Qinghai-Tibetan plateau:How high do Tibetans live?[J].High Alt Med Biol,2001,2(4):489-499.
[8]Beall C M.Two routes to functional adaptation:Tibetan and Andean high-altitude natives[J].Proc Natl Acad Sci USA,2007,104(Suppl 1):8655-8660.
[9]Beall C M.Andean,Tibetan,and Ethiopian patterns of adaptation to high-altitude hypoxia[J].Integr Comp Biol,2006,46(1):18-24.
[10]Wu T,Kayser B.High altitude adaptation in Tibetans[J].High Alt Med Biol,2006,7(3):193-208.
[11]Bigham A W,Kiyamu M,Leon-Velarde F,et al.Angiotensin-converting enzyme genotype and arterial oxygen saturation at high altitude in Peruvian Quechua[J].High Alt Med Biol,2008,9(2):167-178.
[12]Brutsaert T D.Population genetic aspects and phenotypic plasticity of ventilatory responses in high altitude natives[J].Respir Physiol Neurobiol,2007,158(2/3):151-160.
[13]Bigham A,Bauchet M,Pinto D,et al.Identifying signatures of natural selection in Tibetan and Andean populations using dense genome scan data[J].PLoS Genet,2010,6(9):e1001116.
[14]Simonson T S,Yang Y,Huff C D,et al.Genetic evidence for high-altitude adaptation in Tibet[J].Science,2010,329(5987):72-75.
[15]Yi X,Liang Y,Huerta-Sanchez E,et al.Sequencing of 50human exomes reveals adaptation to high altitude[J].Science,2010,329(5987):75-78.
[16]Peng Y,Yang Z,Zhang H,et al.Genetic variations in Tibetan populations and high-altitude adaptation at the Himalayas[J].Mol Biol Evol,2011,28(2):1075-1081.
[17]Xiang K,Ouzhuluobu,Peng Y,et al.Identification of a Tibetan-specific mutation in the hypoxic gene EGLN1and its contribution to high-altitude adaptation[J].Mol Biol Evol,2013,30(8):1889-1898.
[18]吕 坡,范 杰,席焕久.EGLN1基因两个位点多态性与藏族人群高原低氧适应的关系[J].解剖学报,2013,44(3):419-422.
[19]柯金坤,姚宇峰,刘舒媛,等.不同海拔高度低氧环境差异对EPAS1基因多态性的影响[J].中华医学遗传学杂志,2011,28(5):583-588.
[20]柯金坤,姚宇峰,史 磊,等.三个不同海拔藏族群体HIF1A基因九个单核苷酸多态性位点研究[J].中华医学遗传学杂志,2010,27(5):584-589.
[21]Moore L G,Zamudio S,Zhuang J,et al.Analysis of the myoglobin gene in Tibetans living at high altitude[J].High Alt Med Biol,2002,3(1):39-47.
[22]Dupuy D,Aubert I,Duperat V G,et al.Mapping,characterization,and expression analysis of the SM-20human homologue,c1orf12,and identification of a novel related gene,SCAND2[J].Genomics,2000,69(3):348-354.
[23]Jaakkola P,Mole D R,Tian Y M,et al.Targeting of HIF-alpha to the von Hippel-Lindau ubiquitylation complex by O2-regulated prolyl hydroxylation[J].Science,2001,292(5516):468-472.
[24]Aggarwal S,Negi S,Jha P,et al.EGLN1involvement in high-altitude adaptation revealed through genetic analysis of extreme constitution types defined in Ayurveda[J].Proc Natl Acad Sci USA,2010,107(44):18961-18966.
[25]Hirota K,Semenza G L.Regulation of angiogenesis by hypoxia-inducible factor 1[J].Crit Rev Oncol Hematol,2006,59(1):15-26.
[26]李 骞,刘舒媛,林克勤,等.EGLN1基因6 个单核苷酸多态性与高海拔低氧适应的相关性[J].遗传,2013,35(8):992-998.
[27]Mishra A,Mohammad G,Thinlas T,et al.EGLN1variants influence expression and SaO2levels to associate with high-altitude pulmonary oedema and adaptation[J].Clin Sci(London),2013,124(7):479-489.
[28]Buroker N E,Ning X H,Zhou Z N,et al.EPAS1and EGLN1associations with high altitude sickness in Han and Tibetan Chinese at the Qinghai-Tibetan Plateau[J].Blood Cells Mol Dis,2012,49(2):67-73.
[29]Zhao Y,Sun J,Xu P,et al.Intron-mediated alternative splicing of wood-associated NAC transcription factor 1Bregulates cell wall thickening during fiber development in Populus species[J].Plant Physiol,2014,164(2):765-776.
[30]Taylor M S,Devon R S,Millar J K,et al.Evolutionary constraints on the disrupted in Schizophrenia locus[J].Genomics,2003,81(1):67-77.
[31]Jensen C J,Oldfield B J,Rubio J P.Splicing,cis genetic variation and disease[J].Biochem Soc Trans,2009,37(6):1311-1315.
