通过DNA改组技术定向进化赖氨酸脱羧酶基因cadA和ldc
2015-11-11张凯,蔡恒,汪晨
张 凯,蔡 恒,汪 晨
(南京工业大学 生物与制药工程学院,江苏 南京 211800)
通过DNA改组技术定向进化赖氨酸脱羧酶基因cadA和ldc
张 凯,蔡 恒,汪 晨
(南京工业大学 生物与制药工程学院,江苏 南京 211800)
利用DNA改组技术对赖氨酸脱羧酶野生型基因ldc进行随机突变,在大肠杆菌Escherichia coli JM109中构建赖氨酸脱羧酶突变体库。从E.coli JM109和蜂房哈夫尼菌Hafnia alvei AS1.1009中分别克隆出赖氨酸脱羧酶基因cadA和ldc。查询NCBI数据库得知,二者的同源性为75%。分别构建重组质粒pTrc99a-cadA和pTrc99a-ldc,以此2种质粒为模板,经PCR扩增,获得目的基因片段,分析目的基因片段中存在的限制性酶切位点,用多种限制性内切酶碎片化2种基因,切割成不同大小的片段。这些小片段进行不同组合,突变体经过LBXL平板初筛和高效液相色谱(HPLC)复筛,获得1株酶活性提高的赖氨酸脱羧酶突变体,编号为LDC2-16,其比酶活为4 869.86 U/mg(以1 mg总蛋白计),与2种野生型赖氨酸脱羧酶基因表达的酶CadA(1 652.63 U/mg)、Ldc(2 365.93 U/mg)相比,在最适温度37℃、pH 6.0时,突变体的比酶活分别是上述野生型酶的2.95和2.06倍。摇瓶发酵5 h后,目标产物1,5-戊二胺产量从46.9提高至63.9 g/L,提高了36%。
DNA改组;定向进化;赖氨酸脱羧酶;蜂房哈夫尼菌;大肠杆菌
赖氨酸脱羧酶(L-lysine decarboxylase,Ldc,EC 4.1.1.18)是生物体内可逆地催化L-赖氨酸脱羧生成1,5-戊二胺和CO2的高度专一性酶[1]。1,5-戊二胺在农业、医学、化学工业等领域有着广泛的应用。作为一种重要的化工原料,1,5-戊二胺是由可再生原料衍生得到的生物聚酰胺,可以替代传统化工方法生产的生物胺——己二胺[2],能应用于各种聚酰胺产品、聚氨酯、螯合剂和添加剂等,与二元酸进行聚合反应可合成优质高分子材料——新型尼龙[3]。它是一种环保型的、可持续发展型的、耐高温的生物塑料,有着广泛的应用前景[4]。
Ldc存在于 E.coli、尸胺杆菌 (Bacterium cadaveris)和蜂房哈夫尼菌(Hafnia alvei)等微生物中,同时也存在于高等植物中,如黄瓜等。该酶是诱导性胞内酶,需要磷酸吡哆醛作为辅酶促进脱羧反应[5]。
酶的体外定向进化,不需要提前对酶的空间结构以及催化机制进行详细地了解和掌握,只需要通过人为改变基因突变过程中的反应条件,模拟自然进化过程(随机突变、重组和自然选择),在体外进行基因突变、基因重组等操作,进行基因突变体库的构建,根据不同基因的特性来建立高通量筛选方法,最终获得理想的突变酶[6]。DNA改组(DNA shuffling),又称有性PCR[7-8],该方法是将亲本基因进行尽可能多的组合,最终获得理想突变体。不管是从理论角度,还是从实践角度,该方法都比重复寡核苷酸引导进行的诱变及易错PCR的方法等更有利于获得有意义突变,其可使同源性较高的同功能酶进行改组,大大提高基因突变率。
本课题组前期在E.coli JM109中(以pTrc99a为载体)分别单独克隆表达 cadA、ldcC(来自于E.coli)和ldc(来自于H.alvei)3种典型赖氨酸脱羧酶基因,CadA和 LdcC的比酶活分别为1 652和1 329 U/mg;Ldc比酶活为2 366 U/mg(数据未发表)。因此,本研究中,笔者选择2个酶活较高的赖氨酸脱羧酶基因cadA和ldc作为研究对象,分别构建重组质粒pTrc99a-cadA和pTrc99a-ldc,以此2种质粒为模板,经PCR扩增,获得基因片段,继而利用DNA改组等体外分子定向进化技术,对赖氨酸脱羧酶基因ldc进行分子改良,以期获得酶活力提高或酶学性质改善的优良突变菌。
1 材料和方法
1.1 材料
1.1.1 菌株和质粒
H.alvei AS1.1009由天津北洋百川生物技术有限公司惠赠。E.coli JM109、质粒pTrc99a保存于笔者所在实验室,含赖氨酸脱羧酶基因的重组质粒pTrc99a-cadA和pTrc99a-ldc由笔者所在课题组成员构建。
1.1.2 酶和试剂
限制性内切酶、T4 DNA连接酶、rTaq DNA聚合酶等,均购自TaKaRa公司。其他化学试剂为市售国产或进口分析纯。
1.1.3 培养基
液体LB培养基(g/L):蛋白胨10,酵母浸粉5,NaCl 10。121℃湿热灭菌30min。
固体鉴别培养基LBXL:在液体LB培养基的基础上添加0.5%(质量分数)L-赖氨酸和0.01%(质量分数)溴甲酚紫,并添加终浓度为0.3 mmol/L的IPTG对重组质粒进行诱导表达,pH 6.0。
1.1.4 赖氨酸脱羧酶基因引物设计
根据E.coli JM109中赖氨酸脱羧酶基因cadA序列和H.alvei AS1.1009中赖氨酸脱羧酶基因ldc序列,使用Primer Premier5.0软件辅助设计2个基因序列的上下游引物。分别在引物的5′端增加2~3个保护碱基(划线字母),引入限制内切酶酶切位点(加粗斜体字母)BamHⅠ和HindⅢ,以便后期的克隆与表达。引物由南京金斯瑞生物科技有限公司合成。
cadA-1:5′-CGGGATCCATGAACGTTATTGCAATATTG-3′;cadA-2:5′-CCCAAGCTTTTATTTTTTGCTTTCTTCTTTC-3′;ldc-1:5′-CGGGATCCATGAATATCATTGCCATCATGAACG-3′;ldc-2:5′-CCC AAGCTTTTATGACTTCTTCGCCGCTGAT-3′。
1.2 方法
1.2.1 PCR扩增cadA和ldc基因
分别以E.coli JM109和H.alvei AS1.1009的基因组DNA为模板,进行PCR反应。反应体系:模板DNA3μL、上游引物2μL、下游引物2μL、10×Buffer 5μL、MgCl2(2.5 mmol/L)4μL、dNTPs 4μL、rTaq DNA聚合酶0.5μL、加重蒸水至50μL。反应条件:95℃5min;95℃1min,58℃30 s,72℃ 3min,循环30次;72℃10min。
PCR结果用1.0%(质量分数)琼脂糖凝胶电泳进行检测,之后用DNA回收试剂盒(北京庄盟国际生物基因科技公司)回收纯化PCR产物,并经过BamHⅠ和HindⅢ双酶切(37℃、2.5 h),分别连接到pTrc99a载体上,重组质粒 pTrc99a-cadA和pTrc99a-ldc利用 CaCl2转化法转化至大肠杆菌JM109,构建随机突变体库。
1.2.2 DNA改组
1)限制性内切酶法降解DNA及回收小片段以构建获得的重组质粒pTrc99a-cadA和pTrc99aldc为模板,分别用限制性内切酶BamHⅠ和HindⅢ对2个重组质粒进行双酶切,回收cadA和ldc基因片段。分别选择限制性内切酶①XhoⅠ、②EcoRⅠ、③PstⅠ、④ClaⅠ对cadA基因片段进行酶切,⑤SacⅠ、⑥ClaⅠ、⑦PvuⅠ、⑧EcoRⅠ对ldc基因片段进行酶切[9],1.5%琼脂糖凝胶电泳。
2)无引物PCR 上步获得的8种酶切片段分别两两组合作为模板进行一次无引物PCR,即①⑤、①⑥、①⑦、①⑧、②⑤、②⑥、②⑦、②⑧、③⑤、③⑥、③⑦、③⑧、④⑤、④⑥、④⑦和④⑧。反应体系:模板 DNA 3μL、10×Buffer 5μL、MgCl2(2.5 mmol/L)4μL、dNTPs 4μL、rTaq DNA聚合酶0.5μL、加重蒸水至50μL。反应条件:95℃2min;95℃30 s,58℃15 s,72℃ 2.5min,循环40次;72℃10min。1.5%琼脂糖凝胶电泳。再以上步无引物PCR产物混合为1管作为模板,进行2次无引物PCR(反应体系同上步),1.5%琼脂糖凝胶电泳。
3)有引物PCR 以上一步二次无引物PCR产物作为模板,进行有引物PCR,获得大小正确的目的条带,1.5%琼脂糖凝胶电泳。用DNA回收试剂盒(北京庄盟国际生物基因科技公司)回收纯化PCR产物,并经过BamHⅠ和HindⅢ双酶切,分别连接到pTrc99a载体上,重组质粒pTrc99a-LDC利用CaCl2转化法转化至大肠杆菌JM109,构建随机突变体库。
1.2.3 高通量筛选
大量抽提突变文库中重组质粒,CaCl2转化法转化至大肠杆菌JM109,涂布于LB抗性平板。溴甲酚紫是一种酸碱指示剂,其变色范围是pH 5.2(黄色)~6.8(紫色)。1,5-戊二胺是一种具有生物活性的含氮碱,将 LB抗性平板上长出的菌落点于LBXL平板上,培养12 h,菌体产生的目的产物1,5-戊二胺会使得菌体周围的LBXL培养基(黄绿色)变成紫色透明圈,观察紫色透明圈大小,挑取透明圈比野生型大的32个突变株进行比酶活测定。测定上述两轮突变体的酶活,进行进一步验证。经第二轮进化后,获得最佳突变体LDC2-16。
1.2.4 赖氨酸脱羧酶酶活力测定
酶活具体方法参照文献[5,10-11]进行。
1.2.5 温度和pH对赖氨酸脱羧酶活性的影响
在不同温度(23、30、37、44和51℃)下测定酶活力,以未处理的野生型酶液活力为100%,计算各组酶的相对活力。在不同pH(3.0、4.0、5.0、6.0、7.0和8.0)缓冲液(50 mmol/L乙酸钠)配制的50 g/L赖氨酸底物下,测定酶活力,以未处理的野生型酶液活力为100%,计算各组酶的相对活力。
1.2.6 赖氨酸脱羧酶的温度稳定性和pH稳定性
在pH6.0条件下,将酶液在不同的温度(23、30、37、44和51℃)下保温2 h,测定酶活力,以未处理的野生型酶液活力为100%,计算各组酶的相对活力。酶液与上述不同pH(3.0、4.0、5.0、6.0、7.0和8.0)缓冲液(50 mmol/L乙酸钠)于30℃保温2 h,测定酶活力,以未处理的野生型酶液活力为100%,计算各组酶的相对活力。
2 结果与讨论
2.1 PCR扩增cadA和ldc基因结果
首先通过常规PCR扩增方法获得了2个赖氨酸脱羧酶基因:cadA(2 142 bp,来自于 E.coli JM109)和ldc(2 199 bp,来自于H.alvei AS1.1009)的基因片段,琼脂糖凝胶电泳分析结果见图1。查询NCBI数据库得知,2种基因的同源性为75%。胶回收试剂盒回收cadA和ldc基因片段的PCR产物,并经过BamHⅠ和 HindⅢ双酶切、纯化,连接到pTrc99a载体上,获得重组质粒pTrc99a-cadA和pTrc99a-ldc。
2.2 DNA改组结果及鉴定
1)限制性内切酶法降解DNA及回收小片段结果 分别用限制性内切酶①XhoⅠ、②EcoRⅠ、③PstⅠ、④ClaⅠ对 cadA基因片段进行酶切,用⑤SacⅠ、⑥ClaⅠ、⑦PvuⅠ、⑧EcoRⅠ对ldc基因片段进行酶切,琼脂糖凝胶电泳分析,结果见图2。
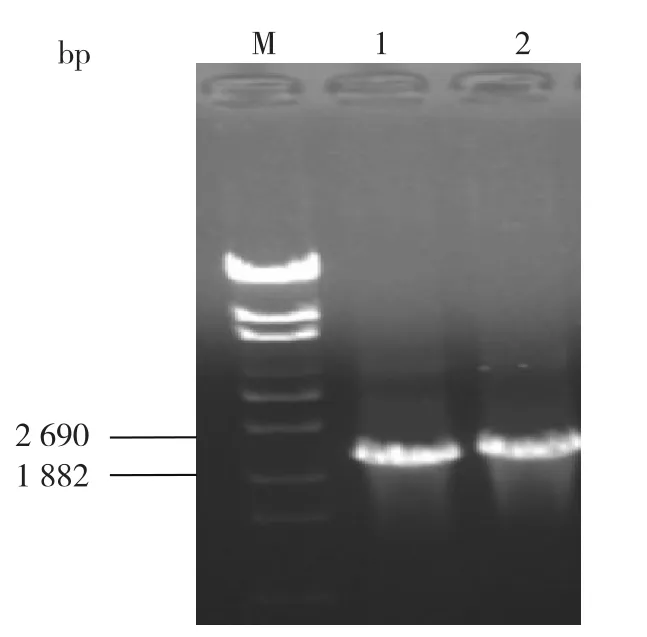
图1 目的基因PCR扩增产物Fig.1 PCR amplification of the two target gene product
2)无引物PCR扩增结果 将由限制性内切酶获得的DNA小片段分别两两组合作为模板进行一次无引物PCR,结果进行琼脂糖凝胶电泳分析,结果见图3。再以上步无引物PCR产物混合为1管作为模板,进行二次无引物PCR,琼脂糖凝胶电泳分析,结果见图4。
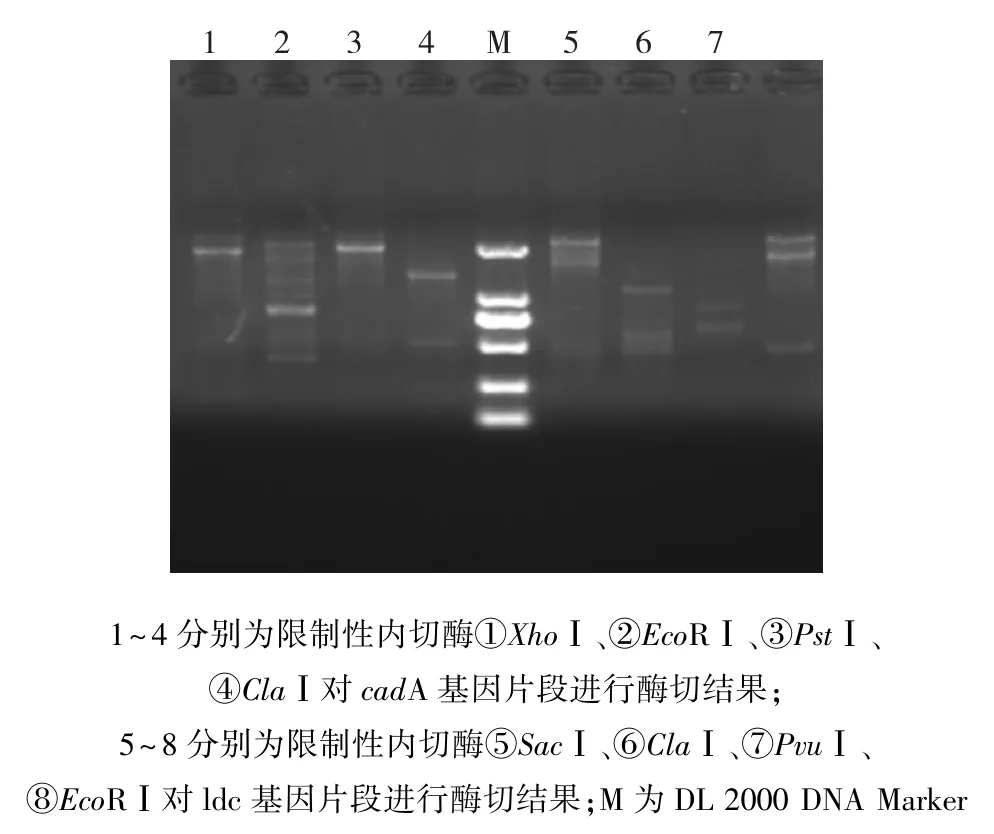
图2 限制性内切酶酶切cadA和ldc基因片段Fig.2 The restrict enzyme digestion of cadA and ldc
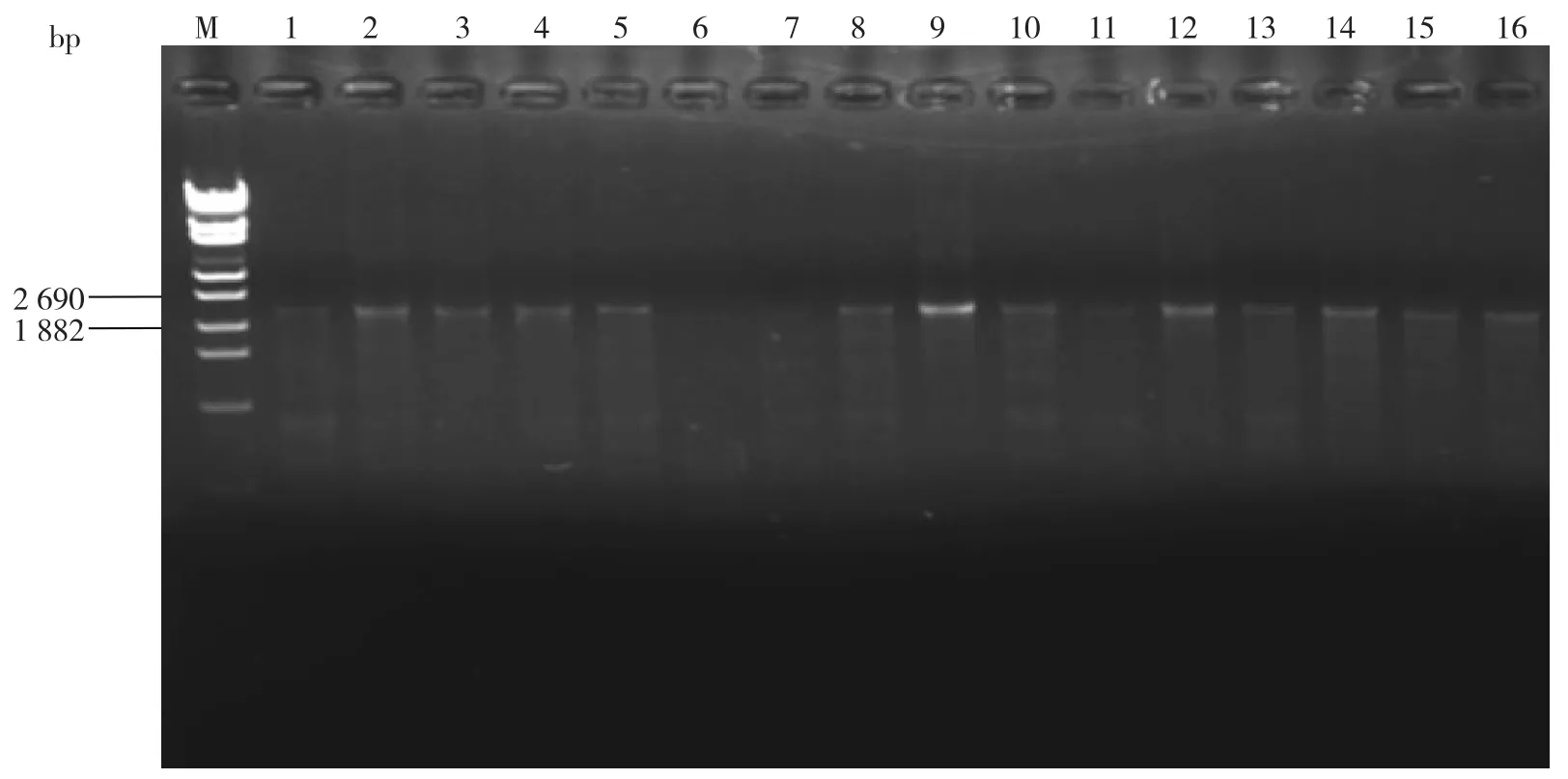
图3 一次无引物PCRFig.3 Primerless PCR for the first time
3)有引物PCR扩增结果 以上一步二次无引物PCR产物作为模板,进行有引物PCR,获得大小正确的2 200 bp的目的条带(图5),琼脂糖凝胶电泳。
2.3 DNA改组突变体库构建
上述DNA改组产物克隆、连接到pTrc99a表达载体上,获得重组质粒pTrc99a-LDC,利用CaCl2转化法转化至大肠杆菌JM109,构建了库容约为5 000的初级突变体库。PCR和双酶切结果验证重组pTrc99a-LDC构建成功(图6)。
经过2轮DNA改组,改组结果较为成功。经过LBXL板初筛和高效液相色谱(HPLC)复筛,成功构建了赖氨酸脱羧酶突变体库,建立了高通量筛选方法,共获得了5 000多个突变体,获得1株酶活性提高的赖氨酸脱羧酶突变体:LDC2-16,其比酶活测定为4 869.86 U/mg(以1 mg总蛋白计),与2种野生型赖氨酸脱羧酶CadA(1 652.63 U/mg)、Ldc(2 365.93 U/mg)相比,在最适温度37℃、pH 6.0时,突变体的比酶活分别是上述野生型酶的2.95和2.06倍。摇瓶发酵5 h后,目标产物1,5-戊二胺产量从46.9 g/L达到了63.9 g/L,提高了36%。Takarashi等[12]利用E.coli JM109过表达cadA基因,通过补料、pH调控等操作,15 h后发酵产量达69 g/L;Martin等[13]以也以C.glutamicum作为1,5-戊二胺生产菌株进行实验,整个过程经过发酵、pH调控、热处理、萃取、蒸馏纯化等步骤,80 h后1,5-戊二胺产量达到72 g/L,是目前研究报道中的最高生产水平。

图4 二次无引物PCRFig.4 Primerless PCR for the second time
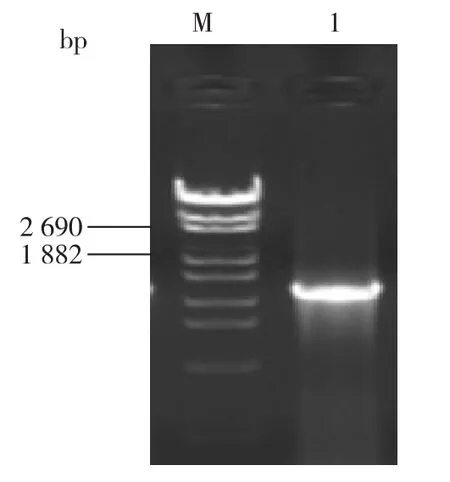
图5 有引物PCRFig.5 PCR with primers
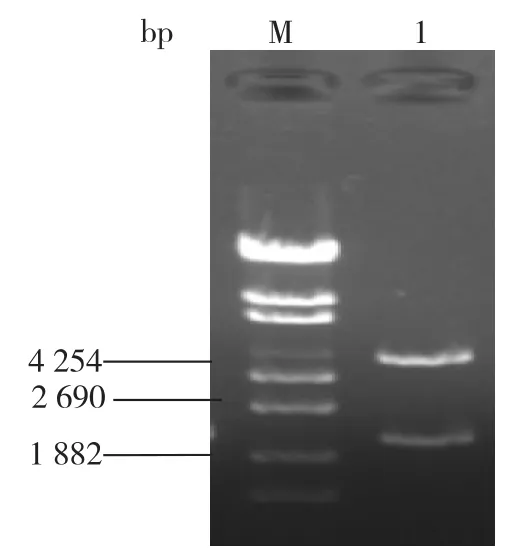
图6 双酶切鉴定重组质粒pTrc99a-LDCFig.6 Double digestion of recombinant plasmid pTrc99a-LDC
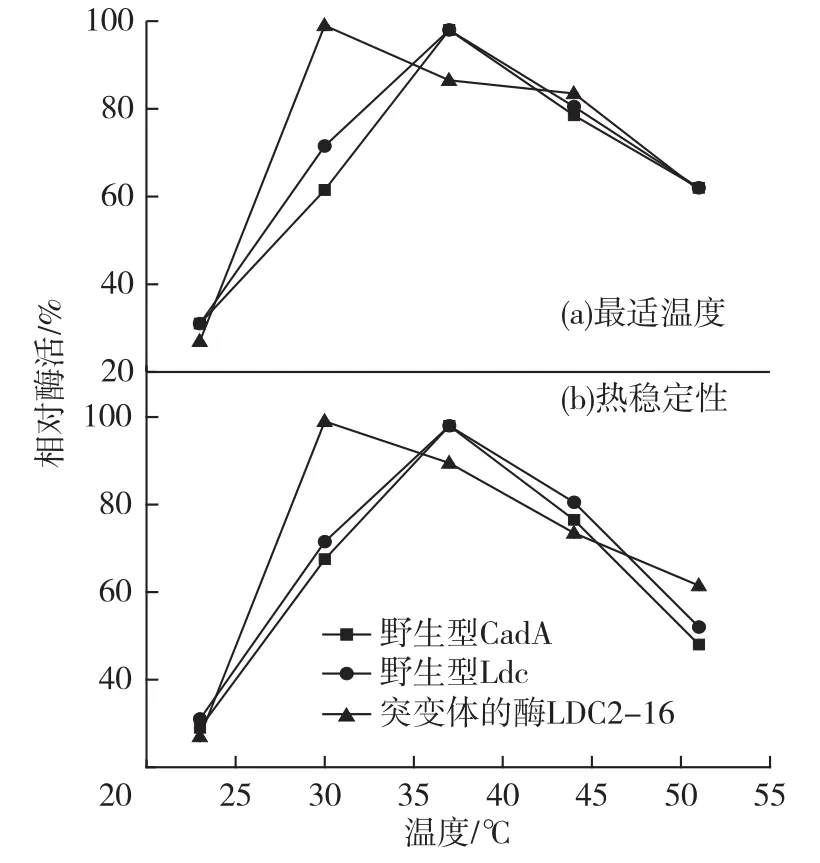
图7 野生型酶CadA、Ldc和突变体的酶LDC2-16的最适温度和温度稳定性Fig.7 The optimal temperature and temperature stability of wild-type CadA,Ldc and mutant LDC2-16
2.4 野生型CadA、Ldc和突变体的酶LDC2-16的最适温度和温度稳定性
图7为野生型 CadA和 Ldc和突变体的酶LDC2-16的最适温度及其温度稳定性。由图7(a)可知:野生型酶的最适温度均为37℃,突变体的酶LDC2-16的最适温度为30℃,同时突变体的酶在45℃时的相对酶活力依旧维持在80%~90%,而野生型酶在该温度下活性已呈现下降趋势。由图7(b)可知:野生型酶在30~45℃稳定性较好,突变体的酶在28~45℃稳定性较好,但在相对较低的温度下,野生型酶的稳定性要稍差于突变体的酶。
2.5 野生型CadA、Ldc和突变体的酶LDC2-16的最适pH和pH稳定性
图8为野生型 CadA和 Ldc及突变体的酶LDC2-16的最适pH及pH稳定性结果。由图8(a)可知:野生型酶的最适pH均为7.0,突变体的酶LDC2-16的最适pH为6.0~7.0,pH范围稍宽。由图8(b)可知:在pH 8.0时,野生型酶活性迅速下降,突变体的酶相对酶活力还维持在50%~60%。无论是pH范围还是pH稳定性,突变体的酶都优于野生型酶。
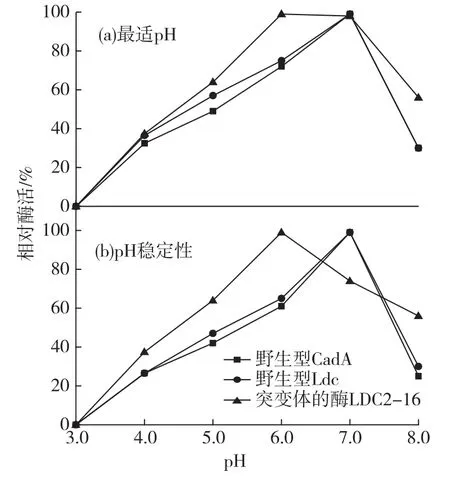
图8 野生型酶CadA、Ldc和突变体的酶LDC2-16的最适pH和pH稳定性Fig.8 The optimal pH and pH stability of wild-type CadA,Ldc and mutant LDC2-16
3 结论
通过体外定向进化技术对赖氨酸脱羧酶进行了分子改造,获得1株酶活性提高的赖氨酸脱羧酶突变体,编号为LDC2-16,其比酶活为4 869.86 U/mg(以1 mg总蛋白计),与2种野生型赖氨酸脱羧酶基因表达的酶 CadA(1 652.63 U/mg)、Ldc(2 365.93 U/mg)相比,在最适温度37℃、pH 6.0时,突变体的比酶活分别是上述野生型酶的2.95和 2.06倍。摇瓶发酵5 h后,目标产物1,5-戊二胺产量从46.9提高至63.9 g/L,提高了36%。后续实验可以利用基因测序、定点突变等方法来具体研究氨基酸突变点对此酶活的影响,进而为解释其蛋白质结构与功能之间的关系奠定坚实的基础。
[1]张凯,蔡恒,汪晨,等.生物法制备1,5-戊二胺的研究进展[J].现代化工,2012,32(10):21-24.
[2]Kiyohiko N,AhuichiE,YukikoM.Enzymaticmethod for producing cadaverine dicarboxylate and its use for the production of nylon:JP,1482055[P].2006-03-01.
[3]牛涛,黎明,张俊环,等.一步法生产1,5-戊二胺谷氨酸棒杆菌基因工程菌的构建[J].中国生物工程杂志,2010,30(8):93-99.
[4]Stefanie K,Weol K J,Hartwig S,et al.Systems-wide metabolic pathway engineering in Corynebacterium glutamicum for bio-based production of diaminopentane[J].Metab Eng,2010,12(4):341-351.
[5]蒋丽丽,吴晓燕,刘毅,等.赖氨酸脱梭酶发酵工艺及其酶学性质[J].精细化工,2006,23(11):1060-1067.
[6]吕晓慧,胡亚冬,胡凤娟,等.基于易错 PCR的假密环菌Armillariella tabescens MAN47 β-甘露聚糖酶耐高温定向进化[J].生物工程学报,2009,25(12):1900-1906.
[7]Stemmer W P C.DNA shuffling by random fragmentation and reassembly:in vitro recombination for molecular evolution[J]. Proc Natl Acad Sci USA,1994,91(1):10747-10751.
[8]Stemmer W P C.Rapid evolution of a protein in vitro by DNA shuffling[J].Nature,1994,370(1):389-391.
[9]Miho K,Kouhei O,Shigeaki H.Novel family shuffling methods for the in vitro ecolution of enzymes[J].Gene,1999,236:159-167.
[10]Bradford M M.A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of proteindye binding[J].Anal Biochem,1976,72(1):248-254.
[11]Phan A P H,Ngo T T,Lenhoff H M.Spectrophotometric assay for lysine decarboxylase[J].Anal Biochem,1982,120(1):193-197.
[12]Takashi M,Hideki S,Masahiro H,et al.Metabolic engineering of Corynebacterium glutamicum for cadaverine fermentation[J]. Biosci Biotechnol Biochem,2007,71(9):2130-2135.
[13]Martin V,Ludwigshafen,Oskar Z,et al.Method for fermentatively producing 1,5-diaminopentane:US,20100292429[P].2010-07-22.
(责任编辑 荀志金)
Directed evolution by DNA shuffling of lysine decarboxylase gene cadA and ldc
ZHANG Kai,CAI Heng,WANG Chen
(College of Biotechnology and Pharmaceutical Enginneering,Nanjing Tech University,Nanjing 211800,China)
We enhanced the activity of two lysine decarboxylase(LDC)cadA and ldc by DNA shuffling through random mutation.One lysine decarboxylase gene cadA from Escherichia coli and the other ldc from Hafnia alvei AS1.1009 were linked into vector pTrc99a.Referring to NCBI database,the nucleotide sequence analysis showed 75%homology between cadA gene and ldc gene.Then,we obtained three recombinant plasmid pTrc99a-cadA and pTrc99a-ldc.As templates,two plasmids were amplified by normal PCR to obtain the genes cadA and ldc.According to the restriction enzyme sites present in the two genes,we used variety of restriction enzyme sites to get endonuclease fragments.Then,the random fragments were purified and reassembled by PCR without primers and the products were amplified by routine PCR.These fragments were successfully reassembled to genes of the same size as LDC.The activity of LDC2-16 can reach to 4 869.86 U/mg total protein,which is 2.95 and 2.06 times as that of the wide type(CadA 1 652.63 U/mg、Ldc 2 365.93 U/mg)under the optimum conditions oftemperature 37℃,pH 6.0.After 5 h fermentation,the target product 1,5-diaminopentane production researched 63.9 g/L from 46.9 g/L with an increase of 36%.
DNA shuffling;directed evolution;lysine decarboxylase;Hafnia alvei;Escherichia coli
Q93
A
1672-3678(2015)05-0020-06
10.3969/j.issn.1672-3678.2015.05.004
2014-03-10
国家重点基础研究发展计划(973计划)(2011CBA00807)
张 凯(1987—),女,江苏徐州人,硕士研究生,研究方向:基因工程和发酵工程;蔡 恒(联系人),副教授,E-mail:cheng@njtech. edu.cn
