中国北方沿海14种经济鱼类和7种鲽科鱼类的PCR-RFLP分析
2015-10-31闫平平齐欣黄大亮王海燕崔妍田卓张丽荣
闫平平齐欣黄大亮*王海燕崔妍田卓张丽荣
(1.大连出入境检验检疫局辽宁大连116600;2.大窑湾出入境检验检疫局)
中国北方沿海14种经济鱼类和7种鲽科鱼类的PCR-RFLP分析
闫平平1齐欣1黄大亮1*王海燕2崔妍1田卓1张丽荣1
(1.大连出入境检验检疫局辽宁大连116600;2.大窑湾出入境检验检疫局)
以中国北方沿海14种经济鱼类(日本下鱵鱼、斑尾复鰕虎鱼、黄尾鰤、鳙鱼、大泷六线鱼、绿鳍马面鲀、大菱鲆、日本笠鳚、绒杜父鱼、牙鲆、斑纹条鳎、长吻红舌鳎、方式云鳚、大西洋鳕)和7种鲽鱼(粗壮拟庸鲽鱼、黄盖鲽、亚洲箭齿鲽、星斑川鲽鱼、马舌鲽、木叶鲽、石鲽)为研究对象,首先对其线粒体16S rRNA基因片段进行PCR扩增及序列同源性比对,随后采用Dde I、Hae III和Nla III 3种限制性内切酶对21种鱼类的细胞色素b基因序列进行限制性片段长度多态性聚合酶链反应(PCR-RFLP)分析,并利用芯片生物分析技术得到酶切产物的特异性图谱和确切片段大小,结果共检出132个电泳条带,实现了21种鱼类的有效鉴别。本文为鱼类种类鉴定和鱼类食品检测提供了科学依据。
经济鱼类;鲽科;PCR-RFLP;16S rRNA;细胞色素b
1 前言
随着海产品消费量的增大,来料加工的鱼种复杂,鱼类产品加工处理中以次充好的现象日益严重,其中最为常见的手段是以低廉鱼类冒充名贵鱼类,这一行为不仅严重地损害了消费者的利益,也对食品检验工作提出了新的挑战。
鱼类品种鉴定通常采用形态学方法,然而有些鱼类由于形态相近并且经加工处理使鱼失去了用于形态辨别的部分,使得鉴定变得十分困难。分子生物学技术为鱼种鉴定提供了新的途径,因为即使鱼类经过加工也能获取较为完整的DNA分子,该类方法具有很好的灵敏度、特异性和可靠性[1,2]。近年来,限制性片段长度多态性聚合酶链反应(PCR-RFLP)法由于具有简便、成本低、通量大等特性,已成功应用于多种鱼类及其产品的鉴定,包括带鱼[3]、鲭[4]、竹?鱼[5]、鳕[6]、金枪鱼[7]和鮟鱇[8]等,同时也为其形态学鉴定提供了辅助手段。
本研究首先采用PCR技术对中国沿海14种经济鱼类和7种鲽科鱼类的16S rRNA基因进行扩增及序列比对分析,随后利用Dde I、Hae IIII和Nla III 3种限制性内切酶对21种鱼类的细胞色素b基因序列进行PCR-RFLP分析,并应用生物芯片技术对酶切产物进行数字化,旨在建立一种简便、快速、可靠性高的鱼类品种鉴定方法。
2 材料与方法
2.1材料
2.1.1试验材料
14种经济鱼类和7种鲽科鱼类:均采自来料加工的深海鱼类产品,并附有捕捞证和卫生证明。本研究所用鱼类均经过大连海洋大学水产养殖中心的鱼种鉴定和确认。
2.1.2试剂
DNA提取试剂盒、PCR-RFLP鱼种鉴定试剂盒、DNA 1000芯片试剂盒:均购自Agilent公司;引物由上海生工公司合成。
2.2方法
2.2.1基因组DNA的提取
取50 mg鱼肉,剪碎后置于离心管中,按照DNA提取试剂盒说明书进行鱼类基因组DNA提取,紫外分光光度计测定DNA浓度和纯度,并于-20℃保存备用。
2.2.216S rRNA基因的PCR扩增及序列比对
16S基因片段扩增用的通用引物序列为:16Sar:5’-cgcctgtttatcaaaaacat-3’[9],16Sbr:5’-ccggtctgaactcaga tcacgt-3’[9]。PCR反应在ABI阶梯PCR仪上进行,25 μL反应体系内含:ddH2O 16.8 μL、10×Taq buffer 2 μL、dNTP 2 μL(2.5 mM)、正反引物各1 μL(10 μM),Taq酶0.2 μL(0.25 U)和模板DNA 2 μL(100 ng)。PCR反应条件:94℃2 min,94℃30 s,52℃40 s,72℃1 min,35个循环;72℃10 min。PCR产物经1%琼脂糖凝胶电泳检测,回收测序。
利用Blast在线工具(http://blast.ncbi.nlm.nih. gov/)对21种鱼测序结果进行同源性序列比对。
2.2.3细胞色素b基因的PCR扩增和酶切
根据Hold等[10]的报道选取464 bp的细胞色素b基因特异性片段作为目的片段,扩增用引物序列为L14735:5'-AAA AACCAC CGT TGT TAT TCA ACT A-3',H15149:5'-GCICCT CAR AAT GAY ATT TGT CCT CA-3'。25 μL反应体系内含:ddH2O 9 μL、2×PCR Master Mix 12.5 μL、正反引物混合液2.5 μL和模板DNA 1 μL(100 ng)。PCR反应条件:95℃5 min,95℃30 s,50℃30 s,72℃30 s,40个循环;72℃7 min。
取2.5 μL扩增产物分别与2.5 μL限制性内切酶Dde I、Hae III或Nla III混匀,37℃酶切2 h以上,加入1 μL 60 mmol/L EDTA溶液,混匀,65℃保温15 min以终止反应。
2.2.4芯片生物技术分析
应用芯片生物技术分别对上文制备的21种鱼类16S rRNA基因扩增产物和细胞色素b基因的酶切产物进行分析。
按照DNA 1000芯片试剂盒说明书在相应芯片孔中依次加入9 μL预染胶、5 μL阳性对照、1μL DNA相对分子质量标准物和1 μL样品;每个样品重复3次。
3 结果与分析
3.116S rRNA基因的PCR扩增及序列比对结果
21种鱼16S rRNA PCR扩增产物大小为600 bp左右,条带单一,可以很好地进行测序分析。测序产物经Genbank数据库比对,结果见表1。

表1 21种鱼16S rRNA基因的PCR产物测序比对结果

(续表1)
3.2细胞色素b基因的PCR扩增
21种鱼类细胞色素b基因的PCR扩增结果均得到464bp大小的片段,条带单一。
3.314种北方沿海经济鱼种和7种鲽科鱼类细胞色素b基因RFLP分析
14种鱼类的PCR-RFLP酶切图谱见图1,共得到84个电泳条带,片段大小见表2;7种鲽科鱼类的PCR-RFLP酶切图谱见图2,共得到48个电泳条带,片段大小见表3。D代表Dde I酶,H代表Hae III酶,N表示Nla III酶。结果显示,只有3种酶的组合才可实现全部鱼类的有效鉴别。试验中所涉及鱼类的酶切位点突变率低,在3次重复实验中每个酶切片段仅相差2-5 bp。
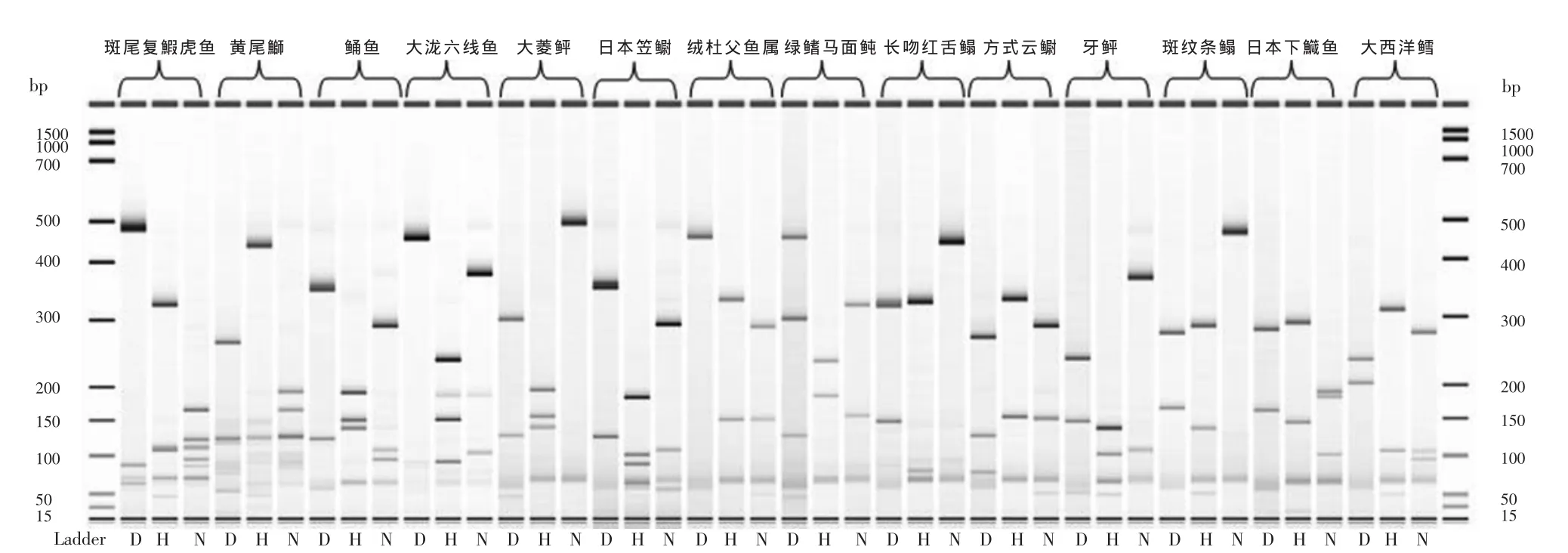
图1 14种海鱼的PCR-RFLP芯片电泳图谱
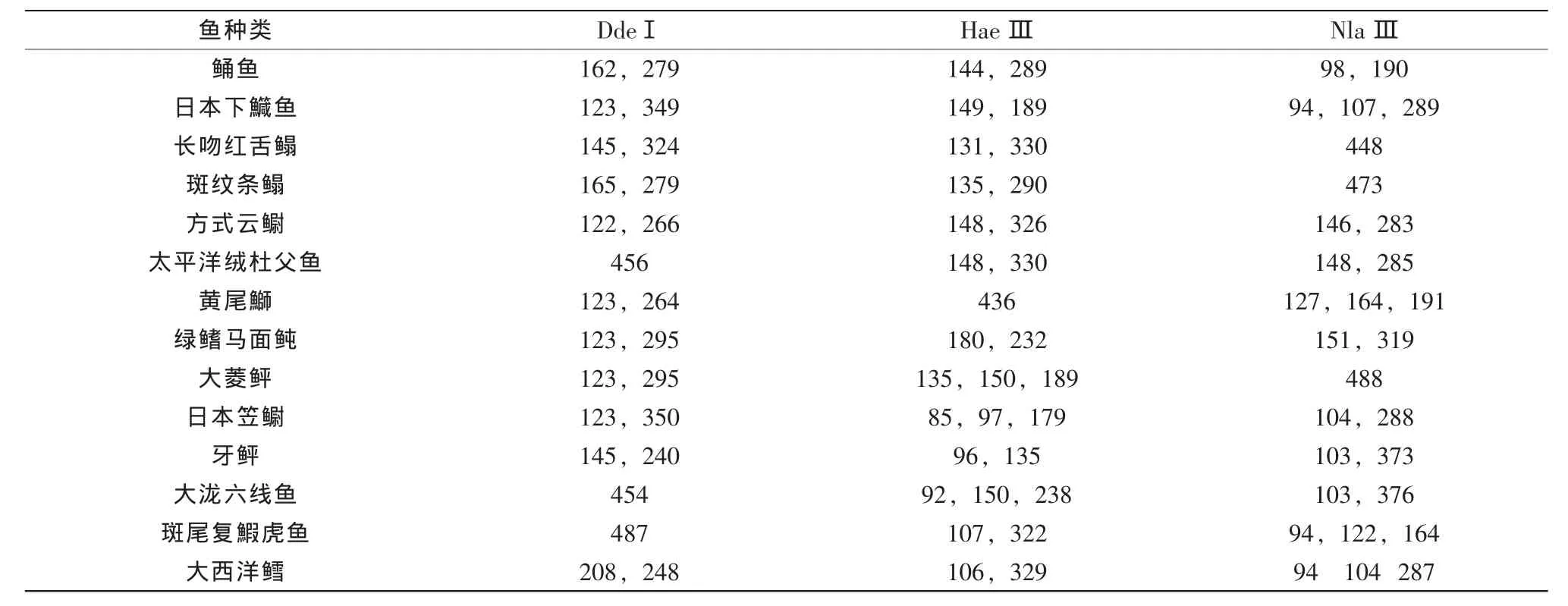
表2 14种海鱼细胞色素b基因PCR扩增片段分别经Dde I、Hae III和N la III酶切的片段大小
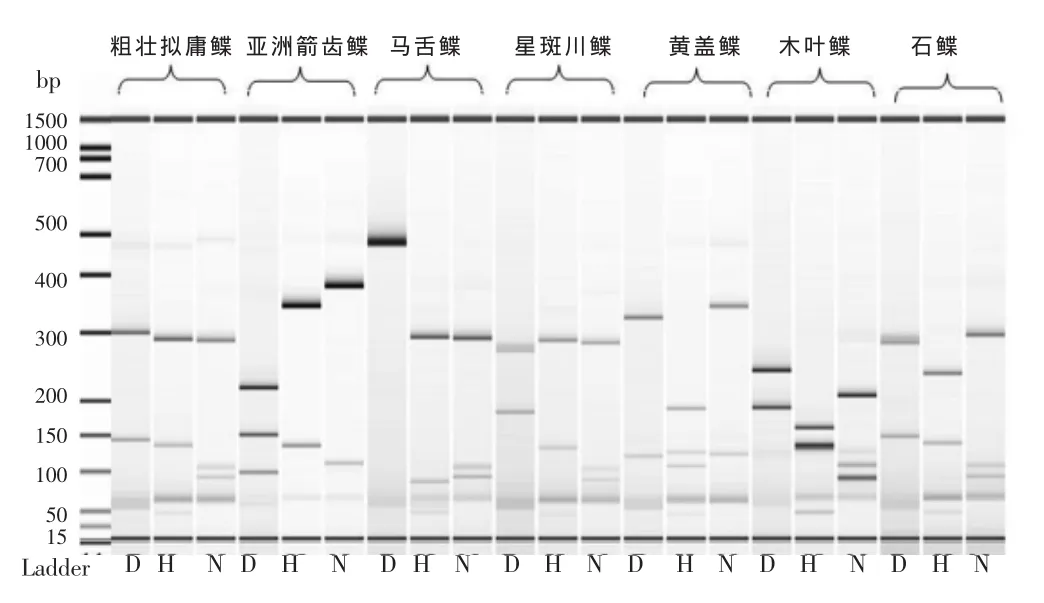
图2 7种鲽鱼PCR扩增片段分别经Dde I、Hae III和Nla III酶切的RFLP片段大小
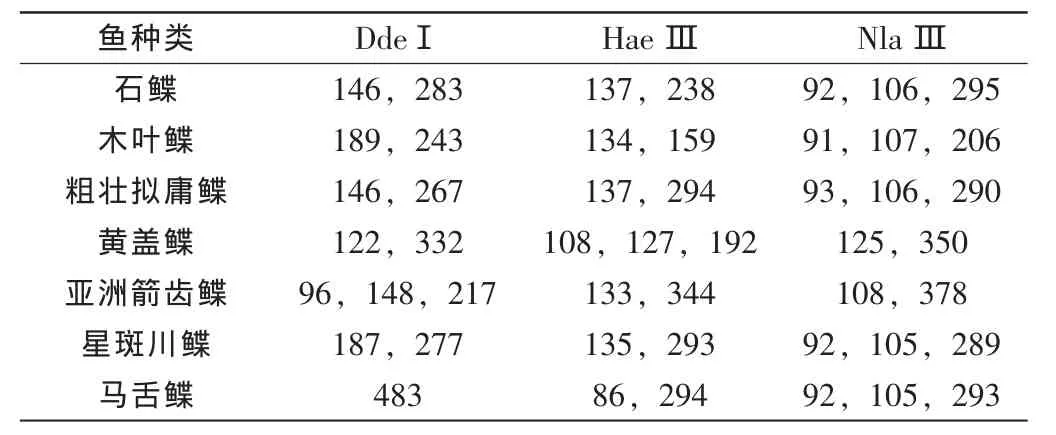
表37 种鲽鱼PCR扩增片段分别经Dde I、Hae III和Nla III酶切的RFLP片段大小
4 讨论
本研究对21种鱼类的16S rRNA基因进行了PCR扩增,并对扩增产物进行了测序及同源性比对分析。良好匹配的可能性百分比为99%;如匹配未有良好,但其反应类型足够接近期望值,仍可清楚鉴定,可信限范围为85%-99%,越接近99%越说明鉴定结果匹配。结果显示,太平洋绒杜父鱼的16S rRNA基因与GenBank数据库中的美洲绒杜父鱼(Hemitripterus americanus)16S rRNA基因(GenBank登录号:AF420458.1)的同源性最高,为94%。本研究所用杜父鱼经大连海洋大学水产养殖中心鉴定为太平洋绒杜父鱼,这一结果与Blast同源性比对结果并不一致,分析构成这一矛盾的原因为GenBank数据库中没有太平洋杜父鱼16S rRNA基因的相关信息,故Blast同源比对结果显示目标序列与同为绒杜父鱼属的美洲绒杜父鱼16S序列相似度最高。美洲绒杜父鱼分布于加拿大和美国东北海域[11],而太平洋绒杜父鱼主要分布于太平洋西北部沿海水体,在中国常见于大连和长岛海域[12],可见本研究最终鉴定结果与鱼类地理分布事实相符。而其他20种鱼类与GenBank数据库中各自相应序列的同源性比对结果均为98%以上。
5 结论
本研究将PCR-RFLP技术与芯片生物分析技术相结合,获得了中国北方14种经济鱼类和7种鲽鱼的PCR-RFLP图谱,所建立的方法重复性好、操作简便、快速,为鱼类的品种鉴定提供了理论参考。整个过程可以在一天内完成,具有较高的推广价值和应用前景。
[1]Zhang J B,Hanner R.Molecular approach to the identification of fish in the South China Sea[J].PLoS One,2012,7(2):30621.
[2]Honardoost M A,Tabatabaeian H,Akbari M,et al.Investigation of sensitivity,specificity and accuracy of Tetra primer ARMS PCR method in comparison with conventional ARMS PCR,based on sequencing technique outcomes in IVS-II-I genotyping of beta thalassemia patients[J].Gene,2014,549(1):1-6.
[3]Chakraborty A,Aranishi F,Iwatsuki Y.Molecular identification of hair tail species(Pisces:Trichiuridae)based on PCRRFLP analysis of the mitochondrial 16S rRNA gene[J].J Appl Genet,2005,46(4):381-385.
[4]Aranishi F.Rapid PCR-RFLP method for discrimination of imported and domestic mackerel[J].Mar Biotechnol(NY),2005,7(6):571-575.
[5]Takashima Y,Morita T,Yamashita M.Complete mitochondrial DNA sequence of Atlantic horse mackerel Treacherous treacherous and molecular identification of two commercially important species T.trachurus and T.japonicas using PCR-RFLP[J].Fish Sci,2006,72:1054-1065.
[6]Akasaki T,Yanagimoto T,Yamakami K,et al.Species identification and PCR-RFLP analysis of cytochrome b gene in cod fish(Order Gadiformes)products[J].J Food Sci,2006,71(3):C190-C195.
[7]Lin W F,Hwang D F.Application of PCR-RFLP analysis on species identification of canned tuna[J].Food Control,2007,18:1050-1057.
[8]Espiñeira M,González-Lavín N,Vieites JM,et al.Authentication of anglerfish species(Lophius spp)by means of polymerase chain reaction-restriction fragment length polymorphism(PCR-RFLP)and forensically informative nucleotide sequencing(FINS)methodologies[J].J Agric Food Chem,2008,56(22):10594-10599.
[9]Wen J,Zeng L,Sun YL,et al.Authentication and traceability of fish maw products from the market using DNA sequencing[J].Food Control,2015,55:185-189.
[10]Hold G L,Russell V J,Pryde S E,et al.Development of a DNA-based method aimed at identifying the fish species present in food products[J].J Agric Food Chem,2001,49(3):1175-1179.
[11]Scotter A J,Kuntz D A,Saul M,et al.Expression and purification of sea raven type II antifreeze protein from Drosophila melanogaster S2 cells[J].Protein Expr Purif,2006,47(2):374-383.
[12]Antonenko D V,Pushchina O I,Solomatov S F,et al.Distribution and some features of biology of sea raven Hemitripterus villous(Hemitripteridae)in the northwestern part of the Sea of Japan[J].J Ichthyol,2010,50(8):640-649.
The Application of PCR-RFLP to 14 Kinds of Economic Fish from North China Coastal and 7 Kinds of Pleuronectidae Fish
Yan Pingping1,Qi Xin1,Huang Daliang1*,Wang Haiyan2,Cui Yan1,Tian Zhuo1,Zhang Lirong1
(1.Dalian Entry-Exit Inspection and Quarantine Bureau,Dalian,Liaoning,116600;2.Dayaowan Entry-Exit Inspection and Quarantine Bureaus)
14 kinds of economic fish(Hyporhamphus sajori,Synechogobius ommaturus,Seriola lalandi,Platycephalus indicus,Hexagrammos otakii,Thamnaconus septentrionalis,Scophthalmus maximus,Chirolophis japonicus,Hemitripterus villosus,Gadus morhua,Zebrias zebrinus,Cynoglossus lighti,Pholis fangi,Paralichthys olivaceus)from North China coastal and 7 kinds of pleuronectidae fish(Hippoglossoides robustus,Limanda aspera,Atheresthes evermanni,Platichthys stellatus,Reinhardtius hippoglossoides,Pleuronichthys cornutus,Kareius bicoloratus)were the objects.First mitochondrial 16s rRNA gene was amplified and homologied of the sequence.Mitochondrial cytochrome b gene of the 21 kinds of fish was analyzed by PCR-RFLP.The diested DNA fragments were analyzed by microfluidic capillary electrophoresis.The total of 132 electrophoresis bandings were detected.The 21 kinds of fish species were fully discriminated.The results of this article provide a scientific basis for the fish species identification and fish food testing.
Economic Fish;Pleuronectidae;PCR-RFLP;16S rRNA;Cytochrome
Q959.4
E-mail:ppy1129@163.com;*通讯作者E-mail:ciqzhhdl@163.com
国家质检总局科技计划项目(2011IK119)
2015-07-08
